卤化物中的卤素键(XB)是共价成键的卤素原子(X=Cl、Br、I,作为XB供给体)与亲核试剂(如路易斯碱等XB受体)之间的相互作用,具有高度的方向性和特异性。近年来,卤素键在多个领域中越来越受到重视,特别是在含有碳卤键(C-X键, 如C-F、C-Cl、C-Br、C-I)的生物分子和药物化合物中,其在多个方面具有显著优势,例如能够增强分子结合亲和力与生物活性、提高反应选择性、改善生物物理性质。此外,还可以通过卤素键介导分子间的相互作用。基于这些优势,卤化物在医药(如激素药物)、杀菌剂及有机合成等领域得到了广泛应用[1]。然而,对于小而复杂的有机分子骨架的特定位点卤化,其化学过程仍然具有挑战性[2]。分子结构上的微小变化可能会对小分子的生物活性和代谢产生深远影响。例如,万古霉素分子中的氯原子被氢原子取代后,其抗菌活性降低约70%[3];蝴蝶霉素中的氯原子被氢原子取代后,其抗菌活性会显著降低甚至基本丧失,导致其不再具有实用价值[4];2-氯硝吡咯菌素的抗真菌活性仅为硝吡咯菌素(具有2个氯原子)的10%[5]。
有机卤化物的合成路线通常包括多个步骤。为了获得较高的化学、区域和立体选择性,往往需要使用保护基团、定向基团或活化基团,并结合复杂且高效的化学催化剂,以此精准控制反应过程,使反应在目标反应位点发生。然而,传统的卤化反应存在诸多弊端,一方面,这类反应通常使用有毒的试剂和溶剂,涉及对环境有害的操作,且需要在恶劣的反应条件下进行[6-7];另一方面,化学卤化反应面临区域选择性差、产率低、产生非目标副产物等问题,通常需要依赖烦琐的化学分离方法[8-9]。值得注意的是,酶促卤化反应在复杂的分子环境中展现出卓越的区域和立体选择性,它无需额外的保护基团或活化基团处理,也避免了双产物的分离问题,为卤化反应提供了一条更为高效且环保的路径[10-11]。基于上述优势,研究者愈发致力于识别、探索并利用生物卤化酶,以开发合成具有不同分子结构化合物的生物途径[12]。
目前已知存在近5 000种天然有机卤化物,且这一数量还在不断增长,这凸显出生物催化剂在催化卤化反应、形成C-X键方面具有重要作用。生物卤化反应由卤化酶催化,这类酶能以F、Cl、Br、I作为卤素供体,生成相应的卤素代谢物。根据不同的催化机制,卤化酶可以分为以下3类[13]。(1)亲电取代型,包括卤化过氧化物酶(血红素依赖型、钒依赖型)和黄素依赖型卤化酶(Fl-Hals)。卤化过氧化物酶是一类最早被发现具有卤化活性的酶,它们首先利用活性位点内产生的过氧化氢与卤化物反应形成次卤化物(HOX,X=Cl、Br、I),随后HOX与底物进行非特异性卤化反应[14]。Fl-Hals是从黄素依赖型单加氧酶(FMOs)超家族进化而来,FMOs通过还原黄素(FADH2)激活分子氧生成氢过氧黄素(FAD-OOH)。Fl-Hals的卤化反应基于FMOs初始激活机制,首先通过FAD-OOH激活反应位点,随后FAD-OOH进一步与卤化物阴离子反应产生次卤化物[15]。然而,在反应过程中,次卤化物在一定程度上缺乏卤化位点的选择可控性。(2)亲核取代型,如S-腺苷-L-甲硫氨酸(SAM)依赖型卤化酶。SAM依赖型卤化酶虽然能够将氟离子引入有机分子中,但底物范围非常有限。(3)自由基型,如非血红素Fe(Ⅱ)/α-酮戊二酸依赖型卤化酶[以下简称Fe(Ⅱ)/α-KG卤化酶]。Fe(Ⅱ)/α-KG卤化酶作为一种生物催化剂,能够在温和条件下通过自由基反应机制,以高区域选择性和高立体选择性将杂原子(如O、N、Cl、N3等)引入未活化的sp3杂化碳[C(sp3)]中心[16-17]。鉴于含有未活化C(sp3)的脂肪族小分子在自然界中广泛存在,Fe(Ⅱ)/α-KG卤化酶在脂肪族化合物的不对称卤化反应中展现出巨大的应用潜力,为生物催化与化学合成相结合形成新型化学键提供了可能。
本文综述Fe(Ⅱ)/α-KG卤化酶的基本特性及催化机制,并简要说明Fe(Ⅱ)/α-KG卤化酶作为生物催化剂在非天然卤化物合成途径等方面的应用,以期通过生物信息学和基因挖掘方法寻找更多结构新颖、生物功能独特的Fe(Ⅱ)/α-KG卤化酶。
1 Fe(Ⅱ)/α-KG卤化酶的基本特性Fe(Ⅱ)/α-KG卤化酶归属于功能强大的Fe(Ⅱ)/α-酮戊二酸依赖加氧酶[Fe(Ⅱ)/α-KGOs]超家族,该家族酶能够催化包括羟基化、卤化、闭环、去饱和、差向异构化、扩环以及环氧化在内的多种氧化转化反应,在众多生物过程中发挥着至关重要的作用[18-19]。相较于传统卤化酶,Fe(Ⅱ)/α-KG卤化酶在催化性能与应用场景上展现出革命性突破,体现在:(1)底物普适性与功能多样性。Fe(Ⅱ)/α-KG卤化酶可作用于脂肪族、芳香族及杂环化合物,可催化卤化、羟基化、环氧化等多种反应。(2)立体选择性的精准控制。Fe(Ⅱ)/α-KG卤化酶通过活性口袋的“手性环境”实现立体化学的精确调控。(3)工程化改造潜力。Fe(Ⅱ)/α-KG卤化酶的模块化结构(α-螺旋束核心+柔性loop区)可为定向进化提供理想模板。
根据生物催化卤化反应过程中反应底物的状态,即与载体蛋白的磷酸泛酰巯基乙胺臂(Ppt臂)相连或游离状态,将Fe(Ⅱ)/α-KG卤化酶分为载体蛋白依赖型(Carrier protein-dependent type)卤化酶和独立型(Free standing type)卤化酶(表 1)。近年来发现了几种载体蛋白依赖型Fe(Ⅱ)/α-KG卤化酶,包括SyrB2[20-21]、BarB1、BarB2[22]、CytC3(来自抗生素γ, γ-dichloroaminobutyrate的生物合成基因簇)[23]、JamE[24]、KtzD(来自Kutzneride生物合成基因簇)[25]、HctB(来自hectochlorin生物合成基因簇)[26]、CmaB(来自coronamic acid生物合成基因簇)、CurA[27]、Thr3[28-29]等。独立型Fe(Ⅱ)/α-KG卤化酶包括WelO5[30]、AmbO5[31]、SaDAH[32]、AdaV[33]、BesD[16]等。载体蛋白依赖型和独立型Fe(Ⅱ)/α-KG卤化酶这两大分支之间的同源性较低,载体蛋白依赖型卤化酶相互间的同源性较高,独立型卤化酶相互间的同源性较低[34]。
| 酶类型
Enzyme type | 酶名称
Enzyme name |
氨基酸序列Amino acid sequence | ||||||||||||||
| 146 | 147 | 148 | 149 | 150 | 151 | 152 | 153 | 154 | 155 | 156 | 157 | 158 | 159 | 160 | ||
| Carrier protein- dependent type | SyrB2 | D | E | G | T | D | - | - | W | H | Q | A | - | - | - | D |
| BarB1 | D | E | G | T | D | - | - | W | H | Q | A | - | - | - | E | |
| BarB2 | D | E | G | T | D | - | - | W | H | Q | A | - | - | - | E | |
| CytC3 | D | E | G | T | D | - | - | W | H | Q | A | - | - | - | D | |
| JamE | K | E | G | F | A | - | - | W | H | Y | - | - | - | - | D | |
| KtzD | D | E | G | T | D | - | - | W | H | Q | A | - | - | - | D | |
| HctB | E | G | E | I | K | - | - | W | H | Q | V | - | - | Y | D | |
| CmaB | N | E | G | T | D | - | - | W | H | Q | A | - | - | - | D | |
| CurA | A | L | G | M | P | G | S | T | A | Y | G | G | L | T | D | |
| Free standing type | WelO5 | - | - | G | Q | S | - | - | P | D | L | K | - | - | - | D |
| AmbO5 | - | - | G | Q | S | - | - | P | D | L | K | - | - | - | D | |
| AdaV | A | M | I | G | D | Q | P | P | G | A | G | - | - | - | D | |
| BesD | Q | R | G | D | T | H | G | W | H | W | G | - | - | - | D | |
| Note: 146-160 represent position 146-160, respectively.In which, 154-156 represent conserved position.Bold letters indicate conserved gene sequences.“-” indicates vacancy. | ||||||||||||||||
2 Fe(Ⅱ)/α-KG卤化酶的催化机制
Fe(Ⅱ)/α-KG卤化酶遵循自由基反应机制,通过与O2结合形成高价和短寿命的FeⅣ=O铁基中间体[35](图 1)。该中间体是一种强氧化剂,可以从未活化的C(sp3)-H键中提取氢,产生底物自由基和FeⅡ-羟基(或氯化物)中间体。卤素原子随后反弹到底物自由基上,生成卤化产物。在催化过程中,Fe(Ⅱ)/α-KG卤化酶和羟化酶都涉及FeⅣ=O中间体的形成,但卤化酶利用卤素-FeⅡ中间体进行氧化,通过卤素的转移实现底物的卤化,而羟化酶则是通过羟基自由基的形成实现底物的羟基化。Fe(Ⅱ)/α-KG卤化酶中卤素的存在改变了催化机制,使得催化过程更倾向于卤化反应而非羟基化反应。
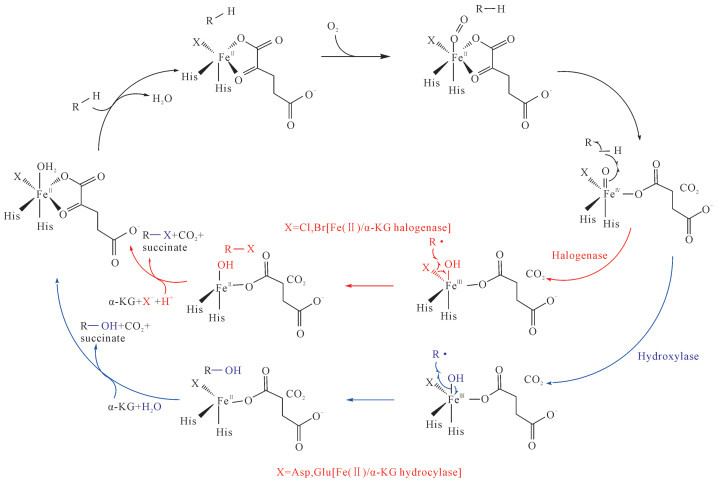
|
| 图 1 Fe(Ⅱ)/α-KG卤化酶和羟化酶催化反应机制 Fig.1 Reaction mechanism of Fe(Ⅱ)/α-KG-dependent halogenase and hydroxylase |
从结构上来说,Fe(Ⅱ)/α-KG卤化酶和羟化酶活性位点的主要差异在于HxG(A)基序。卤化酶中,该基序的甘氨酸或丙氨酸残基被羧酸残基取代,从而使得该酶中的卤化物离子直接配位[36]。底物相对于活性位点FeⅣ=O中间体的空间定位是卤化反应选择性的主要决定因素,而铁中心的氧化还原性质及配体解离行为等协同因素进一步调控反应进程[37]。Srnec等[38]通过低温磁圆二色(LT MCD)光谱与量子化学计算,揭示了α-KG依赖型酶中FeⅣ=O中间体的电子结构及反应机制,其较弱的配体场使FeⅣ=O键在过渡态呈现更强的氧基特性,从而提升内在反应性,进一步印证了电子结构对反应活性的定向影响。此外,Fe(Ⅱ)/α-KG卤化酶在催化过程中也可能引发一些副反应,例如脂肪羟基化、硝化和叠氮化反应,相关现象已有文献报道[39]。
3 Fe(Ⅱ)/α-KG卤化酶的应用研究进展 3.1 载体蛋白依赖型Fe(Ⅱ)/α-KG卤化酶载体蛋白依赖型Fe(Ⅱ)/α-KG卤化酶催化的反应底物都需要通过磷酸泛酰巯基乙胺臂与酰基或肽基载体蛋白共价连接。2005年,首个载体蛋白依赖型Fe(Ⅱ)/α-KG卤化酶SyrB2从丁香假单胞菌Pseudomonas syringae的生物合成途径中被发现[20],底物L-苏氨酸通过载体蛋白SyrB1上共价连接的磷酸泛酰巯基乙胺基团介导,被转运至SyrB2的活性位点[图 2(a)]。Blasiak等[36]首次解析了SyrB2的晶体结构,发现其FeⅡ中心由His116、His235、Cl-、α-KG(双齿配位)及1个水分子构成五配位模式,Cl-取代了经典“2-His-1-羧酸盐”面部三联体中的羧酸配体位点。Matthews等[37]的研究基于该结构证实:底物侧链定位(C4甲基远离Fe=O而靠近Cl-)是控制卤代/羟化选择性的关键机制。Mitchell等[30]将SyrB2分别催化天然底物L-苏氨酸-SyrB1与其不同氨基酸类似物的反应进行比较,表明底物远离氧配体而靠近卤素是确保选择性卤化反应发生的主要结构特征[图 2(b)]。
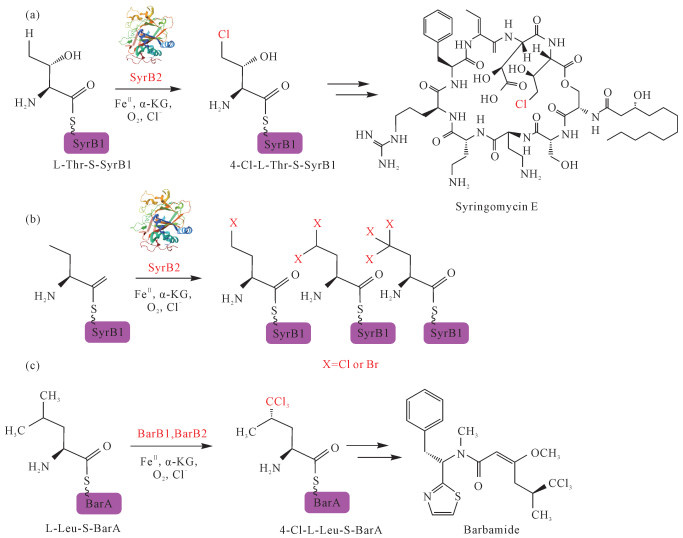
|
| 图 2 Fe(Ⅱ)/α-KG卤化酶SyrB2、BarB1和BarB2催化氨基酸氯化反应 Fig.2 Amino acid chlorination reaction catalyzed by Fe(Ⅱ)/α-KG-dependent halogenase SyrB2, BarB1 and BarB2 enzyme |
研究者通过量子力学/分子力学(QM/MM)计算和分子动力学(MD)模拟相结合的方法,阐明了SyrB2选择性卤化的催化机理[40]。简而言之,SyrB2存在3个相互转换的Cl-FeⅣ-氧异构体,并且只有氧配体指向目标C-H键的异构体在氢原子夺取(H-夺取)过程中具有活性,形成的Cl-FeⅢ-OH/R·中间体是由Arg254氨基酸残基与Cl-FeⅢ-OH的OH配体之间的氢键相互作用决定的。氢键在其中发挥着双重作用:一方面,它能够有效阻止OH参与后续的反弹步骤,从而避免羟基化产物的形成;另一方面,它可以促进Cl-FeⅢ-OH/R·中间体发生异构化,使得氯原子指向烷基自由基。此外,铁的次级配位球(SCS,与铁配体结合相互作用的氨基酸残基)在控制SyrB2卤化酶反应性中起到重要的作用。Wilson等[41]在SyrB2的第104位和121位分别引入了可与铁的水配体和氯配体形成氢键的酪氨酸残基,以此开展相关研究,证实了SCS在控制SyrB2卤化酶反应性中的重要性。基于分子动力学模拟,通过对比两个SyrB2突变体经翻译后修饰(PTM)生成二羟基苯丙氨酸(DOPA)残基的差异,证实了SyrB2中卤代铁异构体采取轴向氯构型的有效性,并突显了SCS对非血红素铁酶(尤其是卤化酶)催化活性的调控作用。此外,在高效软体动物杀灭剂Barbamide的生物合成过程中,Fe(Ⅱ)/α-KG卤化酶BarB1和BarB2通过协同作用能够有效催化附着在肽基载体蛋白BarA上的底物L-亮氨酸C5位甲基进行三氯反应,实现体外催化合成(2S, 4S)-5, 5, 5-三氯亮氨酸[22][图 2(c)]。
3.2 独立型Fe(Ⅱ)/α-KG卤化酶根据反应底物的不同,可将独立型Fe(Ⅱ)/α-KG卤化酶分为三大类:第一类是生物碱卤化酶,第二类是氨基酸卤化酶,第三类是核苷酸卤化酶。
3.2.1 生物碱卤化酶第一个被发现的独立型Fe(Ⅱ)/α-KG卤化酶是来自真枝藻科蓝藻Hapalosiphon welwitschii的WelO5[30](图 3)。WelO5存在于韦氏吲哚啉生物合成途径中,它可以催化12-epi-fisherindole U和12-epi-hapalindole C脂肪碳的选择性单氯化反应,分别生成12-epi-fisherindole G和12-epi-hapalindole E,从而在分子中形成新的立体中心(图 4)。在WelO5-α-KG-底物配合物的结构中,α-KG与底物结合后,FeⅡ中心的空配体位点直接面向底物,12-epi-fisherindole U的C13氯化靶远离卤素配体,靠近FeⅣ=O中间体中氧基的预测位置。由于底物处于远离氧配体的活性卤铁基状态,需要通过色氨酸残基Ser189来调控α-KG的构象重排,以实现选择性卤化反应。AmbO5是通过基因挖掘从蓝藻Fischerella ambigua UTEX 1930中发现的Fe/α-KG卤化酶[31]。AmbO5具有广泛的底物范围,能够对多种生物碱化合物进行位点选择性氯化反应,包括hapalindole[31]、ambiguine[42-43]和fischer indole[44]等生物碱类化合物(图 4)。对AmbO5和WelO5序列的比较分析显示,WelO5的C端片段可能对底物选择性和特异性很重要,用相应的AmbO5序列替换WelO5酶中C端18个残基的片段可以扩大WelO5催化的底物范围[31]。
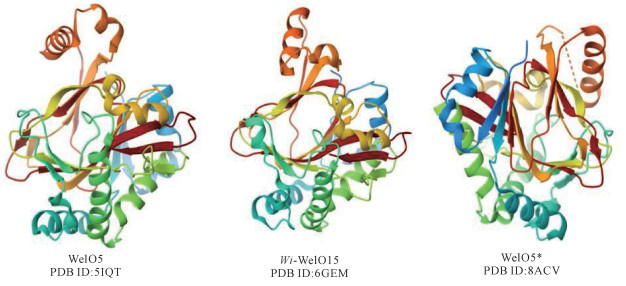
|
| 图 3 生物碱卤化酶结构 Fig.3 Structural of biological alkali halogenase |
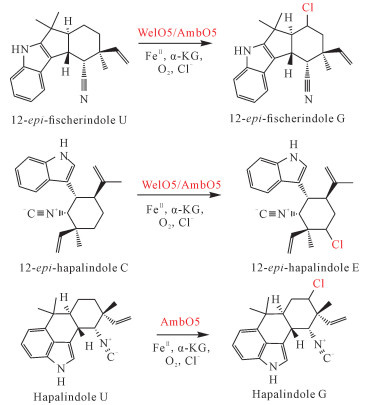
|
| 图 4 Fe(Ⅱ)/α-KG卤化酶WelO5和AmbO5的卤化多样性 Fig.4 Halogenation diversity of Fe(Ⅱ)/α-KG-dependent halogenase WelO5 and AmbO5 enzyme |
通过结构指导的定向进化策略,以NaCl作为氯源,来源于Westiella intricate HT-29-1的Fe(Ⅱ)/α-KG卤化酶Wi-WelO15(图 3)可成功催化未活化的C(sp3)-H键进行化学、区域和立体选择性氯化,显著拓展了其底物谱。该酶变体突破Fe(Ⅱ)依赖型酶氧敏感的限制,可在有氧条件下高效催化非天然对映体hapalindole(其结构中含有酮基而非异氰基功能基团)的温和氯化,并展现出在富电子官能团共存下活化脂肪族C-H键的独特能力,为复杂分子精准功能化及规模化生物催化提供了新范式[45]。此外,基于智能文库设计与机器学习协同策略,Büchler等[46]成功构建了Fe(Ⅱ)/α-KG卤化酶WelO5*(图 3),实现了大环内酯类化合物Soraphen A/C的后期功能化(Late-stage functionalization)。WelO5*活性变体展现出底物混杂性,可催化Soraphen A/C生成氯代与羟基化产物。进一步评估发现,Soraphen氯化衍生物对6种植物病原真菌(小麦叶锈病病原菌Puccinia recondita、小麦壳针孢叶枯病病原菌Septoria tritici、小麦白粉病病原菌Erysiphe graminis、雪霉病病原菌Monographella nivalis、灰霉病病原菌Botrytis cinerea和花生褐斑病病原菌Mycosphaerella arachidis)均表现出显著抑制活性,这为开发绿色抗真菌农用制剂提供了新策略。
3.2.2 氨基酸卤化酶氨基酸作为自然界中的模块化构建单元,不仅参与大分子如蛋白质的合成,还参与小分子天然产物如生物碱和非核糖体肽的生成。基于氨基酸在生物分子细胞生产中的核心作用,卤化氨基酸产物可作为前体,用于进一步转化合成一系列新颖的化合物,如氮杂环、胺类、酮酸和肽等。2019年,Marchand等[16]在卡特兰链霉菌Streptomyces cattleya中鉴定出自由基卤化酶BesD (图 5),该酶可通过γ-位选择性氯化L-赖氨酸生成β-乙炔丝氨酸(βes),从而开辟了一条卤代/烯炔氨基酸生物合成新途径。基于BesD的晶体结构及与Fe(Ⅱ)/α-KG机制类比,推测其催化过程如下:α-KG介导O2氧化FeⅡ生成FeⅣ=O活性中间体,随后夺取底物C-H键中的氢原子,使底物形成自由基中间体,经单电子转移并与氯结合形成C-Cl键。此外,为了发掘更多可能存在的卤化酶,研究者对推定的卤化物酶编码基因序列进行了同源搜索,并根据推定功能的不同,将潜在的同源卤化物酶分为8个类别,命名为HalA-HalH基因簇[17]。实验表明,HalA-HalH中单个卤化酶能够对L-赖氨酸、L-鸟氨酸、L-亮氨酸、L-异亮氨酸等多种氨基酸进行区域选择性氯化(图 6)。这些结果不仅证明了以BesD为代表的自由基卤化物酶家族能够选择性地将C-H键活化成C-X键,从而调控分子的生物活性,还为该酶家族在活性中间体合成方面的应用提供了理论依据,揭示了其在生物化学合成领域的巨大潜力。
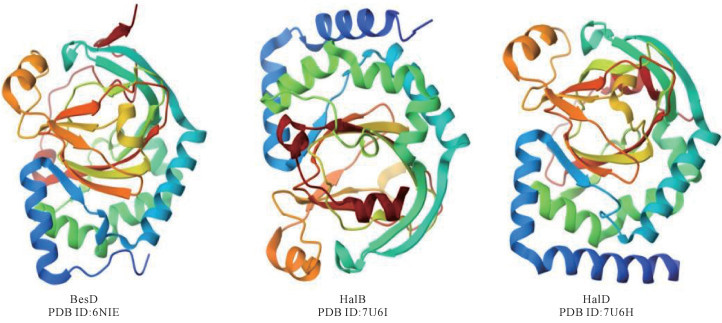
|
| 图 5 氨基酸卤化酶结构 Fig.5 Structural of biological amino acid halogenase |
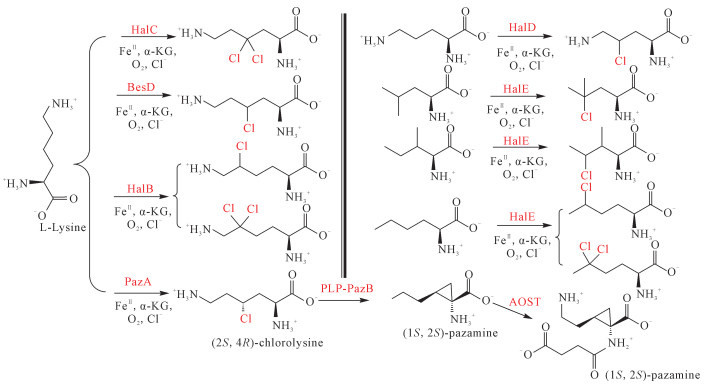
|
| 图 6 Fe(Ⅱ)/α-KG卤化酶BesD及其同源酶催化氨基酸氯化反应 Fig.6 Amino acid chlorination catalyzed by Fe(Ⅱ)/α-KG-dependent halogenase BesD and its homologues |
Li等[47]利用密度泛函理论(DFT)计算,提出BesD氯化反应的详细机理。该机理涉及α-KG脱羧生成Cl-FeⅣ=O中间体,随后Cl-FeⅣ=O发生异构化,FeⅣ=O活性中间体夺取底物上的氢原子,最后氯原子反弹至底物碳自由基上完成氯化过程。值得注意的是,BesD催化L-赖氨酸的溴化和叠氮化反应遵循与氯化反应相同的机理,且总势垒变化不大,这表明引入不同配体(X)对反应速率的影响较小,反映出BesD对阴离子配体(X-)的广谱兼容性。进一步研究发现[48],底物L-赖氨酸与氯离子在BesD/HalB活性位点结合时呈现强正协同效应(协同常数α≈15 500)。同时,结合DFT计算与蛋白质设计,该研究阐明在BesD的SCS中,带负电的E119残基通过氢键形成“静电锁”,阻碍了氯离子或底物的单独结合;仅当氯离子与赖氨酸共结合时,E119才被推离FeⅡ中心,从而重塑活性位点静电环境,协同促进底物与阴离子的精准识别与功能化。Kissman等[49]通过解析和比较Fe(Ⅱ)/α-KG卤化酶HalB、HalD、BesD的晶体结构,阐明选择性卤化产生4-Cl-赖氨酸(BesD)、5-Cl-赖氨酸(HalB)和4-Cl-鸟氨酸(HalD)的酶的选择性基础,实现位点选择性和链长选择性,揭示C末端底物结合“帽子”在定位底物进行C4、C5氯化以及识别赖氨酸、鸟氨酸方面的关键作用。底物结合“帽子”的靶向工程进一步表明,这类酶的选择性可以改变或切换,展示出其在开发适用于生物催化领域的卤化酶方面的潜力。Slater等[50]对L-赖氨酸-4-氯卤化酶与卤化物、α-KG、O2这3个底物结合的动力学途径和热力学关系行了剖析,发现在加入α-KG后,卤化物会配位到辅因子(通常指铁离子),同时,辅因子附近的阳离子L-赖氨酸会结合到酶的特定位点, 这两个结合事件之间表现出强烈的异向协同性。在添加O2后,羟基铁中间体不会将底物困在活性位点,从而显著降低了卤素与L-赖氨酸之间的协同作用。
最近,Sosa等[51]首次发现并报道了含环丙烷结构的非天然氨基酸pazamine及其衍生物pazamide(图 6),并阐明了磷酸吡哆醛(PLP)依赖型酶催化隐蔽卤化和环合的生物合成机制。基于BesD样卤化酶定位偶氮假单胞菌Pseudomonas azotoformans中的pazRABCD基因簇,该簇编码1种调节蛋白(PazR)、1种赖氨酸卤化酶(PazA)、1种丝氨酸羟甲基转移酶类似酶(PazB)和2种假定的氨基酸转运蛋白(PazC/D)。在pazamine生物合成过程中,自由基卤化酶PazA通过氯化L-赖氨酸C4启动该生物合成途径,生成(2S, 4R)-氯赖氨酸,随后(2S, 4R)-氯赖氨酸作为底物被PLP依赖型酶PazB催化,经历C2位的去质子化、C4位的类SN2亲核攻击和氯原子离去等反应步骤,最终形成具有高应变的环丙烷结构化合物(1S, 2S)-pazamine,这一生物合成机制为含非天然氨基酸化合物的生物合成研究提供了参考。
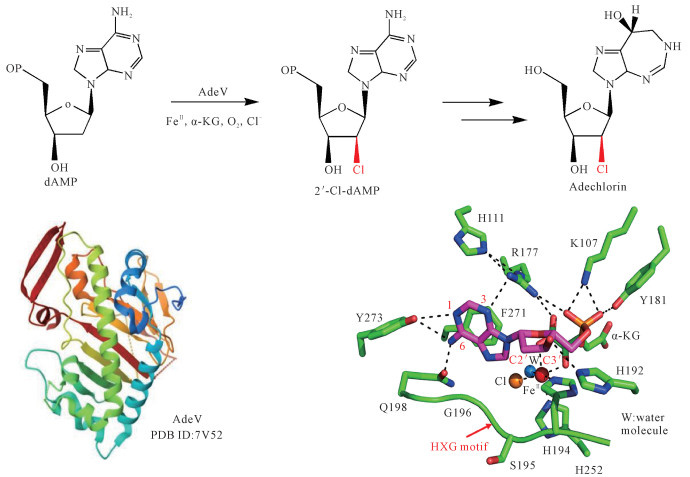
3.2.3 核苷酸卤化酶在药物开发中,核苷/核苷酸类似物因其在抗病毒和抗癌治疗中的显著效果而备受关注。这些化合物大多通过化学合成方法制备,只有少数天然存在的卤化核苷酸从微生物中分离获得。Fe(Ⅱ)/α-KG卤化酶因其能够高效、精准地在核苷酸特定位置引入卤素原子,成为生物催化领域的研究热点。独立型Fe(Ⅱ)/α-KG卤化酶AdeV(后有研究称为AdaV)是目前发现的第一个作用于核苷酸的酶,来源于植物放线菌Actinomadura sp.ATCC 39365参与2′-氯核苷天然产物adechlorin的生物合成过程[33]。AdeV催化不含载体蛋白的2′-脱氧腺苷-5′-单磷酸(dAMP)的FeⅡ、α-KG、O2、Cl-依赖型氯化反应,生成2′-氯-2′-脱氧腺苷-5′-单磷酸(2′-Cl-dAMP),是adechlorin生物合成途径中的关键中间体。为了更好地理解AdeV的催化机理,Dai等[52]解析了AdeV/FeⅡ/Cl和AdeV/FeⅡ/Cl/α-KG的复杂晶体结构,并通过分子对接、突变和生化分析,阐明了AdeV与dAMP相互作用的分子机制(图 7)。氨基酸残基H111可能发挥着重要作用,它通过与自身侧链形成氢键相互作用,来辅助残基R177实现精准定向。随后,在反应过程中,O2取代了原本处于反应体系活性位点上的水分子。与此同时,α-KG发生脱羧反应,形成FeⅣ-oxo中间体,并伴随着oxo配体发生构象重排。FeⅣ-oxo中间体从dAMP中提取1个氢原子,在此过程中,dAMP中C-3′羟基与氨基酸残基H192之间形成氢键,这对dAMP的正确定向以实现有效的卤化反应起着关键作用。最后,dAMP从活性位点中夺取氯离子,从而生成2′-Cl-dAMP。
Zhai等[53]报道了AdaV配合物及其底物和各种AdaV变体的X射线晶体结构,并结合晶体学和生化数据阐明了AdaV的底物特异性和立体选择性的分子机制。同时,还提出了AdaV催化的双重控制机制,其中AdaV变体G196通过创建氯离子结合的铁配位位点在卤化过程中发挥重要作用,而Q203和V269则通过定向FeⅢ-OH中间体来限制氧原子反弹到自由基底物上。此外,Zhai等[53]还通过靶向基因组挖掘鉴定了与2′-chloropentostatin (2′-Cl PTN,一种有效的腺苷脱氨酶抑制剂)相关的天然产物的几个潜在生物合成基因簇,并系统地研究了目标卤化基因在体内的功能作用。通过对3株代表性菌株——小麦微孢菌Microbispora triticiradicis CGMCC 4.7399、曼谷放线动孢菌Actinokineospora bangkokensis NBRC 108932和变弧菌链霉菌Streptomyces varsoviensis CGMCC 4.1431进行发酵培养以及代谢物分析,发现在这些菌株的代谢物中可以检测到2′-Cl PTN,表明该基因簇实际上对相关天然产物的生物合成具有活性。这些结果不仅极大地扩展了关于卤化天然产物生物合成的酶库,也为合理且快速地发现更多AdaV家族酶提供了基础,从而为其作为合成生物学应用中的工具酶开辟了道路。
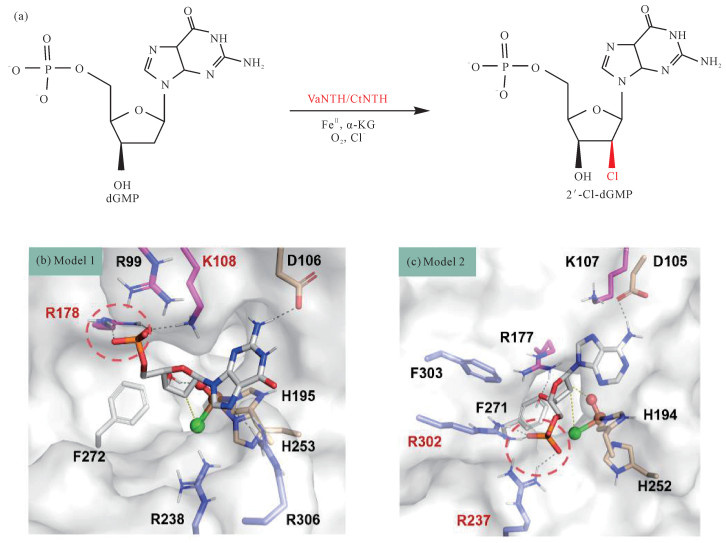
近期,Ni等[54]以已知的卤化酶AdaV为模板,通过序列比对和进化树分析挖掘了2个新的dAMP——VaNTH和CtNTH,它们能够对2′-脱氧鸟苷单磷酸(dGMP)进行卤化[图 8(a)]。此外,新发现的dGMP卤化酶与AdaV的序列相似度高达51%,却表现出截然不同的底物特异性,该特异性来源于核苷酸5′-磷酸基团的结合模式。在CtNTH/dGMP和AdaV/dAMP体系的分子动力学模拟过程中,观察到2种可相互转换的底物结合模式(模式1和模式2),它们在磷酸基的取向上不同[图 8: (b)、(c)]。CtNTH和AdaV均含有两个相似的磷酸结合位点。CtNTH偏好结合模式1,有利于dGMP的卤化反应;而AdaV更偏好结合模式2,有利于dAMP的反应。在CtNTH/dGMP体系的结合模式1中,磷酸基与Arg178和Lys108形成氢键,核碱基部分通过与His195的π-π相互作用以及与Asp106的氢键作用而得以固定,dGMP的脱氧核糖环通过3′羟基和氧基之间的稳定氢键与FeⅡ=O中心紧密结合。分子动力学模拟揭示了核苷酸特异性调控的新机制,研究人员基于此机制成功改造了底物特异性,进而丰富了核苷酸修饰的工具酶种类[55-56]。
4 展望
有机卤化物在自然界中无处不在,并在有机化学合成、农业化学和制药工业中发挥着至关重要的作用。据统计,大约50%的畅销药物和96%的农用化学品都含有共价键连接的卤素原子。这源于卤素原子选择性引入小分子后,能有效调节其生物活性和药代动力学特性。因此,发展高效、高选择性的卤化方法至关重要。值得注意的是,利用Fe(Ⅱ)/α-KG卤化酶催化卤素原子的生物引入,因其优异的区域和立体选择性以及潜在的绿色化学特性,在化学工业中展现出巨大的应用潜力。然而,Fe(Ⅱ)/α-KG卤化酶的酶工程研究尚处于发展阶段,其广泛应用仍受诸多因素限制,主要包括以下4个因素。(1)酶的种类和数量有限。目前报道的Fe(Ⅱ)/α-KG卤化酶种类较少,而且已知酶的活性较低,底物谱相对狭窄,难以满足多样化合成需求,这限制了其在工业应用中的选择范围。(2)催化性能有待提升。现有的Fe(Ⅱ)/α-KG卤化酶在催化效率、区域和立体选择性等方面仍有待提高。(3)酶的结构与功能关系不明确。对于Fe(Ⅱ)/α-KG卤化酶的结构与功能关系,尤其是关键氨基酸残基在催化过程中的作用,仍缺乏深入理解,这限制了基于理性设计的酶工程改造工作的开展,因而难以有效提升其催化性能。(4)酶工程改造策略有限。目前对Fe(Ⅱ)/α-KG卤化酶的改造主要依赖于随机突变和基因筛选,这些方法工作量大、效率低且结果难以预测,缺乏基于酶结构与功能关系的理性改造策略,限制了酶性能的提升。
因此,未来利用Fe(Ⅱ)/α-KG卤化酶生物合成卤化物的研究重点主要在以下4个方面。(1)新酶挖掘与基因合成。通过基因挖掘和序列相似网络分析等技术手段,预测并合成更多潜在的Fe(Ⅱ)/α-KG卤化酶基因,利用异源表达系统实现这些酶的高效表达和纯化,为酶学研究和工业应用提供更多选择。(2)酶结构与功能关系研究。通过计算机辅助深入解析Fe(Ⅱ)/α-KG卤化酶的三维结构,揭示关键氨基酸残基在催化过程中的作用机制;基于结构信息,设计并构建理性改造策略,以提高酶的催化性能、区域和立体选择性。(3)酶工程改造技术优化。发展高通量筛选平台,结合机器学习等技术手段,提高酶改造的效率和准确性;探索定向进化、半理性设计等策略在Fe(Ⅱ)/α-KG卤化酶改造中的应用潜力。(4)工业应用拓展。针对特定合成需求,优化Fe(Ⅱ)/α-KG卤化酶的催化条件,提高其在工业应用中的效率和稳定性;探索该酶在药物合成、农用化学品生产等领域的应用潜力。
此外,人工智能(AI)技术的快速发展也可为Fe(Ⅱ)/α-KG卤化酶的理性设计赋能。基于深度学习与分子动力学模拟,AI技术可精准解析酶的三维构效关系,驱动催化机制动态可视化,并指导突变位点筛选以提升底物选择性及催化效率[57-58]。AI技术通过整合多组学(基因组、蛋白质组)数据,挖掘卤化酶功能进化规律与物种特异性分布特征,可加速新型生物标志物及药物靶点的发现。AI驱动的虚拟酶库构建与活性预测,有助于突破传统实验筛选瓶颈,为卤化酶在合成生物学、药物化学等领域的定向改造提供全景式解决方案。
| [1] |
XU Z J, YANG Z, LIU Y T, et al. Halogen bond: its role beyond drug-target binding affinity for drug discovery and development[J]. Journal of Chemical Information and Modeling, 2014, 54(1): 69-78. |
| [2] |
CANTILLO D, KAPPE C O. Halogenation of organic compounds using continuous flow and microreactor technology[J]. Reaction Chemistry & Engineering, 2017, 2(1): 7-19. |
| [3] |
SUN H H, YEO W L, LIM Y H, et al. Directed evolution of a fluorinase for improved fluorination efficiency with a non-native substrate[J]. Angewandte Chemie International Edition, 2016, 128(46): 14277-14280. |
| [4] |
FRANK A, SEEL C J, GROLL M, et al. Characteriza- tion of a cyanobacterial haloperoxidase and evaluation of its biocatalytic halogenation potential[J]. ChemBioChem, 2016, 17(21): 2028-2032. |
| [5] |
ISUPOV M N, DALBY A R, BRINDLEY A A, et al. Crystal structure of dodecameric vanadium-dependent bromoperoxidase from the red algae Corallina officinalis[J]. Journal of Molecular Biology, 2000, 299(4): 1035-1049. |
| [6] |
CHRÉTIEN J-M, ZAMMATTIO F, LE GROGNEC E, et al. Polymer-supported organotin reagents for regioselective halogenation of aromatic amines[J]. The Journal of Organic Chemistry, 2005, 70(7): 2870-2873. |
| [7] |
HENDERSON S H, WEST R A, WARD S E, et al. Metal-free selective mono-halodecarboxylation of heteroarenes under mild conditions[J]. Royal Society Open Science, 2018, 5(6): 180333. |
| [8] |
INKPEN M S, DU S, DRIVER M, et al. Oxidative purification of halogenated ferrocenes[J]. Dalton Transactions, 2013, 42(8): 2813-2816. |
| [9] |
PAUNOVIC V, PÉREZ-RAMÍREZ J. Catalytic halogenation of methane: a dream reaction with practical scope?[J]. Catalysis Science & Technology, 2019, 9(17): 4515-4530. |
| [10] |
WAGNER C, OMARI M E, KÖNIG G M. Biohalogenation: nature's way to synthesize halogenated metabolites[J]. Journal of Natural Products, 2009, 72(3): 540-553. |
| [11] |
SENN H M. Insights into enzymatic halogenation from computational studies[J]. Frontiers in Chemistry, 2014, 2: 98. |
| [12] |
LATHAM J, BRANDENBURGER E, SHEPHERD S A, et al. Development of halogenase enzymes for use in synthesis[J]. Chemical Reviews, 2017, 118(1): 232-269. |
| [13] |
CROWE C, MOLYNEUX S, SHARMA S V, et al. Halogenases: a palette of emerging opportunities for synthetic biology-synthetic chemistry and C-H functionalisation[J]. Chemical Society Reviews, 2021, 50(17): 9443-9481. |
| [14] |
WEICHOLD V, MILBREDT D, VAN PÉE K H. Specific enzymatic halogenation-from the discovery of halogenated enzymes to their applications in vitro and in vivo[J]. Angewandte Chemie International Edition, 2016, 55(22): 6374-6389. |
| [15] |
MENON B R K, LATHAM J, DUNSTAN M S, et al. Structure and biocatalytic scope of thermophilic flavin-dependent halogenase and flavin reductase enzymes[J]. Organic & Biomolecular Chemistry, 2016, 14(39): 9354-9361. |
| [16] |
MARCHAND J A, NEUGEBAUER M E, ING M C, et al. Discovery of a pathway for terminal-alkyne amino acid biosynthesis[J]. Nature, 2019, 567: 420-424. |
| [17] |
NEUGEBAUER M E, SUMIDA K H, PELTON J G, et al. A family of radical halogenases for the engineering of amino-acid-based products[J]. Nature Chemical Biology, 2019, 15(10): 1009-1016. |
| [18] |
ISLAM M S, LEISSING T M, CHOWDHURY R, et al. 2-oxoglutarate-dependent oxygenases[J]. Annual Review of Biochemistry, 2018, 87: 585-620. |
| [19] |
MARTINEZ S, HAUSINGER R P. Catalytic mecha- nisms of Fe(Ⅱ)- and 2-oxoglutarate-dependent oxyge- nases[J]. Journal of Biological Chemistry, 2015, 290(34): 20702-20711. |
| [20] |
VAILLANCOURT F H, YIN J, WALSH C T. SyrB2 in syringomycin E biosynthesis is a nonheme FeⅡ/α-ketoglutarate- and O2-dependent halogenase[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(29): 10111-10116. |
| [21] |
SRNEC M, SOLOMON E I. Frontier molecular orbital contributions to chlorination versus hydroxylation selectivity in the non-heme iron halogenase SyrB2[J]. Journal of the American Chemical Society, 2017, 139(6): 2396-2407. |
| [22] |
GALONIC D P, VAILLANCOURT F D R H, WALSH C T. Halogenation of unactivated carbon centers in natural product biosynthesis: trichlorination of leucine during barbamide biosynthesis[J]. Journal of the American Chemical Society, 2006, 128: 3900-3901. |
| [23] |
WONG C, FUJIMORI D G, WALSH C T, et al. Structural analysis of an open active site conformation of nonheme iron halogenase CytC3[J]. Journal of the American Chemical Society, 2009, 131: 4872-4879. |
| [24] |
EDWARDS D J, MARQUEZ B L, NOGLE L M, et al. Structure and biosynthesis of the jamaicamides, new mixed polyketide-peptide neurotoxins from the marine cyanobacterium Lyngbya majuscula[J]. Chemistry & Biology, 2004, 11(6): 817-833. DOI:10.3969/j.issn.1001-4160.2004.06.002 |
| [25] |
JIANG W, HEEMSTRA J R, FORSETH R R, et al. Biosynthetic chlorination of the piperazate residue in kutzneride biosynthesis by KthP[J]. Biochemistry, 2011, 50(27): 6063-6072. |
| [26] |
PRATTER S M, LIGHT K M, SOLOMON E I, et al. The role of chloride in the mechanism of O2 activation at the mononuclear nonheme Fe(Ⅱ) center of the halogenase HctB[J]. Journal of the American Chemical Society, 2014, 136(26): 9385-9395. |
| [27] |
CHANG Z, SITACHITTA N, ROSSI J V, et al. Biosynthetic pathway and gene cluster analysis of curacin A, an antitubulin natural product from the tropical marine cyanobacterium Lyngbya majuscula[J]. Journal of Natural Products, 2004, 67(8): 1356-1367. |
| [28] |
FUJIMORI D G, HRVATIN S, NEUMANN C S, et al. Cloning and characterization of the biosynthetic gene cluster for kutznerides[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104: 16498-16503. |
| [29] |
VAILLANCOURT F H, YEH E, VOSBURG D A, et al. Cryptic chlorination by a non-haem iron enzyme during cyclopropyl amino acid biosynthesis[J]. Nature, 2005, 436(7054): 1191-1194. |
| [30] |
MITCHELL A J, ZHU Q, MAGGIOLO A O, et al. Structural basis for halogenation by iron- and 2-oxo-glutarate-dependent enzyme WelO5[J]. Nature Chemical Biology, 2016, 12(8): 636-640. |
| [31] |
HILLWIG M L, ZHU Q, ITTIAMORNKUL K, et al. Discovery of a promiscuous non-heme iron halogenase in ambiguine alkaloid biogenesis: implication for an evolvable enzyme eamily for late-stage halogenation of aliphatic carbons in small molecules[J]. Angewandte Chemie International Edition, 2016, 55(19): 5780-5784. |
| [32] |
KIM C Y, MITCHELL A J, GLINKERMAN C M, et al. The chloroalkaloid (-)-acutumine is biosynthesized via a Fe(Ⅱ)- and 2-oxoglutarate-dependent halogenase in Menispermaceae plants[J]. Nature Communications, 2020, 11(1): 1867. |
| [33] |
ZHAO C H, YAN S, LI Q, et al. An Fe2+- and α-ketoglutarate-dependent halogenase acts on nucleotide substrates[J]. Angewandte Chemie International Edition, 2020, 59(24): 9478-9484. |
| [34] |
王汇滨, 车昌丽, 游松. Fe/α-酮戊二酸依赖型卤化酶在绿色卤化反应中的研究进展[J]. 合成生物学, 2022, 3(3): 545-566. |
| [35] |
AGARWAL V, MILES Z D, WINTER J M, et al. Enzymatic halogenation and dehalogenation reactions: pervasive and mechanistically diverse[J]. Chemical Reviews, 2017, 117(8): 5619-5674. |
| [36] |
BLASIAK L C, VAILLANCOURT F H, WALSH C T, et al. Crystal structure of the non-haem iron halogenase SyrB2 in syringomycin biosynthesis[J]. Nature, 2006, 440: 368-371. |
| [37] |
MATTHEWS M L, NEUMANN C S, MILES L A, et al. Substrate positioning controls the partition between halogenation and hydroxylation in the aliphatic halogenase, SyrB2[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(42): 17723-17728. |
| [38] |
SRNEC M, WONG S D, MATTHEWS M L, et al. Electronic structure of the ferryl intermediate in the α-ketoglutarate dependent non-heme iron halogenase SyrB2:contributions to H atom abstraction reactivity[J]. Journal of the American Chemical Society, 2016, 138(15): 5110-5122. |
| [39] |
MATTHEWS M L, CHANG W C, LAYNE A P, et al. Direct nitration and azidation of aliphatic carbons by an iron-dependent halogenase[J]. Nature Chemical Biology, 2014, 10(3): 209-215. |
| [40] |
HUANG J, LI C, WANG B, et al. Selective chlorination of substrates by the halogenase SyrB2 is controlled by the protein according to a combined quantum mechanics/molecular mechanics and molecular dynamics study[J]. ACS Catalysis, 2016, 6(4): 2694-2704. |
| [41] |
WILSON R H, CHATTERJEE S, SMITHWICK E R, et al. Controllable multi-halogenation of a non-native substrate by SyrB2 iron halogenase[J]. ACS Catalysis, 2024, 14(17): 13209-13218. |
| [42] |
PRICE J C, BARR E W, HOFFART L M, et al. Kinetic dissection of the catalytic mechanism of taurine: α-ketoglutarate dioxygenase (TauD) from Escherichia coli[J]. Biochemistry, 2005, 44(22): 8138-8147. |
| [43] |
GRZYSKA P K, APPELMAN E H, HAUSINGER R P, et al. Insight into the mechanism of an iron dioxygenase by resolution of steps following the FeⅣ=O species[J]. Proceedings of the National Academy of Sciences, 2010, 107(9): 3982-3987. |
| [44] |
DE VISSER S P, KUMAR D. Iron-containing enzy- mes: versatile catalysts of hydroxylation reactions in nature[M]. Cambridge, UK: RSC Publishing, 2011.
|
| [45] |
DUEWEL S, SCHMERMUND L, FABER T, et al. Directed evolution of an FeⅡ-dependent halogenase for asymmetric C(sp3)-H chlorination[J]. ACS Catalysis, 2020, 10(2): 1272-1277. |
| [46] |
BVCHLER J, MALCA S H, PATSCH D, et al. Algorithm-aided engineering of aliphatic halogenase WelO5* for the asymmetric late-stage functionalization of soraphens[J]. Nature Communications, 2022, 13(1): 371. |
| [47] |
LI R N, CHEN S L. Mechanism for the halogenation and azidation of lysine catalyzed by non-heme iron BesD enzyme[J]. Chemistry: An Asian Journal, 2022, 17(17): e202200438. |
| [48] |
SMITHWICK E R, WILSON R H, CHATTERJEE S, et al. Electrostatically regulated active site assembly governs reactivity in nonheme iron halogenases[J]. ACS Catalysis, 2023, 13(20): 13743-13755. |
| [49] |
KISSMAN E N, NEUGEBAUER M E, SUMIDA K H, et al. Biocatalytic control of site-selectivity and chain length-selectivity in radical amino acid halogenases[J]. Proceedings of the National Academy of Sciences, 2023, 120(12): e2214512120. |
| [50] |
SLATER J W, LIN C Y, NEUGEBAUER M E, et al. Synergistic binding of the halide and cationic prime substrate of L-lysine 4-chlorinase, BesD, in both ferrous and ferryl states[J]. Biochemistry, 2023, 62(16): 2480-2491. |
| [51] |
SOSA M B, LEEMAN J T, WASHINGTON L J, et al. Biosynthesis of strained amino acids by a PLP-dependent enzyme through cryptic halogenation[J]. Angewandte Chemie International Edition, 2024, 63(31): e202319344. |
| [52] |
DAI L H, ZHANG X, HU Y M, et al. Structural and functional insights into a nonheme iron- and α-ketoglutarate-dependent halogenase that catalyzes chlorination of nucleotide substrates[J]. Applied and Environmental Microbiology, 2022, 88(9): e0249721. |
| [53] |
ZHAI G Q, GONG R, LIN Y X, et al. Structural insight into the catalytic mechanism of non-heme iron halogenase AdaV in 2'-chloropentostatin biosynthesis[J]. ACS Catalysis, 2022, 12(22): 13910-13920. |
| [54] |
NI J, ZHUANG J Y, SHI Y M, et al. Discovery and substrate specificity engineering of nucleotide halogenases[J]. Nature Communications, 2024, 15(1): 5254. |
| [55] |
NEUGEBAUER M E, KISSMAN E N, MARCHAND J A, et al. Reaction pathway engineering converts a radical hydroxylase into a halogenase[J]. Nature Chemical Biology, 2022, 18(2): 171-179. |
| [56] |
KASTNER D W, NANDY A, MEHMOOD R, et al. Mechanistic insights into substrate positioning that distinguish non-heme Fe(Ⅱ)/α-ketoglutarate-dependent halogenases and hydroxylases[J]. ACS Catalysis, 2023, 13(4): 2489-2501. |
| [57] |
徐沛, 汪卫华, 宁洪伟, 等. 人工智能辅助的酶分子改造应用进展[J]. 生物工程学报, 2024, 40(6): 1728-1741. |
| [58] |
赵永耀. 基于机器学习的酶改造方法研究[D]. 南京: 南京邮电大学, 2023.
|