尽管科学家们在早期预防、诊断和药物研发方面做出了努力,癌症仍然是全球第二大死亡原因[1]。在治疗癌症的药物中,甾体类药物是一类重要的药物,如已上市的临床药物醋酸阿比特龙、雌莫司汀磷酸钠、2-甲氧基雌二醇以及依西美坦等。甾体化合物是一类含有复杂多环结构的药物分子,对维持人体正常生理功能极其重要。药物学家通常采用各种方法对甾体结构进行修饰或改变其甾体骨架,从而有效地改变其生物活性并进一步优化其原有的生物功能,以满足更多的医学需求[2, 3]。
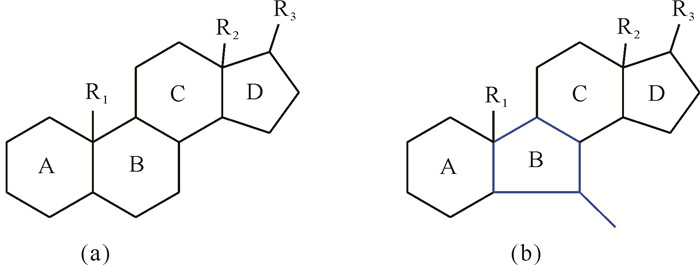
B-Nor甾体化合物可视为自然界中一类罕见的甾体衍生物,与普通甾核结构[6-6-6-5]相比,B-Nor的甾核结构为[6-5-6-5](图 1)。Yokosuka等[4]从燕麦(Avena sativa L.)中分离出12种甾体苷类化合物,其中分离出的B-Nor甾体化合物具有显著的细胞毒活性。Ilkar Erdagi等[5]通过McMurry反应合成B-Nor衍生物,这些衍生物在抗肿瘤等方面表现出潜在的应用前景。
|
| 图 1 普通甾核结构[6-6-6-5](a)和特殊甾核结构[6-5-6-5](b) Fig.1 Ordinary steroid nucleus structure [6-6-6-5](a) and specialized steroid nucleus structure [6-5-6-5](b) |
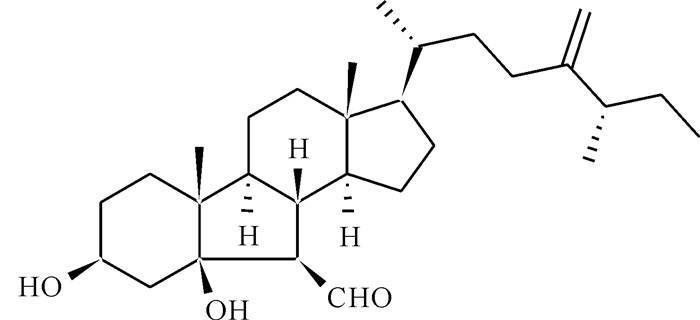
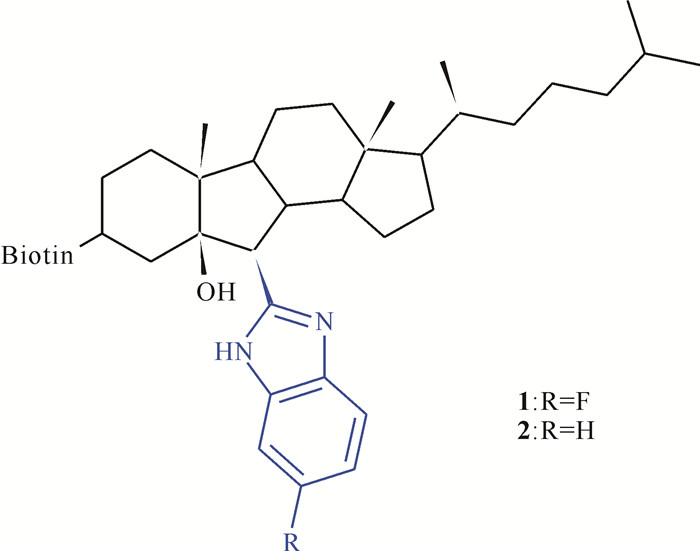
Orostanal[6](图 2)是一种源自日本海洋海绵Stelletta hiwasaensis的化合物,在浓度为10 μg/mL时能有效诱导HL-60细胞凋亡。因此,在前期研究中,本课题组以Orostanal为先导,设计合成了多种系列的B-Nor甾体化合物,并对其抗增殖活性进行筛选[7, 8]。其中,具有胆甾支链结构的B-Nor-6-苯并咪唑基胆甾衍生物展现出显著的细胞毒活性[9]。这些化合物可以通过干扰人卵巢癌细胞(SKOV3细胞)膜结构和细胞代谢功能来抑制细胞增殖[10]。为了深入探讨此类化合物的作用靶点,本课题组将生物素引入胆固醇的3位,获得了一系列胆甾衍生物,并对其进行体外抗肿瘤活性评估[11]。根据前期体外抗肿瘤活性实验数据可知,化合物1和2 (图 3)对SKOV3细胞表现出较好的抑制活性,而对正常肾上皮细胞HEK293T几乎没有毒性,其中化合物1能使SKOV3细胞形态发生变化,将肿瘤细胞增殖阻滞在S期(DNA合成期),并诱导SKOV3细胞进入早期凋亡,在肿瘤细胞的线粒体中诱导促进凋亡的Bax蛋白表达上调,诱导拮抗凋亡的Bcl-2蛋白表达下调,使细胞凋亡[12]。为进一步研究苯并咪唑中不同取代基-F和-H对代谢途径的影响,本研究将化合物1和2分别作用于SKOV3细胞,通过非靶向代谢组学方法分析这两种化合物在SKOV3细胞中的代谢途径,并比较苯并咪唑基团中不同取代基的影响。
|
| 图 2 Orostanal结构 Fig.2 Structure of the Orostanal |
|
| 图 3 化合物1和2结构 Fig.3 Structure of compound 1 and 2 |
1 材料与方法 1.1 试剂与仪器
化合物1和2由本课题组合成[12]。DMEM培养基购自美国Gibco公司;磷酸盐缓冲液(PBS)购自北京索莱宝科技有限公司;胎牛血清、青霉素/链霉素、胰蛋白酶Trypsin EDTA Solution A、胰蛋白酶Trpsin Solution B均购自以色列Biological Industries公司;二氧化碳培养箱MCO-5AC购自日本三洋公司;T25 cm2细胞培养瓶和60 mm细胞培养皿购自比克曼生物试剂有限公司;甲醇、乙腈、乙酸铵和甲酸(纯度为LC-MS级)购自德国CNW Technologies公司;内标:L-2-氯苯丙氨酸(纯度≥98%)购自上海恒柏生物科技有限公司;SKOV3细胞购自上海盖宁生物科技有限公司。液相色谱-串联三重四极杆质谱(LC-MS/MS)分析采用超高效液相色谱(UHPLC)系统(1290,美国Agilent Technologies公司),配备UPLC HSS T3色谱柱(2.1 mm × 100 mm,1.8 μm)及QExactiveTM组合型四极杆OrbitrapTM高分辨质谱仪(QE质谱仪)均购自美国Thermo公司。
1.2 方法 1.2.1 SKOV3细胞的培养配制含有10%胎牛血清、100 U/mL青霉素和0.1 g/L链霉素的DMEM培养基,将SKOV3细胞接种到含有该培养基的培养瓶中,置于37 ℃、5%二氧化碳的二氧化碳培养箱恒温培养至对数生长期备用。
1.2.2 非靶向代谢组学方法(1) 细胞给药及分组
取对数生长期的SKOV3细胞接种到含有DMEM培养基(添加10%胎牛血清、100 U/mL青霉素和0.1 g/L链霉素)的培养皿中,于37 ℃、5%二氧化碳的二氧化碳培养箱中孵育24 h,使其贴壁后进行上药处理,设置对照组和实验组,其中对照组使用未经过药物处理、处于对数生长期的SKOV3细胞所产生的代谢产物,而实验组则是使用浓度均为30 μmol/L的化合物1和化合物2分别作用于SKOV3细胞48 h后的代谢产物。
将浓度均为30 μmol/L的化合物1和化合物2分别作用于SKOV3细胞48 h后,弃去细胞培养基,并用预冷PBS清洗细胞两次。随后加入胰蛋白酶消化SKOV3细胞3 min,加入培养基终止消化后,收集细胞,制备样品。
(2) 提取代谢产物
冷冻干燥后,每个样品加入1 mL萃取溶剂(V乙腈∶V甲醇∶V水=2∶5∶2,含内标L-2-氯苯丙氨酸1 μg/mL)。随后,将样品转移到管中,涡旋30 s,在45 Hz下均质4 min,在冰水浴中超声5 min。均质和超声循环3次,然后在-20 ℃孵育1 h,再以12 000 r/min的速度在4 ℃离心15 min。将上清液转移至LC-MS瓶中,置于-80 ℃低温下保存,以待UHPLC-QE Orbitrap/MS分析。此外,从每个样品中混合等量的上清液制备质量控制(QC)样品。
(3) 色谱质谱测试方法
洗脱剂A为乙腈,洗脱剂B为乙酸铵水溶液(5 mmol/L)。洗脱梯度:1% A,99% B,0 min;1% A,99% B,1 min;99% A,1% B,8 min;99% A,1% B,10 min;1% A,99% B,10.1 min;1% A,99% B,12 min。实验流速为0.5 mL/min,注射量为3 μL。在LC/MS/MS实验过程中,由于QE质谱仪能够在信息依赖基础(IDA)上获取MS/MS光谱,因此实验使用QE质谱仪。在该模式下,根据预先选择的标准,在收集和触发MS/MS光谱采集时,采集软件(Xcalibur 4.0.27, 美国Thermo公司)可以连续评估全扫描测量MS数据。将ESI源条件设置为毛细管温度400 ℃,鞘气流量45 Arb,Aux气流量15 Arb,Full ms分辨率70 000,MS/MS分辨率17 500,NCE模型碰撞能量20/40/60 eV,喷雾电压分别为4.0 kV(正)和-3.6 kV(负)。
1.2.3 数据处理利用内标归一化方法对数据进行分析,将得到的三维数据(峰数、样本名称和归一化峰面积)转移到SIMCA软件包(V14.1, 瑞典Sartorius Stedim data Analytics AB公司)中进行主成分分析(PCA)和正交偏最小二乘法-判别分析(OPLS-DA),PCA显示原始数据分布。从软件中得到的分类参数为R2Y=X和Q2Y=X,采用7次交叉验证方法和排列检验。基于潜在结构判别分析(OPLS-DA)的正交投影构建负荷图(Loading plot),该图显示各变量对两组差异的贡献。为了完善这一分析,首先获得投影中第一主成分(VIP),然后选取VIP值大于“1”的变化代谢物,对剩余变量进行Student′s t检验(P>0.05),剔除两组间变量。用MetaboAnalyst (http://www.metaboanalyst.ca/)和KEGG (http://www.genome.jp/kegg/)等商业数据库进行搜索和分析,数据处理结果由上海百趣生物医学科技有限公司提供。
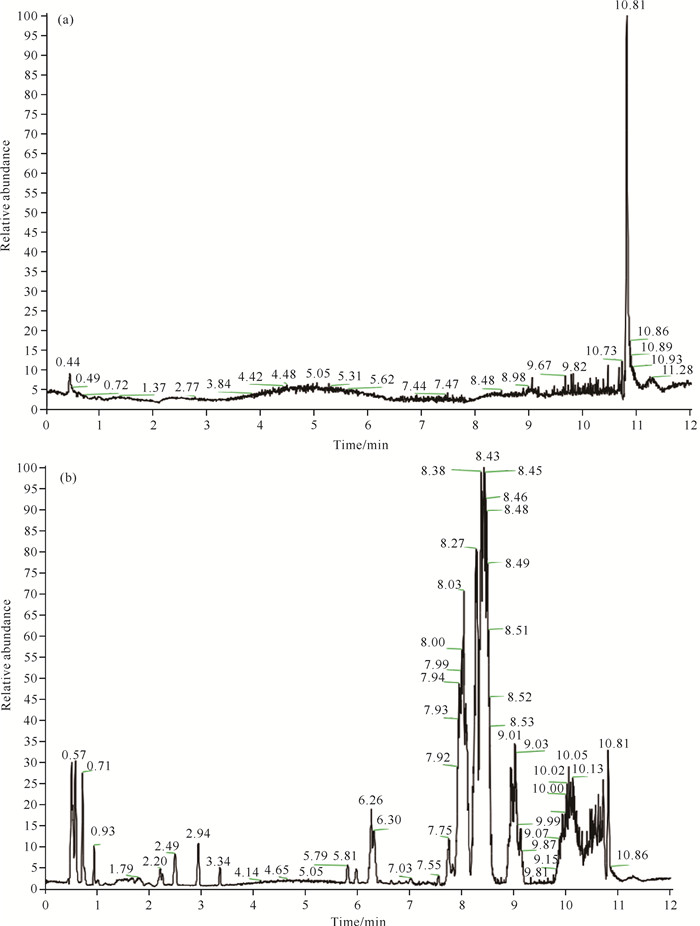
2 结果与分析 2.1 离子流基峰色谱(BPC)经过UHPLC-QE/MS扫描后,得到对照组和QC样品的BPC图。如图 4所示,峰形良好,表明仪器具有良好的分离度;对照样品中无显著色谱峰检出,说明物质残留控制较好,样品间的交叉污染在可控范围内。QC样品色谱峰保留时间和信号强度重叠较好,说明方法具有良好的稳定性和重复性。
|
| 图 4 UHPLC-QE-MS检测空白样品(a)和QC样品(b)的BPC图 Fig.4 BPC plots of UHPLC-QE-MS assay blank sample (a) and QC sample (b) |
2.2 代谢组学
数据经过归一化处理后,进行PCA以揭示其内部结构。通过PCA提取关键的主成分,概括数据的主要特征,从而解释和理解数据中的变量。在得分图上显示组聚类[化合物1组vs对照组,图 5(a)]。OPLS-DA评分图[图 5(b)]清晰区分化合物1组与对照组,且无重叠[R2X=36.1%,R2Y=99.7%,Q2(cum)=73.5%],表明两组之间存在明显的代谢差异。通过置换检验验证模型没有过拟合[图 5(c)]。根据OPLS-DA模型的投影值(VIP>1)和FDR调整后的P值(P < 0.05),识别出31种差异表达代谢物。与对照组相比,化合物1组有2种代谢产物上调,29种代谢产物下调。显著改变的代谢产物包括异金酸、2-羟戊二酸、D-甘露糖-6-磷酸、N-乙酰谷氨酸、磷酸二羟丙酮、CMP-N-乙酰神经氨酸、尿苷5′-二磷酸葡萄糖醛酸和胞苷。热图可视化显示化合物1组和对照组之间存在明显的分离[图 5(d)]。
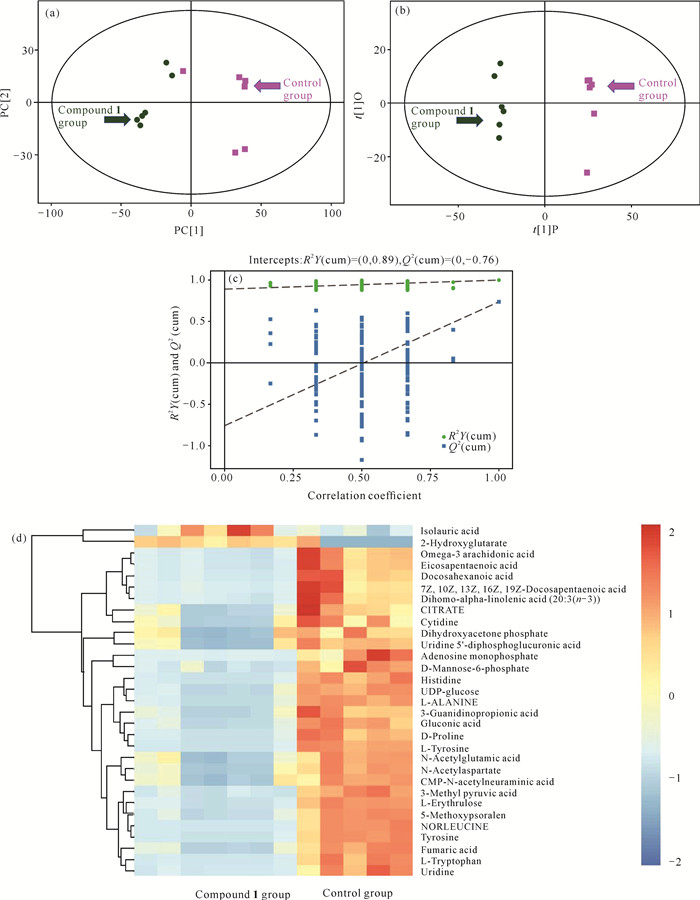
|
| (a) Principal Component Analysis (PCA) Score plots of metabolic profile data for Compound 1 group and control group; (b) Partial least-squares-discriminate analysis (OPLS-DA) score plots of metabolic profiles of compoud 1 and control group; (c) Permutation test on the OPLS-DA model (50 times); (d) Compound 1 hierarchical clustering heat map. 图 5 对照组和化合物1组的样品代谢变化 Fig.5 Analysis of metabolic changes of samples in control group and compound 1 group |
在得分图中,化合物2组表现出明显的组聚类[化合物2组vs对照组,图 6(a)],结果显示其基本处于95%置信区间内。OPLS-DA评分图[图 6(b)]显示化合物2组与对照组区分明显,无重叠,经过药物处理后,两组SKOV3细胞的代谢特征存在明显差异。进行置换检验后发现,原模型R2Y接近1,Q2(cum)的回归线截距小于零,表明本实验建立的模型与样本数据一致,具有良好的稳健性且没有过拟合[图 6(c)]。根据OPLS-DA模型得出的投影值(VIP>1)和FDR调整的P值(P < 0.05),共识别出36种差异表达的代谢产物。与对照组相比,化合物2组中有3种代谢产物上调,33种代谢产物下调。显著改变的代谢产物包括2-羟戊二酸、磷脂酰乙醇胺溶酶16∶0、L-a-溶血磷脂酰丝氨酸、腺苷、尿苷5′-二磷酸葡萄糖醛酸、(+)-12-甲基肉豆蔻酸、柠檬酸盐、胞苷、前列腺素2、油酰乙基酰胺、PS(18∶1(9Z)/0∶0)和N-乙酰谷氨酸。热图可视化进一步显示了化合物2组与对照组之间存在明显的分离[图 6(d)]。
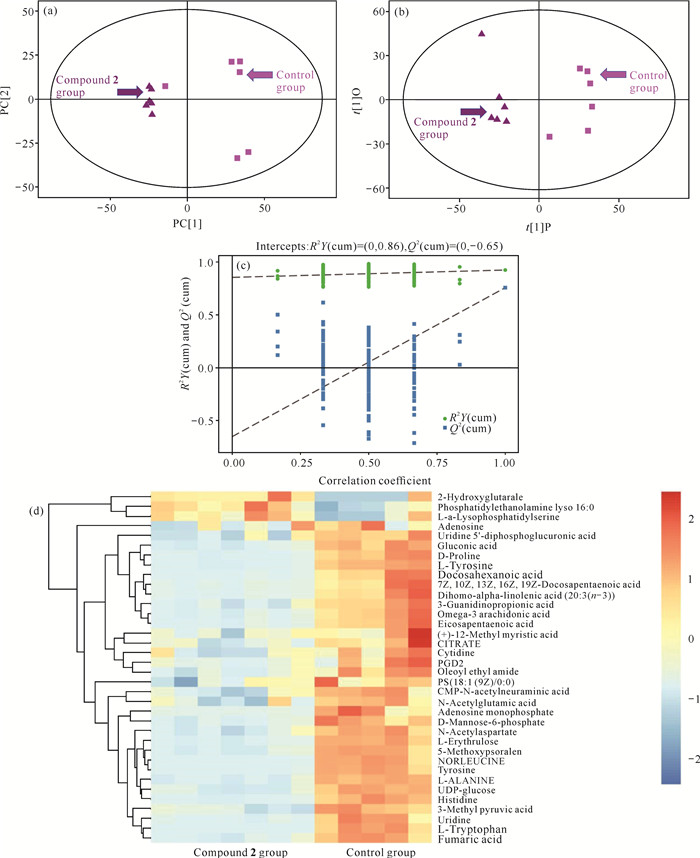
|
| (a) Principal Component Analysis (PCA) score plots of metabolic profile data of compound 2 and control group; (b) Partial least-squares-discriminate analysis (OPLS-DA) score plots of metabolic profiles of compound 2 and control group showing the contribution of variables to the difference between the two groups; (c) Permutation test on the OPLS-DA model (50 times); (d) Compound 2 hierarchical clustering heatmap. 图 6 对照组和化合物2组样品代谢变化分析 Fig.6 Analysis of metabolic changes of samples in control group and compound 2 group |
参考《京都基因与基因组百科全书》[13](KEGG: Kyoto Encyclopedia of Genes and Genomes),通过计算机模拟可生成KEGG通路数据库和KEGG通路图。从图 7(a)可知,化合物1组与对照组相比,氮代谢,丙氨酸、天冬氨酸和谷氨酸代谢,淀粉和蔗糖代谢,氨基糖和核苷酸糖代谢,柠檬酸循环(Citric acid cycle, TCA cycle),氨基酰tRNA代谢差异最大。化合物2组与对照组相比,氮代谢,嘧啶代谢,精氨酸和脯氨酸代谢,氨基酸和核苷酸糖代谢,丙氨酸、天冬氨酸和谷氨酸代谢,淀粉和蔗糖代谢,氨基酰-tRNA代谢和柠檬酸循环差异显著[图 7(b)]。
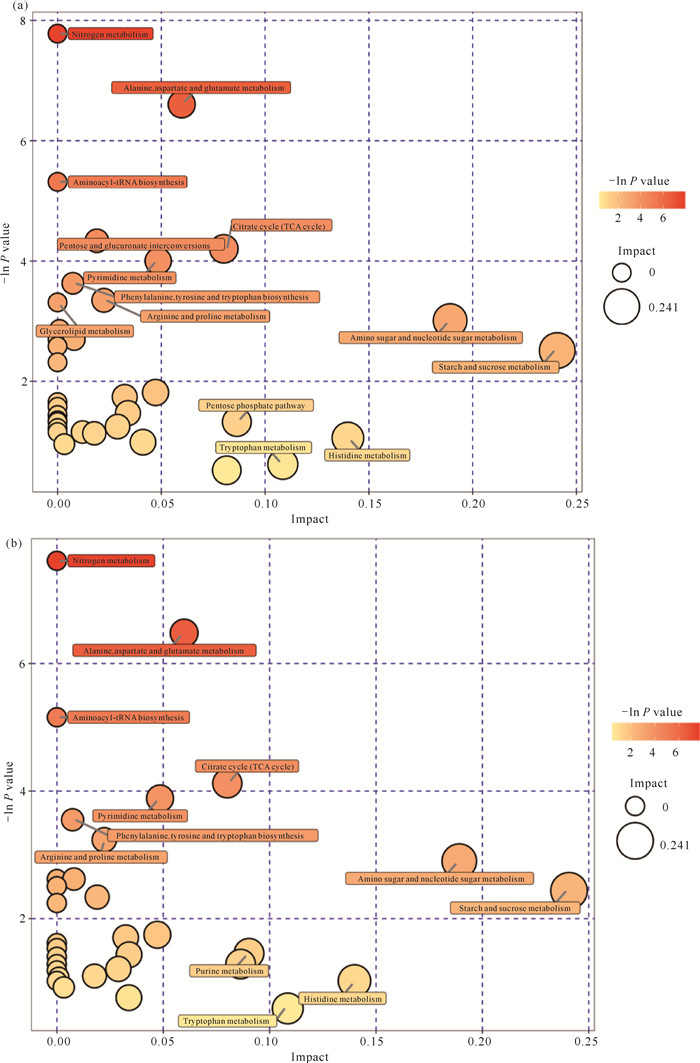
|
| (a) Bubble diagram of metabolic pathway analysis after compound 1 acting on SKOV3 cells; (b) bubble diagram of metabolic pathway analysis after compound 2 acting on SKOV3 cells. 图 7 使用KEGG和MetaboAnalyst进行途径分析的代谢组视图 Fig.7 Metabolome view of pathway analysis using KEGG and MetaboAnalyst |
3 讨论 3.1 丙氨酸、天冬氨酸和谷氨酸代谢
细胞的增殖离不开蛋白质,而蛋白质是由不同种类的氨基酸组成的,异常的氨基酸代谢可能会导致癌症[14]。据报道,丙氨酸、天冬氨酸和谷氨酸的代谢为多种恶性肿瘤生长和生存提供了重要能量来源。此外,谷氨酰胺能够提供用于重要生物物质合成的氮和碳元素,在肿瘤细胞的代谢中具有重要意义[15]。化合物1和2作用于SKOV3细胞后,在丙氨酸、天冬氨酸和谷氨酸的代谢途径中,柠檬酸、N-乙酰-L-天冬氨酸、丙氨酸和富马酸的代谢产物明显减少,柠檬酸的降低未能有效激活β-柠檬酸谷氨酸的合成代谢。同时,N-乙酰-L-天冬氨酸含量减少干扰肿瘤细胞的能量生成,影响其能量供应,从而抑制肿瘤细胞的快速生长和增殖[16],间接影响了嘧啶代谢。然而,丙氨酸和富马酸含量的减少会直接影响天冬氨酸的表达。同时化合物1和2在肿瘤细胞内可有效抑制氮代谢,阻断氨基酸合成和相互转化,因此,细胞增殖过程受到阻滞,进而抑制了肿瘤细胞活性。
3.2 氨基酰tRNA代谢许多疾病的病因都与特定的氨基酰tRNA合成酶有关,如神经病变、癌症、某些代谢疾病和自身免疫性疾病。通过分析化合物的代谢组学途径,化合物1作用于肿瘤细胞后,细胞内部的氨基酰tRNA生物合成过程发生了显著变化,有效抑制了细胞蛋白的合成。由于蛋白质合成受阻,肿瘤细胞增殖过程中断,肿瘤细胞的存活和生长受阻。而经化合物2作用后,在氨基酰tRNA生物合成中,L-丙氨酸、L-组氨酸、L-酪氨酸和L-色氨酸显著下调。这4种氨基酸的减少直接导致其与tRNA结合形成L-丙氨酰-tRNA、L-组氨酰-tRNA、L-酪氨酰-tRNA和L-色氨酰-tRNA的量减少,进而影响这些氨基酸参与的蛋白质合成过程,使肿瘤细胞生长和增殖相关的基因表达受到抑制,并干扰肿瘤细胞生理功能,包括细胞的生长、分裂和各种代谢活动,从而抑制肿瘤细胞的增殖。
3.3 柠檬酸循环线粒体代谢对体内肿瘤的发生发展至关重要[17]。在线粒体中,柠檬酸循环为肿瘤细胞提供能量[18]。化合物1和2作用于SKOV3细胞后,柠檬酸和富马酸含量明显下降,精氨酸生物合成和酪氨酸代谢间接导致富马酸含量下降,从而使柠檬酸循环中断,影响脂肪酸的生物合成和代谢。
3.4 氨基糖和核苷酸糖代谢在氨基糖和核苷酸糖代谢过程中,化合物1和2作用于SKOV3细胞后,UDP-葡萄糖、UDP-葡萄糖酸盐及CMP-N-乙酰神经氨酸盐含量均有明显下降。葡萄糖的减少可导致葡萄糖醛酸酯含量的减少,间接影响抗坏血酸和透明质酸的代谢[19],并间接抑制淀粉和蔗糖的代谢,从而抑制肿瘤细胞的活性。
3.5 精氨酸和脯氨酸代谢精氨酸和脯氨酸代谢是精氨酸、鸟氨酸、谷氨酸、瓜氨酸和脯氨酸的共同代谢途径,是肿瘤代谢变化的关键[20]。脯氨酸是一种重要的氨基酸,可以通过中间代谢产物与谷氨酸相互转化获得,且在细胞内具有多种重要功能,包括稳定蛋白质结构和传导细胞信号等。中间代谢产物P5C由谷氨酸通过P5C合成酶生成[21]。脯氨酸脱氢酶(PRODH)可以将线粒体中的脯氨酸转化为P5C,通过PRODH产生的腺苷三磷酸(ATP)可以维持细胞增殖和体外肿瘤细胞球体的发育[22]。精氨酸是一种促进肿瘤生长的非必需氨基酸,由脯氨酸合成。精氨酸和脯氨酸代谢在调节肿瘤细胞侵袭和肿瘤形成中起着重要作用[23]。经过化合物1和2作用后,精氨酸生物合成代谢中的N-乙酰-L-谷氨酸和富马酸含量显著下降。尿素的合成不能被有效激活,导致精氨酸、鸟氨酸的代谢及柠檬酸循环受到影响,间接影响精氨酸和脯氨酸的代谢途径[24]。
3.6 嘧啶代谢在嘧啶代谢中,尿苷是嘧啶核苷的一种,可能参与肿瘤细胞的核酸合成。胞苷作为嘧啶核苷的组成部分,与尿苷类似,在肿瘤细胞的核酸合成中起作用,其缺乏或代谢异常可能干扰肿瘤细胞的正常生长和分裂。UDP-葡萄糖在细胞代谢中有多种用途:一方面,它可以参与糖基化反应,影响蛋白质的功能和稳定性,这可能与肿瘤细胞表面受体和黏附分子的表达及功能有关,进而影响肿瘤细胞的生长、侵袭和转移。另一方面,UDP-葡萄糖还可能参与细胞内的信号通路调节,对肿瘤细胞的代谢和生存产生影响。化合物2作用于SKOV3细胞后,UDP-葡萄糖、尿苷和胞苷明显下调。肿瘤细胞通常具有较高的增殖速率,需要大量的核酸来支持细胞分裂和生长。尿苷和胞苷的供应减少会影响肿瘤细胞内RNA的合成,进而影响蛋白质的表达和细胞功能。UDP-葡萄糖减少会导致糖基化修饰不足,影响肿瘤细胞表面受体和黏附分子的功能,抑制细胞的增殖和侵袭[25]。
化合物1和2均通过抑制氨基酸代谢、阻断柠檬酸循环、抑制氮代谢及嘧啶代谢等途径来抑制肿瘤细胞增殖,但在具体氨基酸减少种类、氨基糖和核苷酸糖代谢、精氨酸和脯氨酸代谢细节上存在差异,这些差异可能与苯并咪唑取代基不同有关。
综上所述,在非靶向代谢组学分析中,苯并咪唑基团中引入吸电子基团-F原子后,化合物1能有效抑制精氨酸和脯氨酸,使得细胞正常代谢受阻,抑制氮代谢,利用阻断能量代谢的方法抑制RNA代谢,从而抑制肿瘤细胞的氨基酸合成、代谢与增殖活性,进一步中断肿瘤细胞的增殖周期,降低其活性。相较于化合物1,苯并咪唑基团上无取代基团的化合物2可抑制细胞蛋白的合成,进而阻断氨基酸的合成和相互转化,抑制与肿瘤细胞生长和增殖相关的基因表达,影响氮代谢并阻断细胞增殖过程。间接影响肿瘤细胞的能量供应和物质合成,进而影响其RNA的合成以及蛋白质的表达和细胞功能。从结构上看,两个化合物仅存在苯并咪唑取代基中-F和-H的不同,但是在代谢途径上有明显差异,其作用机制值得进一步探讨和研究。
| [1] |
LUO Y, CHENG S D, GAN C F, et al. A new saddle-shaped aza analog of tetraphenylene: atroposelective synthesis and application as a chiral acylating reagent[J]. CCS Chemistry, 2022, 4(9): 2897-2905. DOI:10.31635/ccschem.021.202101486 |
| [2] |
LI E Q, LINDSLEY C W, CHANG J, et al. Molecular skeleton editing for new drug discovery[J]. Journal of Medicinal Chemistry, 2024, 67(16): 13509-13511. DOI:10.1021/acs.jmedchem.4c01841 |
| [3] |
HAMMOUDA M M, ELATTAR K M, RASHED M M, et al. Synthesis, biological activities, and future perspectives of steroidal monocyclic pyridines[J]. RSC Medicinal Chemistry, 2023, 14(10): 1934-1972. DOI:10.1039/D3MD00411B |
| [4] |
YOKOSUKA A, ISHIHARA K, YAMADA T, et al. Steroidal glycosides from the aerial parts of Avena sativa L.and their cytotoxic activity[J]. Journal of Agricultural and Food Chemistry, 2021, 69(48): 14568-14579. DOI:10.1021/acs.jafc.1c05782 |
| [5] |
ILKAR ERDAGI S, YILDIZ U. Synthesis of diosgenin derivatives by A and B ring modifications and low-valent titanium (Ti0)-catalysed McMurry coupling reactions and designing to create novel biological agents[J]. Journal of Molecular Structure, 2022, 1256(15): 132511. |
| [6] |
MIYAMOTO T, KODAMA K, ARAMAKI Y, et al. Orostanal, a novel abeo-sterol inducing apoptosis in leukemia cell from a marine sponge, Stelletta hiwasaensis[J]. Tetrahedron Letters, 2001, 42(36): 6349-6351. DOI:10.1016/S0040-4039(01)01278-3 |
| [7] |
HUANG X T, ZHAN J Y, HUANG Y M, et al. Studies on the interaction between 3-biotinylate-6-benzimidazole B-nor-cholesterol analogs and ct-DNA[J]. New Journal of Chemistry, 2022, 46(19): 9331-9343. DOI:10.1039/D2NJ00896C |
| [8] |
HUANG X T, WU Y L, HUANG Y M, et al. Studies on apoptosis induced by B-norcholesteryl benzimidazole compounds in HeLa cells[J]. Steroids, 2021, 168: 108802. DOI:10.1016/j.steroids.2021.108802 |
| [9] |
CUI J G, QI B, GAN C, et al. Synthesis and in vitro antiproliferative evaluation of some B-norcholesteryl Benzimidazole and Benzothiazole derivatives[J]. Marine Drugs, 2015, 13(4): 2488-2504. DOI:10.3390/md13042488 |
| [10] |
GAN C, HUANG X, WU Y, et al. Untargeted metabolomics study and pro-apoptotic properties of B-norcholesteryl benzimidazole compounds in ovarian cancer SKOV3 cells[J]. The Journal of Steroid Biochemistry and Molecular Biology, 2020, 202: 105709. DOI:10.1016/j.jsbmb.2020.105709 |
| [11] |
ARSHAD T, KHAN K M, RASOOL N, et al. Syntheses, in vitro evaluation and molecular docking studies of 5-bromo-2-aryl benzimidazoles as α-glucosidase inhibitors[J]. Medicinal Chemistry Research, 2016, 25(9): 2058-2069. DOI:10.1007/s00044-016-1614-y |
| [12] |
ZHU Z L, LIU Z P, CUI J G, et al. Apoptosis inducing properties of 3-biotinylate-6-benzimidazole B-nor-cholesterol analogues[J]. Steroids, 2021, 169: 108822. DOI:10.1016/j.steroids.2021.108822 |
| [13] |
KANEHISA M, GOTO S. KEGG: Kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 28(1): 27-30. DOI:10.1093/nar/28.1.27 |
| [14] |
ZHANG Q, LI Q C, LIU S Y, et al. Decreased amino acids in the brain might contribute to the progression of diabetic neuropathic pain[J]. Diabetes Research and Clinical Practice, 2021, 176: 108790. DOI:10.1016/j.diabres.2021.108790 |
| [15] |
LI L, MENG Y, LI Z Y, et al. Discovery and development of small molecule modulators targeting glutamine metabolism[J]. European Journal of Medicinal Chemistry, 2019, 163: 215-242. DOI:10.1016/j.ejmech.2018.11.066 |
| [16] |
MAHEN K K, MARKLEY L, BOGART J, et al. Topical N-phosphonacetyl-l-aspartate is a dual action candidate for treating non-melanoma skin cancer[J]. Experimental Dermatology, 2023, 32(9): 1485-1497. DOI:10.1111/exd.14853 |
| [17] |
CHEN C, ZHENG H, HORWITZ E M, et al. Mitochondrial metabolic flexibility is critical for CD8+ T cell antitumor immunity[J]. Science Advances, 2023, 9(49): eadf9522. DOI:10.1126/sciadv.adf9522 |
| [18] |
CHO S, SONG N, CHOI J Y, et al. Effect of citric acid cycle genetic variants and their interactions with obesity, physical activity and energy intake on the risk of colorectal cancer: results from a nested case-control study in the UK biobank[J]. Cancers, 2020, 12(10): 2939. DOI:10.3390/cancers12102939 |
| [19] |
SALAU V F, ERUKAINURE O L, KOORBANALLY N A, et al. Kolaviron modulates dysregulated metabolism in oxidative pancreatic injury and inhibits intestinal glucose absorption with concomitant stimulation of muscle glucose uptake[J]. Archives of Physiology and Biochemistry, 2023, 129(1): 157-167. DOI:10.1080/13813455.2020.1806331 |
| [20] |
PANOSYAN E H, LIN H J, KOSTER J, et al. In search of druggable targets for GBM amino acid metabolism[J]. BMC Cancer, 2017, 17(1): 162. DOI:10.1186/s12885-017-3148-1 |
| [21] |
CHALECKA M, KAZBERUK A, PALKA J, et al. P5C as an interface of proline interconvertible amino acids and its role in regulation of cell survival and apoptosis[J]. International Journal of Molecular Sciences, 2021, 22(21): 11763. DOI:10.3390/ijms222111763 |
| [22] |
CARVALHO T M, CARDOSO H J, FIGUEIRA M I, et al. The peculiarities of cancer cell metabolism: a route to metastasization and a target for therapy[J]. European Journal of Medicinal Chemistry, 2019, 171: 343-363. DOI:10.1016/j.ejmech.2019.03.053 |
| [23] |
XU S, YAO H, LUO S, et al. A novel potent anticancer compound optimized from a natural oridonin scaffold induces apoptosis and cell cycle arrest through the mitochondrial pathway[J]. Journal of Medicinal Chemistry, 2017, 60(4): 1449-1468. DOI:10.1021/acs.jmedchem.6b01652 |
| [24] |
MATOS A, CARVALHO M, BICHO M, et al. Arginine and arginases modulate metabolism, tumor microenvironment and prostate cancer progression[J]. Nutrients, 2021, 13(12): 4503. DOI:10.3390/nu13124503 |
| [25] |
TEOH S T, OGRODZINSKI M P, LUNT S Y. UDP-glucose 6-dehydrogenase knockout impairs migration and decreases in vivo metastatic ability of breast cancer cells[J]. Cancer Letters, 2020, 492(1): 21-30. |



