2. 广西科学院生物科学与技术研究所,非粮生物质能技术全国重点实验室,国家非粮生物质能源工程技术研究中心,广西南宁 530007
2. National Key Laboratory of Non-Food Biomass Energy Technology, National Engineering Research Center for Non-Food Biorefinery, Institute of Biological Science and Technology, Guangxi Academy of Sciences, Nanning, Guangxi, 530007, China
蚯蚓(Earthworm)属于环节动物门(Annelida)寡毛纲(Oligochaeta)动物,作为土壤生态系统重要物种之一,蚯蚓对提升土壤环境质量、保护土壤生态系统和生物多样性具有重要作用。据统计,2019年之前全世界已发现3 000-3 500种蚯蚓[1-3],其中我国记录在册的有640种[4],是全球报道蚯蚓物种数最丰富的国家之一。中国也是较早利用蚯蚓的国家,其被用作中药材的历史可以追溯到《神农本草经》[5]。此外,《本草纲目》[6]也详细记载并阐述了蚯蚓的药用性能,明确其药性寒凉、味咸,具有降压平喘、解热镇痛、抗凝血、抗血栓、抗肿瘤等药理作用。参环毛蚓(Amynthas aspergillum),亦名参状远盲蚓,系中国南方广泛分布的蚯蚓物种,因主要产于广东、广西两地,故称广地龙。该物种与栉盲环毛蚓(Amynthas pectiniferus)、通俗腔蚓(Metaphire vulgaris)、威廉腔蚓(M.guillemi)一同被收录于《中华人民共和国药典2020年版一部》地龙药材物种名单之中[7]。
长期以来,广地龙的获取主要依赖于野外捕捉。近年来,中药材市场对广地龙的需求呈现增长态势,然而同时却面临着野生资源数量下降及产品在数量和质量方面双提升需求之间的矛盾,这对参环毛蚓药材的生产与发展形成了极大的制约。为了加强野生资源的保护与利用,国家林业和草原局于2023年7月发布了新修订的《有重要生态、科学、社会价值的陆生野生动物名录》[8],上述4个蚯蚓物种(参环毛蚓、栉盲环毛蚓、通俗腔蚓和威廉腔蚓)均被收录其中。因此,开展参环毛蚓遗传资源保护工作,探究其选育与繁殖技术,并构建科学合理的良种培育及生产推广技术体系,是保障参环毛蚓道地药材可持续发展的必由之路。然而,目前多数研究主要集中在参环毛蚓道地药材的药效鉴定[9]以及真伪甄别[10]等方面,仅对皮质远盲蚓(A.corticis)、安德爱胜蚓(Eisenia andrei)、赤子爱胜蚓(E.fetida)和通俗腔蚓4种蚯蚓进行了全基因组测序并完成组装[11-15]。虽然已有的全基因组测序数据为认识蚯蚓多样性及其环境适应能力、再生机制提供了新的视角,但不同物种之间往往差异较大。参环毛蚓作为一个自然种群,具有个体大、野性强、人工养殖条件下易逃逸、繁殖力低等特性,目前暂无大规模人工养殖,因此对其人工驯化、选育有着迫切需求。无论是进行参环毛蚓遗传资源调查、种质资源保护,还是开展参环毛蚓养殖与种群结构优化,都需要依赖组学的研究基础,因而当前参环毛蚓基因组研究工作极为重要。为此,本研究采用流式细胞术和高通量测序技术对参环毛蚓基因组大小进行估算,对所得的基因组数据进行K-mer分析、基因组预测及注释,旨在为后续全基因组精细测序和药效分析提供参考依据和基因资源。
1 材料与方法 1.1 样本的采集与制备本研究中的参环毛蚓样品来自玉林博白传统产区的野外采集。对采集样品形态初步鉴定后,剪取尾部肌肉组织用于基因组DNA提取,进行COXI片段的分子鉴定。PCR反应体系为25 μL,PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s、50 ℃退火30 s,72 ℃延伸1 min,共35个循环;72 ℃延伸10 min。线粒体COXI片段扩增引物为COI-F:ATTCTACAAACCATAAAGACATTGG;COI-R:TACACTTCTGGGTGTCCAAAGAATCA。
1.2 方法 1.2.1 流式细胞术测定参环毛蚓基因组大小将肌肉组织样品置于0.8 mL预冷的mGb解离液[45 mmol/L MgCl2·6H2O、20 mmol/L MOPS、30 mmol/L柠檬酸钠、1% (W:V) 聚乙烯吡咯烷酮(PVP) 40、0.2% (V:V) Tritonx-100、10 mmol/L EDTA-Na2、20 μL/mL β-巯基乙醇]中,切碎后在冰上静置10 min,用40 μm孔径滤网过滤,得到细胞核悬浮液。在细胞核悬浮液中加入碘化丙啶(PI)染液和RNAase溶液,使其工作浓度均为50 μg/mL,混匀后置于冰上避光染色1 h。分别使用鸡血细胞(基因组大小为1.06 Gb)和鹅掌楸(Liriodendron chinense,基因组大小为1.8 Gb)作为内参,将待测样品的悬浮液和内参样品的悬浮液按2∶1比例混合,利用BD FACScalibur流式细胞仪(北京焕彩元合科技有限公司)对染色后的细胞核悬浮液样品上机检测,采用488 nm蓝光激发,检测PI的发射光荧光强度,每次检测收集10 000个颗粒,变异系数(CV)控制在5%以内,使用Modifit 3.0作图分析[16-18],计算当前参环毛蚓的基因组大小,公式如下:待测样品DNA含量=内参DNA含量×待测样品的荧光强度/内参样品的荧光强度。
1.2.2 基因组测序和质量评估通过MGI(华大)平台,构建270-500 bp的小片段文库,对文库进行双端测序。使用SOAPnuke v 2.1.8[19]对原始测序数据进行过滤,去掉低质量、存在接头污染和PCR duplication的测序片段,剩余用于后续分析。
1.2.3 基因组大小、重复序列和杂合率预测选取K值为21,使用Jellyfish[20]计算K-mer分布,使用GenomeScope v 1.0[21]统计K-mer分布曲线,评估基因组大小、重复序列含量和杂合度。
1.2.4 基因组初步组装及其完整性评估使用MaSuRCA v 4.1.1[22]对过滤后的测序数据进行全基因组的组装,以metazoa_odb10数据库为核心基因集,使用BUSCO 5.7.1(http://busco.ezlab.org/)评估组装完整性。
1.2.5 重复序列分析使用同源序列比对(Homolog)和从头预测(De novo)两种方法对重复序列进行注释。具体方法如下:以RepBase v 23.06(https://www.girinst.org/repbase/)作为重复序列的比对数据库,使用RepeatMasker(http://www.repeatmasker.org/)和RepeatProteinMask 4.1.1 (http://www.repeatmasker.org/RepeatModeler/)识别与已知重复序列相似的序列,并对其进行分类。De novo方法主要利用RepeatModeler和LTRharvest[23]先建立De novo重复序列库,通过RepeatMasker对重复序列或转座子自身的序列或结构特征进行构建,使用Tandem repeats finder工具[24]寻找基因组中的串联重复序列。
1.2.6 基因预测和注释使用MAKER v 3.01.03[25]进行基因预测,用于注释的数据集有转录组数据及皮质远盲蚓、秀丽隐杆线虫(Caenorhabditis elegans)、安德爱胜蚓与赤子爱胜蚓近源物种蛋白数据。对参环毛蚓基因组进行blast比对(-p tblastn -e 1e-5 -F -m 8),然后使用GeneWise[26]进行基因预测,从预测结果中随机挑选结构完整的基因2 000个,用于AUGUSTUS[27]与SNAP[28]参数训练。转录组数据首先通过HISAT v 2.2.1[29]进行比对,然后使用StringTie v 2.2.1[30]进行构建,最后使用MAKER进行整合得到最终基因集,以保证最终基因预测的可靠性。KEGG (https://www.genome.jp/kegg/)、SwissProt and TrEMBL数据库(https://www.uniprot.org/)用于基因功能注释。InterProScan (http://www.ebi.ac.uk/interpro/download/)用于GO功能分类分析。
1.2.7 线粒体的组装与系统发育树构建使用GetOrganelle[31]进行线粒体组装,MitoFinder[32]用于线粒体基因组注释。从NCBI数据库中下载当前蚯蚓的线粒体基因组数据,使用PhyloSuite(v 1.2.3)[33]进行线粒体多基因合并后,用于构建线粒体的系统发育树。
1.2.8 基因组的系统发育树构建从NCBI数据库获得皮质远盲蚓、通俗腔蚓与安德爱胜蚓的基因组数据,使用OrthoFinder[34]寻找直系同源基因,MAFFT[35]用于基因序列的多重比对,Gblocks[36]用于去除比对序列差异较大的区域,KaKs_Calculator 3.0[37]用于计算不同直系同源基因间的Ka、Ks及Ka/Ks比值,R用于不同数据的可视化显示。
2 结果与分析 2.1 流式细胞术检测的基因组大小COXI基因的PCR扩增产物大小为659 bp,覆盖COXI基因编码序列(CDS)的38-697位点,占全长的42.8%(图 1)。通过对NCBI数据库的检索,获得与其序列一致性(Identity)最高的物种为参环毛蚓,共获得57条被注释为参环毛蚓的片段,一致性在90.07%到99.22%之间。此外,参环毛蚓与A.longicaeca、加州腔蚓(M.californica)序列一致性较高,分别为85.36%与84.45%。流式细胞术与测序检测表明,使用鸡血细胞作为内参时,计算得到当前参环毛蚓的基因组大小为1.11 Gb,变异系数为0.86%;使用鹅掌楸作为内参时,计算得到其基因组大小为0.98 Gb,变异系数为1.46%。因此估计当前参环毛蚓的基因组大小为1 Gb左右。

|
| 图 1 COXI基因的测序序列 Fig.1 Sequence of COXI gene |
2.2 参环毛蚓基因组测序数据统计及基因组特征
通过对原始测序数据的统计,插入的DNA测序片段平均长度为350 bp,对数据过滤后获得150 bp的reads共44.21 Gb,其中Q20质量的reads占比为98.84%,GC含量为40.34%。使用GenomeScope对K-mer数据进行统计,其拟合度在93.893 2%和98.307 0%之间,表明模型对实际数据的拟合较好。由于只使用了二代测序数据,单倍体基因组的预测长度仅为641 Mb左右,其中重复序列达到329 Mb,占全长的51.3%左右,并且杂合度高(约1.68%),表明种群具有较高的遗传多样性(表 1)。这种杂合度也反映在K-mer频率分布图上(图 2),显示在主峰对应的深度1/2处有着明显的杂合峰。
| 项目 Item |
杂合度/% Heterozygosity/% |
单倍体长度/bp Haploid length/bp |
重复序列长度/bp Repeat length/bp |
独特序列长度/bp Unique length/bp |
拟合度/% Fiting/% |
错误率/% Error rate/% |
| Min | 1.684 91 | 641 566 167 | 329 078 706 | 312 487 461 | 93.893 2 | 0.167 598 |
| Max | 1.686 62 | 641 702 783 | 329 148 781 | 312 554 002 | 98.307 0 | 0.167 598 |
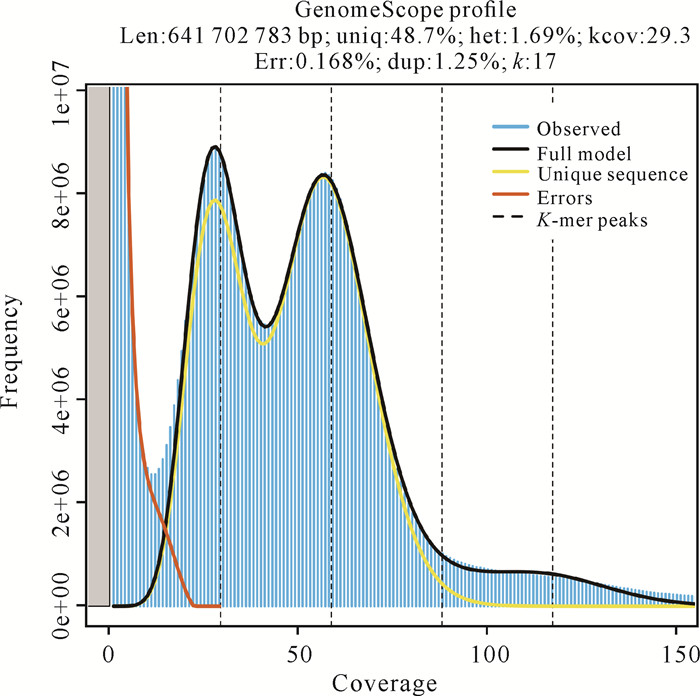
|
| 图 2 K-mer分布图 Fig.2 Distribution map of K-mer |
2.3 参环毛蚓基因组组装和质量评估
使用MaSuRCA对参环毛蚓的二代测序数据进行从头组装,共获得全长约为765.69 Mb的Contig和766.18 Mb的Scaffold,其中Contig与Scaffold序列最长均为69 772 bp,而Contig和Scaffold的N50分别为4 755 bp与5 254 bp(表 2)。BUSCO评估结果显示,基因组序列中组装出76.7%的BUSCO核心基因(表 3)。
| 项目 Item |
Contig | Scaffold | |||
| 片段大小/bp Size/bp |
数量 Number |
片段大小/bp Size/bp |
数量 Number |
||
| N10 | 14 118 | 403 | 15 534 | 3 677 | |
| N20 | 9 927 | 10 610 | 11 015 | 9 621 | |
| N30 | 7 555 | 19 511 | 8 416 | 17 640 | |
| N40 | 5 979 | 30 944 | 6 615 | 27 942 | |
| N50 | 4 755 | 45 341 | 5 254 | 40 976 | |
| N60 | 3 759 | 63 487 | 4 131 | 57 454 | |
| N70 | 2 902 | 86 663 | 3 171 | 78 624 | |
| N80 | 2 105 | 117 570 | 2 299 | 106 875 | |
| N90 | 1 309 | 163 128 | 1 418 | 148 848 | |
| Max length | 69 772 | 69 772 | |||
| Total number | 269 137 | 249 900 | |||
| Total length | 765 693 575 | 766 183 714 | |||
| GC ratio/% | 40.66 | 40.66 | |||
| Note: total number and total length are the number and length of fragments of all assembly contig and scaffold. | |||||
| 类型 Types |
数量 Number |
百分比/% Percentage/% |
| Complete BUSCOs (C) | 732 | 76.7 |
| Complete and single-copy BUSCOs (S) | 631 | 66.1 |
| Complete and duplicated BUSCOs (D) | 101 | 10.6 |
| Fragmented BUSCOs (F) | 133 | 13.9 |
| Missing BUSCOs (M) | 89 | 9.4 |
| Total BUSCO groups searched | 954 |
2.4 参环毛蚓基因组重复序列分析结果
通过Homolog(Trf、RepeatMasker、RepeatProteinmask数据库)与De novo两种方法对参环毛蚓基因组的重复序列进行分析,分别获得40.79、59.77、57.04、365.67 Mb重复序列,对不同方法获取的重复序列合并后共获得总长度为386.28 Mb的重复序列,占全基因组组装序列的50.42%。在该基因组中,重复序列主要以散在重复(Interspersed repeats)为主,因此进一步对重复序列中转座元件(TE)进行不同类型的分类,详细统计数据见表 4。其中,长末端重复序列(Long Terminal Repeated,LTR)是最丰富的重复元件,占基因组的19.86%;其次是长散在重复元件(Long Interspersed Nuclear Elements,LINE),占基因组的12.48%。同时,对不同的TE序列分歧度进行分析,图 3(a)显示了以Repbase为库,通过RepeatMasker注释得到的TE分歧度分布图,其中重复序列主要以低分歧度的DNA转座子为主,其次以LINE为主;而通过De novo方法预测得到的重复序列主要以LTR为主,其次是LINE与DNA转座子[图 3(b)]。
| 转座元件 TE |
长度/bp Length/bp |
占比/% Percentage/% |
| DNA transposons | 64 488 599 | 8.420 |
| LINE | 95 609 592 | 12.480 |
| SINE | 272 483 | 0.036 |
| LTR | 152 164 593 | 19.860 |
| Other | 29 706 | 0.004 |
| Unknown | 168 529 890 | 21.990 |
| Note: SINE means short interspersed nuclear elements. | ||
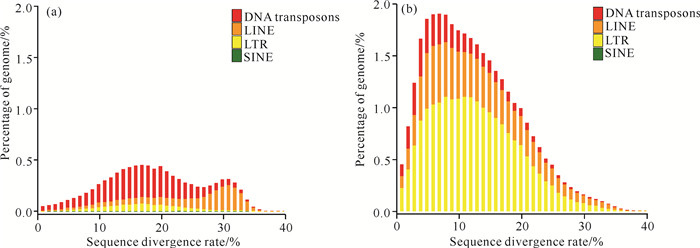
|
| (a) The divergence distribution map of Transposable Elements (TE) obtained through RepeatMasker annotation using Repbase as a library.The x-axis represents the divergence of TE sequences annotated in the genome compared to the corresponding sequences in RepBase, while the y-axis indicates the percentage of TE sequences in the genome at that level of divergence.Different TEs are indicated by different colors.(b) The divergence distribution map of TE predicted by the De novo method. 图 3 4种转座元件分歧度分布 Fig.3 Divergence distribution of four TE sequences |
2.5 参环毛蚓基因预测和注释结果
通过MAKER对基因预测数据集进行整合,共获得27 864个预测基因,其平均基因长度为5 094.78 bp,平均编码序列长度为1 404.87 aa,平均外显子数量为6.16个,平均外显子长度是228.01 bp,平均内含子长度为622.07 bp。对当前基因预测数据集进行功能注释,27 597个基因(99.04%)能在数据库中获得注释信息,其中26 610个基因(95.50%)在NR数据库中获得注释,18 646个基因(66.92%)在KEGG数据库中获得注释,17 305个基因(62.11%)在GO数据库获得注释(表 5)。被KEGG数据库所注释的基因主要分布在信号转导(Signal transduction)、内分泌系统(Endocrine system)和传染疾病(Infectious disease)相关分类中,分别有9 933(35.6%)、5 776(20.7%)与4 718(16.9%)个基因,与细胞膜上的受体、信号分子传导、激素分泌、传递和调节有着重要的关系[图 4(a)]。被GO数据库注释的基因主要分布在细胞过程(Cellular process)、细胞解剖实体(Cellular anatomical entity)和结合(Binding)功能分类中,分别有12 029(43.2%)、12 087(43.3%)与11 241(40.3%)个基因,与细胞内的化学反应、信号传导、信号传导通路和分子间互作相关,详细结果见表 5与图 4(b)。
| 数据库 Database |
注释数量 Number of annotation |
比例/% Percentage/% |
| NR | 26 610 | 95.50 |
| Swissport | 16 755 | 60.13 |
| KEGG | 18 646 | 66.92 |
| TrEMBL | 26 260 | 94.24 |
| Interpro | 23 397 | 83.97 |
| GO | 17 305 | 62.11 |
| Annotated gene | 27 597 | 99.04 |
| Total | 27 864 |
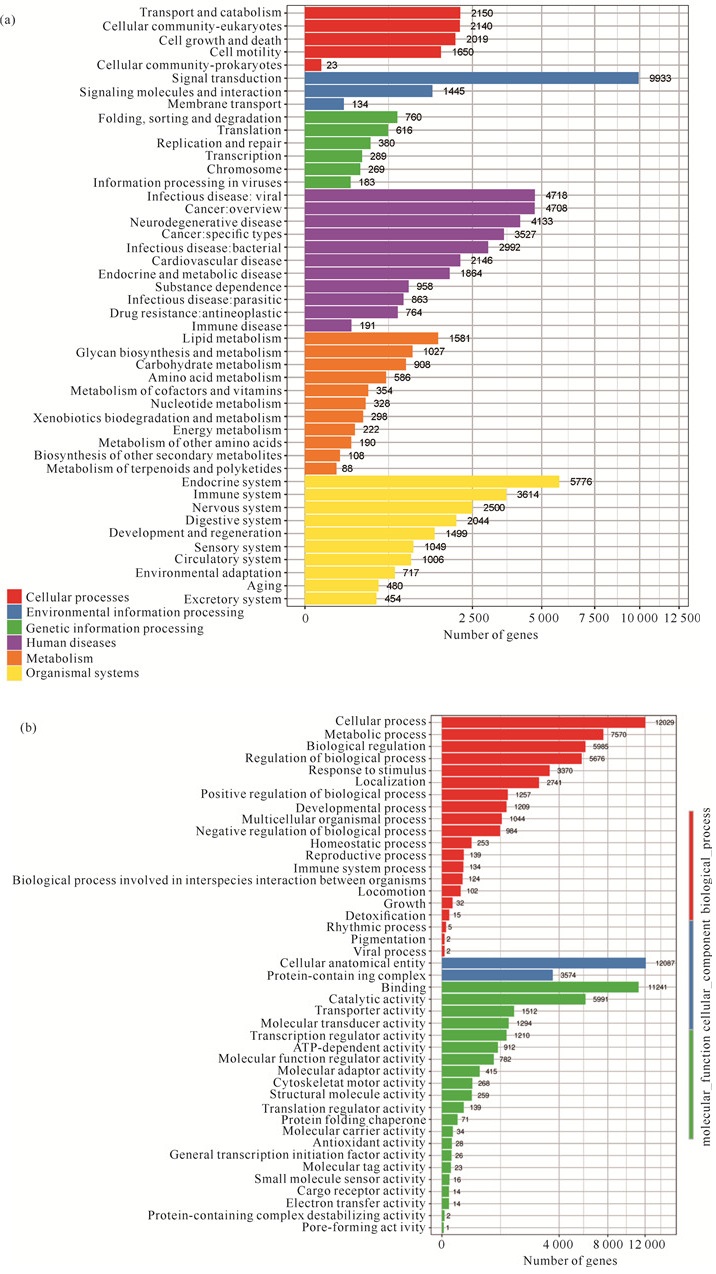
|
| (a) KEGG gene annotation analysis; (b) GO secondary node annotation classification statistics chart. 图 4 参环毛蚓基因预测和注释 Fig.4 Gene prediction and annotation of Amynthas aspergillum |
2.6 参环毛蚓线粒体基因组遗传距离与系统发育分析结果
通过NCBI数据库检索,共获得有完整线粒体基因组的巨蚓科(Megascolecidae)蚯蚓数据106条,归为58个蚯蚓物种。通过基因序列提取,对CytB、COXI等13个基因的种内和种外的遗传距离进行多基因联合,结果如图 5(a)所示,在巨蚓科内,蚯蚓种内的遗传距离(p-distance)有着较宽的分布,从0-15.1%均有,其中壮伟远盲蚓(A.robustus)显示最大的种内差距(15.1%),其次是Duplodicodrilus acinctus(13.6%)、Pheretima kutamaensis(10.9%),而参环毛蚓种内遗传距离为1.9%。不同种间的平均遗传距离为19.5%,参环毛蚓与广布物种加州腔蚓遗传距离较小(16.0%),其次是同属于广西广泛分布的两个蚯蚓物种标记远盲蚓(A.masatakae, 16.2%)和壮伟远盲蚓(16.2%)[图 5(b)]。参环毛蚓与广西广泛分布的蚯蚓其他物种,如远盲属(Amynthas)的简洁远盲蚓(A.gracilis)、毛利远盲蚓(A.morrisi)、皮质远盲蚓的遗传距离分别为19.3%、19.5%与19.8%。此外,参环毛蚓与栉盲环毛蚓、通俗腔蚓、威廉腔蚓的遗传距离均大于平均值19.5%,分别为19.8%、21.0%与20.9%。不同基因的遗传距离计算结果表明,线粒体基因组中不同基因的平均遗传距离有所不同,其中最小的为COX2 (15.1%),最大为ATPase8 (26.4%),这与多基因联合系统发育树的构建结果一致。在种群水平上,本研究测序的个体能很好地与其他参环毛蚓个体聚类,在种间参环毛蚓系统发育关系较为明确(图 6)。

|
| (a) Differences in genetic distance among different earthworm individuals in Megascolecidae; (b) Genetic distance density plot of different mitochondrial genes. 图 5 参环毛蚓线粒体基因组遗传距离与系统发育分析 Fig.5 Genetic distance and phylogenetic analysis of mitochondrial genomes in Amynthas aspergillum |
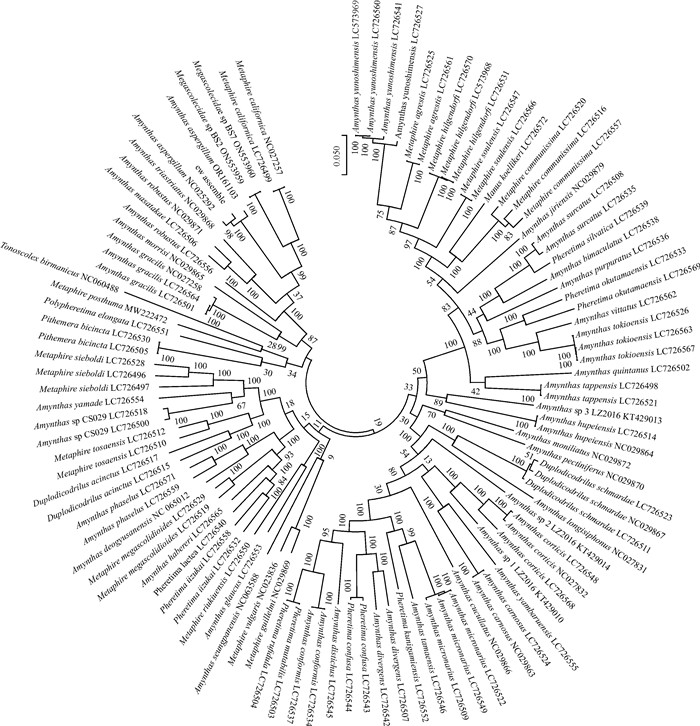
|
| 图 6 巨蚓科蚯蚓内系统发育树 Fig.6 Phylogenetic tree of Megascolecidae earthworms |
2.7 参环毛蚓基因家族鉴定及其遗传距离
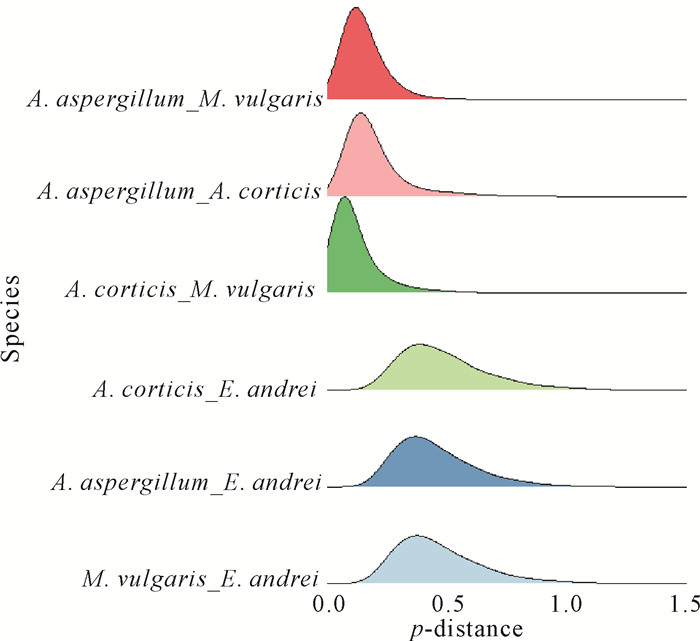
从数据库中分别下载皮质远盲蚓、通俗腔蚓的基因组数据,以安德爱胜蚓作为外群,通过两物种相互之间的直系同源基因的搜索与比对,4个物种基因组共获得37 405个直系同源基因,平均两个物种间有6 234个基因参与遗传距离的分析。结果显示参环毛蚓与通俗腔蚓、皮质远盲蚓平均遗传距离分别为15.64%、20.03%(图 7),与正蚓科(Lumbricidae)物种安德爱胜蚓平均遗传距离为45.32%。这与通过线粒体基因组计算的平均遗传距离不一致,基于线粒体基因计算,参环毛蚓与通俗腔蚓、皮质远盲蚓的平均遗传距离分别为21.00%、19.80%。
|
| 图 7 直系同源基因鉴定及遗传距离分布 Fig.7 Identification of orthologous genes and distribution of genetic distance |
3 讨论
目前,蚯蚓的研究多集中在地理分布、多样性、生态系统服务功能、生态毒理学、天然免疫、药用价值以及生态习性等领域。近年来,有关蚯蚓在土壤碳循环[38, 39]、微生物群落[40-43]、环境污染物降解[44-46]、蚯蚓基因组等方面的研究逐渐增多,特别是多组学技术,如基因组、转录组、单细胞测序、蛋白组学在蚯蚓研究上的应用,使人们对蚯蚓基因组、基因功能、适应与进化、组织再生等方面的认识有了明显的提升[15]。基因组水平上的解析为认识蚯蚓多样性、系统分类、蚯蚓隐存种(Cryptic species)鉴定、新属种发现提供了强大工具,为蚯蚓环境适应能力、再生机制、遗传资源保护与利用、生物资源开发等提供基础数据。目前已对4种蚯蚓(皮质远盲蚓、安德爱胜蚓、赤子爱胜蚓与通俗腔蚓)进行了全基因组测序并完成组装[11-15]。Shao等[13]通过对安德爱胜蚓基因组的研究发现,TE占整个基因组的56.72%,其中DNA转座子、LINE、LTR、SINE、Unknown分别占14.18%、12.42%、2.27%、0.56%、21.55%,LINE2相比其他代表性后生动物物种有显著的扩张,在再生过程中,含有LINE2的差异表达基因(DEGs)的比例显著高于非DEGs。在本研究中,参环毛蚓重复序列占全基因组组装序列的50.42%,具体为DNA转座子(8.42%)、LINE(12.48%)、LTR(19.86%)、SINE(0.036%)、Unkown(21.99%),其中LINE占比与安德爱胜蚓基因组中的相当,这为研究蚯蚓的再生机制提供了新基因数据,后续可通过更多、更完整的蚯蚓基因组比较,阐述蚯蚓物种的再生机制异同。
此外,通过对参环毛蚓基因组的预测共获得27 864个基因,且99.04%基因有功能注释,其中大部分基因与信号转导、内分泌系统、传染疾病相关,这为开发天然药物提供丰富的物质基础,同时对参环毛蚓的深加工与副产物利用有着重要的指导作用,如可以通过对参环毛蚓基因组中蚓激酶相关酶系的挖掘,发现有潜在开发价值的酶系。通过基因组与线粒体基因组的分析表明,参环毛蚓与通俗腔蚓、皮质远盲蚓物种在基因组水平上的平均遗传距离分别为15.64%、20.03%,在线粒体基因组水平上的平均遗传距离分别为21.00%、19.80%,这种线粒体基因组与核基因组之间的遗传距离异质性,可能与皮质远盲蚓基因组三倍体特征有关。基因功能分析显示,皮质远盲蚓基因组扩张的基因家族主要与应激/防御、免疫系统与生殖等功能相关。这类有关蚯蚓组学水平的研究为参环毛蚓的研究提供了丰富的数据,例如皮质远盲蚓作为外来物种,在广西广泛分布,其强大的适应力、免疫与繁殖力等性状对参环毛蚓的人工繁育具有借鉴意义。因此,参环毛蚓的多组学研究对理解蚯蚓的生长、发育、繁殖和环境适应性相关遗传机制有着重要的作用。
基于线粒体基因组的分析表明参环毛蚓与加州腔蚓、标记远盲蚓、壮伟远盲蚓遗传距离最近,分别为16.0%、16.2%、16.2%,这4个物种都在广西地区广泛分布,有着相同的栖居地。此外,加州腔蚓属于腔蚓属(Metaphire),结合之前参环毛蚓与通俗腔蚓、皮质远盲蚓基因组比较分析结果,在核基因组上,参环毛蚓与通俗腔蚓更为接近。以上数据表明,无论是基于形态和解剖特征的传统分类学,还是基于线粒体谱系的分类,单独使用时都有局限性。为更准确地进行物种划分,需要从分子、形态学、生态学等多方面整合信息,获取更多蚯蚓物种的核基因组数据,这将有利于全面厘清蚯蚓物种间的相互关系及进化历程。
4 结论本研究结果表明参环毛蚓基因组为1 Gb左右,其重复序列含量为50.42%,杂合率为1.68%。这些数据显示该种群具有较高的遗传多样性,其基因组属于高重复、高杂合的基因组。鉴于此,在进一步基因组组装中,应适当增加其测序深度,以确保获取更为精准、完整的基因组信息。通过预测,该基因组共获得27 864个基因,且99.04%基因有功能注释,其中大部分基因与信号转导、内分泌系统、传染疾病相关。通对线粒体基因组的分析,参环毛蚓种内遗传距离为1.9%,与广布物种加州腔蚓遗传距离较小(16.0%),其次是标记远盲蚓(16.2%)、壮伟远盲蚓(16.2%)。通过基因组水平的比较,参环毛蚓与通俗腔蚓、皮质远盲蚓平均遗传距离分别为15.64%、20.03%,这与通过线粒体基因组计算的通俗腔蚓(21.00%)、皮质远盲蚓(19.80%)的平均遗传距离不一致,因此不同蚯蚓物种鉴定的分类可能需要更多基因组数据支持。
| [1] |
CSUZDI C. Earthworm species, a searchable database[J]. Opuscula Zoologica Budapest, 2012, 43(1): 97-99. |
| [2] |
PHILLIPS H R P, BACH E M, BARTZ M L C, et al. Global data on earthworm abundance, biomass, diversity and corresponding environmental properties[J]. Scientific Data, 2021, 8(1): 136. DOI:10.1038/s41597-021-00912-z |
| [3] |
PHILLIPS H R P, GUERRA C A, BARTZ M L C, et al. Global distribution of earthworm diversity[J]. Science, 2019, 366(6464): 480-485. DOI:10.1126/science.aax4851 |
| [4] |
蒋际宝, 邱江平. 中国巨蚓科蚯蚓的起源与演化[J]. 生物多样性, 2018, 26(10): 1074-1082. DOI:10.17520/biods.2018105 |
| [5] |
孙星衍, 孙冯翼. 神农本草经[M]. 北京: 人民卫生出版社, 1963.
|
| [6] |
李时珍. 本草纲目[M]. 北京: 人民卫生出版社, 1982.
|
| [7] |
国家药典委员会. 中华人民共和国药典2020年版一部[M]. 北京: 中国医药科技出版社, 2020.
|
| [8] |
国家林业林草局. 有重要生态、科学、社会价值的陆生野生动物名录[EB/OL]. (2023-06-26)[2024-05-22]. https://www.forestry.gov.cn/lyj/1/gkgfxwj/20230626/546059.html.
|
| [9] |
XING Z, GAO H, WANG D, et al. A novel biological sources consistency evaluation method reveals high level of biodiversity within wild natural medicine: a case study of Amynthas earthworms as "Guang Dilong"[J]. Acta Pharmaceutica Sinica B, 2023, 13(4): 1755-1770. DOI:10.1016/j.apsb.2022.10.024 |
| [10] |
张前程, 文红梅, 刘娜, 等. 广地龙特异性引物序列的设计及其混伪品的鉴别[J]. 南京中医药大学学报, 2020, 36(3): 408-413. |
| [11] |
BHAMBRI A, DHAUNTA N, PATEL S S, et al. Large scale changes in the transcriptome of Eisenia fetida during regeneration[J]. PLoS One, 2018, 13(9): e0204234. DOI:10.1371/journal.pone.0204234 |
| [12] |
JIN F, ZHOU Z, GUO Q, et al. High-quality genome assembly of Metaphire vulgaris[J]. PeerJ, 2020, 8: e10313. DOI:10.7717/peerj.10313 |
| [13] |
SHAO Y, WANG X B, ZHANG J J, et al. Genome and single-cell RNA-sequencing of the earthworm Eisenia andrei identifies cellular mechanisms underlying regeneration[J]. Nature Communications, 2020, 11(1): 2656. DOI:10.1038/s41467-020-16454-8 |
| [14] |
WANG X, ZHANG Y, ZHANG Y, et al. Amynthas corticis genome reveals molecular mechanisms behind global distribution[J]. Communications Biology, 2021, 4(1): 135. DOI:10.1038/s42003-021-01659-4 |
| [15] |
翟俊杰, 赵慧峰, 商光申, 等. 蚯蚓基因组学的研究进展: 基于全基因组及线粒体基因组[J]. 生物多样性, 2022, 30(12): 211-221. |
| [16] |
DOLEZEL J, BARTOS J. Plant DNA flow cytometry and estimation of nuclear genome size[J]. Annals of Botany, 2005, 95(1): 99-110. DOI:10.1093/aob/mci005 |
| [17] |
DOLEZEL J, GREILHUBER J, SUDA J. Estimation of nuclear DNA content in plants using flow cytometry[J]. Nature Protocols, 2007, 2(9): 2233-2244. DOI:10.1038/nprot.2007.310 |
| [18] |
PAUL S, ARUMUGAPERUMAL A, RATHY R, et al. Data on genome annotation and analysis of earthworm Eisenia fetida[J]. Data in Brief, 2018, 20: 525-534. DOI:10.1016/j.dib.2018.08.067 |
| [19] |
CHEN Y, CHEN Y, SHI C, et al. SOAPnuke: a MapReduce acceleration-supported software for integrated quality control and preprocessing of high-throughput sequencing data[J]. GigaScience, 2018, 7(1): gix120. |
| [20] |
MARÇAIS G, KINGSFORD C. A fast, lock-free approach for efficient parallel counting of occurrences of k-mers[J]. Bioinformatics, 2011, 27(6): 764-770. DOI:10.1093/bioinformatics/btr011 |
| [21] |
VURTURE G W, SEDLAZECK F J, NATTESTAD M, et al. GenomeScope: fast reference-free genome profiling from short reads[J]. Bioinformatics, 2017, 33(14): 2202-2204. DOI:10.1093/bioinformatics/btx153 |
| [22] |
ZIMIN A V, MARÇAIS G, PUIU D, et al. The MaSuRCA genome assembler[J]. Bioinformatics, 2013, 29(21): 2669-2677. DOI:10.1093/bioinformatics/btt476 |
| [23] |
ELLINGHAUS D, KURTZ S, WILLHOEFT U. LTRharvest, an efficient and flexible software for de novo detection of LTR retrotransposons[J]. BMC Bioinformatics, 2008, 9: 18. DOI:10.1186/1471-2105-9-18 |
| [24] |
BENSON G. Tandem repeats finder: a program to analyze DNA sequences[J]. Nucleic Acids Research, 1999, 27(2): 573-580. DOI:10.1093/nar/27.2.573 |
| [25] |
CANTAREL B L, KORF I, ROBB S M C, et al. MAKER: an easy-to-use annotation pipeline designed for emerging model organism genomes[J]. Genome Research, 2008, 18(1): 188-196. DOI:10.1101/gr.6743907 |
| [26] |
BIRNEY E, CLAMP M, DURBIN R. GeneWise and genomewise[J]. Genome Research, 2004, 14(5): 988-995. DOI:10.1101/gr.1865504 |
| [27] |
STANKE M, WAACK S. Gene prediction with a hidden Markov model and a new intron submodel[J]. Bioinformatics, 2003, 19(Suppl 2): ii215-ii225. |
| [28] |
KORF I. Gene finding in novel genomes[J]. BMC Bioinformatics, 2004, 5(1): 59. DOI:10.1186/1471-2105-5-59 |
| [29] |
KIM D, LANGMEAD B, SALZBERG S L. HISAT: a fast spliced aligner with low memory requirements[J]. Nature Methods, 2015, 12(4): 357-360. DOI:10.1038/nmeth.3317 |
| [30] |
PERTEA M, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nature Biotechnology, 2015, 33(3): 290-295. DOI:10.1038/nbt.3122 |
| [31] |
JIN J J, YU W B, YANG J B, et al. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes[J]. Genome Biology, 2020, 21(1): 241. DOI:10.1186/s13059-020-02154-5 |
| [32] |
ALLIO R, SCHOMAKER-BASTOS A, ROMIGUIER J, et al. MitoFinder: efficient automated large-scale extraction of mitogenomic data in target enrichment phylogenomics[J]. Molecular Ecology Resources, 2020, 20(4): 892-905. DOI:10.1111/1755-0998.13160 |
| [33] |
ZHANG D, GAO F, JAKOVLIC ′ I, et al. PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Molecular Ecology Resources, 2020, 20(1): 348-355. DOI:10.1111/1755-0998.13096 |
| [34] |
EMMS D M, KELLY S. OrthoFinder: phylogenetic orthology inference for comparative genomics[J]. Genome Biology, 2019, 20(1): 238. DOI:10.1186/s13059-019-1832-y |
| [35] |
KATOH K, MISAWA K, KUMA K I, et al. MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform[J]. Nucleic Acids Research, 2002, 30(14): 3059-3066. DOI:10.1093/nar/gkf436 |
| [36] |
CASTRESANA J. Selection of conserved blocks from multiple alignments for their use in phylogenetic analysis[J]. Molecular Biology and Evolution, 2000, 17(4): 540-552. DOI:10.1093/oxfordjournals.molbev.a026334 |
| [37] |
ZHANG Z. KaKs_Calculator 3.0:calculating selective pressure on coding and non-coding sequences[J]. Genomics, Proteomics & Bioinformatics, 2022, 20(3): 536-540. |
| [38] |
NIGUSSIE A, KUYPER T W, BRUUN S, et al. Vermicomposting as a technology for reducing nitrogen losses and greenhouse gas emissions from small-scale composting[J]. Journal of Cleaner Production, 2016, 139: 429-439. DOI:10.1016/j.jclepro.2016.08.058 |
| [39] |
ZHU G, SCHMIDT O, LUAN L, et al. Bacterial keystone taxa regulate carbon metabolism in the earthworm gut[J]. Microbiology Spectrum, 2022, 10(5): e0108122. DOI:10.1128/spectrum.01081-22 |
| [40] |
SWART E, GOODALL T, KILLE P, et al. The earthworm microbiome is resilient to exposure to biocidal metal nanoparticles[J]. Environmental Pollution, 2020, 267: 115633. DOI:10.1016/j.envpol.2020.115633 |
| [41] |
CUI W, GAO P, ZHANG M, et al. Adverse effects of microplastics on earthworms: a critical review[J]. Science of the Total Environment, 2022, 850: 158041. DOI:10.1016/j.scitotenv.2022.158041 |
| [42] |
YANG X, SHANG G, WANG X. Biochemical, transcriptomic, gut microbiome responses and defense mechanisms of the earthworm Eisenia fetida to salt stress[J]. Ecotoxicology and Environmental Safety, 2022, 239: 113684. DOI:10.1016/j.ecoenv.2022.113684 |
| [43] |
ZHU G, CHAO H, SUN M, et al. Toxicity sharing model of earthworm intestinal microbiome reveals shared functional genes are more powerful than species in resisting pesticide stress[J]. Journal of Hazardous Materials, 2023, 446: 130646. DOI:10.1016/j.jhazmat.2022.130646 |
| [44] |
HIRANO T, TAMAE K. Earthworms and soil pollutants[J]. Sensors, 2011, 11(12): 11157-11167. DOI:10.3390/s111211157 |
| [45] |
DU Y, SHANG G, ZHAI J, et al. Effects of soybean oil exposure on the survival, reproduction, biochemical responses, and gut microbiome of the earthworm Eisenia fetida[J]. Journal of Environmental Sciences, 2023, 133: 23-36. DOI:10.1016/j.jes.2022.07.022 |
| [46] |
GUDETA K, KUMAR V, BHAGAT A, et al. Ecological adaptation of earthworms for coping with plant polyphenols, heavy metals, and microplastics in the soil: a review[J]. Heliyon, 2023, 9(3): e14572. DOI:10.1016/j.heliyon.2023.e14572 |



