2. 广东连南板洞省级自然保护区管理处,广东清远 513300
2. Management Bureau of Guangdong Liannan Bandong Provincial Nature Reserve, Qingyuan, Guangdong, 513300, China
叶绿体是植物进行光合作用的主要场所,拥有独立的基因组,它和线粒体基因组、细胞核基因组共同构成植物的三大类遗传系统[1]。与线粒体基因组、细胞核基因组相比,叶绿体基因组具有基因组较小(约150 kb)和结构稳定的特点。一个典型的叶绿体基因组包含一个大单拷贝(Large Single Copy, LSC)区、一个小单拷贝(Small Single Copy, SSC)区和两个反向重复(Inverted Repeat, IR)区[2]。在具有光合作用的植物中,大多数叶绿体基因组含有110-130个不同的基因,其中包括大约80个蛋白编码基因,30个tRNA基因和4个rRNA基因[3]。已有研究表明,对叶绿体基因组特征和系统发育进行分析,有助于开发DNA条形码的和厘清近缘物种之间的亲缘关系,进而为物种鉴定和群体遗传学研究奠定基础[4]。
青钱柳[Cyclocarya paliurus (Batalin) Iljinsk.]又名青钱李、山麻柳等,隶属于胡桃科(Juglandaceae DC.ex Perleb)青钱柳属(Cyclocarya Iljinsk.),是我国特有的单种属植物[5]。青钱柳为高大落叶乔木,主要分布于我国湖南、浙江、江西和贵州等15个省[6]。《中国中药资源志要》[7]中记载,青钱柳具有重要的药用价值,其树皮、树叶等具有清热解毒、止痛的功效[8]。近年来,大量研究表明青钱柳的叶片中富含多糖、黄酮类等次生代谢产物,具有降血糖、降血脂、抗衰老和抗炎等功效[9]。目前,有关青钱柳的研究多集中于化学成分、药效成分分析以及资源培育等方面[10],而有关叶绿体基因组的研究较少,仅Hu等[11]基于Illumina Hiseq测序技术首次发表了青钱柳的叶绿体基因组。Qu等[12]同样基于二代测序技术获得四倍体和二倍体青钱柳的叶绿体基因组,四倍体青钱柳的叶绿体基因组与二倍体的相差167 bp。然而,上述研究主要以数据的形式发表,并未深入分析青钱柳的叶绿体基因组特征。
胡桃科包含马尾树亚科(Rhoipteleoideae Reveal)、黄杞亚科(Engelhardioideae Iljinsk.)和胡桃亚科(Juglandoideae Eaton)。该分类体系自确立发表后,国内外学者长期关注其系统发育研究,其中胡桃亚科包含化香树族(Platycaryeae Nakai)、山核桃族(Caryeae Koidz.)以及胡桃族(Juglandeae Rchb.)。化香树族仅包含化香树属(Platycarya Siebold & Zucc.)植物;胡桃族中包含青钱柳属、胡桃属(Juglans L.)和枫杨属(Pterocarya Kunth),三属之间的系统发育关系尚未明确。国内外许多学者基于不同的分子数据集构建了胡桃科的系统发育树,结果存在不同的拓扑结构[13-18]。如Li等[14]基于4个叶绿体基因片段和2个核基因片段构建胡桃科系统发育树,结果显示山核桃属(Carya Nutt.)嵌套在胡桃族中,与胡桃属聚为一个分支,而青钱柳属与枫杨属聚为姐妹分支。Zhang等[15]测序了17个胡桃科植物的6个叶绿体基因片段和2个核基因片段,系统发育分析结果表明青钱柳属与枫杨属、胡桃属共同形成的分支呈姐妹群关系,后与化香树属聚为一支,而山核桃属与喙核桃属(Annamocarya A.Chev.)相互嵌套位于基部位置。Larson-Johnson[17]使用5个基因片段构建了包含37个物种的胡桃科系统发育树,结果同样支持青钱柳属与枫杨属亲缘关系最近,而胡桃属与山核桃属聚为一支。Mu等[18]采用RAD测序技术和叶绿体基因组序列对19个胡桃科植物进行系统发育分析,结果显示胡桃族中核基因数据集和叶绿体基因组数据集存在明显的系统发育冲突,核基因数据集构建的系统发育树中青钱柳属与胡桃属、枫杨属共同形成的分支呈姐妹群关系,而叶绿体基因组数据集结果为青钱柳属与枫杨属亲缘关系最近,后与胡桃属聚为一支。不同的遗传数据具有不同的进化历史,这可能是导致胡桃科系统发育研究中出现不同拓扑结构的主要原因。
本研究以青钱柳为材料,利用Illumina二代测序技术对其叶绿体全基因组进行测序,并基于叶绿体基因组蛋白编码核苷酸序列,结合已发表的胡桃科物种的叶绿体基因组数据,对青钱柳叶绿体基因组进行特征分析和系统发育分析,以期为该种的种质资源保护和开发利用提供基础数据。
1 材料与方法 1.1 样品采集、DNA提取及叶绿体基因组测序组装和注释2022年10月,笔者前往广东省清远市连南瑶族自治县板洞省级自然保护区天然林,采集青钱柳植株的新鲜健康叶片,以硅胶干燥保存,凭证标本存放于华南农业大学林学与风景园林学院植物标本馆(CANT),标本号为qyln0218。使用天根生物科技有限公司的植物DNA基因组提取试剂盒提取基因组总DNA,采用Illumina HiSeqPE150双端测序策略进行测序,至少获得5 Gb的数据。使用SOAPnuke软件[19]对测序得到的原始数据(Raw data)进行过滤,获得高质量的测序数据(Clean data)。使用Python软件GetOrganelle v1.6.3a(https://github.com/Kinggerm/GetOrganelle)进行叶绿体基因组从头组装,参考基因组选择公开发表的青钱柳(MW118603.1)叶绿体基因组[20]。使用Bandage软件[21]和Geneious v11.1.5软件[22]对组装得到的scaffold序列进行手动拼接,最终得到完整的青钱柳叶绿体基因组序列。
使用PGA软件对组装得到的完整的叶绿体基因组进行注释[23],同样以公开发表的青钱柳(MW118603.1)叶绿体基因组作为参考基因组,后续使用Geneious v11.1.5软件对注释结果进行手动校正,相似度参数(Similarity)设置大于80%。将得到的基因组注释信息以及基因组序列文件提交至NCBI的GenBank数据库中,后续使用在线软件OGDRAW v1.3.1(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)绘制青钱柳叶绿体基因组图谱[24]。
1.2 比较叶绿体基因组分析结合NCBI数据库中公开发表的胡桃科叶绿体基因组数据,分析了包括喙核桃属1种[25]、山核桃属6种[26, 27]、青钱柳属1种、黄杞属(Engelhardia Lesch.ex Blume)3种[28, 29]、胡桃属5种、化香树属2种[30]、枫杨属5种[18]及马尾树属(Rhoiptelea Diels & Hand.-Mazz.)1种[31]在内的8属24种植物26个叶绿体基因组。将获得的叶绿体基因组Genbank格式文件导入基因组结构分析软件Mauve[32]进行共线性比对,采用默认宽度和图形表现形式,并利用内置的MUSCLE 3.6[33]进行分析。同时,使用在线软件IRscope进行叶绿体基因组IR边界分析[34]。此外,使用在线软件mVISTA进行叶绿体全基因组变异区块分析,选择公开发表的青钱柳(MW118603.1)作为参照对象,通过Geneious v11.1.5软件[35]获得该基因组的注释文件,选择软件内置的LAGAN进行多序列全局比对。
1.3 密码子偏好性分析使用Python脚本“get_annotated_regions_from_gb.py”(https://github.com/Kinggerm/PersonalUtilities/)提取青钱柳的蛋白编码基因核苷酸序列,并对蛋白编码基因序列进行筛选,确保每条序列长度不小于300 bp,且每条序列均包含起始密码子“M”和终止密码子“*”,同时剔除重复序列[36]。使用Python脚本“concatenate_fasta.py”(https://github.com/Kinggerm/PersonalUtilities/)将符合要求的蛋白编码基因核苷酸序列合并,最终得到用于后续分析的编码序列(Coding Sequence, CDS)数据集。使用CodonW v1.4.2软件(https://sourceforge.net/projects/codonw/)和在线软件CUSP(http://emboss.toulouse.inra.fr/cgi-bin/emboss/cusp)计算青钱柳叶绿体基因组蛋白编码基因核苷酸序列的有效密码子(ENc)、相对同义密码子使用度(RSCU)、密码子总GC含量(GCall)以及第1、2、3位密码子的GC含量(GC1、GC2、GC3)。
1.4 简单重复序列分析利用在线软件REPuter(https://bibiserv.cebitec.uni-bielefeld.de/reputer/)[37]识别青钱柳叶绿体全基因组的散在重复序列,参数设置为Maximum Computed Repeats=50 bp;Minimal Repeat Size=30 bp;Hamming Distance=3[38]。使用在线软件Tandem repeats finder(https://tandem.bu.edu/trf/),选择默认参数鉴定青钱柳叶绿体全基因组的串联重复序列[39]。使用Perl脚本MISA检测青钱柳叶绿体基因组的简单重复序列(Simple Sequence Repeat, SSR)。软件参数设置为单核苷酸重复次数(momo-)≥10,二核苷酸重复次数(di-)≥5,三核苷酸重复次数(tri-)≥4,四核苷酸重复次数(tetra-)、五核苷酸重复次数(penta-)和六核苷酸重复次数(hexa-)≥3,若两个SSR之间的距离小于100 bp,则认为是复合简单重复序列(compound-)[40-42]。
1.5 系统发育分析选择桦木属(Betula L.)、鹅耳枥属(Carpinus L.)和铁木属(Ostrya Scop.)6个种作为外类群[43]。结合胡桃科26个叶绿体基因组序列,使用Geneious v11.1.5软件提取它们共有的叶绿体编码蛋白基因核苷酸序列(CDS数据集),用于后续构建系统发育树。使用MUSCLE v3.8.31软件[33]对CDS数据集进行序列比对,使用IQ-TREE v2.0.3软件[44]构建基于最大似然法的系统发育树,自举检验值设置为1 000。
2 结果与分析 2.1 青钱柳叶绿体基因组基本特征分析如图 1所示,青钱柳叶绿体基因组全长为161 291 bp,GC含量为36.00%,与其他胡桃科植物的叶绿体基因组相似,青钱柳叶绿体基因组为典型的四分体结构,包括1个LSC区(90 367 bp, 33.60%)和1个SSC区(18 590 bp,29.70%)以及把它们分隔开的2个IR区(26 167 bp, 42.50%)(表 1)。PGA软件注释结果显示,青钱柳叶绿体基因组共注释得到114个基因,其中包括80个蛋白编码基因,4个rRNA编码基因和30个tRNA编码基因(表 1和表 2)。按照基因的功能可以将注释得到基因分为4大类,包括光合系统基因(Genes for photosynthesis, 45个)、自我复制基因(Genes for self-replication, 56个)、其他基因(Other genes, 5个)和未知功能基因(Genes of unknown function,5个)。其中20个基因含有2份拷贝,包括7个编码蛋白基因(ndhB、rpl2、rpl23、rps7、ycf1、ycf2、ycf15),4个rRNA编码基因(rrn4.5S、rrn5S、rrn16、rrn23S)和9个tRNA编码基因(trnA-UGC、trnG-GCC、trnI-GAU、trnK-UUU、trnL-CAA、trnM-CAU、trnN-GUU、trnR-ACG、trnV-GAC),rps12基因存在反式剪接的情况。
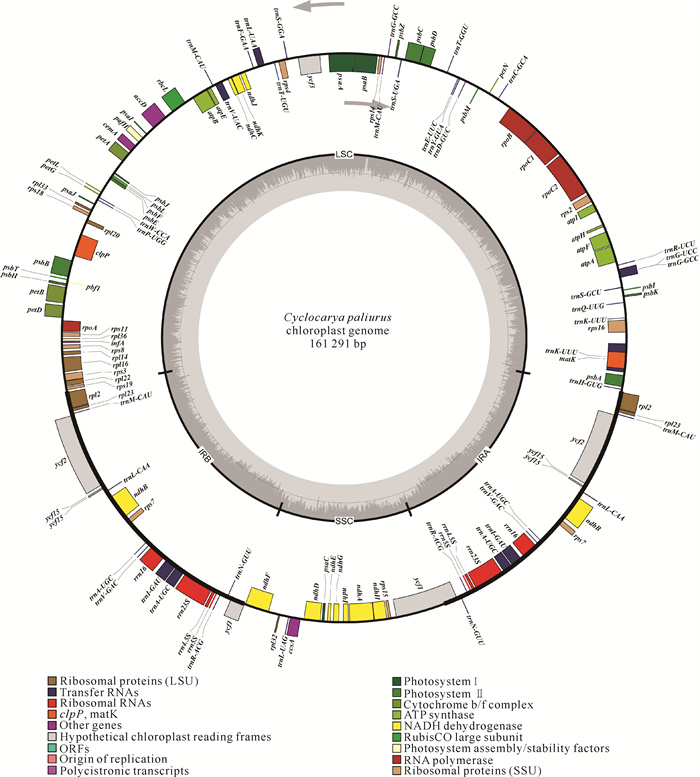
|
| 图 1 青钱柳叶绿体基因组图谱 Fig.1 Chloroplast genome map of Cyclocarya paliurus (Batalin) Iljinsk. |
| 物种 Species |
GenBank登录号 GenBanklogin numbers |
基因组长度/bp Chloroplast genome length/bp |
基因组GC含量/% Chloroplast genome GC content/% |
LSC区长度/bp LSC length/bp |
SSC区长度/bp SSC length/bp |
IR区长度/bp IR length/bp |
GC含量/% GC content/% |
基因 Gene |
编码蛋白基因 CDS |
转运RNA tRNA |
核糖体RNA rRNA |
||
| LSC | GC | IR | |||||||||||
| C.paliurus | MW118603.1 | 160 938 | 36.00 | 90 221 | 18 593 | 26 062 | 33.60 | 29.70 | 42.50 | 113 | 79 | 30 | 4 |
| C.paliurus | MW531677.2 | 161 105 | 36.00 | 90 391 | 18 590 | 26 062 | 33.50 | 29.70 | 42.50 | 113 | 79 | 30 | 4 |
| C.paliurus | PP722698 | 161 291 | 36.00 | 90 367 | 18 590 | 26 167 | 33.60 | 29.70 | 42.50 | 114 | 80 | 30 | 4 |
| 基因分类 Category of gene |
基因分组 Group of gene |
基因名称 Name of gene |
| Gene for photosynthesis | ATP synthase | atpA, atpB, atpE, atpF, atpH, atpI |
| Cytochrome b6/f complex | petA, petB, petD, petG, petL, petN | |
| NADH dehydrogenase | ndhA, ndhB, ndhC, ndhD, ndhE, ndhF, ndhG, ndhH, ndhI, ndhJ, ndhK | |
| Photosystem Ⅰ | psaA, psaB, psaC, psaI, psaJ | |
| Photosystem Ⅱ | psbA, psbB, psbC, psbD, psbE, psbF, psbH, psbI, psbJ, psbK, psbL, psbM, psbN, psbT, psbZ | |
| ATP-dependent protease subunit p | clpP | |
| RuBPcase large subunit | rbcL | |
| Genes for self-replication | Ribosomal protein (SSU) | rps2, rps3, rps4, rps7, rps8, rps11, rps14, rps15, rps16, rps18, rps19 |
| Ribosomal protein (LSU) | rpl2, rpl14, rpl16, rpl20, rpl22, rpl23, rpl32, rpl33, rpl36 | |
| Ribosomal RNAs | rrn16, rrn26S, rrn4.5S, rrn5S | |
| RNA polymerase | rpoA, rpoB, rpoC1, rpoC2 | |
| Transfer RNAs | trnA-UGC, trnC-GCA, trnD-GUC, trnE-UUC, trnF-GAA, trnG-GCC, trnG-UCC, trnH-GUG, trnI-GAU, trnK-UUU, trnL-CAA, trnL-UAA, trnL-UAG, trnM-CAU, trnN-GUU, trnP-UGG, trnQ-UUG, trnR-ACG, trnR-UCU, trnS-GCU, trnS-GGA, trnS-UGA, trnT-GGU, trnT-UGU, trnV-GAC, trnV-UAC, trnW-CCA, trnY-GUA | |
| Other genes | Subunit of acetyl-CoA-carboxylase | accD |
| Envelop membrane protein | cemA | |
| Maturase | matK | |
| C-type cytochrome synthesis ccsA gene | ccsA | |
| Translation initiation lactor IF-1 | infA | |
| Genes of unknown functions | Hypothetical chloroplast reading frames | ycf1, ycf2, ycf3, ycf4, ycf15 |
2.2 叶绿体基因组比较分析
参考已发表的青钱柳叶绿体基因组,使用mVISTA在线分析网站对胡桃科8属24种植物的26个叶绿体基因组序列的差异程度进行评估。如图 2所示,不同物种之间的叶绿体基因组序列有一定程度的分化,编码区序列比非编码区保守,各个区域的变异率为LSC>SSC>IR。共线性分析结果表明,24种胡桃科植物的叶绿体基因组完全共线性,无基因重排和倒位现象(图 3)。
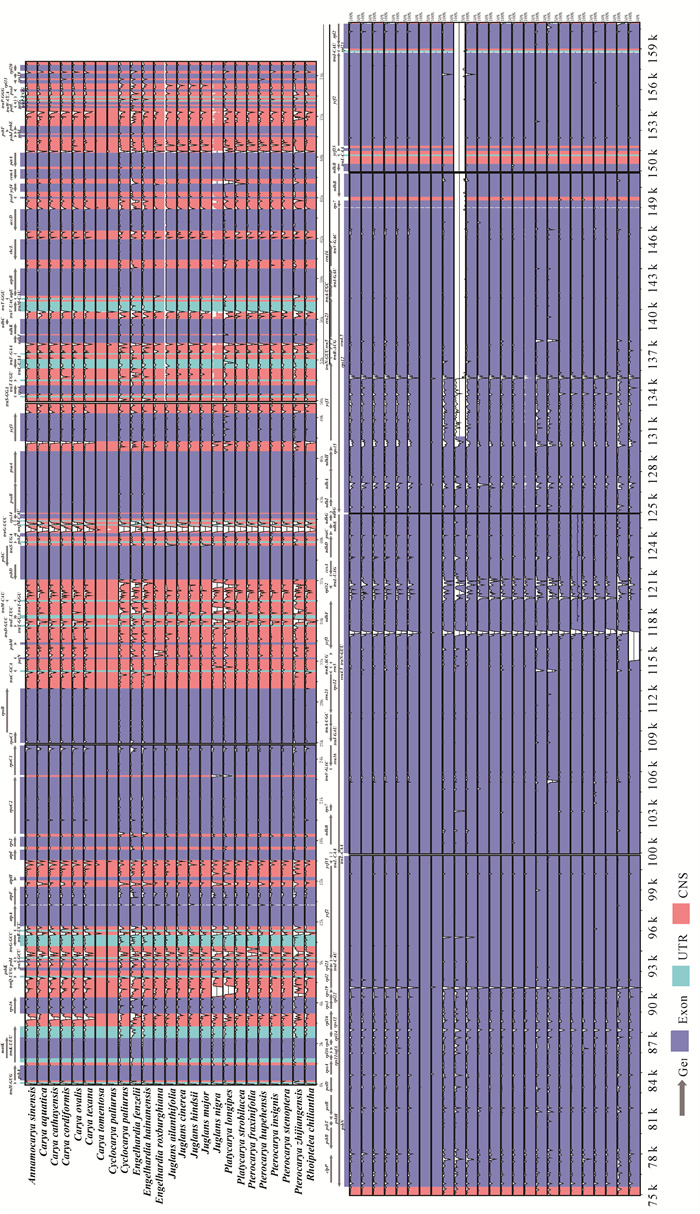
|
| 图 2 胡桃科24种植物的26个叶绿体基因组可视化比对 Fig.2 Visual comparison of 26 chloroplast genomes from 24 species of Juglandaceae DC.ex Perleb |

|
| 图 3 24种胡桃科植物的26个叶绿体基因组共线性分析 Fig.3 Synteny analysis of 26 chloroplast genomes from 24 species of Juglandaceae DC.ex Perleb |
对胡桃科8属24种植物的26个叶绿体基因组的IR边界分析显示(图 4),其4个边界,即JLB(LSC/IRb)、JSB(IRb/SSC)、JSA(SSC/IRa)和JLA(IRa/LSC)的基因位置存在差异。在JLB边界有2种类型,青钱柳属、化香树属、胡桃属[除樗叶胡桃(Juglans ailantifolia Carrière)]、黄杞属[除海南黄杞(Engelhardia hainanensis P.Y.Chen)]、马尾树属植物的rps19基因位于LSC区域,距离IRb区域有2-13 bp,其余物种中均发现rps19基因跨越了JLB边界。在JSB边界同样有2种类型,ycf1基因在青钱柳属、水山核桃[Carya aquatica (F.Michx.) Nutt.ex Elliott]、马尾树(Rhoiptelea chiliantha Diels & Hand.-Mazz.)、枫杨属、化香树属、樗叶胡桃和海南黄杞中,跨越了JSB边界,跨越长度为5-4 728 bp,其余物种的ycf1基因位于IRb区域。在JLA边界中,仅化香树的rpl2基因跨越了边界304 bp,其余物种的rpl2基因位于IRa区域。

|
| 图 4 24种胡桃科植物的26个叶绿体基因组边界差异分析 Fig.4 Analysis of 26 chloroplast genome boundary differences from 24 species of Juglandaceae DC.ex Perleb |
2.3 青钱柳叶绿体基因组同义密码子的偏好性
由表 3可知,提取并筛选青钱柳的蛋白CDS,共获得50条编码序列,其总GC含量为37.79%,各个编码基因的GC含量变化区间为30.92%(ndhF)-44.84% (rps11)。蛋白编码核苷酸序列GC含量大于40.00%的基因数量有14个,范围为40.05%(atpE)- 44.84% (rps11)。青钱柳叶绿体基因组蛋白编码基因密码子GC1、GC2和GC3含量分别为46.50%(33.05%-58.61%)、38.20%(28.26%-56.83%)和28.67%(20.96%-36.55%),不同位置GC含量的分布是不均匀的,该结果同时表明青钱柳叶绿体基因组的密码子多以A或T结尾。青钱柳叶绿体基因组50个编码基因的有效密码子数(ENc)的范围为37.54 (rps14) -59.91 (ycf3),平均值为48.68。除rps14、rps18和cemA基因外,其余基因的ENc值均大于40;其中ycf3基因高达59.91,接近61, 说明青钱柳叶绿体基因组编码区的密码子整体偏性较弱。
| 基因 Gene |
ENc值 ENc value |
GC含量/% GC content/% |
|||
| GCall | GC1 | GC2 | GC3 | ||
| CDS | 48.68 | 37.79 | 46.50 | 38.20 | 28.67 |
| psbA | 42.13 | 42.28 | 49.72 | 43.50 | 33.62 |
| matK | 50.72 | 33.27 | 39.21 | 31.88 | 28.71 |
| atpA | 47.24 | 39.96 | 55.31 | 39.76 | 24.80 |
| atpF | 53.72 | 36.40 | 45.41 | 32.43 | 31.35 |
| atpI | 44.30 | 36.42 | 49.19 | 36.69 | 23.39 |
| rps2 | 46.71 | 36.71 | 42.19 | 40.51 | 27.43 |
| rpoC2 | 48.59 | 36.41 | 45.78 | 36.46 | 27.00 |
| rpoC1 | 50.06 | 39.01 | 50.95 | 37.30 | 28.78 |
| rpoB | 47.55 | 38.04 | 49.86 | 38.01 | 26.25 |
| psbD | 46.87 | 42.37 | 52.54 | 43.22 | 31.36 |
| psbC | 46.05 | 43.72 | 54.98 | 46.10 | 30.09 |
| rps14 | 37.54 | 40.59 | 43.56 | 47.52 | 30.69 |
| psaB | 47.43 | 40.36 | 48.84 | 42.86 | 29.39 |
| psaA | 49.63 | 42.25 | 51.93 | 43.54 | 31.29 |
| ycf3 | 59.91 | 38.86 | 46.15 | 38.46 | 31.95 |
| rps4 | 50.00 | 38.12 | 50.50 | 37.62 | 26.24 |
| ndhJ | 46.45 | 39.20 | 50.94 | 37.74 | 28.93 |
| ndhK | 52.02 | 37.28 | 42.86 | 42.86 | 26.12 |
| ndhC | 47.60 | 34.71 | 46.28 | 33.88 | 23.97 |
| atpE | 51.64 | 40.05 | 48.51 | 39.55 | 32.09 |
| atpB | 44.26 | 41.94 | 56.50 | 42.68 | 26.63 |
| rbcL | 47.24 | 43.63 | 58.61 | 43.70 | 28.57 |
| accD | 48.79 | 34.74 | 41.68 | 34.67 | 27.86 |
| ycf4 | 50.50 | 38.56 | 43.24 | 40.54 | 31.89 |
| cemA | 39.66 | 32.75 | 37.83 | 28.26 | 32.17 |
| petA | 50.95 | 39.75 | 52.48 | 36.02 | 30.75 |
| rps18 | 38.22 | 34.64 | 37.25 | 41.18 | 25.49 |
| rpl20 | 41.63 | 32.20 | 33.05 | 41.53 | 22.03 |
| clpP | 51.90 | 41.29 | 57.87 | 37.06 | 28.93 |
| psbB | 46.62 | 43.09 | 54.62 | 46.37 | 28.29 |
| petB | 40.24 | 39.35 | 48.61 | 41.67 | 27.78 |
| rpoA | 49.36 | 33.94 | 46.08 | 31.63 | 24.10 |
| rps11 | 44.09 | 44.84 | 51.80 | 56.83 | 25.90 |
| rps8 | 47.32 | 36.79 | 40.74 | 40.74 | 28.89 |
| rpl14 | 45.92 | 37.67 | 53.66 | 37.40 | 21.95 |
| rps3 | 44.86 | 35.45 | 49.55 | 34.55 | 22.27 |
| rpl22 | 44.46 | 31.94 | 35.33 | 37.13 | 23.35 |
| rpl2 | 52.44 | 43.52 | 51.27 | 48.00 | 31.27 |
| ycf2 | 53.30 | 37.47 | 41.59 | 34.27 | 36.55 |
| ndhB | 48.15 | 37.23 | 41.33 | 38.79 | 31.58 |
| rps7 | 45.54 | 41.03 | 52.56 | 44.87 | 25.64 |
| ycf1 | 48.99 | 32.65 | 40.32 | 28.70 | 28.93 |
| ndhF | 43.50 | 30.92 | 35.66 | 34.58 | 22.52 |
| ccsA | 47.71 | 32.39 | 34.17 | 36.99 | 26.02 |
| ndhD | 46.70 | 34.91 | 40.43 | 36.69 | 27.61 |
| ndhE | 49.32 | 32.67 | 41.58 | 34.65 | 21.78 |
| ndhG | 45.08 | 35.40 | 44.07 | 34.46 | 27.68 |
| ndhI | 40.60 | 32.93 | 41.32 | 36.53 | 20.96 |
| ndhA | 43.84 | 34.07 | 43.41 | 37.64 | 21.15 |
| ndhH | 46.80 | 37.56 | 50.25 | 35.79 | 26.65 |
| Note: GC1, GC2 and GC3 represent the GC content of the first, second and third bits of the codon, respectively.GCall represents the total GC content of codons.ENc indicates the number of effective codons. | |||||
在青钱柳叶绿体基因组的相对同义密码子使用度(RSCU)分析中,剔除不编码氨基酸的TAG、TAA和TGA终止密码子,以及仅编码蛋氨酸(Met)和色氨酸(Trp)且没有使用偏性的ATG和TGG唯一密码子。最终共得到59种密码子,编码19种氨基酸,总密码子数量为18 652个。由图 5和表 4可见,编码异亮氨酸(Ile)的AUU密码子数量最多,为865个,编码半胱氨酸(Cys)的UGC密码子数量最少,仅为55个。参与编码氨基酸的密码子中,RSCU>1的密码子有28个,其中27个以A(11)或U(16)结尾,占比96.43%;RSCU < 1的密码子有30个,其中28个以C(16)或G(12)结尾;编码亮氨酸(Leu)的密码子UUA,其RSCU=2;以上结果表明青钱柳叶绿体基因组的蛋白编码基因偏好使用A和U结尾的密码子(表 4)。
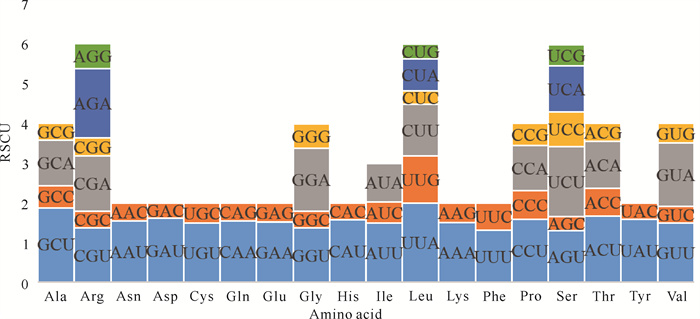
|
| 图 5 青钱柳叶绿体基因组密码子RSCU分布情况 Fig.5 RSCU distribution of codons in the chloroplast genome of Cyclocarya paliurus (Batalin) Iljinsk. |
| 氨基酸 Amino acid |
密码子 Codon |
数目 Number |
RSCU |
| Phe | UUU | 708 | 1.31 |
| UUC | 372 | 0.69 | |
| Leu | UUA | 692 | 2.00 |
| UUG | 414 | 1.19 | |
| CUU | 446 | 1.29 | |
| CUC | 120 | 0.35 | |
| CUA | 279 | 0.80 | |
| CUG | 129 | 0.37 | |
| Ile | AUU | 865 | 1.49 |
| AUC | 312 | 0.54 | |
| AUA | 566 | 0.97 | |
| Val | GUU | 402 | 1.49 |
| GUC | 115 | 0.43 | |
| GUA | 429 | 1.59 | |
| GUG | 133 | 0.49 | |
| Thr | ACU | 402 | 1.67 |
| ACC | 168 | 0.70 | |
| ACA | 286 | 1.19 | |
| ACG | 106 | 0.44 | |
| Ala | GCU | 527 | 1.88 |
| GCC | 157 | 0.56 | |
| GCA | 319 | 1.14 | |
| GCG | 119 | 0.42 | |
| Ser | UCU | 426 | 1.76 |
| UCC | 212 | 0.87 | |
| UCA | 282 | 1.16 | |
| UCG | 132 | 0.54 | |
| Gly | GGU | 479 | 1.37 |
| GGC | 151 | 0.43 | |
| GGA | 551 | 1.58 | |
| GGG | 213 | 0.61 | |
| Tyr | UAU | 595 | 1.59 |
| UAC | 152 | 0.41 | |
| Cys | UGU | 162 | 1.49 |
| UGC | 55 | 0.51 | |
| Pro | CCU | 323 | 1.59 |
| CCC | 147 | 0.72 | |
| CCA | 231 | 1.14 | |
| CCG | 111 | 0.55 | |
| His | CAU | 382 | 1.57 |
| CAC | 104 | 0.43 | |
| Gln | CAA | 538 | 1.55 |
| CAG | 155 | 0.45 | |
| Asn | AAU | 699 | 1.55 |
| AAC | 204 | 0.45 | |
| Asp | GAU | 639 | 1.61 |
| GAC | 156 | 0.39 | |
| Arg | CGU | 257 | 1.37 |
| CGC | 80 | 0.43 | |
| CGA | 259 | 1.38 | |
| CGG | 84 | 0.45 | |
| AGA | 330 | 1.76 | |
| AGG | 117 | 0.62 | |
| Lys | AAA | 700 | 1.51 |
| AAG | 226 | 0.49 | |
| Ser | AGU | 314 | 1.30 |
| AGC | 88 | 0.36 | |
| Glu | GAA | 784 | 1.52 |
| GAG | 248 | 0.48 |
2.4 青钱柳叶绿体基因组重复序列分析
通过在线软件REPuter和Tandem repeats finder计算青钱柳叶绿体基因组中的长重复序列。结果表明,青钱柳叶绿体基因组共检测到136个长重复序列,每个重复序列长度至少为30 bp,包括87个串联重复序列、19个正向重复序列、26个回文重复序列和4个反向重复序列。其中串联重复序列占比最多,为64.93%,回文重复序列次之(19.40%),反向重复序列最少(2.98%)。大部分串联重复序列(62,71.26%)位于LSC区,其次是IR区(20,22.99%)。然而,青钱柳叶绿体基因组中并无发现互补重复序列。
在青钱柳叶绿体基因组中共检测到82个SSR位点,主要分布在LSC区(68,82.93%),其次是SSC区(10,12.20%)和IR区(4,4.87%)。青钱柳叶绿体基因组中共包含101个SSR位点,包括单核苷酸重复、双核苷酸重复、三核苷酸重复、四核苷酸重复、五核苷酸重复和六核苷酸重复6种类型,数目分别为70、20、4、4、2、1和16(表 5)。其中单核苷酸重复占比最大,为69.31%,且全部由A/T组成。由A/T组成的SSR位点占总数的94.06%,表明青钱柳叶绿体基因组的SSR中,碱基A和T具有明显优势(表 5)。
| SSR类型(数量) SSR type(number) |
重复序列 Repeat sequence |
重复次数 Number of replications |
总数 Total amount |
||||||||||||
| 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | |||
| Mononucleotide | A | 13 | 9 | 4 | 1 | 5 | 1 | 33 | |||||||
| T | 14 | 9 | 6 | 5 | 3 | 37 | |||||||||
| Dinucleotide | AG | 1 | 1 | ||||||||||||
| AT | 6 | 1 | 2 | 1 | 10 | ||||||||||
| CT | 1 | 1 | |||||||||||||
| TA | 4 | 1 | 2 | 7 | |||||||||||
| TC | 1 | 1 | |||||||||||||
| Trinucleotide | ATA | 1 | 1 | ||||||||||||
| ATT | 1 | 1 | 2 | ||||||||||||
| GTA | 1 | 1 | |||||||||||||
| Tetranucleotide | AATA | 2 | 2 | ||||||||||||
| GAAA | 1 | 1 | |||||||||||||
| TGAA | 1 | 1 | |||||||||||||
| Pentanucleotide | AATAA | 1 | 1 | ||||||||||||
| ATATA | 1 | 1 | |||||||||||||
| Hexanucleotide | TTATAT | 1 | 1 | ||||||||||||
2.5 系统发育分析
为明确青钱柳的系统发育位置,选择桦木属、鹅耳枥属和铁木属6个种作为外类群,结合NCBI数据库公开发表的胡桃科26个叶绿体基因组数据,构建系统发育树,结果显示胡桃属、枫杨属,青钱柳属、化香树属、黄杞属和马尾树属各自均聚为一个分支,且拥有很高的支持率(BS=100),青钱柳与NCBI数据库公开发表其他青钱柳数据聚为一个单系群,且支持率为100。青钱柳属与胡桃属、枫杨属共同形成的分支成姐妹群关系且支持率为100,后与化香树属聚为一支(BS=100)(图 6)。
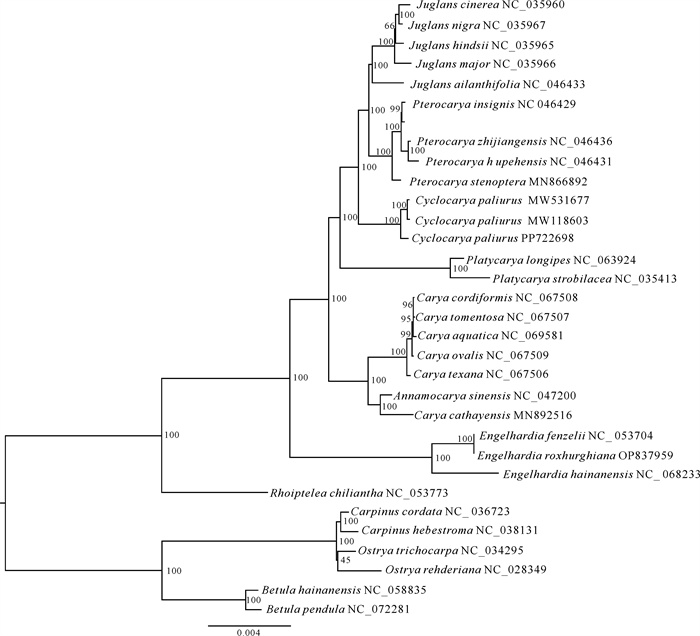
|
| 图 6 基于77个叶绿体基因组蛋白编码基因序列使用最大似然法构建30个物种系统发育树 Fig.6 Phylogenetic tree of 30 species based on 77 chloroplast genome protein coding genes sequence by maximum likelihood method |
3 讨论 3.1 青钱柳叶绿体基因组特征分析
本研究测序了组装中国特有珍贵树种青钱柳的叶绿体基因组序列。与绝大部分陆生植物的叶绿体基因组一致,青钱柳叶绿体基因组同样具有高度保守的特征[45],其长度为161 291 bp,GC含量为36.0%,这与其他公开发表的胡桃科的叶绿体基因组的长度(158 459-161 713 bp)和GC含量(35.8%-36.2%)相符[46, 47]。青钱柳叶绿体基因组各区的GC含量差异较大(表 1),其中IR区GC含量最高,为42.50%,这是由于该区域中包含GC含量较高的rRNA导致[48]。与NCBI数据库公开发表的青钱柳基因数据相比,存在806 bp的碱基差异(图 2),其中LSC区差异最多(496 bp),IR区次之(238 bp),SSC区最少(67 bp),且主要分布在基因间隔区中。青钱柳叶绿体基因组共注释得到114个基因,包括80个蛋白编码基因、4个rRNA和30个tRNA。NCBI数据库前期公开发表的青钱柳叶绿体基因组注释信息中缺失了infA蛋白编码基因,植物的infA基因翻译起始因子IF1是叶绿体中蛋白质翻译起始的重要组分[49],这可能是注释误差导致的。
尽管青钱柳的叶绿体基因组结构和基因顺序高度保守,但在IR/SC区域连接处,检测到存在差异。IR区的扩张与收缩可能会引起叶绿体基因组的大小出现差异[45]。在青钱柳叶绿体基因组的JSA边界分析中,其IR区扩张至ycf1基因(5 813 bp),进而引起IR区长度的扩增(26 167 bp),扩增的序列长度与公开发表的青钱柳叶绿体基因组相比增加了105 bp。结合叶绿体基因组可视化比对结果分析发现,公开发表的青钱柳叶绿体基因组的ycf1基因缺失105 bp,这可能是由于测序误差导致(图 2)。在JLA边界中,除化香树外,其余胡桃科物种rpl2基因均保留在IR区中,而化香树的rpl2基因有304 bp序列扩张至LSC区中,其IR区的收缩进而导致长度缩短至14 754 bp,这与前人的研究结果一致[50]。IR区的扩张在印加树属(Inga Mill.)[51]、牻牛儿苗属(Erodium L′Hér.ex Aiton)[52]和兜兰属(Paphiopedilum Pfitzer)[53]中均有报道。Palmer等[54]推测IR区的扩张有助于稳定叶绿体基因组的结构,然而具体的稳定机制目前尚不清楚。
简单重复序列(SSR)又称微卫星序列,是指1-5个核苷酸为重复单位且首尾相连形成的核苷酸序列,其在生物体的基因组中广泛存在。已有研究表明,SSR在群体遗传学和系统进化等领域发挥着重要作用[55-57]。本研究在青钱柳叶绿体基因组中共鉴定出101个SSR位点,其中,85.37%的位点分布在基因间隔区中,14.63%的位点分布在蛋白编码基因中(atpB、cemA、rpl22、rpoB、rpoC1、rpoC2和ycf1)。101个SSR位点中,单核苷酸SSR占比最高,这与大多数被子植物的重复序列特征相似,表明单核苷酸重复可能提供更多的系统发育信息[4]。在青钱柳植物的叶绿体基因组中,由A/T组成的SSR位点占总数的94.06%。植物叶绿体基因组SSR标记富含A/T重复,这在不同植物类群中均已得到证明,这也表明几乎所有植物叶绿体基因组偏好使用A和T碱基[58],同时具有较高的A/T重复类SSR的区域,其遗传多样性较高。本研究鉴定的重复序列(表 5)可作为青钱柳植物的分子标记,有助于后续青钱柳的物种鉴定和群体遗传学研究。
青钱柳叶绿体基因组蛋白编码基因密码子GC3为28.67%,由此可看出其密码子使用偏好性与其他被子植物相似,偏好使用碱基A和T结尾。密码子偏好性是物种对所处环境长期适应的表现,在生物中广泛存在;已有研究表明不同物种具有不同的密码子偏好特性,通过对不同植物的密码子偏好性研究,能为物种基因表达水平的预测、进化起源的研究等领域提供重要的数据基础[59, 60]。同义密码子相对使用度(RSCU),是指编码同一氨基酸时,某一特定密码子在所有同义密码子中出现的概率,能够直观反映密码子的偏好性,当某一密码子的RSCU>1时,表明该密码子的使用频率较高[61, 62]。在青钱柳叶绿体基因组中,共鉴定到28个密码子的RSCU>1,且其中27个以A(11)或U(16)结尾,占比96.43%;这一结果表明青钱柳叶绿体基因组的蛋白编码基因偏好使用A和U为结尾的密码子,这也与其他胡桃科植物(胡桃属、枫杨属和山核桃属等)[47]以及大多数被子植物的叶绿体基因组密码子使用偏好性一致[36, 63]。
3.2 系统发育分析以鹅耳枥属、铁木属和桦木属中的6种植物作为外类群,基于叶绿体基因组蛋白编码核苷酸序列构建最大似然系统发育树(图 6),系统发育树的各节点支持率为64%-100%。Manos等[13]结合分子数据(ITS基因片段和叶绿体基因片段)和形态特征,建立仅包含化香树属的化香树族,且位于胡桃科的基部位置,与其余胡桃科植物呈姐妹群关系。然而后续Zhang等[15]和Xiang等[16]基于不同基因片段推断的胡桃科系统发育结果,却显示化香树族与胡桃族亲缘关系更近。本研究结合NCBI数据库公开发表的胡桃科24个物种的26个叶绿体基因组数据推断的系统发育结果同样支持化香树族与胡桃族亲缘关系最近(BS=100),本研究结果与Mu等[18]使用核基因数据集推断的系统发育结果与一致。
胡桃族中包含青钱柳属、枫杨属和胡桃属,目前基于不同的分子数据集推断的系统发育结果存在争议,3个属之间的系统发育关系主要为胡桃属位于胡桃族的基部位置,以及青钱柳属位于胡桃族基部位置两种情况。Mu等[18]推测,造成这一系统冲突的原因主要是数据类型的选择,当使用核基因数据集推断胡桃族系统发育关系时,胡桃属与青钱柳属、枫杨属共同形成的分支呈姐妹群关系;当使用叶绿体基因组时,青钱柳属则位于胡桃族的基部位置。本研究基于叶绿体基因组构建的系统发育树中,同样显示青钱柳属位于胡桃族的基部位置。近年来,在植物系统发育研究中,大量核质冲突现象被研究学者们发现,并提出造成这一现象主要是受不完全谱系分选、基因流、基因渐渗以及杂交导致的叶绿体捕获等原因影响[64],如杨属(Populus L.)[65]、乌蔹莓属(Causonis Raf.)[66]以及唐棣属(Amelanchier Medik.)[67]等。然而本研究由于缺少核基因数据,无法进一步探讨胡桃族中出现的核质冲突现象。结合化石证据,目前青钱柳属植物最古老的化石可追溯到古新世晚期[68],而胡桃属和枫杨属最古老的化石可追溯到中新世中期[18],故本研究推测,青钱柳属可能是胡桃族中较为原始的类群,即支持青钱柳属位于胡桃族基部位置的论断。
4 结论本研究利用高通量测序技术成功组装了青钱柳的叶绿体基因组,进一步明确了其结构特征。通过重复序列鉴定,确认青钱柳叶绿体基因组存在101个SSR位点。密码子偏好性分析显示,青钱柳叶绿体基因组蛋白编码基因密码子偏好使用A和U为结尾的密码子。基于蛋白编码基因核苷酸序列进行的胡桃科系统发育推断中,支持化香树族与胡桃族呈姐妹群关系,推测青钱柳属可能与胡桃属、枫杨属共同形成的分支呈姐妹群关系。同时,胡桃族中存在核质冲突现象,建议未来结合核基因数据对胡桃科进行进一步的系统发育分析,以明确在科水平下各物种之间的分类关系。
| [1] |
JARVIS P, LÓPEZ-JUEZ E. Biogenesis and homeostasis of chloroplasts and other plastids[J]. Nature Reviews Molecular Cell Biology, 2013, 14(12): 787-802. DOI:10.1038/nrm3702 |
| [2] |
LI H T, LUO Y, GAN L, et al. Plastid phylogenomic insights into relationships of all flowering plant families[J]. BMC Biology, 2021, 19(1): 232. DOI:10.1186/s12915-021-01166-2 |
| [3] |
DOUGLAS S E. Plastid evolution: origins, diversity, trends[J]. Current Opinion in Genetics & Development, 1998, 8(6): 655-661. |
| [4] |
刘潮, 韩利红, 彭悦, 等. 黄丹木姜子叶绿体基因组特征分析[J]. 南方农业学报, 2022, 53(1): 12-20. DOI:10.3969/j.issn.2095-1191.2022.01.002 |
| [5] |
FANG S, WANG J, WEI Z, et al. Methods to break seed dormancy in Cyclocarya paliurus (Batal) Iljinskaja[J]. Scientia Horticulturae, 2006, 110(3): 305-309. DOI:10.1016/j.scienta.2006.06.031 |
| [6] |
中国科学院中国植物志编辑委员会. 中国植物志: 第二十一卷[M]. 北京: 科学出版社, 1979: 150.
|
| [7] |
中国药材公司. 中国中药资源志要[M]. 北京: 科学出版社, 1994.
|
| [8] |
聂小华, 吴聪聪, 林胜利, 等. 青钱柳中活性物质及其功能特性研究进展[J]. 浙江工业大学学报, 2022, 50(2): 222-227. DOI:10.3969/j.issn.1006-4303.2022.02.017 |
| [9] |
王凯平, 李文蕊, 宋梦姿, 等. 青钱柳的化学成分、药理作用及产品开发研究进展[J]. 中国医院药学杂志, 2023, 43(18): 2100-2104. |
| [10] |
方升佐. 青钱柳产业发展历程及资源培育研究进展[J]. 南京林业大学学报(自然科学版), 2022, 46(6): 115-126. |
| [11] |
HU Y H, YAN J, FENG X, et al. Characterization of the complete chloroplast genome of wheel wingnut (Cyclocarya paliurus), an endemic in China[J]. Conservation Genetics Resources, 2017, 9(2): 273-275. DOI:10.1007/s12686-016-0671-3 |
| [12] |
QU Y, WANG Q, YU Y, et al. Comparative analysis of the complete chloroplast genome between tetraploidy and diploidy of Cyclocarya paliurus (Batall.) Iljinskaja[J]. Mitochondrial DNA Part B: Resources, 2021, 6(9): 2669-2671. DOI:10.1080/23802359.2021.1964402 |
| [13] |
MANOS P S, STONE D E. Evolution, phylogeny, and systematics of the Juglandaceae[J]. Annals of the Missouri Botanical Garden, 2001, 88(2): 231-269. DOI:10.2307/2666226 |
| [14] |
LI R Q, CHEN Z D, LU A M, et al. Phylogenetic relationships in Fagales based on DNA sequences from three genomes[J]. International Journal of Plant Sciences, 2004, 165(2): 311-324. DOI:10.1086/381920 |
| [15] |
ZHANG J B, LI R Q, XIANG X G, et al. Integrated fossil and molecular data reveal the biogeographic diversification of the eastern Asian-eastern North American disjunct hickory genus (Carya Nutt.)[J]. PLoS One, 2013, 8(7): e70449. DOI:10.1371/journal.pone.0070449 |
| [16] |
XIANG X G, WANG W, LI R Q, et al. Large-scale phylogenetic analyses reveal fagalean diversification promoted by the interplay of diaspores and environments in the Paleogene[J]. Perspectives in Plant Ecology, Evolution and Systematics, 2014, 16(3): 101-110. DOI:10.1016/j.ppees.2014.03.001 |
| [17] |
LARSON-JOHNSON K. Phylogenetic investigation of the complex evolutionary history of dispersal mode and diversification rates across living and fossil Fagales[J]. New Phytologist, 2016, 209(1): 418-435. DOI:10.1111/nph.13570 |
| [18] |
MU X Y, TONG L, SUN M, et al. Phylogeny and divergence time estimation of the walnut family (Juglandaceae) based on nuclear RAD-Seq and chloroplast genome data[J]. Molecular Phylogenetics and Evolution, 2020, 147: 106802. DOI:10.1016/j.ympev.2020.106802 |
| [19] |
CHEN Y X, CHEN Y S, SHI C, et al. SOAPnuke: a MapReduce acceleration-supported software for integrated quality control and preprocessing of high-throughput sequencing data[J]. GigaScience, 2018, 7(1): gix120. |
| [20] |
JIN J J, YU W B, YANG J B, et al. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes[J]. Genome Biology, 2020, 21(1): 241. DOI:10.1186/s13059-020-02154-5 |
| [21] |
WICK R R, SCHULTZ M B, ZOBEL J, et al. Bandage: interactive visualization of de novo genome assemblies[J]. Bioinformatics, 2015, 31(20): 3350-3352. DOI:10.1093/bioinformatics/btv383 |
| [22] |
KEARSE M, MOIR R, WILSON A, et al. Geneious Basic: an integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics, 2012, 28(12): 1647-1649. DOI:10.1093/bioinformatics/bts199 |
| [23] |
QU X J, MOORE M J, LI D Z, et al. PGA: a software package for rapid, accurate, and flexible batch annotation of plastomes[J]. Plant Methods, 2019, 15: 50. DOI:10.1186/s13007-019-0435-7 |
| [24] |
GREINER S, LEHWARK P, BOCK R. Organellar- GenomeDRAW (OGDRAW) version 1.3.1:expanded toolkit for the graphical visualization of organellar genomes[J]. Nucleic Acids Research, 2019, 47(W1): W59-W64. DOI:10.1093/nar/gkz238 |
| [25] |
JI Y, ZHANG W, LI D, et al. The complete chloroplast genome sequence of Annamocarya sinensis (Juglandaceae), an Endangered species endemic to Yunnan Province, China[J]. Mitochondrial DNA Part B: Resources, 2020, 5(3): 2021-2023. DOI:10.1080/23802359.2020.1756477 |
| [26] |
SHEN J, LI X, CHEN X, et al. The complete chloroplast genome of Carya cathayensis and phylogenetic analysis[J]. Genes, 2022, 13(2): 369. DOI:10.3390/genes13020369 |
| [27] |
LUO J, CHEN J, GUO W, et al. Reassessment of Annamocarya sinesis (Carya sinensis) taxonomy through concatenation and coalescence phylogenetic analysis[J]. Plants, 2021, 11(1): 52. DOI:10.3390/plants11010052 |
| [28] |
WU Y M, MU X Y, QIN Y H. The first complete chloroplast genome in Engelhardia sensu stricto, Engelhardia hainanensis Chen: genome characterization and its phylogenetic relationships within the family Juglandaceae[J]. Mitochondrial DNA Part B: Resources, 2023, 8(4): 479-483. DOI:10.1080/23802359.2023.2196359 |
| [29] |
LING L Z, ZHANG S D. Characterization of the complete chloroplast genome of Engelhardia roxburghiana (Juglandaceae)[J]. Mitochondrial DNA Part B: Resources, 2019, 5(1): 250-251. |
| [30] |
DONG W P, XU C, LI W, et al. Phylogenetic resolution in Juglans based on complete chloroplast genomes and nuclear DNA sequences[J]. Frontiers in Plant Science, 2017, 8: 1148. DOI:10.3389/fpls.2017.01148 |
| [31] |
GENG Y F, HU G X, WANG S, et al. Complete chloroplast genome of the threatened Rhoiptelea chiliantha (Juglandaceae s.l.)[J]. Conservation Genetics Resources, 2019, 11(3): 317-319. DOI:10.1007/s12686-018-1021-4 |
| [32] |
DARLING A C E, MAU B, BLATTNER F R, et al. Mauve: multiple alignment of conserved genomic sequence with rearrangements[J]. Genome Research, 2004, 14(7): 1394-1403. DOI:10.1101/gr.2289704 |
| [33] |
EDGAR R C. MUSCLE: multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research, 2004, 32(5): 1792-1797. DOI:10.1093/nar/gkh340 |
| [34] |
AMIRYOUSEFI A, HYVÖNEN J, POCZAI P. IR- scope: an online program to visualize the junction sites of chloroplast genomes[J]. Bioinformatics, 2018, 34(17): 3030-3031. DOI:10.1093/bioinformatics/bty220 |
| [35] |
FRAZER K A, PACHTER L, POLIAKOV A, et al. VISTA: computational tools for comparative genomics[J]. Nucleic Acids Research, 2004, 32(Web Server issue): W273-W279. |
| [36] |
毛立彦, 黄秋伟, 龙凌云, 等. 7种睡莲属植物叶绿体基因组密码子偏好性分析[J]. 西北林学院学报, 2022, 37(2): 98-107. DOI:10.3969/j.issn.1001-7461.2022.02.13 |
| [37] |
KURTZ S, CHOUDHURI J V, OHLEBUSCH E, et al. REPuter: the manifold applications of repeat analysis on a genomic scale[J]. Nucleic Acids Research, 2001, 29(22): 4633-4642. DOI:10.1093/nar/29.22.4633 |
| [38] |
CHEN Y Q, ZHONG H, ZHU Y, et al. Plastome structure and adaptive evolution of Calanthe s.l.species[J]. PeerJ, 2020, 8: e10051. DOI:10.7717/peerj.10051 |
| [39] |
BENSON G. Tandem repeats finder: a program to analyze DNA sequences[J]. Nucleic Acids Research, 1999, 27(2): 573-580. DOI:10.1093/nar/27.2.573 |
| [40] |
THIEL T, MICHALEK W, VARSHNEY R, et al. Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L.)[J]. Theoretical and Applied Genetics, 2033, 106(3): 411-422. |
| [41] |
BEIER S, THIEL T, MVNCH T, et al. MISA-web: a web server for microsatellite prediction[J]. Bioinformatics, 2017, 33(16): 2583-2585. DOI:10.1093/bioinformatics/btx198 |
| [42] |
卢政阳, 于凤扬, 肖月娥, 等. 北陵鸢尾叶绿体基因组及其特征分析[J]. 草地学报, 2023, 31(6): 1656-1664. |
| [43] |
DING Y M, PANG X X, CAO Y, et al. Genome structure-based Juglandaceae phylogenies contradict alignment-based phylogenies and substitution rates vary with DNA repair genes[J]. Nature Communications, 2023, 14(1): 617. DOI:10.1038/s41467-023-36247-z |
| [44] |
NGUYEN L T, SCHMIDT H A, VON HAESELER A, et al. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Molecular Biology and Evolution, 2015, 32(1): 268-274. DOI:10.1093/molbev/msu300 |
| [45] |
RUHLMAN T A, JANSEN R K. Plastid genomes of flowering plants: essential principles[J]. Methods in Molecular Biology, 2021, 2317: 3-47. |
| [46] |
YANG Y Y, QU X J, ZHANG R, et al. Plastid phylogenomic analyses of Fagales reveal signatures of conflict and ancient chloroplast capture[J]. Molecular Phylogenetics and Evolution, 2021, 163: 107232. DOI:10.1016/j.ympev.2021.107232 |
| [47] |
ZENG Y J, SHEN L W, CHEN S Q, et al. Codon usage profiling of chloroplast genome in Juglandaceae[J]. Forests, 2023, 14(2): 378. DOI:10.3390/f14020378 |
| [48] |
HUOTARI T, KORPELAINEN H. Complete chloroplast genome sequence of Elodea canadensis and comparative analyses with other monocot plastid genomes[J]. Gene, 2012, 508(1): 96-105. DOI:10.1016/j.gene.2012.07.020 |
| [49] |
MILLEN R S, OLMSTEAD R G, ADAMS K L, et al. Many parallel losses of infA from chloroplast DNA during angiosperm evolution with multiple independent transfers to the nucleus[J]. The Plant Cell, 2001, 13(3): 645-658. DOI:10.1105/tpc.13.3.645 |
| [50] |
MO Z, LOU W, CHEN Y, et al. The chloroplast genome of Carya illinoinensis: genome structure, adaptive evolution, and phylogenetic analysis[J]. Forests, 2020, 11(2): 207. DOI:10.3390/f11020207 |
| [51] |
DUGAS D V, HERNANDEZ D, KOENEN E J M, et al. Mimosoid legume plastome evolution: IR expansion, tandem repeat expansions, and accelerated rate of evolution in clpP[J]. Scientific Reports, 2015, 5: 16958. DOI:10.1038/srep16958 |
| [52] |
BLAZIER J C, JANSE R K, MOWER J P, et al. Variable presence of the inverted repeat and plastome stability in Erodium[J]. Annals of Botany, 2016, 117(7): 1209-1220. DOI:10.1093/aob/mcw065 |
| [53] |
GUO Y Y, YANG J X, BAI M Z, et al. The chloroplast genome evolution of Venus slipper (Paphiopedilum): IR expansion, SSC contraction, and highly rearranged SSC regions[J]. BMC Plant Biology, 2021, 21(1): 248. DOI:10.1186/s12870-021-03053-y |
| [54] |
PALMER J D, THOMPSON W F. Chloroplast DNA rearrangements are more frequent when a large inverted repeat sequence is lost[J]. Cell, 1982, 29(2): 537-550. DOI:10.1016/0092-8674(82)90170-2 |
| [55] |
梁明山, 曾宇, 周翔, 等. 遗传标记及其在作物品种鉴定中的应用[J]. 植物学通报, 2001, 36(3): 257-265. DOI:10.3969/j.issn.1674-3466.2001.03.001 |
| [56] |
杨梦婷, 黄洲, 干建平, 等. SSR分子标记的研究进展[J]. 杭州师范大学学报(自然科学版), 2019, 18(4): 429-436. DOI:10.3969/j.issn.1674-232X.2019.04.013 |
| [57] |
KANTETY R V, LA ROTA M, MATTHEWS D E, et al. Data mining for simple sequence repeats in expressed sequence tags from barley, maize, rice, Sorghum and wheat[J]. Plant Molecular Biology, 2002, 48(5/6): 501-510. DOI:10.1023/A:1014875206165 |
| [58] |
KUANG D Y, WU H, WANG Y L, et al. Complete chloroplast genome sequence of Magnolia kwangsiensis (Magnoliaceae): implication for DNA barcoding and population genetics[J]. Genome, 2011, 54(8): 663-673. DOI:10.1139/g11-026 |
| [59] |
梁菲菲. 密码子偏性的影响因素及研究意义[J]. 畜牧与饲料科学, 2010, 31(1): 118-119. DOI:10.3969/j.issn.1672-5190.2010.01.058 |
| [60] |
李平, 白云凤, 冯瑞云, 等. 籽粒苋苹果酸酶(NAD-ME)基因密码子偏好性分析[J]. 应用与环境生物学报, 2011, 17(1): 12-17. |
| [61] |
SHARP P M, TUOHY T M, MOSURSKI K R. Codon usage in yeast: cluster analysis clearly differentiates highly and lowly expressed genes[J]. Nucleic Acids Research, 1986, 14(13): 5125-5143. DOI:10.1093/nar/14.13.5125 |
| [62] |
SHI S L, XIA R X. Codon usage in the Iflaviridae family is not diverse though the family members are isolated from diverse host taxa[J]. Viruses, 2019, 11(12): 1087. DOI:10.3390/v11121087 |
| [63] |
WANG Z, XU B, LI B, et al. Comparative analysis of codon usage patterns in chloroplast genomes of six Euphorbiaceae species[J]. PeerJ, 2020, 8(1): e8251. |
| [64] |
DUAN L, FU L, CHEN H F. Phylogenomic cytonuclear discordance and evolutionary histories of plants and animals[J]. Science China Life Sciences, 2023, 66(12): 2946-2948. DOI:10.1007/s11427-023-2456-6 |
| [65] |
WANG M C, ZHANG L, ZHANG Z Y, et al. Phylogenomics of the genus Populus reveals extensive interspecific gene flow and balancing selection[J]. The New Phytologist, 2020, 225(3): 1370-1382. DOI:10.1111/nph.16215 |
| [66] |
YU J, ZHAO H, NIU Y, et al. Distinct hybridization modes in wide- and narrow-ranged lineages of Causonis (Vitaceae)[J]. BMC Biology, 2023, 21(1): 209. DOI:10.1186/s12915-023-01718-8 |
| [67] |
LIU B B, CAMPBELL C S, HONG D Y, et al. Phylogenetic relationships and chloroplast capture in the Amelanchier-Malacomeles-Peraphyllum clade (Maleae, Rosaceae): evidence from chloroplast genome and nuclear ribosomal DNA data using genome skimming[J]. Molecular Phylogenetics and Evolution, 2020, 147: 106784. DOI:10.1016/j.ympev.2020.106784 |
| [68] |
MANCHESTER S R, DILCHER D L. Pterocaryoid fruits (Juglandaceae) in the Paleogene of North America and their evolutionary and biogeographic significance[J]. American Journal of Botany, 1982, 69(2): 275-286. DOI:10.1002/j.1537-2197.1982.tb13258.x |



