2. 自然资源部北部湾滨海湿地生态系统野外科学观测研究站, 广西北海 536015
2. Observation and Research Station of Coastal Wetland Ecosystemin Beibu Gulf, Ministry of Natural Resources, Beihai, Guangxi, 536015, China
短叶茳芏(Cyperus malaccensis subsp.monophyllus)属莎草科(Cyperaceae)莎草属(Cyperus),曾命名为C.malaccensis var.brevifolius[1],是我国东南至南方沿海分布面积较大的原生滨海盐沼草本植物,但其耐盐能力较差,常见于有淡水注入的潮间带淤泥质滩涂,常与红树林混生(图 1),因其植株密集,根系稠密,在保滩护岸、促淤造陆、固碳、阻留悬浮物和改良土壤等方面具有较高的生态价值。与其原种茳芏(C.malaccensis)的主要形态区别在于茳芏的地上茎顶部的3片苞片极展开,长于花序,短叶茳芏则相反[1]。短叶茳芏主要分布在我国华南沿海和日本,我国集中分布区主要有福建闽江口[2]、福建泉州湾、广东珠江河口、广西廉州湾、广西钦州茅尾海滩涂等[3]。

|
| 图 1 短叶茳芏的生境、花序及苞片 Fig.1 Habitat, inflorescence and bract of C.malaccensis subsp.monophyllus |
滨海盐沼、红树林和海草生态系统可高效固碳,是“蓝色碳汇”的重要组成部分,具有减缓气候变化的潜在作用。有研究指出,“盐沼草(以茳芏和短叶茳芏为主要种类)——红树林协同生态修复系统”可有效突破红树造林技术瓶颈[4, 5]。国内学者开展了较全面的关于短叶茳芏的碳、氮循环过程,温室气体收支及重金属富集规律等方面的研究,也有学者对茳芏和短叶茳芏的群落分布动态变化开展了研究[6-8]。短叶茳芏及其原种茳芏,以及莎草属的其他物种在形态学上极为相近,较难区分,但尚未有学者利用分子生物学技术开展其物种鉴定、系统发生关系和遗传多样性的研究。叶绿体基因组具有重组率较低、后代遗传稳定等特点,相比于核基因组,是揭示物种系统发生关系的有效手段[9, 10]。莎草科是一个庞大的单子叶植物类群,全球有5 600种,莎草属有约950种[11],但在Genbank中仅上传了不到20个莎草属物种的叶绿体全基因组序列(截至2024年8月),其中不乏形态分类有争议的物种。为了厘清莎草属、莎草科的系统发生关系,亟需补充该属物种的叶绿体基因组信息。本研究以短叶茳芏的叶片为研究材料,采用Illumina高通量测序技术,对短叶茳芏叶绿体全基因组进行测序和组装,并利用生物信息技术对其结构特征进行分析,构建莎草科的系统发育进化树,为莎草属植物的进化和系统发育研究,以及后续开展物种鉴定和遗传多样性的研究提供分子基础信息。
1 材料与方法 1.1 材料样品采自广西钦州市钦南区茅尾海(108°34′43.80″E,21°52′10.83″N),选取健康植株的新鲜嫩叶用于DNA提取,将带有花果的植株制成标本用于后期的形态鉴定。标本存放于广西海洋科学院(广西红树林研究中心)的标本室。
1.2 基因组DNA的提取及测序从样品的新鲜健康叶片中提取总基因组DNA,送至上海凌恩生物科技有限公司,经检测合格后,采用Illumina NovaSeq 6000双末端测序策略进行建库测序。
1.3 基因组组装和注释使用Trimmomatic v 0.38[12]对原始数据进行质量剪切,过滤质量比较低的数据。基因组序列通过NOVOPlasty v 4.2软件[13]进行组装。使用GeSeq软件[14]预测叶绿体基因组中的蛋白质编码(PCG)、转达RNA(tRNA)和核糖体RNA(rRNA)基因。最后,使用OGDRAW v 1.3.1软件[15]绘制短叶茳芏叶绿体基因组图谱。短叶茳芏基因组序列已上传GenBank(登录号:OR438648)。
1.4 SSR分析通过MISA在线软件[16]进行短叶茳芏叶绿体基因组的简单重复序列(Simple Sequence Repeat,SSR)分析,参数设置[17]如下:(1)1-10,2-6,3-5,4-5,5-5和6-5,即1个碱基重复≥10次;2个碱基重复≥6次;3/4/5/6个碱基均重复≥5次。(2)两个SSR的最小距离设置为100 bp,如果距离小于100 bp,两个SSR序列则组成一个复合微卫星。
1.5 重复序列分析短叶茳芏叶绿体基因组的重复序列采用REPuter在线软件[18](http://bibiserv.techfak.uni-bielefeld.de/reputer)进行分析,包括正向重复(Forward repeats)、反向重复(Reverse repeats)、互补重复(Complement repeats)和回文重复(Palindromic repeats)。参数设置:汉明距离(Hamming distance)设置为0,即重复序列完全一致,长度大于30 bp。
1.6 SNP检测及注释利用MUMmer软件[19](v3.23)分析潜在单核苷酸多态性(Single Nucleotide Polymorphism,SNP)位点;以茳芏的叶绿体基因组作为参考,将参考序列SNP位点两侧各100 bp的序列提取出来,然后利用组装软件BLAT v35在组装结果中进行比对,验证SNP位点。若比对长度小于101 bp,则认为该SNP为不可信SNP,被删除;若重复被比对上多次,则被认为是重复区域的SNP,也将被去除,最终获得可靠的SNP。
1.7 叶绿体基因组比较及系统发育分析从Genbank下载莎草属5个物种茳芏(C.malaccensis,OR238391)、香附子(C.rotundus,MT473237)、高秆莎草(C.exaltatus,MW123055)、水蜈蚣(C.aromaticus,MT937178)、碎米莎草(C.iria,MW123056)的叶绿体全基因组序列并进行基因组序列和结构比较。从GenBank下载12个莎草科植物的叶绿体全基因组序列,包括茳芏、香附子、高秆莎草、水蜈蚣、碎米莎草、细莞(Isolepis setacea,MW041568)、Eleocharis cellulosa,MN985041、荸荠(E.dulcis,MN967018)、翼果薹草(Carex neurocarpa,KU238086)、团穗薹草(Carex agglomerata,MT795185)、宽叶薹草(Carex siderosticta,KP751906)和割鸡芒(Hypolytrum nemorum,KU207098),并以禾本科植物高粱(Sorghum bicolor,NC008602)作为外群, 基于叶绿体SNP矩阵构建系统发育树,并按照1.6节描述的方法获得SNP位点[20]。利用PhyML v 3.0软件(http://www.atgc-montpellier.fr/phyml/)构建系统进化树,采用最大似然法,分支的可信度基于Bootstrap重复抽样1 000次,其他参数选择默认设置。
2 结果与分析 2.1 测序数据利用高通量测序平台Illumina NovaSeq 6000测得Raw data 3.44 G,GC含量为37.73%,Q20为94.01%,Q30为86.75%,去除低质量reads,得到Clean data 3.12 G,有效数据的平均GC含量为37.58%,有效数据的Q20和Q30分别为95.51%和89.00%,测序数据质量合格。
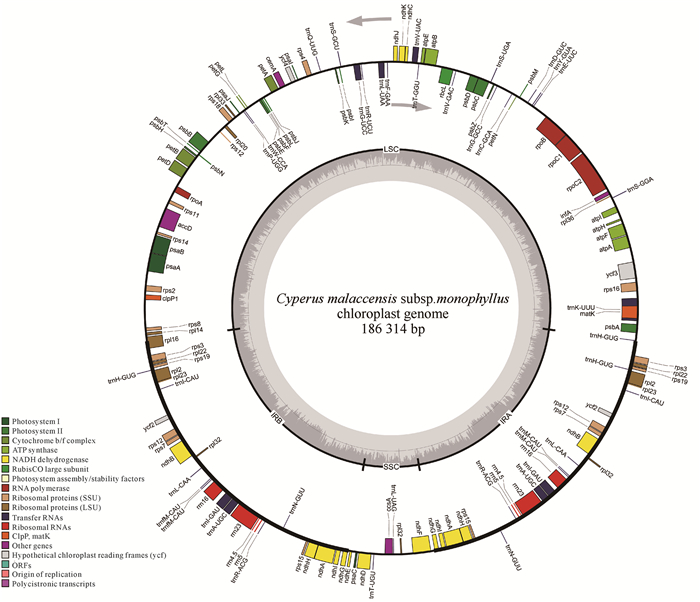
2.2 短叶茳芏叶绿体基因大小短叶茳芏叶绿体全基因组呈环状双链,序列全长为186 314 bp,GC含量为33.17%,包含大单拷贝(LSC)区、小单拷贝(SSC)区及两个反向重复序列(IRA、IRB)区,其中LSC起止位置为1-101 162,长度为101 162 bp;SSC起止位置为138 584-148 893,长度为10 310 bp;IRA和IRB长度均为37 421 bp,起止位置分别为148 894-186 314和101 163-138 583(图 2)。
|
| Genes drawn on the outside of the circle are transcribed in a counterclockwise direction, while genes drawn on the inside of the circle are transcribed in a clockwise direction. 图 2 短叶茳芏叶绿体基因组图谱 Fig.2 Genome map of Cyperus malaccensis subsp.monophyllus chloroplast |
2.3 短叶茳芏叶绿体基因注释、基因功能及分类
短叶茳芏叶绿体基因组共注释了141个基因,主要分为3大类,即94个蛋白质编码(PCG)基因,39个转运RNA(tRNA)基因,8个核糖体RNA(rRNA)基因。27个基因在IR区重复,包括15个蛋白质编码基因(rps3、rpl22、rps19、rpl2、rpl23、ycf2、rps12、rps7、ndhB、rpl16、rps15、ndhH、ndhA、ndhI、ndhG),8个tRNA基因(trnN-GUU、trnR-ACG、trnA-UGC、trnI-GAU、trnH-GUG、trnfM-CAU、trnL-CAA、trnI-CAU),4个rRNA基因(rrn4.5、rrn5、rrn16、rrn23)。此外,根据功能进行分类,又可分为光合作用相关基因、自我复制相关基因、其他蛋白编码基因和未知功能基因。其中,与光合作用相关基因有49个,可进一步细分为6个组,包括光合系统Ⅰ、光合系统Ⅱ、NADH脱氢酶、细胞色素复合物、ATP合酶和二磷酸核酮糖羧化酶大亚基;自我复制相关基因有69个,可进一步细分为5个组,分别是核糖体大亚基蛋白、核糖体小亚基蛋白、RNA聚合酶亚基、rRNA基因和tRNA基因;其他蛋白编码基因有6个;未知功能基因有4个(表 1)。
| 基因分类 Category of genes |
基因分组 Group of genes |
基因名称 Name of genes |
| Photosynthesis genes | Photosystem Ⅰ | psaA,psaB,psaC,psaI,psaJ |
| Photosystem Ⅱ | psbA,psbB,psbC,psbD,psbE,psbF,psbH,psbI,psbJ,psbK,psbL,psbM,psbN,psbT,psbZ | |
| NADH dehydrogenase | ndhA,ndhB,ndhC,ndhD,ndhE,ndhF,ndhG,ndhH,ndhI,ndhJ,ndhK | |
| Cytochrome b/f complex | petA,petB,petD,petG,petL,petN | |
| ATP synthase | atpA,atpB,atpE,atpF,atpH,atpI | |
| Large subunit of Rubisco | rbcL | |
| Self-replication genes | Large subunits of ribosome | rpl14,rpl16,rpl2,rpl20,rpl22,rpl23,rpl32,rpl33,rpl36 |
| Small subunits of ribosome | rps11,rps12,rps14,rps15,rps16,rps18,rps19,rps2,rps3,rps4,rps7,rps8 | |
| RNA polymerase | rpoA,rpoB,rpoC1,rpoC2 | |
| rRNA | rrn4.5,rrn5,rrn16,rrn23 | |
| tRNA | trnA-UGC,trnC-GCA,trnD-GUC,trnE-UUC,trnF-GAA,trnG-GCC,trnG-UCC,trnH-GUG,trnI-CAU,trnI-GAU,trnK-UUU,trnL-CAA,trnL-UAA,trnL-UAG,trnM-CAU,trnN-GUU,trnP-UGG,trnQ-UUG,trnR-ACG,trnR-UCU,trnS-GCU,trnS-GGA,trnS-UGA,trnT-GGU,trnT-UGU,trnV-GAC,trnV-UAC,trnW-CCA,trnY-GUA,trnfM-CAU | |
| Other genes | Maturase | matK |
| Protease | clpP1 | |
| Envelope membraneprotein | cemA | |
| Acetyl-CoA carboxylase | accD | |
| c-type cytochrome synthesis gene | ccsA | |
| Translation initiation factor | infA | |
| Genes of unknown function | ORF of unknown function | ycf2,ycf2,ycf3,ycf4 |
2.4 叶绿体基因组SSRs分析
通过对短叶茳芏叶绿体全基因组序列进行MISA分析,共检测出105个符合条件的SSR位点,均由A或T组成,表明SSRs的碱基组成偏向使用A/T碱基。其中包括91个单核苷酸重复基序、13个二核苷酸重复基序、1个三核苷酸重复基序,未发现四核苷酸/五核苷酸/六核苷酸重复基序(表 2)。在所有SSRs中,最多的重复基序是A/T,然后依次是AT/AT和ATA,这些重复基序从碱基数量来看,具有A/T碱基偏好性,这与短叶茳芏叶绿体全基因组AT含量占比高一致(62.42%)。从分布区段上看,SSRs的主要分布在LSC区,有73个SSR位点;其次是IR区(IRA和IRB各有13个);SSC区最少,仅有6个SSR位于该区段。
| SSR重复基序 SSR repeat motifs |
长度/bp Length/bp |
SSR数量/个 Number of SSRs |
合计占比/% Percentage of total/% |
| Mononucleotide | |||
| A/T | 10 | 39 | 86.67 |
| 11 | 21 | ||
| 12 | 15 | ||
| 13 | 3 | ||
| 14 | 5 | ||
| 15 | 1 | ||
| 16 | 3 | ||
| 17 | 2 | ||
| 19 | 1 | ||
| 21 | 1 | ||
| Dinucleotide | |||
| AT/AT | 6 | 5 | 12.38 |
| 7 | 4 | ||
| 8 | 3 | ||
| 17 | 1 | ||
| Trinucleotide | |||
| AAT/ATT | 5 | 1 | 0.95 |
2.5 长重复序列分析
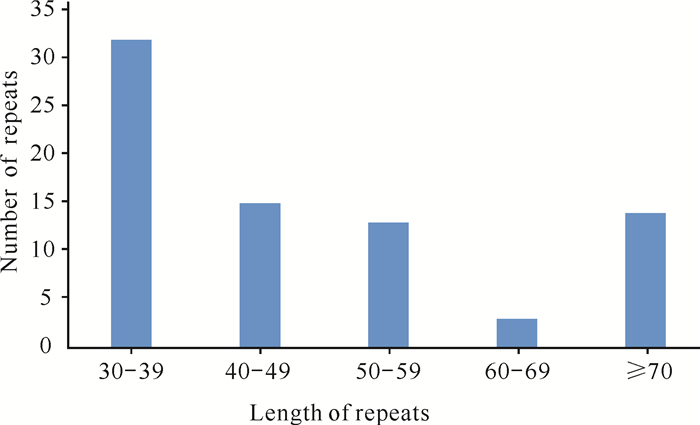
利用REPuter在线软件对短叶茳芏的叶绿体基因组进行重复序列分析,共鉴定出77个长重复序列(图 3),包括41个正向重复序列、4个反向重复序列、30个回文重复序列和2个互补重复序列。短叶茳芏叶绿体基因组重复序列长度主要为30-60 bp(图 3),占全部重复序列的78%。这些重复序列可为开发种群进化标记提供基础。
|
| 图 3 长重复序列长度范围分布 Fig.3 Distribution of length ranges of long repeats sequences |
2.6 SNP分析
以茳芏的叶绿体全基因组作为参照,对短叶茳芏的叶绿体全基因组序列进行SNP分析,共鉴定到183个SNP位点,包括位于基因区的SNP位点37个和位于基因间区的SNP位点146个。其中,基因区包括17个同义突变、20个非同义突变(表 3)。
| 项目 Items |
SNP数量 SNP number |
占比/% Percentage/% |
|
| CDS region | Synonymous | 17 | 9.3 |
| Nonsynonymous | 20 | 10.9 | |
| Intergenic region | 146 | 79.8 | |
| Total SNP | 183 | 100.0 | |
2.7 叶绿体基因组比较分析
选取莎草属6个物种的叶绿体基因组进行比较分析[21, 22],发现短叶茳芏与其原种茳芏的叶绿体基因组结构基本相同。莎草属6个物种叶绿体基因组长度为182 986-187 401 bp,其中水蜈蚣叶绿体基因组长度最长,香附子最短;6个物种的基因组GC含量相差不大,为33.16%-33.26%;短叶茳芏和茳芏具有相同的基因组分和数量,而香附子的基因总数均少于另外5个同属植物(表 4)。短叶茳芏和茳芏的基因总数和编码蛋白质基因的数量均高于其他4种,说明其叶绿体在生长过程中能行使更多的功能。
| 物种名称 Name of species |
Genbank ID | 基因组长度/bp Genome length/bp |
GC含量/% GC content/% |
LSC长度/bp LSC length/bp |
SSC长度/bp SSC length/bp |
IR长度/bp IR length/bp |
基因总数 Total number of genes |
蛋白质基因数量 Number of protein genes |
rRNA基因数量 Number of rRNA genes |
tRNA基因数量 Number of tRNA genes |
| C.malaccensis subsp.monophyllus | OR438648 | 186 314 | 33.17 | 101 162 | 10 310 | 37 421 | 141 | 94 | 8 | 39 |
| C.malaccensis | OR238391 | 186 098 | 33.18 | 100 934 | 10 296 | 37 434 | 141 | 94 | 8 | 39 |
| C.iria | MW123056 | 185 697 | 33.16 | 99 360 | 10 267 | 38 035 | 135 | 89 | 8 | 38 |
| C.exaltatus | MW123055 | 187 139 | 33.18 | 100 780 | 10 271 | 38 044 | 135 | 90 | 8 | 41 |
| C.rotundus | MT937176 | 182 986 | 33.26 | 100 733 | 10 315 | 35 969 | 133 | 84 | 8 | 41 |
| C.aromaticus | MT937178 | 187 401 | 33.13 | 101 085 | 10 496 | 37 910 | 136 | 87 | 8 | 41 |
2.8 系统发育分析
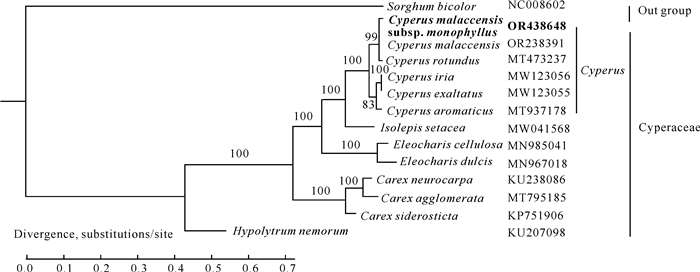
为进一步研究短叶茳芏的遗传背景和分类学关系,将短叶茳芏叶绿体基因组序列与其他12个莎草科植物的叶绿体基因组序列进行多重比对后,以高粱作为外群,基于叶绿体SNP矩阵构建系统发育树(图 4)。结果显示,莎草属(Cyperus)、荸荠属(Eleocharis)和薹草属(Carex)的单系性得到强烈支持(支持率均为100%),细莞属(Isolepis)与莎草属形成姐妹群,并得到100%支持,共同与荸荠属构成姐妹群,支持率为100%。莎草属中,短叶茳芏与其原种茳芏、香附子聚为一支,亲缘关系较近,支持率为99%,从形态学上来看,这3种也更为相似。碎米莎草、高秆莎草和水蜈蚣聚为另一支,但水蜈蚣聚为一支的支持率仅为83%。
|
| 图 4 基于叶绿体全基因组序列最大似然法构建的14个物种的系统发育树 Fig.4 Phylogenetic tree of 14 species constructed based on the maximum likelihood method of chloroplast genome sequence |
3 讨论
短叶茳芏为茳芏的亚种,是海湾河口滩涂绿化的优良植物。二者是我国南方沿海分布面积最大的2种原生滨海盐沼草本植物,也是典型的盐沼植物,在保滩护岸、促淤造陆、阻留悬浮物和改良土壤等方面具有较高的生态价值。近年来,研究者们对短叶茳芏的研究主要集中在环境、生态、湿地、土壤等多个学科[6, 23-25]。
叶绿体基因组序列是植物鉴定和建立物种间遗传关系的一种通用工具。目前,短叶茳芏的叶绿体基因组尚未有研究者报道,同属的也仅有茳芏[26]、碎米莎草[22]、油莎草(C.esculentus)[27]等被报道。本研究基于Illumina高通量测序技术获得莎草科莎草属植物短叶茳芏的叶绿体基因组全序列,并首次通过生物信息学手段对其进行分析研究。结果显示,短叶茳芏叶绿体基因组全长为186 314 bp,为典型的四分体结构,通过比较短叶茳芏与其他5个同属植物叶绿体基因组序列,发现上述植物的叶绿体基因组大小相近(182 986-187 401 bp),说明属间基因组大小具有一定的稳定性[28-30]。短叶茳芏叶绿体基因组GC含量为33.17%,共注释了141个基因,所编码的基因类别、数量及排列顺序与另外5种同属植物差异不大,同时具有高度相似的GC含量(33.16%-33.26%)。此外,短叶茳芏的基因组总长度、LSC和SSC长度均大于茳芏,但短叶茳芏的IR区要短于茳芏,说明短叶茳芏的IR区存在收缩现象。
叶绿体基因组中常用来鉴定物种分类的分子条形码有matK、rcbL和trnH-psbA,但并不能区分短叶茳芏及其原种茳芏(相似性分别为99.94%、99.93%、99.91%)。植物通用的分子条形码SSR是一种高效的分子标记,具有数量多、重现性高、显性遗传、单亲遗传和相对保守等优点,是在种群和个体水平上进行物种鉴定和遗传变异评估的最佳选择。Ren等[27]对油莎草进行长重复序列分析,获得49个长重复序列,包括15个正向重复序列和34个回文重复序列。本研究的短叶茳芏叶绿体基因组中共检测到77个长重复序列(长度主要为30-60 bp),其中41个正向重复序列、4个反向重复序列、30个回文重复序列和2个互补重复序列。短叶茳芏绿体基因组的SSR位点共有105个,出现频率最高的为单核苷酸重复基序,占SSR总数的86.67%,SSR位点数量与其他同属植物差别较大,远少于油莎草[27](396个),推测可能是不同物种在鉴定SSR位点时软件设置参数不完全一致导致的,如油莎草SSR鉴定设置的单核苷酸到六核苷酸的重复次数依次为8、5、4、3、3、3,而本研究设置的参数是10、6、5、5、5、5。本研究填补了莎草属植物短叶茳芏SSR位点研究的空白,为短叶茳芏分子标记的开发和莎草属物种鉴定提供了分子基础信息。
SNP是指由单个核苷酸的变异所引起的DNA序列多态性。在基因组DNA中,任何碱基都有可能发生突变,因此SNP既可能位于编码基因上,也有可能位于非编码序列上,在编码区内的SNP(coding SNP,cSNP)因其可能影响个体的功能而备受关注。本研究通过对短叶茳芏的叶绿体基因组进行SNP挖掘分析,共鉴定到183个SNP位点,包括位于基因区的SNP位点37个和位于基因间区的SNP位点146个。其中,基因区包括17个同义突变、20个非同义突变。这些SNP位点可为短叶茳芏的物种鉴定、遗传多样性分析和物种溯源等研究提供技术基础资料。
为进一步研究短叶茳芏的遗传背景和分类学关系,从NCBI数据库公开的植物中选取了包括短叶茳芏在内的13个莎草科植物进行聚类分析。结果显示短叶茳芏与香附子、茳芏聚为一支,分支支持率达到99%,是亲缘关系最近的3个物种。水蜈蚣与另两种莎草属物种碎米莎草、高秆莎草聚为一支的支持率仅83%。经进一步核实,目前水蜈蚣在《中国植物志》中的拉丁名已修订为Kyllinga polyphylla,属莎草科水蜈蚣属。Fan等[21]在2024年5月发表的论文中对短叶水蜈蚣(Cyperus brevifolius)叶绿体基因组进行系统发育分析,显示C.brevifolius属于莎草属,但支持率为74%。说明莎草属物种依据形态特征进行的分类学分析和基于基因序列的系统发育关系分析存在一定的矛盾,甚至已发表的论文和已上传Genbank的数据也存在着诸多混淆之处。因此,对形态相似的物种进行基于叶绿体基因组的测序,开发筛选出有效的分子条形码,对系统进化以及物种鉴定等领域的研究具有理论和实践意义。
本研究对短叶茳芏的叶绿体基因组进行了组装和注释,分析了基因组特征并构建了系统发育树,挖掘了一批SSR和SNP标记,为莎草属植物的物种鉴定及亲缘关系分析提供了进一步的分子基础资料,为莎草属植物物种后续的系统发育研究及植物资源开发提供理论基础。本研究仅采集了钦州茅尾海一地的短叶茳芏,后续研究也将从沿海各地采集短叶茳芏及茳芏的样品,对挖掘的SSR和SNP进行设计筛选和验证,开发有效的分子标记。
| [1] |
中国科学院中国植物志委员会. 中国植物志: 第十一卷莎草料[M]. 北京: 科学出版社, 1961.
|
| [2] |
ZENG A, HU W, ZENG C, et al. Litter decomposition and nutrient dynamics of native species (Cyperus malaccensis) and alien invasive species (Spartina alterniflora) in a typical subtropical estuary (Min River) in China[J]. Estuaries and Coasts, 2020, 43(7): 1873-1883. DOI:10.1007/s12237-020-00744-x |
| [3] |
潘良浩, 史小芳, 曾聪, 等. 广西滨海盐沼生态系统研究现状及展望[J]. 广西科学, 2017, 24(5): 453-461. |
| [4] |
何斌源, 赖廷和, 潘良浩, 等. 盐沼草: 白骨壤混种减轻污损动物危害的生物防治效果研究[J]. 广西植物, 2014, 34(2): 203-211. |
| [5] |
何斌源, 赖廷和, 王欣, 等. 盐沼草对桐花树人工林污损动物危害的生物防治研究[J]. 广西科学, 2013, 20(3): 185-192. |
| [6] |
林晓雪, 黄佳芳, 李慧, 等. 闽江河口芦苇沼泽和短叶茳芏沼泽生态系统含碳温室气体的年收支[J]. 生态学报, 2022, 42(22): 9186-9198. |
| [7] |
牟晓杰, 刘兴土, 仝川, 等. 闽江河口短叶茳芏湿地CH4和N2O排放对氮输入的短期响应[J]. 环境科学, 2012, 33(7): 2482-2489. |
| [8] |
宋萍, 封磊, 洪伟, 等. 闽江河口湿地土壤植物系统Zn、Cd的分布特征[J]. 福建林学院学报, 2009, 29(1): 17-22. |
| [9] |
陆奇丰, 黄至欢, 骆文华. 极小种群濒危植物广西火桐、丹霞梧桐的叶绿体基因组特征[J]. 生物多样性, 2021, 29(5): 586-595. |
| [10] |
BIRKY C W JR. Uniparental inheritance of mitochondrial and chloroplast genes: mechanisms and evolution[J]. Proceedings of the National Academy of Sciences, 1995, 92(25): 11331-11338. DOI:10.1073/pnas.92.25.11331 |
| [11] |
TAHERI Y, HERRERA-BRAVO J, HUALA L, et al. Cyperus spp.: a review on phytochemical composition, biological activity, and health-promoting effects[J]. Oxidative Medicine and Cellular Longevity, 2021, 2021(1): 4014867. DOI:10.1155/2021/4014867 |
| [12] |
BOLGER A M, LOHSE M, USADEL B. Trimmomatic: a flexible trimmer for illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. |
| [13] |
NICOLAS D, PATRICK M, GUILLAUME S. NOVOPlasty: de novo assembly of organelle genomes from whole genome data[J]. Nucleic Acids Research, 2017, 45(4): e18. |
| [14] |
TILLICH M, LEHWARK P, PELLIZZER T, et al. GeSeq-versatile and accurate annotation of organelle genomes[J]. Nucleic Acids Research, 2017, 45(W1): W6-W11. |
| [15] |
GREINER S, LEHWARK P, BOCK R. Organellar Genome DRAW (OGDRAW) version 1.3.1:expanded toolkit for the graphical visualization of organellar genomes[J]. Nucleic Acids Research, 2019, 47(W1): W59-W64. |
| [16] |
BEIER S, THIEL T, MVNCH T, et al. MISA-web: a web server for microsatellite prediction[J]. Bioinformatics, 2017, 33(16): 2583-2585. |
| [17] |
谢海坤, 焦健, 樊秀彩, 等. 基于高通量测序组装'赤霞珠'叶绿体基因组及其特征分析[J]. 中国农业科学, 2017, 50(9): 1655-1665. |
| [18] |
KURTZ S, CHOUDHURI J V, OHLEBUSCH E, et al. REPuter: the manifold applications of repeat analysis on a genomic scale[J]. Nucleic Acids Research, 2001, 29(22): 4633-4642. |
| [19] |
KURTZ S, PHILLIPPY A, DELCHER A L, et al. Versatile and open software for comparing large genomes[J]. Genome Biology, 2004, 5(2): 1-9. |
| [20] |
LI D M, ZHAO C Y, LIU X F. Complete chloroplast genome sequences of Kaempferia galanga and Kaempferia elegans: molecular structures and comparative analysis[J]. Molecules, 2019, 24(3): 474. |
| [21] |
FAN X, YUAN K, ZHENG Y. The complete chloroplast genome and phylogenetic analysis of Cyperus brevifolius (Rottb.) Hassk.1844 (Cyperaceae)[J]. Mitochondrial DNA Part B, 2024, 9(5): 621-624. |
| [22] |
YANG L L, NIU J Q, TANG W W. The complete chl- oroplast genome of pioneering plant Cyperus iria L.(Cyperaceae) in ecological restoration[J]. Mitochondrial DNA Part B, 2021, 6(4): 1335-1336. |
| [23] |
姚钦予, 孙志高, 胡星云, 等. 氮负荷增强对闽江口短叶茳芏湿地植物-土壤系统氮累积与分配的影响[J]. 生态学报, 2024, 44(11): 4831-4843. |
| [24] |
张鹏飞, 孙志高, 陈冰冰, 等. 闽江河口芦苇与短叶茳芏空间扩展对湿地系统磷赋存特征的影响[J]. 应用生态学报, 2020, 31(12): 4258-4266. |
| [25] |
王华, 孙志高, 李家兵, 等. 闽江口芦苇与短叶茳芏湿地土壤无机硫形态分布特征及其影响因素[J]. 生态学报, 2019, 39(13): 4921-4932. |
| [26] |
PAN L, CHEN K, LIAO X. The complete chloroplast genome and phylogenetic analysis of Cyperus malaccensis Lam (Cyperaceae)[J]. Mitochondrial DNA Part B, 2024, 9(1): 114-118. |
| [27] |
REN W, GUO D, XING G, et al. Complete chloroplast genome sequence and comparative and phylogenetic analyses of the cultivated Cyperus esculentus[J]. Diversity, 2021, 13(9): 405. |
| [28] |
段春燕, 张凯, 段义忠. 长柄扁桃叶绿体基因组比较[J]. 生物工程学报, 2020, 36(12): 4921-4932. |
| [29] |
阳秀玫, 陈亮明, 禹霖, 等. 华中樱叶绿体基因组特征与分子标记[J]. 中南林业科技大学学报, 2024, 44(1): 175-184. |
| [30] |
杨芳. 七里香蔷薇叶绿体基因组测序及结构分析[J]. 基因组学与应用生物学, 2019, 38(8): 3586-3594. |



