2. 自然资源部北部湾滨海湿地生态系统野外科学观测研究站,广西北海 536015
2. Observation and Research Station of Coastal Wetland Ecosystem in Beibu Gulf, Ministry of Natural Resources, Beihai, Guangxi, 536015, China
在全球生物多样性丧失的背景下,生物监测在明确生物的多样性以及生态系统管理、保护方面发挥着重要的作用[1]。传统的环境生物监测方法作为侵入性技术,通常需要大量的捕捞和调查工作,可能对生物个体、种群及其栖息地带来负面影响。环境DNA(environmental DNA,eDNA)技术从环境中分离、提取DNA并结合宏条形码(Metabarcoding)技术来分析物种信息,是一种非侵入性的调查方法,在过去的十年里被广泛应用于水生生态系统中鱼类、无脊椎动物和微生物的生物资源评估以及生物多样性调查方面。继2023年1月4日起正式实施环境DNA生物监测与评估的3项技术标准后,江苏省地方标准《淡水生物环境DNA监测技术方法》(DB32/T 4539-2023)于2023年10月22日起正式实施。2024年6月,美国白宫科学技术政策办公室发布了一份《美国国家水环境DNA战略》(National Aquatic Environmental DNA Strategy),旨在推进快速、低成本和高效的eDNA技术,以了解海洋生物及其变化[2]。尽管环境DNA技术发展迅速并逐步成熟,但是该技术仍存在着一定的不足[3, 4]。例如,eDNA释放后在自然环境中会随着时间的延长而逐步衰减,但是其在环境中存活的时间相对较长,并可沿着水流长距离转移[5],容易造成假阳性结果,从而带来错误的判断[6]。在监测稀有野生物种和控制生物入侵时,选择一种能有效区分活体生物(代谢活跃)或死亡(休眠)生物的技术方法往往特别重要[7, 8]。寻找一种与eDNA相似、周转率更快的分子,如环境RNA(environmental RNA,eRNA),可能是解决上述问题的有效途径。由于RNA的单链结构以及内源性、外源性RNA酶的普遍存在,环境中RNA在体外的降解速度快于DNA。当RNA从生物体释放至环境中后会在一定时间内迅速降解[5, 9],这一特性有助于减少环境残留RNA对检测结果的影响,降低假阳性率,从而有效地指示与采样点密切相关的“当下状态”的生物组合[10]。基于这一特点,eRNA技术应用于生物监测可以弥补eDNA技术的一些不足。
相较于eDNA技术来说,eRNA技术是一种新兴的生物多样性监测手段,目前该技术还未被广泛应用。因此,本文将详细阐述eRNA技术的方法学研究、目前的应用情况、优势以及局限性,并利用文献计量学对该技术的国内外研究进展进行量化分析,展望其未来研究方向。
1 eRNA技术的概述eRNA是环境中的RNA,通常是由生物体以细胞形式、囊泡形式或游离形式释放至周围环境中[11]。eRNA在环境中得以保存的原因尚未完全清楚,推测可能存在几种机制,如囊泡、细胞区室、病毒蛋白质衣壳、生物膜或其他结合分子提供了对RNA的保护[5]。eRNA技术是指提取环境中的RNA、反转录后进行PCR扩增和高通量测序,并用生物信息学分析样本中包含的生物遗传信息,进一步解析物种构成的一种技术。在分子生物学研究中,DNA揭示了一个物种所拥有的基因,而RNA则解释了哪些基因在工作、哪些基因是沉默的。RNA只能由活跃的生物体产生,是一种非常脆弱的分子,在环境中释放时会因为自发水解以及丰富的RNA酶的存在而迅速降解,因此其在环境样本中所能提取的数量曾被认为不足以指示生物的多样性。然而越来越多的证据表明,eRNA在陆地和水生环境中有丰富的表达[5, 12, 13],科学家们甚至从化石中提取到了病毒、酵母、植物和动物的RNA,生物体释放至环境中的RNA量足以解析群落组成和分析基因表达[5, 14]。因此,eRNA技术作为一种新兴的技术手段,在评估生态系统和生态水平的快速变化、海洋/淡水生物安全、活跃生物和死亡生物体区分等方面受到了研究人员的广泛关注[9, 15]。
2 eRNA技术检测结果的影响因素研究者对影响eDNA降解的生物和非生物因素开展了多方面的研究,例如pH值[16-18]、水温[17, 19]、盐度[20]、紫外线[17]、微生物活动[21]、生物量[19]和DNA特性[22, 23],但对于影响eRNA降解的因素认识尚处于起步阶段。复杂的自然环境中,各种因素都会导致eRNA的降解。eRNA在环境中的丰度和持久性可能取决于多个因素:释放的速度和机制、分子结构(分子长度、构象)、运输机制(游离,或者与粒子、细胞、细胞器、囊泡膜、衣壳蛋白结合)、非生物环境(光、氧气、pH值、盐度、湿度、底物)和生物环境(生物群落的种类组成、微生物群落对有机质的分解)。
eRNA的降解是由核酸酶引起,生物体内的内源核酸酶和微生物活动产生的核酸外切酶均可将eRNA分解成小片段直至检测不出。环境因素的变化会直接影响相关酶的水解反应[24],从而影响eRNA的降解速率,例如温度、紫外线和pH值等,其中温度的影响最为显著[17]。高温(>50 ℃)会直接导致DNA/RNA变性和降解,虽然不乏一些生物体如嗜热生物体可以在高温下存活[25],但是绝大多数生物难以抵抗高温。温度已被证明是影响eDNA降解的关键因素之一,相比之下,eRNA受温度的影响要小得多[26-30]。Qian等[10]以中国对虾(Fenneropenaeus chinensis)为研究对象,发现移除水族缸中生物体后,eDNA比eRNA更易受温度的影响,eDNA随着温度的降低衰减速率明显下降,25 ℃、8 h后即检测不到eDNA,而在10 ℃、383 h后eDNA浓度才降解到检测限度以下;另外,eRNA在10、15、20、25 ℃ 4个温度下的降解速率差异较小,能够检测到eRNA的时间分别为22、22、14、11 h,表现出较好的稳定性,提示eRNA在低温下比eDNA具有更强的时效性,可以有效减少因eDNA残留时间过长而导致的错误结果。
碱性条件下,由于2′-OH的存在,游离形式的RNA比DNA更容易发生碱性水解而不稳定[31, 32],然而无论样品pH值如何,eRNA都可以在环境中保存足够长的时间,足以被检测和提取。酸度对eDNA降解影响的研究已有较多报道[16-18],但eRNA受pH值影响的研究还较少。Kagzi等[33]在受控的水箱内评估去除实验动物后,eRNA和eDNA在不同pH值条件下的降解速率,结果发现在各种pH值梯度下,eDNA均比eRNA能保留更长的时间跨度并具备更低的衰减速率,但是eDNA在中性和碱性条件下的衰减速率明显高于酸性条件,而eRNA的衰减速率在不同pH值条件下并无显著差异。这一结果可能是因为eRNA是以一种复杂的状态受到细胞、细胞器或其他结合分子等的保护,而不是以游离的状态直接暴露于核酸酶的作用下[5]。
eRNA的释放还与物种相关,即物种与物种间的生理状态及应激状态存在差异。前述有关中国对虾的研究中,COI基因的eDNA和eRNA在24 h内的释放量基本一致[10]。Wood等[34]研究了海洋无脊椎动物缨鳃虫(Sabella spallanzanii)和柄海鞘(Styela clava)的eRNA产生和降解,发现COI mRNA释放到环境的浓度明显低于eDNA,但eRNA的衰减速率常数较eDNA没有显著差异,且两者的变化趋势一致,移除生物体后,eRNA在13 h时仍能被检测到。Marshall等[35]发现贻贝eRNA在不同条件下的衰减速率常数有差异,其脱落及降解率与生物体的丰度、生物量呈正相关,COI mRNA和H2B mRNA释放至环境的初始浓度大于eDNA;另外,不同基因的eRNA降解率存在显著差异,rRNA在环境中停留时间比mRNA更长,其中16S rRNA在第6天和第10天时能检测到的概率分别是92.31%和69.23%,18S rRNA在第10天时能检测到的概率是100%,而H2B mRNA在48 h时能检测到的概率仅23.08%,COI mRNA在72 h后未能被检测出。一些物种的eRNA浓度在释放初期甚至可以高于相应的eDNA,移除生物体后,其释放至水环境的mRNA在13-72 h仍可以被检测出[34-36]。不同基因的eRNA的释放和降解速率不同,提示在用eRNA研究生物多样性时,可根据不同的研究目的选择不同的条形码基因作为检测对象。
另外,提取的eRNA如果混有eDNA则可能会影响检测结果。李龙召等[37]通过eRNA技术评价沉积物中纤毛虫分子多样性时发现,反转录之前先纯化RNA,可去掉eDNA污染,能够获得更加准确且具有生命活性的底栖生物群落信息。
了解eRNA技术检测结果的影响因素有助于研究人员根据现场研究目标合理地选择调查方法以及合适的靶标基因,这在早期发现入侵物种或监测稀有/濒危种群等情况下尤其重要[33]。
3 eRNA技术在不同水域生态系统中的研究 3.1 淡水生态系统在研究湖泊鱼类群落组成时,Littlefair等[38]发现能够从水中提取eRNA,并可像eDNA一样提供可靠的物种组成信息,这两种分子在描述鱼类群落组成方面的差异相对较小,并且eRNA的易降解性可以更好地检测当下的生物组合,避免外源性DNA的带入,从而正确地检测出更多已知的、存在于湖泊中的物种。Miyata等[13]分别使用eRNA技术、eDNA技术和传统监测方法对河流中的鱼类进行监测,结果发现eRNA技术与传统监测方法检测得到的丰度的相关性要高于eDNA技术,并认为eRNA技术比eDNA技术能够更好地量化实际居住在特定区域的鱼类丰度。
3.2 海洋生态系统Laroche等[39]利用eDNA/eRNA技术在海区不同类型的勘探钻井点对底栖生物进行监测,认为eDNA/eRNA技术组合适宜用作生物多样性评估的独立方法或作为基于形态监测方法的补充。Lejzerowicz等[40]通过对深海底栖有孔虫的多样性研究发现,基于eDNA的方法和基于eRNA的方法所获得的研究结果具有相似的分类组成,认为eRNA更适合于研究当下活跃的群落成分。Pochon等[41]在评估鲑鱼养殖场对海底沉积物有孔虫富集度的影响时发现,总计有349个操作分类单元(OTUs)来自eDNA和eRNA的数据集,其中从eDNA和eRNA样本分别检测出64和43个特有OTUs,另有242个共有OTUs在两者中均被检测到。eRNA不仅被用于生物监测和生物多样性评估方面,而且还可被用于生态毒理学方面的研究。如Greco等[42]利用eRNA分析沉积物中不同浓度铬对物种多样性的影响,结果发现eRNA技术监测可得出有孔虫多样性与沉积物中的铬浓度呈显著负相关的结论,并指出eRNA技术应用于解析重金属铬浓度对小型动物多样性的影响要优于eDNA技术。
4 eRNA技术的优势与局限 4.1 eRNA/eDNA技术与传统方法比较在传统的调查方法中,目标生物的存在或丰度是通过捕获或记录个体来确定的,具有侵入性大、耗时、费力等缺点[43],而eRNA和eDNA在自然环境中普遍存在,可以来自动物的粪便、尿液、黏液以及脱落细胞[5],通过检测释放到环境中的RNA或DNA来识别个体,不需要破坏生物栖息地,同时又较少依赖形态专业知识[44, 45]。但目前eRNA和eDNA技术也存在一些问题,如难以获得物种的发育时期、性别比例等信息,且在样本提取检测时又因为没有标准的流程,往往容易出现人为污染或样本间的交叉污染,导致假阳性结果[46]。
4.2 与eDNA技术比较死亡的生物体仍会产生大量的DNA,但RNA主要由具有生理活性的活体生物体产生,因此与eDNA相比,高周转率的eRNA更能反映生物的活跃情况以及生物停留时间的长短[5],从而推断出生物“实时”的生态状态。因此,相对于eDNA技术可能发生的高假阳性率,eRNA可以更好地反映生物体的代谢是否活跃[5, 9, 39]。
作为一种监测技术,监测的灵敏度、准确度、覆盖度都是考量的关键点。由表 1可知,在不同生态系统中利用eRNA和eDNA技术进行物种多样性监测所得的结果存在差异。例如,Miyata等[13]对日本那珂川流域的淡水鱼类分别进行了基于eDNA和eRNA的多样性检测,结果显示eDNA技术共检测到83种鱼类,出现了多种本不应该生活在淡水河流中的海水鱼类,相对于eRNA技术来说假阳性结果更多,认为eRNA相比eDNA可以更好地作为量化特定区域实际栖息鱼类丰度的指标。Littlefair等[38]利用eDNA和eRNA对加拿大安大略省西北部淡水研究基地国际可持续发展研究所(IISD)实验湖地区的生物群落进行分析,结果发现与往年传统监测方法所得的结果相比,eDNA技术的平均样本覆盖率为0.882,eRNA技术的平均样本覆盖率为0.863,但是eRNA技术真阳性率明显高于eDNA技术,能正确检测出湖泊中更多的、已知存在的物种,说明eRNA技术适合作为eDNA技术的一种补充手段来描述活跃的生物群落。Guardiola等[47]通过eRNA和eDNA 2种技术方法研究深海沉积物中生物群落的多样性,发现eDNA技术和eRNA技术获得了不同的群落组成成分,并由此认为它们应被视为互补工具,并尽可能同时使用。Laroche等[14]在用eRNA技术衡量Alpha多样性时发现其对环境的影响具有更强的响应,而eDNA技术则善于检测对群落组成的影响。
| 区域 Region |
环境样本 Environ-mental sample |
主要检测对象 Main detection object |
eDNA技术检测结果 Test results of eDNA technology |
eRNA技术检测结果 Test results of eRNA technology |
eRNA技术检测的优势 Advantages of eRNA technology detection |
参考文献 References |
| North-West Italy(Piedmont administrative region) | Soil cores | Fungal community | 2 404 OTUs detected in DNA samples | 1 811 OTUs detectedin RNA samples | The eRNA technologyperformed better in ana-lysing active microbial communities | [12] |
| Nakagawa-oohashi and Shin-nakabashi on the Naka river in Japan | Water samples | Fishes | 83 species were detected | 55 species were detected | Correlation with abun-dance in the ecologicalsurvey was higher foreRNA than for eDNA | [13] |
| New Zealand′s NorthIsland in the SouthTaranaki Bight | Sediment samples | Ecological community | 18 564 OTUs obtained for bacteria, 5 379OTUs for eukaryotes | 15 752 OTUs obtained for bacteria, 6 241OTUs for eukaryotes | If funds, time and expertise allow, be assessed using both eDNA and eRNA products | [14] |
| IISD ExperimentalLakes Area (IISD-ELA), a freshwaterresearch facility innorthwest Ontario, Canada | Water samples | Ecological community | The average samplecoverage for eDNAwas 0.882 | The average samplecoverage for eRNAwas 0.863 | The rate of true positives was significantly higher than the eDNA results, and more species known to be present in the lake were correctly detected | [38] |
| Chinook salmon farms in the Marlborough Sounds, New Zealand | Surface sediment | Benthic foraminifera | A total of 349 OTUs were obtained, ofwhich 64 were detected only in eDNA samples | A total of 349 OTUs were obtained, ofwhich 43 were detected only in eRNA samples | There were stronger correlations among the macrofauna count data and environmental data for the eRNA data compared with the eDNA data | [41] |
| Blanes Canyon (Western MediterraneanSea, NE Iberian Peninsula) and the adjacent open slope | Deep-sea sediment | Ecological community | A total of 4 151 OTUs were obtained, 3 542 were detected in eDNA samples | A total of 4 151 OTUs were obtained, 2 931 were detected in eRNA samples | The eRNA technologyperformed better in analysing Nematoda | [47] |
| Southeastern coast of Minami-Torishima Island in the North Pacific Ocean | Sediment cores | Meiofauna | 305 OTUs detectedonly in eDNA | 2 064 OTUs were detected only in eRNAs | eRNA technology detects more OTUs than eDNA technology | [48] |
| Auckland ViaductHarbor, New Zealand | Plate samples andwatersamples | Ecological community | 3 671 ASVs were obtained in eDNA samples | 1 872 ASVs were obtained in eRNA samples | eRNA technology is better for detecting “live” signals in environmental samples | [49] |
虽然eRNA在环境中检测分子的时效性要优于eDNA,但是eRNA的时效性也会因为eRNA的快速降解而造成假阴性的问题。研究人员使用eRNA技术监测某水体中的生物时,如无法检测出eRNA,他们则认为该物种已经离开或不再栖息在该生境中,而事实可能并非如此[10]。因此,不建议直接用eRNA代替eDNA监测[10],有必要使用多重方法,综合分析检测结果,才可能避免这一问题的出现。
5 基于文献检索的国内外eRNA技术研究现状通过研究不同时间段某一领域的发文量,能较为直观地体现该领域研究趋势的变化,因此使用Web of Science核心合集数据库(网址为https://apps.webofknowledge.com)检索2015年1月至2024年3月发表的、主题为“environmental RNA,eRNA”的文献,经人工校正去除与主题不相关的文献,共检索出54篇论文。采用VOSviewer软件[50]进行科学知识图谱绘制,对文献发表国家和核心关键词进行共现与聚类分析。其中,有关eRNA的英文文章在2015年仅1篇,表明该领域的研究这时候才刚起步(图 1);随后发文数量逐年增加,2021-2023年年均发文数量超过10篇。
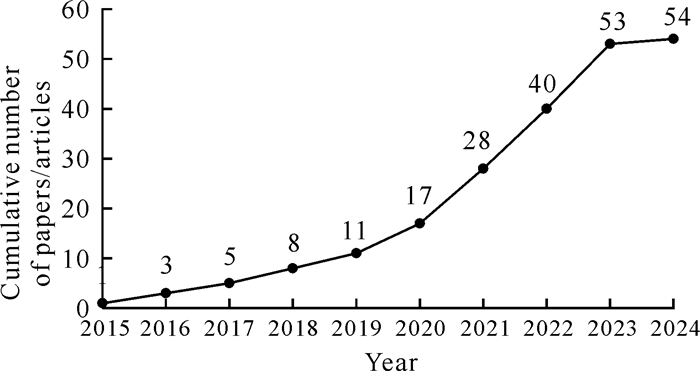
|
| 图 1 eRNA研究论文累计发文量统计 Fig.1 Cumulative publication statistics of research papers about eRNA |
采用VOSviewer软件对eRNA发文国家进行合作网络可视化,分析结果见图 2,其中圆圈越大表示发文越多。按国家分类,来自美国、新西兰和日本的研究机构发文量排名前3,分别发表14、12、11篇(表 2)。美国的发文量明显高于其他国家,占总发文量的25.93%;新西兰次之,占比22.22%。在中国知网中搜索“环境RNA”得到的文献数为3篇,其中1篇为2022年发表,另外两篇发表时间为2023年,说明国内的eRNA研究处于刚起步阶段。不同国家论文的影响力和质量以篇均被引频次来衡量,篇均被引频次越高,说明该国家的发文水平和质量越高,该统计数据如表 2所示, 从中可以看出新西兰的篇均被引频次最高。
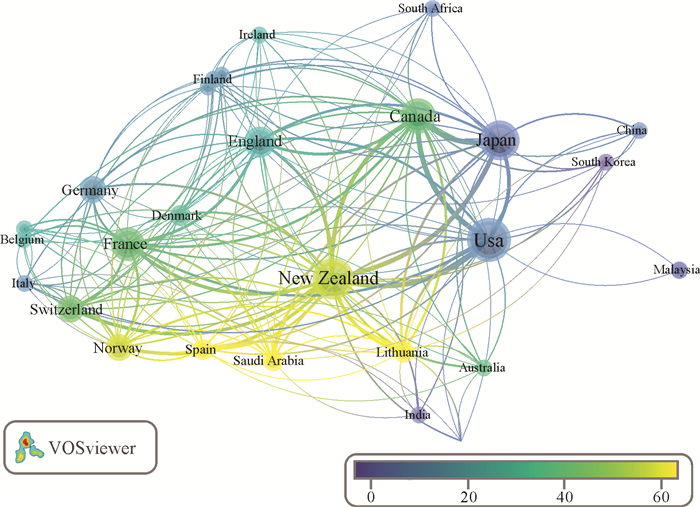
|
| 图 2 eRNA研究文献发表国家合作网络图谱 Fig.2 Mapping of national collaborative networks for the publication of research paper focusing eRNA |
| 排名 Ranking |
国家 Nation |
发文量/篇 Number of publications/articles |
篇均被引频次/次 Citation frequency of per article/time |
| 1 | USA | 14 | 10.64 |
| 2 | New Zealand | 12 | 53.58 |
| 3 | Japan | 11 | 7.18 |
| 4 | Canada | 8 | 37.25 |
| 5 | France | 7 | 37.86 |
关键词是一篇论文的精髓和核心,是对论文主题的高度凝练,出现频次高的关键词可以看作是研究领域的研究热点。利用VOSviewer软件对eRNA研究相关文献关键词进行共现和聚类分析,可以发现该领域的热点和焦点。“biomonitoring”“biodiversity”“degradation”“extracellular DNA”“diversity”5个关键词频次最高,其中“biomonitoring”出现78次,“degradation”出现61次,并在图谱中与其他关键词关系密切(图 3),这表明对eRNA的生物多样性监测及eRNA是否容易降解的研究是最热门的。从关键词共现聚类视图中可以看出,有关eRNA的研究主要呈现3个聚类:聚类一(红色标注)关注eRNA技术的可行性问题,如环境中遗留的RNA能在环境中保存多久、是否容易降解,等等;聚类二(绿色标注)关注eRNA技术在生物监测领域已开展的实际应用;聚类三(蓝色标注)的研究主要集中在eDNA/eRNA技术在小型底栖动物多样性解读上的优势。
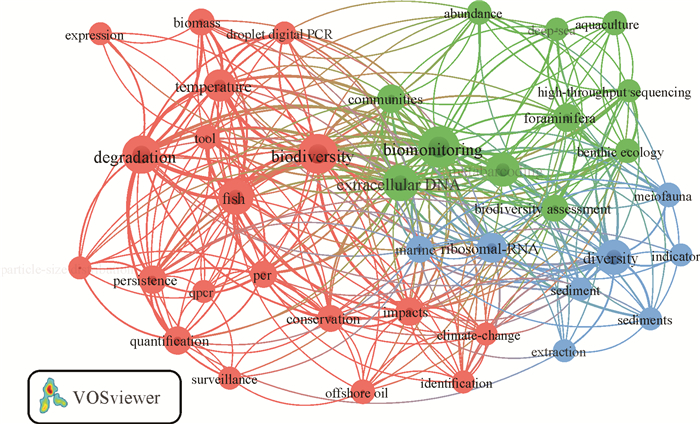
|
| 图 3 eRNA相关论文核心关键词共现聚类视图 Fig.3 Core keywords co-occurrence clustering view of eRNA related papers |
6 展望
eRNA和eDNA技术同为新兴的非侵入性监测技术,对环境破坏性小,准确性高,是传统生态调查方法的有效补充,但两种技术各有利弊。eRNA技术能够灵敏检测到生物体释放至环境中的遗传信息的变化,且由于RNA快速降解的特性,相对于eDNA技术来说可提高生物监测的准确性和分辨率。但是,正因为eRNA易受环境影响而降解,容易丢失有效信息,并且与eDNA技术相比,eRNA的样品采集、处理等过程更为复杂且需更加谨慎。由此可见,eRNA技术不能完全替代eDNA技术,二者应互相结合、优势互补,从而有效减少误差,提高检测的准确性。目前,通过eRNA技术开展生物多样性的研究主要集中在美国、新西兰和日本等发达国家,而国内基于eRNA技术的研究基本上空白。随着分子生物学技术的不断发展,国内外科研工作者需提高认知,继续关注未来eRNA技术的改进和优化、跨领域合作与应用拓展、数据整合与分析平台建设等方面的工作,提高生物监测结果的准确性和可靠性。
| [1] |
KELLY R P, PORT J A, YAMAHARA K M, et al. Harnessing DNA to improve environmental management[J]. Science, 2014, 344(6191): 1455-1456. DOI:10.1126/science.1251156 |
| [2] |
KELLY R P, LODGE D M, LEE K N, et al. Toward a national eDNA strategy for the United States[J]. Environmental DNA, 2024, 6(1): e432. DOI:10.1002/edn3.432 |
| [3] |
DEJEAN T, VALENTINI A, DUPARC A, et al. Persistence of environmental DNA in freshwater ecosystems[J]. PLoS One, 2011, 6(8): e23398. DOI:10.1371/journal.pone.0023398 |
| [4] |
JANE S F, WILCOX T M, MCKELVEY K S, et al. Distance, flow and PCR inhibition: eDNA dynamics in two headwater streams[J]. Molecular Ecology Resources, 2015, 15(1): 216-227. DOI:10.1111/1755-0998.12285 |
| [5] |
CRISTESCU M E. Can environmental RNA revolutionize biodiversity science?[J]. Trends in Ecology & Evolution, 2019, 34(8): 694-697. |
| [6] |
DEINER K, ALTERMATT F. Transport distance of invertebrate environmental DNA in a natural river[J]. PLoS One, 2014, 9(2): e88786. DOI:10.1371/journal.pone.0088786 |
| [7] |
DARLING J A, MAHON A R. From molecules to management: adopting DNA-based methods for monitoring biological invasions in aquatic environments[J]. Environmental Research, 2011, 111(7): 978-988. DOI:10.1016/j.envres.2011.02.001 |
| [8] |
MERKES C M, MCCALLA S G, JENSEN N R, et al. Persistence of DNA in carcasses, slime and avian feces may affect interpretation of environmental DNA data[J]. PLoS One, 2014, 9(11): e113346. DOI:10.1371/journal.pone.0113346 |
| [9] |
POCHON X, ZAIKO A, FLETCHER L M, et al. Wanted dead or alive? Using metabarcoding of environmental DNA and RNA to distinguish living assemblages for biosecurity applications[J]. PLoS One, 2017, 12(11): e0187636. DOI:10.1371/journal.pone.0187636 |
| [10] |
QIAN T, SHAN X, WANG W, et al. Effects of temperature on the timeliness of eDNA/eRNA: a case study of Fenneropenaeus chinensis[J]. Water, 2022, 14(7): 1155. DOI:10.3390/w14071155 |
| [11] |
YATES M C, DERRY A M, CRISTESCU M E. Environmental RNA: a revolution in ecological resolution?[J]. Trends in Ecology & Evolution, 2021, 36(7): 601-609. |
| [12] |
ADAMO M, VOYRON S, CHIALVA M, et al. Metabarcoding on both environmental DNA and RNA highlights differences between fungal communities sampled in different habitats[J]. PLoS One, 2020, 15(12): e0244682. DOI:10.1371/journal.pone.0244682 |
| [13] |
MIYATA K, INOUE Y, AMANO Y, et al. Fish environmental RNA enables precise ecological surveys with high positive predictivity[J]. Ecological Indicators, 2021, 128: 107796. DOI:10.1016/j.ecolind.2021.107796 |
| [14] |
LAROCHE O, WOOD S A, TREMBLAY L A, et al. Metabarcoding monitoring analysis: the pros and cons of using co-extracted environmental DNA and RNA data to assess offshore oil production impacts on benthic communities[J]. PeerJ, 2017, 5: e3347. DOI:10.7717/peerj.3347 |
| [15] |
VEILLEUX H D, MISUTKA M D, GLOVER C N. Environmental DNA and environmental RNA: current and prospective applications for biological monitoring[J]. The Science of the Total Environment, 2021, 782: 146891. DOI:10.1016/j.scitotenv.2021.146891 |
| [16] |
SEYMOUR M, DURANCE I, COSBY B J, et al. Acidity promotes degradation of multi-species environmental DNA in lotic mesocosms[J]. Communications Biology, 2018, 1: 4. DOI:10.1038/s42003-017-0005-3 |
| [17] |
STRICKLER K M, FREMIER A K, GOLDBERG C S. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms[J]. Biological Conservation, 2015, 183: 85-92. DOI:10.1016/j.biocon.2014.11.038 |
| [18] |
TSUJI S, YAMANAKA H, MINAMOTO T. Effects of water pH and proteinase K treatment on the yield of environmental DNA from water samples[J]. Limnology, 2017, 18(1): 1-7. DOI:10.1007/s10201-016-0483-x |
| [19] |
JO T, MURAKAMI H, YAMAMOTO S, et al. Effect of water temperature and fish biomass on environmental DNA shedding, degradation, and size distribution[J]. Ecology and Evolution, 2019, 9(3): 1135-1146. DOI:10.1002/ece3.4802 |
| [20] |
DELL'ANNO A, CORINALDESI C. Degradation and turnover of extracellular DNA in marine sediments: ecological and methodological considerations[J]. Applied and Environmental Microbiology, 2004, 70(7): 4384-4386. DOI:10.1128/AEM.70.7.4384-4386.2004 |
| [21] |
ZULKEFLI N S, KIM K H, HWANG S J. Effects of microbial activity and environmental parameters on the degradation of extracellular environmental DNA from a eutrophic lake[J]. International Journal of Environmental Research and Public Health, 2019, 16(18): 3339. DOI:10.3390/ijerph16183339 |
| [22] |
JO T, MURAKAMI H, MASUDA R, et al. Rapid degradation of longer DNA fragments enables the improved estimation of distribution and biomass using environmental DNA[J]. Molecular Ecology Resources, 2017, 17(6): e25-e33. |
| [23] |
TURNER C R, BARNES M A, XU C C Y, et al. Particle size distribution and optimal capture of aqueous macrobial eDNA[J]. Methods in Ecology and Evolution, 2014, 5(7): 676-684. DOI:10.1111/2041-210X.12206 |
| [24] |
LINDAHL T. Instability and decay of the primary structure of DNA[J]. Nature, 1993, 362(6422): 709-715. DOI:10.1038/362709a0 |
| [25] |
LÓPEZ-GARCÍA P, PHILIPPE H, GAIL F, et al. Autochthonous eukaryotic diversity in hydrothermal sediment and experimental microcolonizers at the Mid-Atlantic Ridge[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(2): 697-702. |
| [26] |
HOFREITER M, SERRE D, POINAR H N, et al. Ancient DNA[J]. Nature Reviews Genetics, 2001, 2(5): 353-359. DOI:10.1038/35072071 |
| [27] |
ZHU B. Degradation of plasmid and plant DNA in water microcosms monitored by natural transformation and real-time polymerase chain reaction (PCR)[J]. Water Research, 2006, 40(17): 3231-3238. DOI:10.1016/j.watres.2006.06.040 |
| [28] |
CORINALDESI C, BEOLCHINI F, DELL'ANNO A. Damage and degradation rates of extracellular DNA in marine sediments: implications for the preservation of gene sequences[J]. Molecular Ecology, 2008, 17(17): 3939-3951. DOI:10.1111/j.1365-294X.2008.03880.x |
| [29] |
POTÉ J, ACKERMANN R, WILDI W. Plant leaf mass loss and DNA release in freshwater sediments[J]. Ecotoxicology and Environmental Safety, 2009, 72(5): 1378-1383. DOI:10.1016/j.ecoenv.2009.04.010 |
| [30] |
FU X H, WANG L, LE Y Q, et al. Persistence and renaturation efficiency of thermally treated waste recombinant DNA in defined aquatic microcosms[J]. Journal of Environmental Science and Health.Part A, Toxic/Hazardous Substances & Environmental Engineering, 2012, 47(13): 1975-1983. |
| [31] |
BERNHARDT H S, TATE W P. Primordial soup or vinaigrette: did the RNA world evolve at acidic pH?[J]. Biology Direct, 2012, 7: 4. DOI:10.1186/1745-6150-7-4 |
| [32] |
OIVANEN M, KUUSELA S, LÖNNBERG H. Kinetics and mechanisms for the cleavage and isomerization of the phosphodiester bonds of RNA by Brønsted acids and bases[J]. Chemical Reviews, 1998, 98(3): 961-990. DOI:10.1021/cr960425x |
| [33] |
KAGZI K, HECHLER R M, FUSSMANN G F, et al. Environmental RNA degrades more rapidly than environmental DNA across a broad range of pH conditions[J]. Molecular Ecology Resources, 2022, 22(7): 2640-2650. DOI:10.1111/1755-0998.13655 |
| [34] |
WOOD S A, BIESSY L, LATCHFORD J L, et al. Release and degradation of environmental DNA and RNA in a marine system[J]. Science of the Total Environment, 2020, 704: 135314. DOI:10.1016/j.scitotenv.2019.135314 |
| [35] |
MARSHALL N T, VANDERPLOEG H A, CHAGANTI S R. Environmental (e) RNA advances the reliability of eDNA by predicting its age[J]. Scientific Reports, 2021, 11(1): 2769. DOI:10.1038/s41598-021-82205-4 |
| [36] |
TSURI K, IKEDA S, HIROHARA T, et al. Messenger RNA typing of environmental RNA (eRNA): a case study on zebrafish tank water with perspectives for the future development of eRNA analysis on aquatic vertebrates[J]. Environmental DNA, 2021, 3(1): 14-21. DOI:10.1002/edn3.169 |
| [37] |
李龙召, 黄平平, 徐奎栋, 等. DNA/RNA提取及处理方法对评价沉积物中纤毛虫分子多样性的影响[J]. 海洋科学, 2022, 46(7): 52-60. |
| [38] |
LITTLEFAIR J E, RENNIE M D, CRISTESCU M E. Environmental nucleic acids: a field-based comparison for monitoring freshwater habitats using eDNA and eRNA[J]. Molecular Ecology Resources, 2022, 22(8): 2928-2940. DOI:10.1111/1755-0998.13671 |
| [39] |
LAROCHE O, WOOD S A, TREMBLAY L A, et al. A cross-taxa study using environmental DNA/RNA metabarcoding to measure biological impacts of offshore oil and gas drilling and production operations[J]. Marine Pollution Bulletin, 2018, 127: 97-107. DOI:10.1016/j.marpolbul.2017.11.042 |
| [40] |
LEJZEROWICZ F, VOLTSKY I, PAWLOWSKI J. Identifying active foraminifera in the Sea of Japan using metatranscriptomic approach[J]. Deep Sea Research Part Ⅱ: Topical Studies in Oceanography, 2013, 86: 214-220. |
| [41] |
POCHON X, WOOD S A, KEELEY N B, et al. Accurate assessment of the impact of salmon farming on benthic sediment enrichment using foraminiferal metabarcoding[J]. Marine Pollution Bulletin, 2015, 100(1): 370-382. DOI:10.1016/j.marpolbul.2015.08.022 |
| [42] |
GRECO M, LEJZEROWICZ F, REO E, et al. Environmental RNA outperforms eDNA metabarcoding in assessing impact of marine pollution: a chromium-spiked mesocosm test[J]. Chemosphere, 2022, 298: 134239. DOI:10.1016/j.chemosphere.2022.134239 |
| [43] |
SEYMOUR M. Rapid progression and future of environmental DNA research[J]. Communications Biology, 2019, 2: 80. DOI:10.1038/s42003-019-0330-9 |
| [44] |
EVANS N T, SHIREY P D, WIERINGA J G, et al. Comparative cost and effort of fish distribution detection via environmental DNA analysis and electrofishing[J]. Fisheries, 2017, 42(2): 90-99. DOI:10.1080/03632415.2017.1276329 |
| [45] |
SIGSGAARD E E, CARL H, M∅LLER P R, et al. Monitoring the near-extinct European weather loach in Denmark based on environmental DNA from water samples[J]. Biological Conservation, 2015, 183: 46-52. DOI:10.1016/j.biocon.2014.11.023 |
| [46] |
赵明, 赵梦迪, 马春艳, 等. 环境DNA在水域生态中的研究进展[J]. 中国水产科学, 2018, 25(4): 714-720. |
| [47] |
GUARDIOLA M, WANGENSTEEN O S, TABERLET P, et al. Spatio-temporal monitoring of deep-sea communities using metabarcoding of sediment DNA and RNA[J]. PeerJ, 2016, 4: e2807. DOI:10.7717/peerj.2807 |
| [48] |
KITAHASHI T, SUGIME S, INOMATA K, et al. Meiofaunal diversity at a seamount in the Pacific Ocean: a comprehensive study using environmental DNA and RNA[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2020, 160: 103253. DOI:10.1016/j.dsr.2020.103253 |
| [49] |
VON AMMON U, WOOD S A, LAROCHE O, et al. Linking environmental DNA and RNA for improved detection of the marine invasive fanworm Sabella spallanzanii[J]. Frontiers in Marine Science, 2019, 6: 621. DOI:10.3389/fmars.2019.00621 |
| [50] |
VAN ECK N J, WALTMAN L. Software survey: VOSviewer, a computer program for bibliometric mapping[J]. Scientometrics, 2010, 84(2): 523-538. DOI:10.1007/s11192-009-0146-3 |



