2. 广西科学院, 广西海洋科学院(广西红树林研究中心), 广西红树林保护与利用重点实验室, 广西北海 536007
2. Guangxi Key Laboratory of Mangrove Conservation and Utilization, Guangxi Academy of Marine Science (Guangxi Mangrove Research Center), Guangxi Academy of Sciences, Beihai, Guangxi, 536007, China
经济贝类是重要的食品来源,也是海洋生态系统中不可或缺的部分。我国科研团队已经对一些重要的经济贝类开展了大量的遗传研究,包括基因组、转录组、表观遗传和分子标记等,如栉孔扇贝(Chlamys farreri)[1]、近江牡蛎(Crassostrea ariakensis)[2]、硬壳蛤(Mercenaria mercenaria)[3]、缢蛏(Sinonovacula constricta)[4]等全基因组精细参考图谱的陆续发布。贝类转录组测序的研究为功能基因、分子标记开发提供了数据支撑,加速了贝类遗传育种研究的进程:通过转录组测序得到了温度胁迫下马氏珠母贝(Pinctada fucata martensii)和缢蛏的差异表达基因,为双壳贝类对高低温胁迫的响应机制提供了理论基础[5, 6];通过转录组测序获得了泥蚶(Tegillarca granosa)耐低氧的候选基因[7];利用转录组学分析可以研究贝类在不同毒性胁迫下的免疫应答机制,了解其机体抗氧化酶系统及解毒代谢机制[8, 9]。对贝类不同组织和不同发育阶段进行转录组解读,可了解其器官分化的相关基因[10, 11]和变态发育过程中的关键基因[12]。
弓獭蛤(Lutraria rhynchaena Jonas, 1844,同物异名Lutraria arcuata Reeve, 1854)隶属于软体动物门(Mollusca)瓣鳃纲(Lamellibranchia)帘蛤目(Veneroida)蛤蜊科(Mactridae)獭蛤属(Lutraria)[13, 14]。弓獭蛤广泛分布在我国、日本、菲律宾和越南等沿海国家,在我国主要分布在广东、海南、广西和台湾沿海[13, 14],栖息于潮间带至水深80 m的浅海泥沙质海底,与獭蛤属的大獭蛤(L.maxima)、施氏獭蛤(L.sieboldii)和澳洲獭蛤(L.australis)等形态相似,在我国沿海居民皆俗称它们为象鼻螺。象鼻螺个体较大,每只成年个体质量90-120 g,我国自1997年开始通过浅海开放式底播方式人工养殖象鼻螺,养殖范围主要集中在南方,2020年北海的象鼻螺产量达6万多吨[15]。2022年,北海象鼻螺获得了中华人民共和国农产品地理标志登记证书(中华人民共和国农业农村部公告第532号)。
因弓獭蛤具有可观的经济价值,国内外学者对其开展了多方面的研究,国外关于弓獭蛤的研究主要由越南学者发表。越南学者发布了基于第三代纳米孔和第二代Illumina NovaSeq6000平台测序组装的弓獭蛤基因组草图,基因组大小为545-547 Mbp[16],但此后未有基于该基因组的拓展研究发表。研究者对越南弓獭蛤的地理分布、繁育技术、线粒体基因组、微卫星等也做了一些报道[17-19]。目前国内关于弓獭蛤研究的报道主要局限于苗种繁育技术[20]、受精卵的细胞学观察[21]、重金属响应[22]等。有研究者对弓獭蛤使用了高浓度的镉胁迫,发现其不同组织对重金属的胁迫响应存在显著差异,但是并未进行分子生物学层面的深入研究。总体来说,国内目前对弓獭蛤的遗传育种、功能基因表达等方面研究还处于起步阶段。
本研究以弓獭蛤的肌肉(斧足)、外套膜、鳃、肝胰腺、虹吸管、雌性性腺及雄性性腺7种组织为研究对象,采用RNA-seq测序技术进行转录组测序,通过数据组装、各组织表达量的比较和对差异表达基因注释获得弓獭蛤各组织的基因表达信息,为进一步开展弓獭蛤遗传育种、分子标记、功能基因的挖掘等方面研究提供数据参考。
1 材料与方法 1.1 材料测序用的弓獭蛤贝(其贝壳见图 1)采集于北海市金不换水产有限公司位于广西北海市侨港镇(109°9′30.85″N,21°18′2.92″E)的天然海域养殖场,均为成年活贝,平均湿重为(109.4±30.6) g、壳长为(92.5±17.8) mm、壳高为(47.1±3.8) mm、壳宽为(31.9±3.9) mm。取斧足、外套膜、鳃、肝胰腺、虹吸管、雌性性腺及雄性性腺7种组织,每种组织取9个个体样本,设置3个生物学重复,每3种组织样本混合后提取RNA。实时荧光定量PCR(Real Time qPCR)验证实验用的弓獭蛤贝购自北海市南珠市场。

|
| 图 1 弓獭蛤贝壳 Fig.1 Shell of L.rhynchaena |
1.2 方法 1.2.1 RNA的提取及检测
使用MiniBEST通用RNA提取试剂盒(TAKARA公司)提取各组织总RNA,提取步骤按照试剂盒说明书进行。使用NanoDrop分光光度计(Thermo Fisher Scientific公司)检测总RNA纯度,使用Qubit 2.0 (Thermo Fisher Scientific公司)检测总RNA浓度,琼脂糖凝胶电泳检测总RNA完整性。
1.2.2 文库构建及转录组测序检测合格的总RNA送上海凌恩生物科技有限公司,通过带有Oligo (dT)的磁珠富集具有polyA尾巴的真核mRNA后,采用Illumina Truseq RNA sample prep Kit方法完成7种组织共21个样品的文库构建,经质检合格后开展基于Illumina HiSeq 2500的RNA-seq测序。
1.2.3 生物信息学分析使用FastQC对测序得到的原始数据进行质量评估,使用trimmomatic软件(v0.36)去除reads接头序列、将序列末端(3′端)质量较低(质量值小于20)的碱基修剪掉,去除含N比率超过10%的reads,舍弃长度小于75 bp的序列,过滤后得到高质量的测序数据(Clean reads)。利用Trinity软件(v2.8.6)进行无参考转录组从头组装得到非冗余基因(Unigene)并预测开放阅读框(ORF)。用Salmon(v1.1.0)软件将质控后得到的高质量序列和拼接结果进行比对(Mapping)。将组装的序列分别在NR(Non-Redundant protein sequence database)、GO(Gene Ontology)、eggNOG(evolutionary genealogy of genes: Non-supervised Orthologous Groups)、KEGG(Kyoto Encyclopedia of Genes and Genomes)及Swiss-Prot等数据库中进行基因功能注释。
使用RESM v1.3.3软件计算各转录本在样本中的表达丰度,根据RPKM(Reads Per Kilobase per Million mapped reads)判断基因表达量,以P<0.05和|log2(FoldChange)|≥1为标准筛选差异表达基因(Differentially Expressed Genes,DEGs)。通过对不同组织之间DEGs进行GO和KEGG通路富集分析,确定DEGs参与的生化过程和信号转导途径。
1.2.4 实时荧光定量PCR验证分析随机选取9个差异基因进行实时荧光定量PCR实验,以判断RNA-seq数据结果是否可信。经前期实验筛选内参后,以β-actin作为内参基因。利用Primer premier 5.0软件设计引物如表 1所示。使用PrimeScripTM RT reagent Kit with gDNA Eraser试剂盒(TAKARA公司)将总RNA反转录合成cDNA。以反转录产物为模板,使用TB Green Premix Ex Taq Ⅱ(Tli RNaseH Plus)试剂盒(TAKARA公司)在QuantStudioTM3实时荧光定量PCR仪(Thermo Fisher Scientific公司)上进行基因表达相对定量分析。PCR反应体系为20 μL,PCR程序参照产品说明书进行。样品进行3次生物学重复和3次技术重复,用2-ΔΔCt方法计算基因的相对表达量。
| 基因注释 Gene annotation |
差异表达基因(序列号) DEGs (ID) |
正向引物序列(5′→3′) Forward primer sequence (5′→3′) |
反向引物序列(5′→3′) Reverse primer sequence (5′→3′) |
| β-actin | Reference gene | TGTTCCAGCCATCTTTCCTCGG | TGCATTCTGTCGGCAATACCTGG |
| Paramyosin-like isoform X4 | TRINITY_DN24180_ c2_g1 |
CCTGTTATCGTGTTGGACGCAGTC | GGCGATGAGCCCCTGTAGACATT |
| Uncharacterized protein LOC128233073 gene |
TRINITY_DN32671_ c2_g1 |
AAACATGTGGTCACGCAAAGGACG | CCCAGGGGATGGCAAAAGTCTCT |
| Beta-tubulin family protein | TRINITY_DN39293_ c3_g10 |
TGGTGCTGGCAACAACTGGGC | CCACCACCGAGGGAGTGAGTTAGC |
| Myosin, essential light chain, adductor muscle- like |
TRINITY_DN41770_ c1_g1 |
ATGAGCGGTTTGACTGAAGGCG | AACGATGGCATTTGTTGGGTTATG |
| Cytochrome b | TRINITY_DN43434_ c17_g1 |
GGGGATTGGACGAATGGTTTAACG | GCCTATCAAGCTCGACGGGGTCT |
| Uncharacterized protein LOC123546623 |
TRINITY_DN26713_ c0_g5 |
TTTCAGATTTTTGCTCTTTCGGCG | GCAACCATCCTTCCACTGAGCG |
| Unknown gene | TRINITY_DN1528_ c0_g1 |
ACGAAGGGGCAAATGTAAAGACCG | CAGCTGGCCGTTTTCATTGGTT |
| Unknown gene | TRINITY_DN11355_ c0_g1 |
AAAACAGCCTTCGAATCAGGGGT | TCCGTGCTTTCTGCAACCACAC |
| Hypothetical protein KUTeg_013128 |
TRINITY_DN10044_ c0_g2 |
TCACAAGACACAACGGTACAGGGG | CATTCGTGTTCGGTCCAGCATTC |
2 结果与分析 2.1 转录组测序结果 2.1.1 测序数据及转录本拼接
测序原始数据及质控数据统计结果如表 2所示。原始数据、有效数据、有效碱基为3个生物学重复样品之和,质控后Q20和Q30碱基比例为3个生物学重复样品的平均值。共测得1 039 382 252条raw reads,质控过滤后得到939 197 518条clean reads,各组织有效碱基数从15.39至29.26 G不等。各组织Phred数值分别大于20、30的碱基占总体碱基的百分比均分别大于等于96.93%和91.96%,数据质量达到后续分析条件。
| 样品 Sample |
原始数据 Raw reads |
有效数据 Clean reads |
有效碱基/G Clean bases/G |
Q20碱基比例/% Q20 base ratio/% |
Q30碱基比例/% Q30 base ratio/% |
| Muscles | 162 017 978 | 147 372 742 | 21.66 | 97.86 | 94.17 |
| Mantles | 156 425 372 | 142 236 382 | 20.92 | 97.28 | 93.27 |
| Gills | 124 370 610 | 110 431 134 | 16.05 | 97.13 | 92.54 |
| Hepatopancreases | 120 987 368 | 106 082 674 | 15.39 | 96.93 | 91.96 |
| Siphons | 124 436 732 | 112 546 950 | 16.52 | 97.86 | 93.92 |
| Female gonads | 132 136 526 | 122 619 518 | 18.12 | 98.22 | 94.97 |
| Male gonads | 218 997 666 | 197 908 118 | 29.26 | 97.75 | 93.85 |
2.1.2 基因功能注释
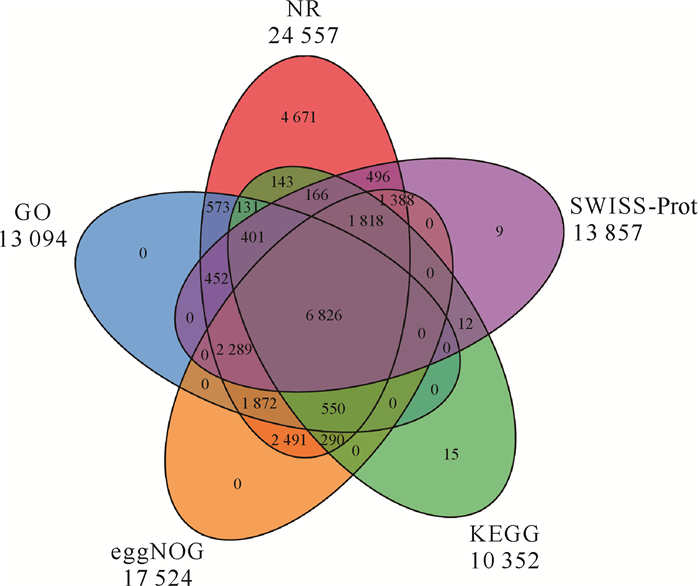
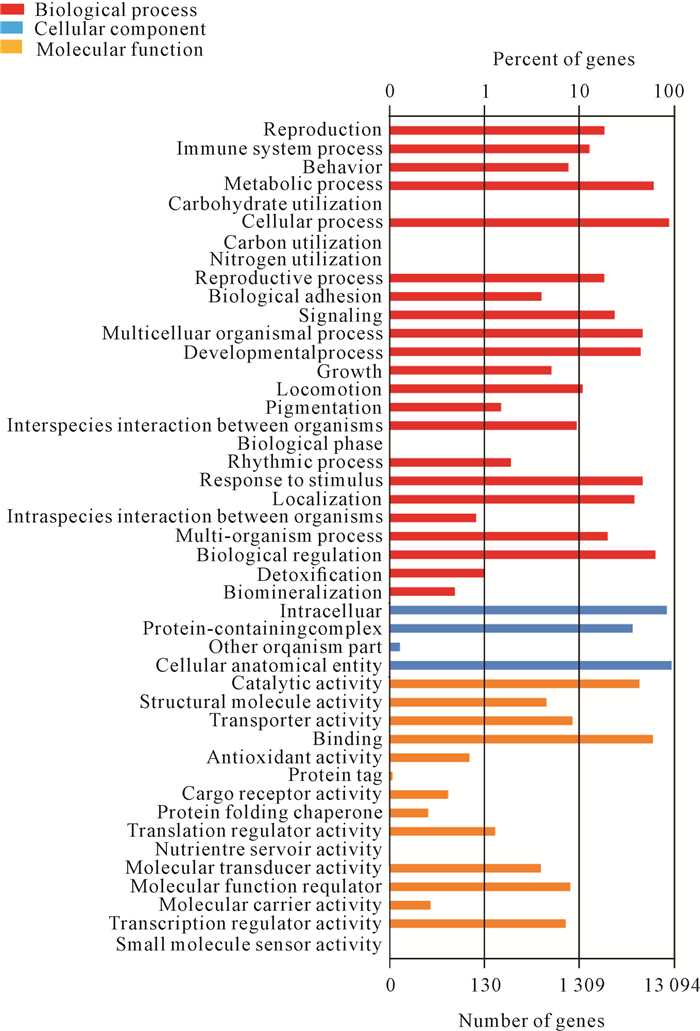
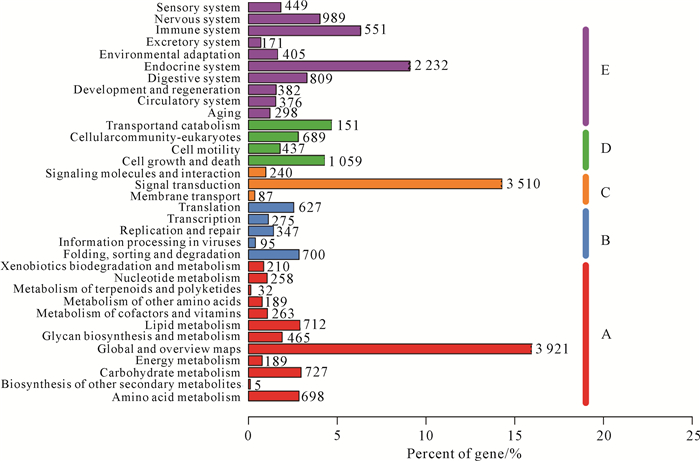
拼接得到56 773条Unigene,N50为1 373,平均长度为1 033.12 bp。将预测基因的蛋白序列在NR、GO、eggNOG、KEGG和SWISS-Prot等数据库中比对,分别得到24 557、13 094、17 524、10 352和13 857条注释信息(各数据库注释韦恩图见图 2)。根据GO注释将基因功能分为3大类别:生物过程(BP)、细胞成分(CC)和分子功能(MF),细分为45个分支(图 3)。通过eggNOG数据库进行基因对应的COG(Clusters of Orthologous Groups of proteins)注释,按注释结果进行功能分类得到了25个功能类别(图 4),其中注释基因数量最多的功能为信号转导机制,其次是功能未知的基因,然后是翻译后修饰、蛋白转运及伴侣蛋白。通过KEGG数据库得到10 352条注释信息,涵盖了5个功能大类:新陈代谢、遗传信息处理、环境信息处理、细胞过程和有机系统的生物过程通路(图 5)。通过SWISS-Prot数据库得到13 857条注释信息,注释结果可用作基因功能描述的参考。经5个数据库比对共有24 593条Unigene获得注释,注释率为43.32%。
|
| 图 2 5个数据库注释基因数量韦恩图 Fig.2 Venn diagram of the number of annotated Unigenes in five databases |
|
| 图 3 GO注释基因功能分类 Fig.3 GO functional classification of annotated genes |

|
| 图 4 COG注释基因功能分类 Fig.4 COG functional classification of annotated genes |
|
| A: Metabolism; B: Genetic information processing; C: Environmental information processing; D: Cellular processes; E: Organismal systems. 图 5 KEGG注释基因功能分类及数量 Fig.5 KEGG functional classification and number of annotated genes |
2.2 弓獭蛤不同组织间的差异分析 2.2.1 不同组织基因表达分析
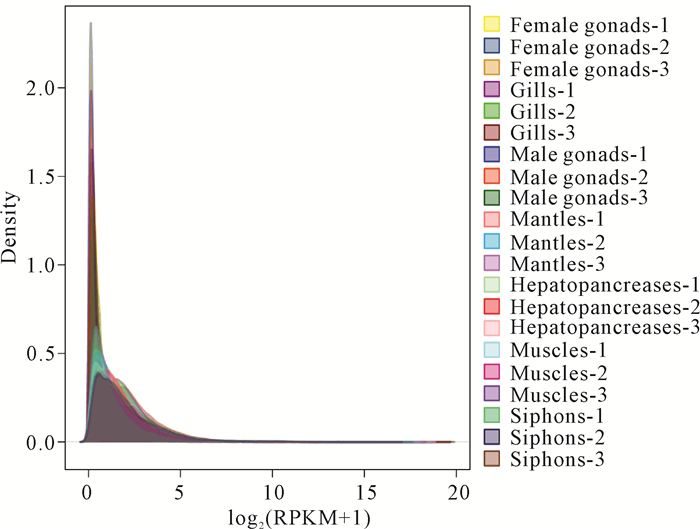
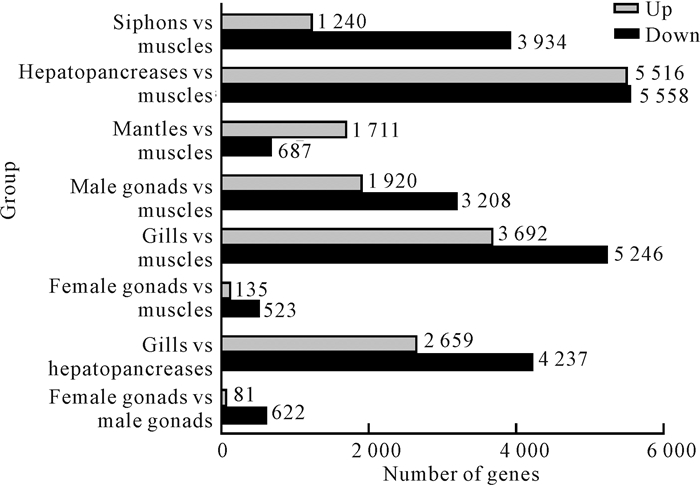
RPKM能够消除基因长度和测序数据分析中的基因表达水平估算产生的影响。基于RPKM估算基因转录本丰度,7种组织中基因表达数量由多到少依次为肝胰腺>鳃>外套膜>雄性性腺>肌肉>虹吸管>雌性性腺(图 6)。在此基础之上,对雌性性腺与雄性性腺、鳃与肝胰腺及各组织与肌肉组织之间的表达基因进行差异比对,肝胰腺vs肌肉组织获得差异基因数量最多,有11 074条,其次是鳃vs肌肉组织有8 938条,鳃vs肝胰腺有6 896条DEGs,虹吸管vs肌肉组织有5 174条DEGs,雄性性腺vs肌肉组织有5 128条DEGs,外套膜vs肌肉组织有2 398条DEGs,雌性性腺vs雄性性腺有703条DEGs,雌性性腺vs肌肉组织有658条DEGs,其各自上调与下调的差异基因数量如图 7所示。
|
| 图 6 不同样品表达水平密度曲线 Fig.6 Expression level density curves of different samples |
|
| 图 7 组织间差异基因数量 Fig.7 Number of DEGs between tissues |
2.2.2 组织间差异基因的GO富集分析
对差异基因进行GO注释,各组织间DEGs的GO条目富集在生物过程、细胞成分和分子功能3大类上的数量分布情况如表 3所示。
| 组别 Group |
GO富集数量 GO enrich number |
生物过程富集数量 Biological process enrich number |
细胞成分富集数量 Cellular component enrich number |
分子功能富集数量 Molecular function enrich number |
| Female gonads vs male gonads | 150 | 98 | 36 | 16 |
| Gills vs hepatopancreases | 1 502 | 1 159 | 111 | 232 |
| Female gonads vs muscles | 270 | 184 | 70 | 16 |
| Gills vs muscles | 1 368 | 931 | 235 | 202 |
| Male gonads vs muscles | 846 | 562 | 178 | 106 |
| Mantles vs muscles | 487 | 363 | 57 | 67 |
| Hepatopancreases vs muscles | 1 143 | 812 | 179 | 152 |
| Siphons vs muscles | 1 195 | 818 | 211 | 166 |
雌性性腺vs雄性性腺的DEGs富集程度较显著的GO条目主要包括纤毛移动(Cilium movement)、轴动蛋白复合体组装(Axonemal dynein complex assembly)、基因丝装配(Axoneme assembly)、9+2运动纤毛(9+2 motile cilium)和基因丝(Axoneme)。鳃vs肝胰腺的DEGs富集程度较显著的GO条目主要为甘露糖结合(Mannose binding)、磺基转移酶活性(Sulfotransferase activity)、角质细胞增殖的调控(Regulation of keratinocyte proliferation)和碳水化合物结合(Carbohydrate binding)。雌性性腺vs肌肉组织的DEGs富集程度较显著的GO条目主要为内质网靶向蛋白(Protein targeting to ER)、内质网蛋白定位的建立(Establishment of protein localization to endoplasmic retic)及游离胞浆核糖体(Cytosolic ribosome)。鳃vs肌肉组织的DEGs富集程度显著的GO条目为共质体(Symplast)和盐胁迫响应(Response to salt stress)。雄性性腺vs肌肉组织DEGs富集的GO条目主要有多聚核糖体(Polysomal ribosome)、SRP依赖协同翻译蛋白靶向膜(SRP-dependent cotranslational protein targeting to membrane)、色素体(Plastid)及协同翻译蛋白靶向膜(Cotranslational protein targeting to membrane)。外套膜vs肌肉组织的DEGs富集程度显著的GO条目主要有凋亡细胞的识别(Recognition of apoptotic cell)、伪足膜(Pseudopodium membrane)和吞噬作用、识别(Phagocytosis, recognition)。肝胰腺vs肌肉组织的DEGs富集程度较显著的GO条目包括共质体、色素体和外封结构(External encapsulating structure)。虹吸管vs肌肉组织的DEGs富集程度较显著的GO条目有质体间质(Plastid stroma)和多聚核糖体。
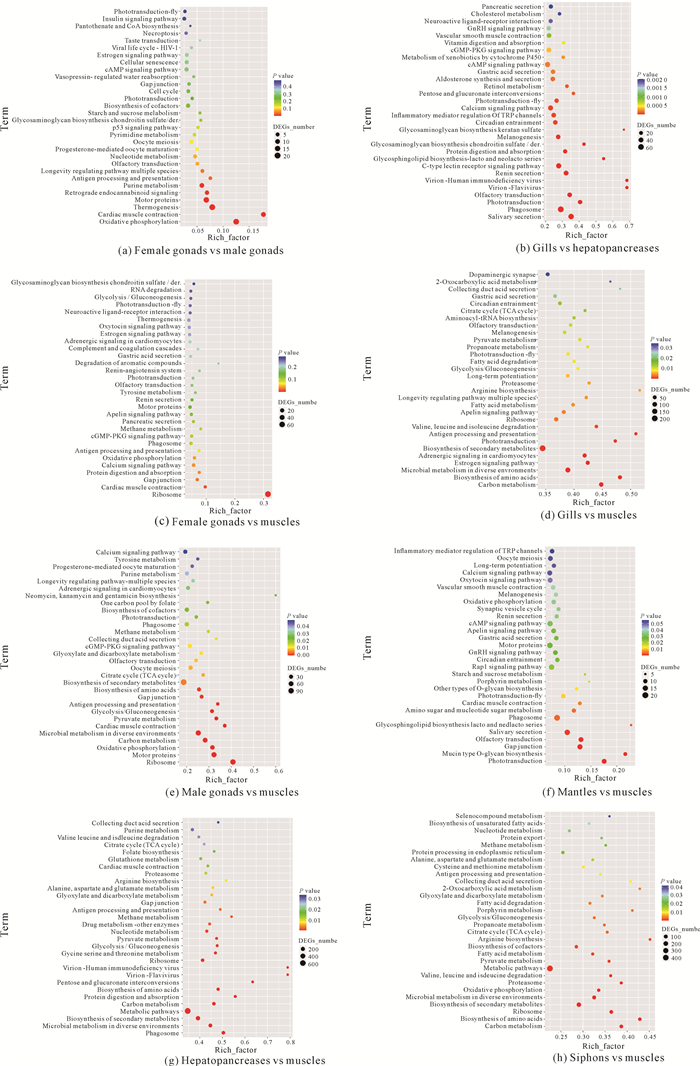
2.2.3 组织间差异基因的KEGG富集分析对弓獭蛤各组织转录组的DEGs经过KEGG通路注释分析显示(图 8),雌性性腺vs肌肉组织差异基因所注释的显著富集(P<0.05)的通路仅6条,富集的DEGs最多的通路是核糖体(Ribosome),其次是钙信号通路(Calcium signaling pathway)。此外,在心肌收缩(Cardiac muscle contraction)、间隙连接(Gap junction)、蛋白质的消化和吸收(Protein digestion and absorption)、氧化磷酸化通路(Oxidative phosphorylation)均有差异基因显著富集。雄性性腺vs肌肉组织差异基因所注释的显著富集(P<0.05)的通路有30条,其中富集DEGs较多的通路主要为次生代谢物的生物合成(Biosynthesis of secondary metabolites)、核糖体(Ribosome)、不同环境下微生物的代谢(Microbial metabolism in diverse environments)及马达蛋白(Motor proteins)等通路,核糖体、马达蛋白、氧化磷酸化和碳代谢等通路的富集程度较高。雌性性腺vs雄性性腺富集通路中,显著富集(P<0.05)的通路只有8条,富集DEGs数量最多的通路是生热作用(Thermogenesis),其次是与能量代谢相关的氧化磷酸化通路。其中逆行内源性大麻素信号通路(Retrograde endocannabinoid signaling)仅在雌雄性腺间的差异基因中存在显著富集,因此该通路可能参与了弓獭蛤性腺的分化过程。
|
| 图 8 差异表达基因KEGG富集通路气泡图 Fig.8 KEGG enrichment pathway bubble maps of DEGs |
作为参与运动的肌肉类组织,外套膜、虹吸管与肌肉组织间的差异基因主要在生物活动、生物合成及代谢相关的通路存在显著富集。外套膜vs肌肉组织差异基因所注释的显著富集(P<0.05)的通路有26条,各通路富集的DEGs数量较均匀,其中富集程度最强的有光传导(Phototransduction)、粘蛋白O型-聚糖生物合成(Mucin type O-glycan biosynthesis)、间隙连接、嗅觉转导(Olfactory transduction)及唾液分泌(Salivary secretion)等通路。虹吸管vs肌肉组织差异基因所注释的显著富集(P<0.05)的通路有33条,其中富集DEGs最多的主要有代谢通路(Metabolic pathways)、次生代谢物的生物合成和不同环境下微生物的代谢。富集程度最高的通路主要为碳代谢(Carbon metabolism)、氨基酸的生物合成(Biosynthesis of amino acids)及核糖体。
鳃vs肌肉组织差异基因所注释的显著富集(P<0.05)的通路有40条,其中富集DEGs最多的通路主要有次生代谢物的生物合成、不同环境下微生物的代谢、碳代谢等通路。肝胰腺vs肌肉组织差异基因所注释的显著富集(P<0.05)的通路有35条,其中富集DEGs最多且富集程度强的通路主要为代谢通路、次生代谢物的生物合成、不同环境下微生物的代谢和吞噬体(Phagosome)。鳃vs肝胰腺差异基因所注释的显著富集(P<0.05)的通路有63条,其中富集DEGs最多的通路主要是代谢通路,其次是吞噬体,在唾液分泌、吞噬体及光传导等通路的富集程度较其他通路更显著。根据差异基因富集的通路分析,弓獭蛤的鳃和肝胰腺主要负责机体的消化吸收、免疫应答等重要生理活动。
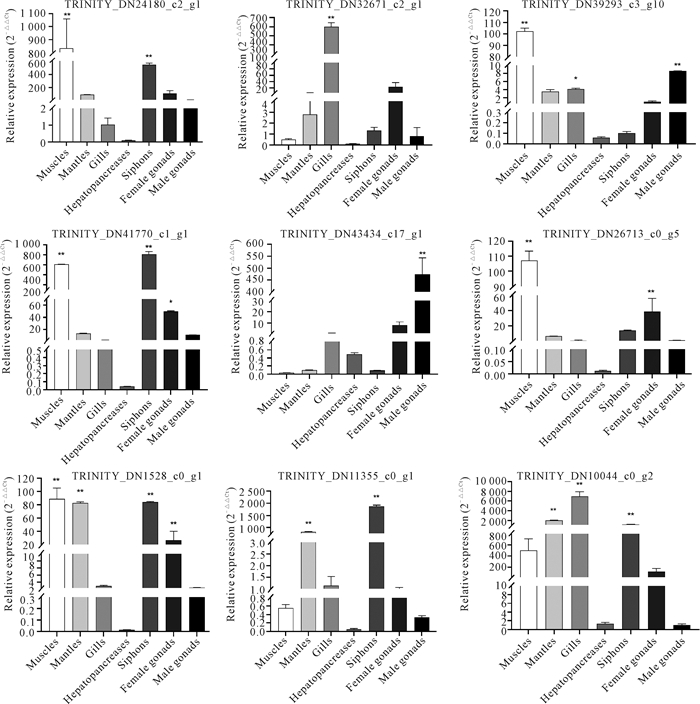
2.2.4 实时荧光定量PCR验证为验证所送组织转录组测序结果的可靠性,从获得的全部DEGs中随机挑选了9个进行实时荧光定量PCR验证,通过2-ΔΔCt计算出相对表达量,结果显示TRINITY_DN32671_c2_g1仅在鳃中具有极显著表达,TRINITY_DN24180_c2_g1、TRINITY_DN39293_c3_g10、TRINITY_DN41770_c1_g1等基因在弓獭蛤肌肉组织中具有极显著表达(图 9),与测序结果(表 4)基本趋于一致,表明弓獭蛤各组织转录组测序结果可信。
|
| *indicates that the tissue relative expression was significantly different from that of the control group (P < 0.05);** indicates that the tissue relative expression have extremely significant difference with the control group (P < 0.01). 图 9 9个随机挑选的差异基因在7种组织中的相对表达量的实时荧光定量PCR验证(内参为β-actin) Fig.9 Relative expression levels of selected nine differential genes randomly in seven tissues verified by real-time fluorescence quantitative PCR (normalized to β-actin) |
| 差异基因 DEGs |
肌肉 Muscles |
外套膜 Mantles |
鳃 Gills |
肝胰腺 Hepatopan- creases |
虹吸管 Siphons |
雌性性腺 Female gonads |
雄性性腺 Male gonads |
| TRINITY_ DN24180_c2_g1 |
6 560.19± 2 838.42 |
837.23± 226.67 |
62.30± 17.55 |
6.78± 0.69 |
5 588.52± 3 136.44 |
13.59± 10.64 |
5.95± 5.10 |
| TRINITY_ DN32671_c2_g1 |
0.02± 0.03 |
0.47± 0.64 |
212.81± 43.50 |
0.02± 0.03 |
0.06± 0.09 |
0.10± 0.07 |
0.33± 0.37 |
| TRINITY_ DN39293_c3_g10 |
1 065.38± 524.85 |
49.23± 27.13 |
193.32± 30.73 |
286.46± 139.35 |
4.00± 3.63 |
2.38± 1.69 |
12.68± 10.45 |
| TRINITY_ DN41770_c1_g1 |
13 918.97± 4 153.05 |
1 883.14± 872.16 |
133.45± 18.98 |
19.24± 5.87 |
31 367.27± 8 587.22 |
35.22± 28.44 |
22.71± 23.53 |
| TRINITY_ DN43434_c17_g1 |
127.87± 20.63 |
28 251.31± 39 790.45 |
759.15± 787.38 |
90 753.38± 111 305.82 |
11 379.46± 15 953.93 |
270.58± 367.23 |
749 182.67± 18 057.58 |
| TRINITY_ DN26713_c0_g5 |
1 236.06± 532.67 |
75.63± 53.45 |
0.69±0.30 | 0.48±0.14 | 71.81±24.66 | 2.77±2.82 | 1.94±1.87 |
| TRINITY_ DN1528_c0_g1 |
15.12± 7.29 |
2.50±2.01 | 0.26± 0.37 |
0.00±0.00 | 3.86±0.51 | 0.00±0.00 | 0.00±0.00 |
| TRINITY_ DN11355_c0_g1 |
1.30±0.68 | 353.29± 33.60 |
4.60±3.18 | 0.46±0.44 | 7 776.53± 4 838.19 |
0.14±0.20 | 0.00±0.00 |
| TRINITY_ DN10044_c0_g2 |
189.32± 26.18 |
194.49± 75.46 |
471.82± 70.20 |
1.20±0.40 | 109.29± 42.27 |
0.77±0.57 | 0.75±0.54 |
3 讨论 3.1 转录组技术在双壳贝类中的应用
多组学技术和分子生物学技术已经广泛应用于水产品的育种、繁殖、病害鉴定、杂交种鉴定等方面。在我国贝类养殖中具有传统优势的物种,如牡蛎、扇贝等,均已构建有丰富的全基因组、转录组、蛋白组、小RNA组及表观遗传组资源库。对经济贝类进行多组学资源库的构建和研究对推动养殖业的优良种选育和产业的可持续发展有着重要的作用。
转录组可揭示特定组织在特定状态下的基因表达情况,多应用于组织间差异、环境胁迫或毒理条件下的差异表达和功能基因筛选。有研究者对虾夷扇贝(Mizuhopecten yessoensis)的外套膜进行转录组测序,获得16 816个Unigenes[23];对海湾扇贝(Argopecten irradians)发育阶段个体进行转录测序,获得70 929个Unigenes[24];从牛角江珧(Atrina pectinata)的外套膜、鳃、性腺、后闭壳肌和肝胰腺5个组织中提取RNA,转录测序获得127 263个Unigenes[25]。
弓獭蛤作为我国南方重要的养殖经济贝类,是北海市的地理标志农产品,其多组学研究还比较匮乏。本研究利用RNA-seq技术对成体弓獭蛤的7种组织进行转录组测序,旨在丰富弓獭蛤的基因数据库资源。虽然NCBI上已有越南学者上传的基于肌肉组织测序的弓獭蛤基因组,但因獭蛤属的几个种形态学极为相似较难区分,因此本文对测序数据采用无参考转录组从头拼接的方法进行组装。对7种不同组织的转录组进行测序分析,比研究单一组织或较少组织在胁迫条件下的基因变化获得的数据更加全面和可信,也为后期进一步开展弓獭蛤遗传、生理、养殖等研究建立数据库资源。
本研究通过RNA-seq技术共获得939 197 518条clean reads。组装及ORF查找后获得了56 773个Unigenes,N50为1 373,平均长度1 033.12 bp,将其放入5个公共数据库中进行对比,有24 593条基因得到注释信息。通过各组织间差异基因的比较发现,肝胰腺vs肌肉组织和鳃vs肌肉组织的差异基因数量较多。
3.2 KEGG通路雌性性腺vs肌肉组织富集的胰岛素信号通路(Insulin signaling pathway)和卵母细胞成熟(Oocyte meiosis)均与弓獭蛤性腺的发育相关,这两条通路在纵肋织纹螺(Nassarius variciferus)[26]和钝缀锦蛤(Tapes conspersus) [27]中也被提出与性腺发育有关。鳃vs肝胰腺和鳃vs肌肉组织之间在唾液分泌、胃酸分泌通路、碳水化合物、蛋白质的消化吸收等通路有显著富集,这表明鳃是弓獭蛤的消化器官之一。肝胰腺vs鳃及肝胰腺vs肌肉组织间存在吞噬体、病毒体-黄病毒、TRP通道的炎症介质调节和药物代谢-其他酶等通路的显著富集,可证明肝胰腺是负责抵抗疾病、免疫和解毒的关键组织,且经对比在肝胰腺中存在甘氨酸、丝氨酸、苏氨酸、丙氨酸、天冬氨酸和谷氨酸等多种必需氨基酸的代谢通路。外套膜与肌肉组织存在光传导通路的富集,因此推测外套膜会对外界光照强度刺激产生应答,此外,贝类外套膜中表达的基因参与色素沉着、金属转录和免疫[28],在本研究中,参与黑色素生成的关键限速酶的酪氨酸酶(Tyrosinase)[29, 30]在外套膜中显著高表达。
3.3 功能注释在不同物种的同一组织、同一物种的不同组织之间的转录组表达往往存在相似之处也存在差异,本次对所获得的基因和两两组织间的DEGs进行GO功能注释,结果表明均在细胞过程(Cellular process)、生物调节(Biological regulation)、细胞结构体(Cellular anatomical entity)、结合(Binding)等功能上有较多基因富集,这与泥蚶[7]、钝缀锦蛤[27]和缢蛏[31]等贝类具有相似性。本研究弓獭蛤注释率为43.32%,与大部分双壳贝类注释率相当,但与其他研究较深的常见经济动物相比,注释率较低,这表明在贝类中仍有许多基因或序列的功能等待发掘。
双壳贝类对重金属镉暴露有显著的物种、时间和剂量效应的差异[32],本研究中,参与贝类重金属镉胁迫响应的抗氧化酶系的相关基因如谷胱甘肽过氧化物酶(Glutathione peroxidase)、硫氧还蛋白(Thioredoxin)、超氧化物歧化酶(Superoxide dismutase)、金属硫蛋白(Metallothionein)、谷胱甘肽硫转移酶(Glutathiones S-transferase)、过氧化氢酶(Catalase)、谷氧还蛋白(Glutaredoxin)等基因在鳃、肝胰腺、外套膜和虹吸管有较高表达量。贝类免疫主要以体液免疫和血细胞免疫的方式进行[33],在本研究的结果中,参与体液免疫的溶酶体(Biogenesis of lysosome-related organelles complex)、toll样受体(toll-like receptor)、髓样分化因子(Myeloid differentiation primary response 88)、同种异体移植炎症因子(Allograft inflammatory factor)等相关基因以及凝集素(Lectin)等多集中于鳃、肝胰腺和外套膜表达,而血细胞免疫相关的基因如巨噬细胞相关基因和受体等多集中分布于鳃和外套膜中。
通过对转录组数据的分析,本研究在弓獭蛤的雌雄性腺、肌肉组织、鳃和肝胰腺中分别得到了大量与弓獭蛤性状、生长发育、生理代谢及免疫调节相关的通路及功能基因,但这些通路和基因在弓獭蛤各组织内具体的表达和调控机制仍待进一步研究。
4 结论多组学研究在经济贝类的遗传育种等方面发挥着重要作用。本研究基于第二代高通量RNA-seq测序技术获得我国南方一种重要的养殖贝类弓獭蛤的转录组序列数据﹐通过组装拼接得到了56 773条预测基因。利用BLAST软件比对NR、GO、eggNOG、KEGG和SWISS-Prot数据库﹐进行了功能基因注释﹐共有24 593条基因得到注释。通过组织间的两两对比获得了雌性性腺vs雄性性腺(703条)、鳃vs肝胰腺(6 896条)、雌性性腺vs肌肉(658条)、鳃vs肌肉(8 938条)、雄性性腺vs肌肉(5 128条)、外套膜vs肌肉(2 398条)、肝胰腺vs肌肉(11 074条)和虹吸管vs肌肉组织(5 174条)的差异表达基因。对各组之间DEGs进行GO注释和KEGG富集分析,讨论了各组之间可能存在的功能差异。本研究为开展弓獭蛤分子遗传学研究提供了基因资源,也为进一步开展弓獭蛤功能基因挖掘、遗传育种研究和分子标记的开发提供了基础参照数据。
| [1] |
LI Y L, SUN X Q, HU X, et al. Scallop genome reveals molecular adaptations to semi-sessile life and neurotoxins[J]. Nature Communications, 2017, 8(1): 1721. DOI:10.1038/s41467-017-01927-0 |
| [2] |
WU B, CHEN X, YU M J, et al. Chromosome-level genome and population genomic analysis provide insights into the evolution and environmental adaptation of Jinjiang oyster Crassostrea ariakensis[J]. Molecular Ecology Resources, 2022, 22(4): 1529-1544. DOI:10.1111/1755-0998.13556 |
| [3] |
SONG H, GUO X M, SUN L N, et al. The hard clam genome reveals massive expansion and diversification of inhibitors of apoptosis in Bivalvia[J]. BMC Biology, 2021, 19(1): 15. DOI:10.1186/s12915-020-00943-9 |
| [4] |
RAN Z S, LI Z Z, YAN X J, et al. Chromosome-level genome assembly of the razor clam Sinonovacula constricta (Lamarck, 1818)[J]. Molecular Ecology Resources, 2019, 19(6): 1647-1658. DOI:10.1111/1755-0998.13086 |
| [5] |
WANG Q H, LIU Y, ZHENG Z, et al. Adaptive respon- se of pearl oyster Pinctada fucata martensii to low water temperature stress[J]. Fish & Shellfish Immunology, 2018, 78(7): 310-315. |
| [6] |
孔祥辉, 王莎莎, 董迎辉, 等. 缢蛏急性高温胁迫应答主要候选基因的表达特征分析[J]. 渔业科学进展, 2022, 43(2): 194-203. |
| [7] |
张阳, 金铭, 刘宏星, 等. 泥蚶低氧胁迫后血细胞转录组及耐低氧相关基因的筛选与分析[J]. 海洋科学, 2023, 47(1): 34-44. |
| [8] |
MOREIRA R, BALSEIRO P, PLANAS J V, et al. Transcriptomics of in vitro immune-stimulated hemocytes from the Manila clam Ruditapes philippinarum using high-throughput sequencing[J]. PLoS One, 2012, 7(4): e35009. DOI:10.1371/journal.pone.0035009 |
| [9] |
ZHANG J J, LI H J, QIN Y J, et al. Identification of fu- nctional genes involved in Cd2+ response of Chinese surf clam (Mactra chinensis) through transcriptome sequencing[J]. Environmental Toxicology and Pharmacology, 2016, 41: 113-120. DOI:10.1016/j.etap.2015.11.006 |
| [10] |
LI H L, LIU J G, HUANG X T, et al. Characterization, expression and function analysis of DAX1 gene of scallop (Chlamys farreri Jones and Preston 1904) during its gametogenesis[J]. Journal of Ocean University of China, 2014, 13(4): 696-704. DOI:10.1007/s11802-014-2299-9 |
| [11] |
MA Y W, YE Y Y, YAO R H, et al. Transcriptome sequencing analysis of sex-related genes in the gonads of Mytilus unguiculatus[J]. Fishes, 2023, 8(9): 456. DOI:10.3390/fishes8090456 |
| [12] |
ZHANG J B, XIONG X W, DENG Y W, et al. Integrated application of transcriptomics and metabolomics provides insights into the larval metamorphosis of pearl oyster (Pinctada fucata martensii)[J]. Aquaculture, 2021, 532(1): 736067. |
| [13] |
徐凤山, 张素萍. 中国海产双壳类图志[M]. 北京: 科学出版社, 2008.
|
| [14] |
李琪. 中国近海软体动物图志[M]. 北京: 科学出版社, 2019.
|
| [15] |
陆威. "北海象鼻螺"申报农产品地理标志[J]. 农家之友, 2021(1): 37. |
| [16] |
THAI B T, LEE Y P, GAN H M, et al. Whole genome assembly of the snout otter clam, Lutraria rhynchaena, using Nanopore and Illumina Data, benchmarked against bivalve genome assemblies[J]. Frontiers in Genetics, 2019, 10: 1158. DOI:10.3389/fgene.2019.01158 |
| [17] |
THAI B T, TAN M H, LEE Y P, et al. Characterisation of 12 microsatellite loci in the Vietnamese commercial clam Lutraria rhynchaena Jonas 1844 (Heterodonta: Bivalvia: Mactridae) through next-generation sequencing[J]. Molecular Biology Reports, 2016, 43(5): 391-396. DOI:10.1007/s11033-016-3966-2 |
| [18] |
GAN H M, TAN M H, THAI B T, et al. The complete mitogenome of the marine bivalve Lutraria rhynchaena Jonas 1844 (Heterodonta: Bivalvia: Mactridae)[J]. Mitochondrial DNA Part A, DNA Mapping, Sequencing, and Analysis, 2016, 27(1): 335-336. |
| [19] |
HAO D M, SY D T, TUYET D T A, et al. Distribution and density of Lutraria rhynchaena Jonas, 1844 relate to sediment while reproduction shows multiple peaks per year in Cat Ba-Ha Long Bay, Vietnam[J]. Open Life Sciences, 2020, 15(1): 721-734. DOI:10.1515/biol-2020-0072 |
| [20] |
邹杰, 杨家林. 弓獭蛤健康苗种繁育技术[J]. 科学养鱼, 2013(1): 43-44. |
| [21] |
郑淑雅, 饶小珍, 陈昭娜. 弓獭蛤受精及早期卵裂过程核相变化的细胞学观察[J]. 福建师范大学学报(自然科学版), 2014, 30(1): 98-103. |
| [22] |
何智能, 巫冷蝉, 王宏玲, 等. 弓獭蛤(Lutraria arcuata) 的重金属富集特征及其对镉胁迫的生理响应[J]. 广西科学院学报, 2023, 39(2): 169-178. |
| [23] |
于佐安, 李大成, 李华琳, 等. 感染"褐色沉积症"虾夷扇贝的外套膜转录组测序及差异表达基因分析[J]. 中国水产科学, 2023, 30(3): 284-296. |
| [24] |
TENG W, CONG R H, QUE H Y, et al. De novo transcriptome sequencing reveals candidate genes involved in orange shell coloration of bay scallop Argopecten irradians[J]. Journal of Oceanology and Limnology, 2018, 36(4): 1408-1416. DOI:10.1007/s00343-018-7063-3 |
| [25] |
SUN X J, LI D M, LIU Z H, et al. De novo assembly of pen shell (Atrina pectinata) transcriptome and screening of its genic microsatellites[J]. Journal of Ocean University of China, 2017, 16(5): 882-888. DOI:10.1007/s11802-017-3274-z |
| [26] |
梁书东. 纵肋织纹螺(Nassarius variciferus) 性腺发育相关基因的筛选及胚胎发育研究[D]. 沈阳: 沈阳农业大学, 2021.
|
| [27] |
连昌朋. 钝缀锦蛤性腺发育和生殖周期、转录组学研究及形态性状对活体质量的影响分析[D]. 南宁: 广西大学, 2023.
|
| [28] |
DING J, ZHAO L, CHANG Y, et al. Transcriptome sequencing and characterization of Japanese scallop Patinopecten yessoensis from different shell color lines[J]. PLoS One, 2015, 10(2). |
| [29] |
CHANG T S. Natural melanogenesis inhibitors acting through the down-regulation of tyrosinase activity[J]. Materials (Basel), 2012, 5(9): 1661-1685. DOI:10.3390/ma5091661 |
| [30] |
MAO J, ZHANG X, ZHANG W, et al. Genome-wide identification, characterization and expression analysis of the MITF gene in Yesso scallops (Patinopecten yessoensis) with different shell colors[J]. Gene, 2019, 688: 155-162. DOI:10.1016/j.gene.2018.11.096 |
| [31] |
CAO W, DONG Y H, GENG Y S, et al. Comprehensive analysis of whole-transcriptome profiles in response to acute hypersaline challenge in Chinese razor clam Sinonovacula constricta[J]. Biology, 2023, 12(1): 106. DOI:10.3390/biology12010106 |
| [32] |
ZHAN J F, SUN T, WANG X H, et al. Meta-analysis reveals the species-, dose- and duration-dependent effects of cadmium toxicities in marine bivalves[J]. Science of the Total Environment, 2023, 859(Pt 2): 160164. |
| [33] |
ALLAM B, RAFTOS D. Immune responses to infec- tious diseases in bivalves[J]. Journal of Invertebrate Pathology, 2015, 131(12): 121-136. |



