2. 海南热带海洋学院, 热带海洋生物资源利用与保护教育部重点实验室, 海南三亚 572022;
3. 承德医学院蚕业研究所, 河北承德 067000;
4. 河北石油职业技术大学, 河北承德 067000;
5. 唐山海都水产食品有限公司, 河北唐山 063000;
6. 河北省海水鱼产业技术研究院, 河北唐山 063000;
7. 承德医学院, 河北医工结合国际联合研究中心, 河北承德 067000;
8. 锦州奥鸿药业有限责任公司, 辽宁锦州 121013
2. Key Laboratory of Utilization and Conservation for Tropical Marine Bioresources, Ministry of Education, Hainan Tropical Ocean University, Sanya, Hainan, 572022, China;
3. Institute of Sericulture, Chengde Medical University, Chengde, Hebei, 067000, China;
4. Hebei Petroleum University of Technology, Chengde, Hebei, 067000, China;
5. Tangshan Haidu Aquatic Food Co., Ltd., Tangshan, Hebei, 063000, China;
6. Research Institute of Hebei Marine Fish Industry Technology, Tangshan, Hebei, 063000, China;
7. Hebei International Research Center of Medical Engineering, Chengde Medical University, Chengde, Hebei, 067000, China;
8. Jinzhou Aohong Pharmaceutical Co., Ltd., Jinzhou, Liaoning, 121013, China
遗传多样性指的是生物群体内部及不同群体之间的可遗传变异的总和,是生物多样性的核心内容[1-4],可以反映物种或种群的遗传变异水平、生存能力、进化潜力以及适应能力。物种或种群的遗传多样性越丰富,也就意味着其包含的遗传变异越多,生存能力、进化能力以及适应能力也就越强[5]。此外,遗传多样性还是研究物种或种群遗传结构的核心,而分析物种或种群的遗传结构对其开发利用、育种改良以及保护管理均具有重要的理论意义[6-10]。
卤虫(Artemia saline)又称盐水丰年虾、丰年虫,属于节肢动物门(Arthropoda)甲壳纲(Crustasea)鳃亚足纲(Branchiopoda)无甲目(Anostraca)卤虫科(Artemiidae)卤虫属(Artemia)的一种小型甲壳动物[11]。卤虫含有丰富的营养物质,是水产经济动物苗种生产中极佳的鲜活饵料,在养殖名贵观赏水族动物、河蟹育苗、海水仔稚鱼培育中均有广泛的应用[12-14]。此外,卤虫还具有繁殖速度快、适应性强及生活周期短等特点,是基础理论研究中良好的实验动物[15]。
卤虫在世界各地(南北极地区除外)的很多高盐水体均有分布,分布地多达55个国家350多个地区[16]。我国卤虫资源丰富,分布在沿海省份的盐田以及西藏、新疆、青海、内蒙古、河北、山西等地的盐湖中。渤海湾地区是中国沿海地区卤虫及其休眠卵的主要产地,包括河北省的唐山市、沧州市,辽宁省的营口市,天津市的汉沽区、塘沽区以及山东省的滨州市、潍坊市、东营市等[17]。其中,河北省沧州市和山东省滨州市不仅出产卤虫原料,而且已经发展成为卤虫卵的主要集散地,每年均有来自全国各地的大量卤虫原料经过这两个地方加工后流入市场。
为满足我国水产养殖业对卤虫的需求,1991年旧金山卤虫(Artemia franciscana)被引入中国渤海湾,随着旧金山卤虫的引入,渤海湾地区的卤虫产量急剧增加[18]。历经三十多年的引种和定殖,旧金山卤虫在中国境内已有广泛的分布,但渤海湾地区旧金山卤虫种群的遗传多样性及种群遗传结构却鲜有报道[19],仅发现有关河北唐山地区的旧金山卤虫种群遗传学研究[20]。由于研究样本严重缺乏,在旧金山卤虫引入渤海湾地区后,我国对其遗传结构的现状及变化趋势知之甚少,这也对旧金山卤虫资源的保护带来了威胁。
基于此,本研究通过采集河北省沧州市黄骅市的卤虫种群样本,利用COⅠ基因作为分子标记,分析该种群的遗传多样性,并将其与旧金山卤虫大盐湖种群和旧金山湾种群进行比较,拟探讨黄骅市旧金山卤虫种群的遗传结构在新栖息地的适应性变化,为更合理、有效地保护旧金山卤虫资源提供重要的数据支持,并为旧金山卤虫引入至我国其他地区的可行性提供了重要的理论基础。
1 材料与方法 1.1 样品采集与DNA提取2020年9月于河北省沧州市黄骅市南排河镇辛立灶村(38°22′ N,117°40′ E)采集33个旧金山卤虫卵样品。采集到的卤虫卵使用Chelex-100 (Bio-Rad, USA)试剂盒提取基因组DNA,提取方法参考试剂盒说明书[21, 22]。
1.2 COⅠ基因序列扩增及测序使用COⅠ基因通用引物1/2COⅠ_Fol-F (5′-ATTCTACGAATCACAAGGATATTGG-3′)和1/2COⅠ_Fol-R (5′-TACACTTCAGGATGGCCAAAAAATCA-3′)作为引物[23],对样本基因组DNA的COⅠ基因序列进行扩增。PCR扩增采用20 μL体系,包括2×EasyTaq®PCR SuperMix 10 μL,DNA模板1 μL,正反向引物各0.5 μL,ddH2O 8 μL。PCR扩增程序:预变性95 ℃ 5 min;变性94 ℃ 45 s, 退火55 ℃ 60 s, 延伸72 ℃ 60 s,35个循环;末延伸72 ℃ 10 min,4 ℃保存。扩增产物的大小和纯度用1.5%的琼脂糖凝胶电泳检测,得到目的长度片段的PCR扩增产物送至北京擎科生物科技股份有限公司进行测序。
1.3 数据分析将测序结果与美国国立生物技术信息中心(NCBI)BLAST数据库(https://blast.ncbi.nlm.nih.gov/Blast.cgi)内储存的卤虫COⅠ基因片段序列数据进行比对,利用Mega7[24]进行DNA信息位点分析,包括碱基组成、多态位点、变异位点、核苷酸多态性。旧金山卤虫大盐湖种群(以下简称“大盐湖种群”,GSL)与旧金山卤虫旧金山湾种群(以下简称“旧金山湾种群”,SFB)的线粒体基因组COⅠ片段序列数据来源于2013年的NCBI数据库(https://www.ncbi.nlm.nih.gov/nucleotide/,表 1)。使用Network program v5.0进行单倍型网络分析,估算旧金山卤虫黄骅种群(以下简称“黄骅种群”,HU)与大盐湖种群和旧金山湾种群的遗传分化。利用软件Arlequin v3.5[25]计算群体的单倍型数(h)、单倍型比(Hr)、单倍型多样性(Hd)、核苷酸(基因)多样性(Pi)、平均核苷酸差异数(k)、多态位点数(S)、群体遗传分化指数(Fst)并进行分子方差分析(AMOVA)。
| 种群 Population | 样本数量分布 Number of samples distribution | GenBank登录号 GenBank accession number |
| GSL (29 samples) |
1 | KF662960 |
| 2 | KF662968 | |
| 21 | KF662970 | |
| 2 | KF662971 | |
| 2 | KF662976 | |
| 1 | KF662977 | |
| SFB (37 samples) |
26 | KF662960 |
| 6 | KF662968 | |
| 4 | KF662970 | |
| 1 | KF662975 |
2 结果与分析 2.1 COⅠ基因序列分析
本研究共获取黄骅种群COⅠ基因序列33条,长度为709 bp,通过与NCBI核苷酸数据库内储存的卤虫COⅠ基因DNA片段序列进行比对,确定本研究获取的DNA片段均为旧金山卤虫。黄骅种群样本的COⅠ基因序列具有7个变异位点,其中简约信息位点1个,自裔位点6个;而大盐湖种群样本的COⅠ基因序列具有11个变异位点,其中简约信息位点7个,自裔位点4个;旧金山湾种群样本的COⅠ基因序列具有7个变异位点,其中简约信息位点6个,自裔位点1个。
2.2 遗传多样性分析由表 2可知,多态位点数和总变异位点数这两个指标的数值在3个种群中均相同,黄骅种群、大盐湖种群、旧金山湾种群数值分别为6、11、7,表现为黄骅种群 < 旧金山湾种群 < 大盐湖种群,其中黄骅种群与旧金山湾种群接近。黄骅种群的单倍型多样性为0.324,低于大盐湖(0.475)和旧金山湾种群(0.480)。黄骅种群的核苷酸多样性(0.000 87)明显低于大盐湖种群(0.002 78)和旧金山湾种群(0.003 20),黄骅种群的平均核苷酸差异数(0.523)也明显低于大盐湖种群(1.680)和旧金山湾种群(1.946),这些指标与单倍型多样性的结果表现的趋势一致。黄骅种群的中性检验指标Tajima′s D值为-1.802,并不显著。
| 种群 Population |
样本数量 Number of samples | 多态位点数 Number of polymorphic sites | 总变异位点数 Site number of total variation | 单倍型数 Number of haplotypes |
单倍型多样性 Haplotype diversity | 核苷酸多样性 Nucleotide diversity | 平均核苷酸差异数 Average number of nucleotide differences | Tajima′s D中性检验值 Tajima′s D neutral test value |
| HU | 33 | 6 | 6 | 4 | 0.324 | 0.000 87 | 0.523 | -1.802 |
| GSL | 29 | 11 | 11 | 6 | 0.475 | 0.002 78 | 1.680 | -1.297 |
| SFB | 37 | 7 | 7 | 4 | 0.480 | 0.003 20 | 1.946 | 0.451 |
与大盐湖种群和旧金山湾种群相比,黄骅种群中的单倍型多样性和核苷酸多样性数值相对较低,表明相较于美洲本土的旧金山卤虫种群,引入并定殖于中国河北省沧州市黄骅市南排河镇辛立灶村的旧金山卤虫种群的遗传多样性相对较低。
2.3 单倍型聚类分析基于COⅠ基因序列进行群体遗传结构分析,共有99个旧金山卤虫样本,3个种群共检测到9种单倍型(H1-H9),其中H1、H5、H6为主要的单倍型,分别占总数的35.35%(35个样本)、27.27%(27个样本)和25.25%(25个样本)(图 1,表 3)。由表 3可知,H1型分别由77.14%的黄骅种群、5.71%的大盐湖种群和17.14%的旧金山湾种群组成;H5型分别由3.70%的大盐湖种群和96.30%的旧金山湾种群组成;H6型分别由84.00%的大盐湖种群和16.00%的旧金山湾种群组成。
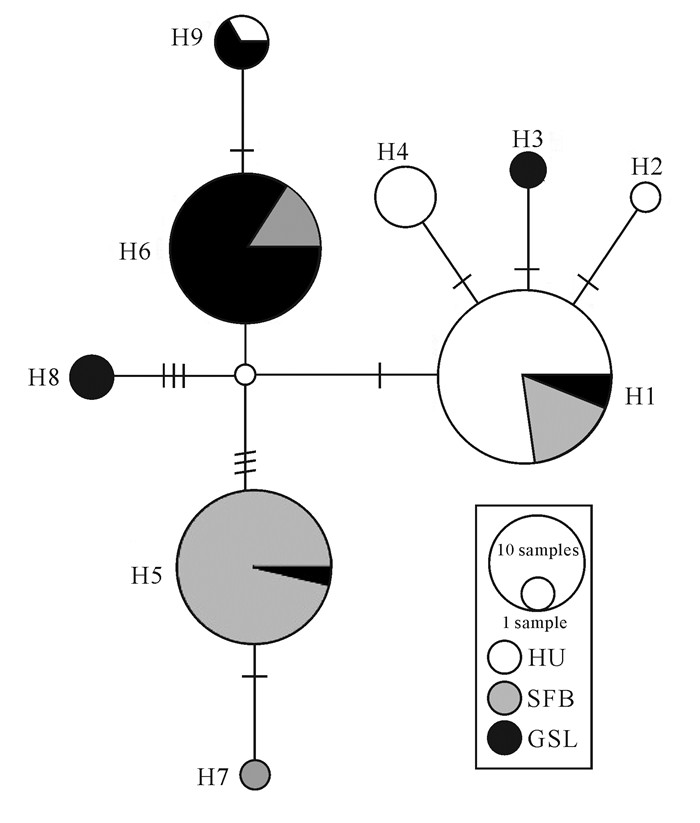
|
| 图 1 基于COⅠ基因序列构建的黄骅种群、大盐湖种群和旧金山湾种群的单倍型网络图 Fig.1 Haplotype networks of Huanghua population, the Great Salt Lake population, and the San Francisco Bay population constructed based on COⅠ gene sequences |
| 单倍型 Haplotype |
种群 Population |
样本数量 Number of samples |
比例/% Proportion/% |
| H1 | HU | 27 | 77.14 |
| GSL | 2 | 5.71 | |
| SFB | 6 | 17.14 | |
| H2 | HU | 1 | 100.00 |
| H3 | GSL | 1 | 100.00 |
| H4 | HU | 4 | 100.00 |
| H5 | GSL | 1 | 3.70 |
| SFB | 26 | 96.30 | |
| H6 | GSL | 21 | 84.00 |
| SFB | 4 | 16.00 | |
| H7 | SFB | 1 | 100.00 |
| H8 | GSL | 2 | 100.00 |
| H9 | HU | 1 | 33.33 |
| GSL | 2 | 66.67 | |
| Total | 99 |
黄骅种群分别分布在H1、H2、H4、H9型中,主要单倍型为H1型(81.82%),其次为H4型(12.12%),再次为H2型(3.03%)和H9型(3.03%)。大盐湖种群分别分布在H1、H3、H5、H6、H8、H9型中,主要单倍型为H6型(72.41%);其次为H1(6.90%)、H8(6.90%)、H9(6.90%)型;再次为H3(3.45%)、H5(3.45%)型。旧金山湾种群分别分布在H1、H5、H6、H7型中,主要单倍型为H5型(70.27%),其次为H1型(16.22%),再次为H6型(10.81%),最后为H7型(2.70%)(表 3)。各种群的单倍型分布模式表明,3个种群的主要单倍型类型并不相同,表明3个种群之间有明显的遗传分化。
2.4 群体遗传结构分析由表 4可知,3个种群两两之间均存在显著的遗传分化(P=0.000 1)。黄骅种群与大盐湖种群、旧金山湾种群间的遗传分化指数均为0.64,大盐湖种群与旧金山湾种群间的遗传分化指数为0.56。3个旧金山卤虫种群的分子方差分析结果(表 5)表明,在3个种群的总遗传变异中,有61.04%的遗传变异来源于3个种群间的遗传变异,而仅有38.96%的遗传变异来源于各种群内个体间的遗传变异,表明这3个旧金山卤虫种群间具有明显的遗传分化。
| 黄骅种群 HU |
大盐湖种群 GSL |
旧金山湾种群 SFB |
|
| HU | 0.00 | 0.000 1 | 0.000 1 |
| GSL | 0.64 | 0.00 | 0.000 1 |
| SFB | 0.64 | 0.56 | 0.00 |
| 变异来源 Source of variation | 自由度 Degree of freedom |
平方和 Sum of squares | 变异组成 Variance compon-ents | 变异百分比/% Variation percen-tage/% |
| Among populations | 2 | 73.112 | 1.091 99 | 61.04 |
| Within populations | 96 | 66.908 | 0.696 96 | 38.96 |
| Total | 98 | 140.020 | 1.788 95 | 100.00 |
3 讨论 3.1 黄骅种群群体遗传多样性
遗传多样性是生物种群进化的动力,也是种群适应环境能力的直接体现,因此被认为是影响种群生存和发展的前提[26, 27]。外来物种的遗传多样性和遗传结构受到奠基者效应、瓶颈效应、有性生殖比例等多种因素的影响,物种或种群的遗传多样性可以反映其在入侵和定殖过程中的一些特殊的历史事件[26]。因此,对物种或种群的遗传多样性及遗传结构进行研究,不仅可以分析物种或种群的生存能力、进化潜力以及适应能力,还可以用于分析外来物种的入侵机理和路径[26]。
本研究基于COⅠ基因序列对比探究了黄骅种群、大盐湖种群和旧金山湾种群的遗传多样性,在多态位点数、总变异位点数这两个指标上,黄骅种群与旧金山湾种群接近,均低于大盐湖种群;在核苷酸多样性、平均核苷酸差异数以及单倍型多样性等遗传变异指标中,黄骅种群均低于大盐湖种群和旧金山湾种群;在单倍型聚类分析中,3个类群的主要单倍型类型并不相同,表明3个种群之间有明显的遗传分化。本研究认为,相比于美洲本土的旧金山卤虫大盐湖种群和旧金山湾种群,引种并定殖于河北省沧州市黄骅市的旧金山卤虫的遗传多样性相对较低,这一结果与越南Vinh Chau地区定殖的旧金山卤虫的遗传多样性情况类似[28]。这可能是由于奠基者效应造成的,即黄骅种群仅由引入种群的少量个体繁衍而成;也可能是该种群在定殖后经历了瓶颈效应,导致种群大小在短时间内迅速下降,从而导致后代遗传多样性降低[29-31]。
3.2 黄骅种群种源现如今亚洲旧金山卤虫种群的种源不仅包括人类从旧金山卤虫的两个主要种源地大盐湖种群和旧金山湾种群的多次引入,还包括人类从中美洲以及欧洲种群的引入[31]。本研究发现,黄骅种群的主要单倍型是H1型,而大盐湖种群和旧金山湾种群的主要单倍型分别为H6型和H5型,即黄骅种群的主要单倍型与大盐湖种群、旧金山湾种群均存在差异,具有显著的遗传分化;并且这3个旧金山卤虫种群具有的总遗传变异主要来源于3个种群间的遗传变异,表明黄骅种群并非是从大盐湖种群和旧金山湾种群这两个旧金山卤虫主要种源地引种、繁衍而成,而可能是由旧金山卤虫的其他次生种源地(本土种源地引入定殖后再向外引种的地区)如中美洲或欧洲引种、繁衍而成。
4 结论本研究通过对河北省沧州市黄骅市南排河镇辛立灶村旧金山卤虫种群的COⅠ基因进行测序,并将测序结果与大盐湖种群、旧金山湾种群的COⅠ基因序列数据进行比对,从而分析比较这3个旧金山卤虫种群的遗传多样性与遗传结构的差异。本研究发现,相比于大盐湖种群和旧金山湾种群,黄骅种群的遗传多样性相对较低,推测可能是旧金山卤虫在黄骅引种、定殖过程中经历过奠基者效应和瓶颈效应;此外,黄骅种群与大盐湖种群、旧金山湾种群存在显著的遗传分化,表明黄骅种群并非直接从大盐湖种群和旧金山湾种群引种、繁衍而成。
由于旧金山卤虫的商品化由来已久,很多地区包括越南Vinh Chau地区、澳大利亚Port Hedland地区、伊朗Urmia湖以及渤海湾部分地区等都已大量生产卤虫及卤虫卵,这也使旧金山卤虫的栖息地的分布极易受到养殖生产等人为因素的干扰。因此推测黄骅种群可能是经过美洲本土种源地扩张之后形成的次生种源地的再次引种。因此,为进一步丰富黄骅种群的遗传多样性,可从大盐湖种群或旧金山湾种群再次引种,从而提高该种群的生存能力、进化潜力和适应能力,进而保证该种群的延续与发展。
致谢
特别感谢海南热带海洋学院生态环境学院王沛政教授为本研究提供实验设施及实验条件。
| [1] |
郝云庆, 罗晓波, 王晓玲. 濒危植物五小叶槭(Acer pentaphyllum Diels)天然种群遗传多样性的ISSR标记分析[J]. 四川大学学报(自然科学版), 2019, 56(1): 161-166. |
| [2] |
杨舒婷, 马晓娜, 白晓霖, 等. 极小种群野生植物巴郎山杓兰的CDDP遗传多样性分析[J]. 四川大学学报(自然科学版), 2022, 59(6): 155-163. |
| [3] |
ELLEGREN H, GALTIER N. Determinants of genetic diversity[J]. Nature Reviews Genetics, 2016, 17(7): 422-433. DOI:10.1038/nrg.2016.58 |
| [4] |
彭羽, 卿凤婷, 米凯, 等. 生物多样性不同层次尺度效应及其耦合关系研究进展[J]. 生态学报, 2015, 35(2): 577-583. |
| [5] |
许如意. 卤虫转录组EST-SSR标记开发及群体遗传多样性研究[D]. 天津: 天津科技大学, 2019.
|
| [6] |
HEDRICK P W. Recent developments in conservation genetics[J]. Forest Ecology and Management, 2004, 197: 3-19. DOI:10.1016/j.foreco.2004.05.002 |
| [7] |
杨梅, 张敏, 师守国, 等. 武当木兰种群遗传结构的ISSR分析[J]. 林业科学, 2014, 50(1): 76-81. |
| [8] |
NEI M. Estimation of average heterozygosity and genetic distance from a small number of individuals[J]. Genetics, 1978, 89(3): 583-590. DOI:10.1093/genetics/89.3.583 |
| [9] |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic-linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314-331. |
| [10] |
WUYUN T N, AMO H, XU J S, et al. Population str-ucture of and conservation strategies for wild Pyrus ussuriensis Maxim.in China[J]. PLoS One, 2015, 10(8): e0133686. DOI:10.1371/journal.pone.0133686 |
| [11] |
LAVENS P, SORGELOOS P. Manual for the culture and use of brine shrimp Artemia in aquaculture[M]. Chent, Belgium: State University of Chent, 1986: 319.
|
| [12] |
SORGELOOS P, DHERT P, CANDREVA P. Use of the brine shrimp, Artemia spp., in marine fish larviculture[J]. Aquaculture, 2001, 200(1/2): 147-159. |
| [13] |
苏秀榕, 李太武, 欧阳芬, 等. 卤虫卵营养价值的研究[J]. 营养学报, 1997(1): 121-123. |
| [14] |
AZRA M N, NOOR M I M, BURLAKOVS J, et al. Trends and new developments in Artemia research[J]. Animals, 2022, 12(18): 2321. DOI:10.3390/ani12182321 |
| [15] |
VANHAECKE P, TACKAERT W, SORGELOOS P. The biogeography of Artemia: an updated review[J]. Izwo Collected Reprints, 1987, 1: 129-155. |
| [16] |
何为. 1999-2000年度我国沿海卤虫产业调查与分析[J]. 上海水产大学学报, 2000(3): 264-267. |
| [17] |
曲若竹. 两性生殖卤虫的遗传多样性与进化[D]. 大连: 辽宁师范大学, 2003.
|
| [18] |
张波, 郭金昌. 旧金山湾卤虫(Artemia franciscana)在中国渤海湾地区的引种[J]. 海湖盐与化工, 1993, 22(3): 7-9. |
| [19] |
ZHENG B, SUN S C. Review of the biogeography of Artemia Leach, 1819 (Crustacea: Anostraca) in China[J]. International Journal of Artemia Biology, 2013, 3(1): 20-50. |
| [20] |
付春正, 王晖, ASEM A, 等. 基于COI基因的入侵物种旧金山卤虫遗传多样性与遗传分化研究[J]. 河北渔业, 2022(2): 1-6, 32. |
| [21] |
ASEM A, EIMANIFAR A, LI W, et al. Phylogeography and population genetic structure of an exotic invasive brine shrimp, Artemia Leach, 1819 (Crustacea: Anostraca), in Australia[J]. Australian Journal of Zoology, 2018, 66(6): 307-316. |
| [22] |
ASEM A, EIMANIFAR A, SUN S C. Genetic variation and evolutionary origins of parthenogenetic Artemia (Crustacea: Anostraca) with different ploidies[J]. Zoologica Scripta, 2016, 45(4): 421-436. |
| [23] |
MUÑOZ J, GÓMEZ A, GREEN A J, et al. Phylogeography and local endemism of the native Mediterranean brine shrimp Artemia salina (Branchiopoda: Anostraca)[J]. Molecular Ecology, 2008, 17(13): 3160-3177. |
| [24] |
KUMAR S, STECHER G, TAMURA K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2015, 33(7): 1870-1874. |
| [25] |
EXCOFFIER L, LISCHER H E. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. |
| [26] |
桂富荣, 郭建英, 万方浩. ISSR分子标记在入侵植物研究中的应用[J]. 应用生态学报, 2007, 18(4): 919-927. |
| [27] |
胡玉婷, 江河, 段国庆, 等. 基于线粒体Cyt b基因的皖南山区温州光唇鱼种群遗传结构[J]. 中国农学通报, 2017, 33(32): 121-126. |
| [28] |
KAPPAS I, ABATZOPOULOS T J, VAN H N, et al. Genetic and reproductive differentiation of Artemia franciscana in a new environment[J]. Marine Biology, 2004, 146(1): 103-117. |
| [29] |
褚栋, 张友军, 万方浩. 分子标记技术在入侵生态学研究中的应用[J]. 应用生态学报, 2007, 18(6): 1383-1387. |
| [30] |
李忠超, 王小兰. 保护生物学中若干术语的理解和辨析(2)[J]. 生物学通报, 2005, 40(11): 3-5. |
| [31] |
EIMANIFAR A, STAPPEN G V, MARDEN B, et al. Artemia biodiversity in Asia with the focus on the phylogeography of the introduced American species Artemia franciscana Kellogg, 1906[J]. Molecular Phylogenetics and Evolution, 2014, 79: 392-403. |



