美蓝拟相手蟹(Parasesarma eumolpe)原隶属于相手蟹科(Sesarminae)的近相手蟹属(Perisesarma),现已归属于拟相手蟹属(Parasesarma)[1-3],分布于马来西亚、新加坡、泰国、文莱,以及中国南部沿海的广西、广东和海南等省区,是一种栖息于沿海红树林生态系统中的相手蟹类[4, 5]。国外已对美蓝拟相手蟹进行了食性、体色、形态及分子系统学等方面的研究[2, 5-7],而国内仅对其地理分布和系统发育做了初步研究[4, 8],目前尚无关于其群体遗传多样性方面的研究报道。
动物线粒体DNA由于具有因高碱基突变率而使其进化速度快的特征,成为了研究蟹类起源进化、系统发育、群体遗传等的一种有效分子遗传标记[9]。迄今为止,在蟹类群体遗传多样性的研究中,线粒体DNA的细胞色素氧化酶亚基Ⅰ (Cytochrome Oxidase Ⅰ, COⅠ)基因是使用较多且有效的分子标记[10-13]。美蓝拟相手蟹的分布具有中国大陆与马来西亚、新加坡、泰国的跨海地理分布格局特征[4, 5],适合用于揭示在较大的生态地理格局上遗传谱系演化的系统地理学规律。本研究以中国南部沿海红树林生态系统中8个美蓝拟相手蟹群体为研究对象,利用线粒体COⅠ基因片段序列,研究其群体遗传多样性,探讨现有群体分布格局形成的历史原因和演化过程。研究结果将有助于揭示我国红树林潮间带相手蟹类群体遗传结构和分子系统地理学规律,为蟹类的群体遗传学研究和红树林生态系统中蟹类的多样性保护研究提供基础资料。
1 材料与方法 1.1 材料2012年7月至2013年6月,本研究团队对中国南部沿海福建、广东、广西、海南等省区天然红树林区域的蟹类资源进行调查,并对调查区域中发现美蓝拟相手蟹分布的8个调查地点进行样品采集,共采集95个个体(表 1),采集地点基本覆盖了美蓝拟相手蟹在中国南部沿海的分布范围,采集的样本于95%乙醇固定保存备用。
| 群体 Populations |
样本数 No.of samples |
经纬度 Longitude & latitude |
| Fangchenggang, Guangxi | 15 | 108°14′7.9″ E, 21°37′6.2″ N |
| Leizhou, Guangdong | 10 | 109°44′9.7″ E, 20°48′13.2″ N |
| Xuwen, Guangdong | 3 | 110°7′19.3″ E, 20°14′47.7″ N |
| Dongzhaigang, Hainan | 19 | 110°33′34.4″ E, 19°58′34.2″ N |
| Qinglangang, Hainan | 16 | 110°48′7.9″ E, 19°35′39.2″ N |
| Sanya, Hainan | 6 | 109°37′7.0″ E, 18°13′27.3″ N |
| Dongfang, Hainan | 19 | 108°38′15.2″ E, 19°12′58.4″ N |
| Yangpu, Hainan | 7 | 109°15′15.0″ E, 19°46′8.8″ N |
1.2 方法
取美蓝拟相手蟹步足肌肉组织约20 mg,采用试剂盒(OMEGA公司)提取DNA,获得的DNA经检测合格后于-20 ℃保存备用。扩增所用引物为通用引物[14],引物序列如下。COIH2198:5′-TAAACTTCAGGGTGACCAAAAAATCA - 3′,COIL1490:5′-GGTCAACAAATCATAAAGATATTGG - 3′。PCR扩增反应体系如下:25 μL 2×Taq10 MasterMix(北京奥赛博科技发展有限公司),2 μL的模板DNA,2 μL的上下游引物,超纯水补足到50 μL。PCR反应条件为94 ℃预变性2 min,35个循环(94 ℃变性45 s,47 ℃退火50 s,72 ℃延伸1 min),最后72 ℃延伸7 min。获得的PCR产物经检测合格后,送至广州英潍捷基贸易有限公司进行纯化和双向测序(ABI 3730XL测序仪)。
1.3 数据分析DNA序列均由DNAstar v7.1软件[15]进行编辑、校对、比对和分析。运用DnaSP v5软件[16]检测多态位点数、简约信息位点数以及单倍型数目。采用MEGA 7.0软件[17]基于Kimura-2P计算遗传距离,以弧边招潮(Uca arcuata,基因登录号:MK611607)为外群构建单倍型邻接关系树(NJ),采用1 000次重抽样评估其可靠性。采用Network 5.0软件构建单倍型网络关系图,分析单倍型谱系结构情况。
采用Arlequin 3.5软件[18]计算单倍型多样度(h)和核苷酸多样度(π),进行分子方差分析(Analysis of Molecular Variance, AMOVA,重复抽样次数设置为5 000),以及计算群体间的遗传分化指数Fst值(重复抽样次数设置为10 000)。群体的历史动态评估由核苷酸不配对分布和中性检验进行分析,中性假说是否成立由Tajima′s D值[19]和Fu′s Fs值[20]来判别,根据获得的群体扩张前的θ0值、群体扩张后的θ1值和群体扩张参数τ值进行核苷酸不配对分布分析[21]。群体扩张事件发生的时间用公式t=τ/2u计算,群体发生扩张的时间用t表示,DNA序列长度的突变速率用u表示。蟹类线粒体COⅠ基因2.3%/MY(Million Years,百万年)的核苷酸分歧速率已被诸多研究采用[22-24],美蓝拟相手蟹群体的扩张时间亦采用2.3%/MY的核苷酸分歧速率来估算。
2 结果与分析 2.1 序列多态性分析每个个体均获得长度为631 bp的COⅠ基因序列,没有插入/缺失位点。序列比对共发现50个变异位点,其中简约信息位点16个,共计51处核苷酸替换被这些同义突变位点所定义(44处为转换,7处为颠换)。美蓝拟相手蟹95个个体共定义了49个单倍型(单倍型Hap1-Hap49,GenBank登陆号:MT827024-MT827072),这些单倍型序列的A、T、G、C碱基平均含量分别为28.5%、39.1%、17.3%、15.1%,G+C含量(32.4%)明显低于A+T含量(67.6%)。
美蓝拟相手蟹总群体的单倍型多样度为0.876 8±0.032 5,核苷酸多样度为0.042 2±0.026 3。8个地理群体中,单倍型多样度最高的是徐闻群体(1.000 0±0.272 2),最低的是东寨港群体(0.672 5±0.119 0);核苷酸多样度最高的和最低的也分别是徐闻群体(0.053 3±0.047 8)和东寨港群体(0.029 2±0.020 7)(表 2)。由于部分群体样品数量较少,可能会对统计结果产生一定影响。
| 群体 Populations |
个体数 No.of samples |
单倍型数 No.of haplotypes |
单倍型多样度(h) Haplotype diversity (h) |
核苷酸多样度(π) Nucleotide diversity (π) |
Tajima′s D | Fu′s Fs | 核苷酸不配对分布 Nucleotide mismatch distribution |
||||||
| D | P | Fs | P | τ | θ0 | θ1 | |||||||
| Fangchenggang | 15 | 10 | 0.895 2±0.070 4 | 0.039 2±0.026 3 | |||||||||
| Leizhou | 10 | 8 | 0.955 6±0.059 4 | 0.051 1±0.033 8 | |||||||||
| Xuwen | 3 | 3 | 1.000 0±0.272 2 | 0.053 3±0.047 8 | |||||||||
| Dongzhaigang | 19 | 8 | 0.672 5±0.119 0 | 0.029 2±0.020 7 | |||||||||
| Qinglangang | 16 | 13 | 0.950 0±0.048 5 | 0.046 8±0.030 2 | |||||||||
| Sanya | 6 | 5 | 0.933 3±0.121 7 | 0.048 0±0.034 8 | |||||||||
| Dongfang | 19 | 12 | 0.900 6±0.058 6 | 0.044 5±0.028 7 | |||||||||
| Yangpu | 7 | 5 | 0.857 1±0.137 1 | 0.045 7±0.032 5 | |||||||||
| All | 95 | 49 | 0.876 8±0.032 5 | 0.042 2±0.026 3 | -2.503 | <0.001 | -27.236 | <0.001 | 2.029 | 0.000 | 335.000 | ||
2.2 单倍型分布及其关系
美蓝拟相手蟹的49个单倍型中,出现了1个分布于除徐闻群体以外各群体的主体单倍型(Hap2),其所占频率为34.7%;有40个单倍型只在1个个体中被检测到,所占频率为42.1%,表明主体单倍型比较原始,可能是其祖先单倍型;有4个单倍型分别在2个个体中被发现;其他均为群体间共享的单倍型(表 3)。
| 单倍型 Haplotypes |
群体Populations | 合计 Total |
|||||||
| 防城港 Fangchenggang |
雷州 Leizhou |
徐闻 Xuwen |
东寨港 Dongzhaigang |
清澜港 Qinglangang |
三亚 Sanya |
东方 Dongfang |
洋浦 Yangpu |
||
| Hap1 | 1 | 1 | |||||||
| Hap2 | 5 | 2 | 11 | 4 | 2 | 6 | 3 | 33 | |
| Hap3 | 1 | 1 | |||||||
| Hap4 | 1 | 1 | 2 | 4 | |||||
| Hap5 | 1 | 2 | 3 | ||||||
| Hap6 | 2 | 1 | 3 | ||||||
| Hap7 | 1 | 1 | |||||||
| Hap8 | 1 | 1 | |||||||
| Hap9 | 1 | 1 | |||||||
| Hap10 | 1 | 1 | |||||||
| Hap11 | 1 | 1 | |||||||
| Hap12 | 1 | 1 | |||||||
| Hap13 | 1 | 1 | |||||||
| Hap14 | 1 | 1 | |||||||
| Hap15 | 2 | 2 | |||||||
| Hap16 | 1 | 1 | |||||||
| Hap17 | 1 | 1 | |||||||
| Hap18 | 1 | 1 | 2 | ||||||
| Hap19 | 1 | 1 | |||||||
| Hap20 | 1 | 1 | |||||||
| Hap21 | 2 | 1 | 1 | 4 | |||||
| Hap22 | 1 | 1 | |||||||
| Hap23 | 1 | 1 | 2 | ||||||
| Hap24 | 1 | 1 | |||||||
| Hap25 | 1 | 1 | |||||||
| Hap26 | 1 | 1 | 2 | ||||||
| Hap27 | 1 | 1 | |||||||
| Hap28 | 1 | 1 | |||||||
| Hap29 | 1 | 1 | |||||||
| Hap30 | 1 | 1 | |||||||
| Hap31 | 1 | 1 | |||||||
| Hap32 | 1 | 1 | |||||||
| Hap33 | 1 | 1 | |||||||
| Hap34 | 1 | 1 | |||||||
| Hap35 | 1 | 1 | |||||||
| Hap36 | 1 | 1 | |||||||
| Hap37 | 1 | 1 | |||||||
| Hap38 | 1 | 1 | |||||||
| Hap39 | 1 | 1 | |||||||
| Hap40 | 1 | 1 | |||||||
| Hap41 | 1 | 1 | |||||||
| Hap42 | 1 | 1 | |||||||
| Hap43 | 1 | 1 | |||||||
| Hap44 | 1 | 1 | |||||||
| Hap45 | 1 | 1 | |||||||
| Hap46 | 1 | 1 | |||||||
| Hap47 | 1 | 1 | |||||||
| Hap48 | 1 | 1 | |||||||
| Hap49 | 1 | 1 | |||||||
| Total | 15 | 10 | 3 | 19 | 16 | 6 | 19 | 7 | 95 |
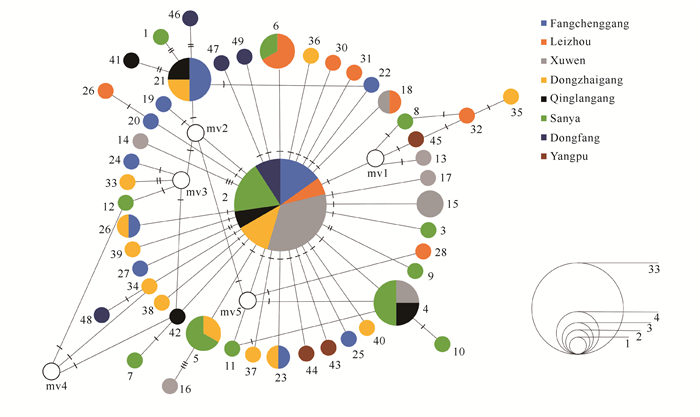
基于美蓝拟相手蟹49个单倍型构建的邻接关系树,其拓扑结构显示没有明显的单倍型类群分化出来,即群体之间尚未形成显著的分支谱系,单倍型聚类没有表现出地域性(图 1)。采用中介网络法构建的美蓝拟相手蟹单倍型网络关系图,其拓扑结构与邻接关系树类似,亦明显表现出49个单倍型,没有分化出明显的地域性分支,而是呈现出明显的星状结构,即以占频率34.7%的主体单倍型Hap2为中心,其余单倍型呈星状排列在其周围(图 2)。

|
| Uca arcuate (Ua) served as an outgroup.Bootstrap supports greater than or equal to 50% are shown. 图 1 基于Kimura-2P构建的美蓝拟相手蟹COⅠ基因单倍型邻接树 Fig.1 Neighbor-joining tree of COⅠ gene haplotypes constructed using Kimura-2P distance for P.eumolpe |
|
| The different color shades refer to different populations, sizes of circles indicate frequencies of haplotype, the numbers 1-49 represent haplotype ordinals, and number of dash line on the lines presents the number of mutant numbers. 图 2 美蓝拟相手蟹单倍型网络关系图 Fig.2 Network relationship diagram for haplotypes of P.eumolpe |
2.3 群体遗传结构
进行AMOVA时,先设置美蓝拟相手蟹8个群体作为一个整体,得到群体间的遗传差异为0.48%,群体内的遗传差异为99.52%,群体间的遗传差异很小,群体间的固定指数为0.004 84(P>0.05)(表 4),表明美蓝拟相手蟹8个群体的遗传变异主要来自群体内而非群体间。此外,将8个群体按地域分为两广(防城港群体、雷州群体、徐闻群体)和海南(东寨港群体、清澜港群体、三亚群体、东方群体、洋浦群体)两个组群进行AMOVA,结果显示,组群间的遗传差异为-0.45%,群体内的遗传差异为99.74%,组群间的遗传差异非常小(ΦCT=0.004 46,P>0.05)(表 4),表明遗传差异主要来自群体内。
| 组群 Group |
分组与变异来源 Source of grouping and variation |
自由度 df |
变异组成 Variation components |
变异百分比/% Percentage of variation/% |
固定指数 Fixation index |
P值 P value |
| One gene pool | Among populations | 7 | 0.005 11 | 0.48 | 0.004 84 | 0.304 01 |
| Within populations | 87 | 1.052 07 | 99.52 | |||
| Two gene pools (Fangchenggang, Leizhou, Xuwen) (Dongzhaigang, Qinglangang, Sanya, Dongfang, Yangpu) | Among groups | 1 | - 0.004 70 | -0.45 | ΦCT=-0.004 46 | 0.657 87 |
| Among populations within groups | 6 | 0.007 43 | 0.70 | ΦSC=0.007 01 | 0.261 97 | |
| Within populations | 87 | 1.052 07 | 99.74 | ΦST=0.002 58 | 0.281 52 |
群体遗传分化指数Fst值显示,8个地理群体间遗传分化指数值为-0.045 94-0.096 26(表 5),呈现出中低度遗传分化水平,统计检验的P值除了东寨港群体分别与三亚群体、防城港群体之间存在显著性差异外(P<0.05),其他群体间差异不显著(P>0.05)。
| 群体 Populations |
防城港 Fangchenggang |
雷州 Leizhou |
徐闻 Xuwen |
东寨港 Dongzhaigang |
清澜港 Qinglangang |
三亚 Sanya |
东方 Dongfang |
洋浦 Yangpu |
| Fangchenggang | 0.252 25 | 0.180 18 | 0.000 00 | 0.639 64 | 0.810 81 | 0.207 21 | 0.621 62 | |
| Leizhou | 0.017 14 | 0.585 59 | 0.243 24 | 0.639 64 | 0.441 44 | 0.747 75 | 0.954 95 | |
| Xuwen | 0.083 00 | -0.039 12 | 0.090 09 | 0.342 34 | 0.180 18 | 0.468 47 | 0.405 41 | |
| Dongzhaigang | 0.053 60 | 0.012 62 | 0.085 68 | 0.252 25 | 0.027 03 | 0.279 28 | 0.261 26 | |
| Qinglangang | -0.010 98 | -0.013 36 | -0.008 50 | 0.003 88 | 0.531 53 | 0.891 89 | 0.873 87 | |
| Sanya | -0.045 94 | 0.006 62 | 0.060 24 | 0.096 26 | -0.005 75 | 0.639 64 | 0.477 48 | |
| Dongfang | 0.013 73 | -0.015 82 | 0.031 30 | 0.007 37 | -0.016 76 | -0.014 27 | 0.594 59 | |
| Yangpu | -0.017 12 | 0.029 29 | 0.016 85 | 0.026 02 | -0.025 18 | -0.024 39 | -0.012 93 |
2.4 群体历史动态
美蓝拟相手蟹核苷酸不配对分布图为单峰状,并且与群体扩张模型下的预期分布密切吻合(图 3),也与星状网络图一致(图 2)。此外,Fs值(-27.236,P<0.001)和D值(-2.503,P<0.001)的中性检验结果与核苷酸不配对分布结果也一致(表 2),不仅Fs检验值为负值且统计检验显著偏离中性,D值也都是负值并且统计检验也都是显著的。上述所有结果表明美蓝拟相手蟹在历史上发生过明显的群体扩张事件。美蓝拟相手蟹核苷酸不配对分布的τ值为2.029,按照COⅠ基因2.3%/MY的分歧速率计算,其群体扩张时间大约为14万年前。

|
| 图 3 美蓝拟相手蟹群体核苷酸不配对分布图 Fig.3 Nucleotide mismatch distribution map of P.eumolpe population |
3 讨论 3.1 群体遗传多样性
单倍型多样度(h)与核苷酸多样度(π)是在分子水平上衡量群体遗传多样性的重要指标[25]。美蓝拟相手蟹8个群体表现为高水平的单倍型多样度(0.876 8±0.032 5)和核苷酸多样度(0.042 2 ± 0.026 3),符合高h和高π的模式(h>0.5,π>0.005)[26],说明美蓝拟相手蟹曾经具有较大而有效的群体数量,在长期演化过程中较稳定,对沿海环境变化和自然选择压力有较强的适应能力,未出现过瓶颈效应现象,因而呈现出较高的遗传多样性水平。与基于COⅠ分子标记的其他潮间带蟹类相比,美蓝拟相手蟹的群体遗传多样性指数高于环纹招潮[Uca annulipes(h=0.207, π=0.000 4)][27]、双齿拟相手蟹[P.bidens (h=0.495, π=0.001 33)][12]、近亲拟相手蟹[P.affine(h=0.508 9, π=0.001 126)][28],与天津厚蟹(Helice tientsinensis)和侧足厚蟹[H.latimera(h=0.787, π=0.002 1)][10]处于相近水平,说明不同蟹类的群体遗传多样性水平存在一定差异,亦反映出不同物种对不同环境的适应潜力也不同。
3.2 群体遗传结构遗传分化指数Fst是衡量群体遗传分化程度的重要指标,当0<Fst≤0.05时,表明群体间遗传分化水平很低;当0.05<Fst≤0.15时,表明群体间发生中度遗传分化;当0.15<Fst≤0.25时,表明群体间存在高度遗传分化;当Fst>0.25时,群体间发生极高的遗传分化[29]。美蓝拟相手蟹8个群体间的Fst值为-0.045 94-0.096 26,表明各群体间存在不同程度的遗传分化。东寨港群体与三亚、徐闻、防城港群体间的Fst值处于0.05-0.15,说明它们之间存在中等程度的遗传分化;而大部分群体间的遗传分化指数Fst值均小于0.05,并且各群体间的遗传分化水平与地理距离没有呈现出明显的相关关系,提示美蓝拟相手蟹各群体间存在一定的基因交流。AMOVA结果表明,美蓝拟相手蟹8个群体间的遗传变异水平明显低于群体内,可见群体之间无显著的群体遗传差异;单倍型网络关系图也支持上述结论,主体单倍型几乎出现在所有群体中,表明各群体在历史上存在较强的基因交流[30]。
诸多海洋无脊椎动物具有浮游期,浮游幼虫可以借助洋流进行扩散,使不同地理群体之间能保持基因交流,从而产生遗传同质化[31, 32]。相手蟹科蟹类通常会在涨潮期间释放幼虫,而其幼虫发育通常会持续近1个月,甚至更长时间[33, 34],美蓝拟相手蟹与其他相手蟹科蟹类一样,较长的幼虫浮游期可能是其具备较强潜在扩散能力的重要因素。幼体浮游期长,加上潮汐动力及洋流运输作用,可能使美蓝拟相手蟹不同地理群体之间基因交流频繁,群体间缺少遗传分化,也就很难在群体之间产生独立的群体遗传结构。有关潮间带蟹类的研究表明,天津厚蟹和侧足厚蟹群体之间存在较显著的遗传差异,可能是与其地理群体之间存在天然障碍如长江冲淡水有关[10]。隋宥珍等[35]在对黄渤、渤海、东海、南海口虾蛄(Oratosquilla oratoria)种群遗传结构的研究中认为其种群遗传结构模式可能与栖息地海洋环境条件以及自身的生活史特征有关,长江淡水注入而形成的天然屏障同样也阻碍了口虾蛄幼体分布的连续性。而在中国南部沿海,可能缺乏明显影响群体扩散的天然屏障,同样的现象也出现在双齿近相手蟹[12]、近亲拟相手蟹[28]以及其他海洋生物如可口革囊星虫(Phascolosoma esculenta)[36]、金钱鱼(Scatophagus argus)[37]、长鳍篮子鱼(Siganus canaliculatus)[38]等群体中。
3.3 群体历史动态研究表明,如果核苷酸不配对分布呈明显的单峰,意味着该群体经历过近期的群体扩张事件[21, 39, 40]。美蓝拟相手蟹的核苷酸不配对分布图为单峰状,观测值与预期值分布一致,并且单倍型网络关系图呈星状分布,进一步表明美蓝拟相手蟹经历了群体快速扩张事件。在过去的几十万年里,西北太平洋气候的剧烈变迁,对生活于该地区生物群落的演化产生了很大的影响;更新世冰期,特别是末次冰盛期,西北太平洋海平面变动剧烈,逼迫海洋生物栖息地发生剧烈变化[41]。单倍型网络关系图、核苷酸不配对分析和中性检验所揭示的遗传信息,均支持美蓝拟相手蟹在历史上曾发生过群体扩张事件,扩张时间大约在14万年前的观点。其他生活于潮间带或浅海区域的蟹类群体扩张时间与美蓝拟相手蟹相近,它们均在更新世冰期发生过群体扩张事件[10, 12, 28, 42]。这些蟹类形成的系统地理分布格局模式可能与更新世冰期-间冰期气候变化有密切关系。Shahdadi等[43]对西太平洋河口蟹类的遗传结构研究发现双齿拟相手蟹复合群体在日本到印度尼西亚西巴布亚的整个分布范围内存在潜在遗传结构,这个复合群体至少由5个不同的进化枝组成,更新世冰期海平面退缩和洋流可能是其复合群体形成5个进化枝的主要成因。可见,西北太平洋更新世冰期和间冰期的交替,剧烈影响着海洋流动的模式以及海水水温、盐度等,而这些因素的变化必然影响潮间带及浅海区蟹类的群体遗传结构与系统地理分布格局。
4 结论本研究基于线粒体COⅠ基因对中国南部沿海美蓝拟相手蟹8个群体进行遗传结构和群体历史动态研究,结果表明美蓝拟相手蟹不同群体之间无显著的遗传差异,单倍型邻接关系树和网络关系图结果均未表现出明显的地理谱系结构,核苷酸不配对分布图为单峰状,表明美蓝拟相手蟹在历史上经历了群体快速扩张事件,扩张时间大约在14万年前。幼体浮游阶段具较强的潜在扩散能力,再加上群体扩张事件,可能是美蓝拟相手蟹能进行频繁的基因交流导致群体间无显著遗传分化的重要原因。由于采集的美蓝拟相手蟹群体数量有限,数据分析存在一定局限性。今后,应扩大研究范围,调查美蓝拟相手蟹在中国沿海广东以北区域是否有分布,加入国外美蓝拟相手蟹种群标本,并尽可能增加某些种群的数量,运用多种分子标记手段,对其遗传多样性进行深入研究,从而更全面地阐释其分子系统地理分布格局。
| [1] |
NG P K L, GUINOT D, DAVIE P J F. Systema Brachyurorum: part Ⅰ.An annotated checklist of extant Brachyuran crabs of the world[J]. The Raffles Bulletin of Zoology (Suppl.), 2008, 17: 1-286. |
| [2] |
ADNAN S, SCHUBART C D. Taxonomic review of Perisesarma (Decapoda: Brachyura: Sesarmidae) and closely related genera based on morphology and molecular phylogenetics: new classification, two new genera and the questionable phylogenetic value of the epibranchial tooth[J]. Zoological Journal of the Linnean Society, 2017, 182(3): 517-548. |
| [3] |
SHAHDADI A, FRATINI S, SCHUBART C D. Taxonomic reassessment of Parasesarma (Crustacea: Brachyura: Decapoda: Sesarmidae) based on genetic and morphological comparisons, with the description of a new genus[J]. Zoological Journal of the Linnean Society, 2020, 190(4): 1123-1158. DOI:10.1093/zoolinnean/zlaa025 |
| [4] |
杨明柳, 徐敬明, 吴斌, 等. 北部湾红树林蟹类多样性初步研究[J]. 四川动物, 2014, 33(3): 347-352, 357. |
| [5] |
SHAHDADI A, NG P K L, SCHUBART C D. Morphological and phylogenetic evidence for a new species of Parasesarma De Man, 1895 (Crustacea: Decapoda: Brachyura: Sesarmidae) from the Malay Peninsula, previously referred to as Parasesarma indiarum (Tweedie, 1940)[J]. The Raffles Bulletin of Zoology, 2018, 66: 739-762. |
| [6] |
HUANG H, TODD P, YEO D C J. Inter- and intra-specific variation in the facial colours of Perisesarma eumolpe and Perisesarma indiarum (Crustacea: Brachyura: Sesarmidae)[J]. Hydrobiologia, 2008, 598(1): 361-371. DOI:10.1007/s10750-007-9169-z |
| [7] |
YA B P, YEO D C J, TODD P A. Feeding ecology of two species of Perisesarma (Crustacea: Decapoda: Brachyura: Sesarmidae) in Mandai mangroves, Singapore[J]. Journal of Crustacean Biology, 2008, 28(3): 480-484. DOI:10.1651/07-2835R.1 |
| [8] |
徐岩, 潘红平, 阎冰, 等. 基于线粒体COⅠ和16S rRNA基因的中国沿海相手蟹系统发育研究[J]. 海洋学报, 2019, 41(8): 63-71. DOI:10.3969/j.issn.0253-4193.2019.08.008 |
| [9] |
徐敬明. 蟹类线粒体DNA的研究与应用[J]. 中国海洋大学学报(自然科学版), 2006, 36(6): 879-884. DOI:10.16441/j.cnki.hdxb.2006.06.006 |
| [10] |
XU J M, YANG F, SUN S C. Population genetic structure and phylogeography of the mud-flat crabs Helice tientsinensis and Helice latimera along the coast of China seas based on mitochondrial DNA[J]. African Journal of Biotechnology, 2012, 11(16): 3738-3750. |
| [11] |
HAN Z Q, ZHENG W, CHEN G B, et al. Population genetic structure and larval dispersal strategy of portunid crab Charybdis bimaculatain in Yellow sea and East China sea[J]. Mitochondrial DNA, 2015, 26(3): 402-408. DOI:10.3109/19401736.2013.840592 |
| [12] |
ZHOU H L, XU J M, YANG M L, et al. Population genetic diversity of sesarmid crab (Perisesarma bidens) in China based on mitochondrial DNA[J]. Mitochondrial DNA A DNA Mapp Seq Anal, 2016, 27(5): 3255-3262. |
| [13] |
REN G J, MIAO G D, MA C Y, et al. Genetic structure and historical demography of the blue swimming crab (Portunus pelagicus) from southeastern sea of China based on mitochondrial COⅠ gene[J]. Mitochondrial DNA A DNA Mapp Seq Anal, 2018, 29(2): 192-198. |
| [14] |
FOLMER O, BLACK M, WR H, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ⅰ from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology, 1994, 3(5): 294-299. |
| [15] |
DURFEE T, SCHWEI T E. DNASTAR's next-generation software[M]. Weinheim: WILEY-VCH Verlag GmbH & Co.KgaA, 2008.
|
| [16] |
LIBRADO P, ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [17] |
KUMAR S, STECHER G, TAMURA K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [18] |
EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
| [19] |
TAJIMA F. Statistical-method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595. DOI:10.1093/genetics/123.3.585 |
| [20] |
FU Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147(2): 915-925. DOI:10.1093/genetics/147.2.915 |
| [21] |
ROGERS A R, HARPENDING H. Population growth makes waves in the distribution of pairwise genetic differences[J]. Molecular Biology and Evolution, 1992, 9(3): 552-569. |
| [22] |
GOPURENKO D, HUGHES J M, KEENAN C P. Mitochondrial DNA evidence for rapid colonization of the Indo-West Pacific by the mud crab Scylla serrata[J]. Marine Biology, 1999, 134(2): 227-233. DOI:10.1007/s002270050541 |
| [23] |
DANIELS S R, STEWART B A, GOUWS G, et al. Phylogentic relationships of the southern African freshwater crab fauna (Decapoda: Potamonautidae: Potamonautes) derived from multiple data sets reveal biogeographic patterning[J]. Molecular Phylogenetics and Evolution, 2003, 25(3): 511-523. |
| [24] |
PFEILER E, HURTADO L A, KNOWLES L L, et al. Population genetics of the swimming crab Callinectes bellicosus (Brachyura: Portunidae) from the eastern Pacific Ocean[J]. Marine Biology, 2005, 146(3): 559-569. DOI:10.1007/s00227-004-1463-y |
| [25] |
NEI M, LI W H. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proceedings of the National Academy of Sciences of the United States of America, 1979, 76(10): 5269-5273. |
| [26] |
GRANT W S, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [27] |
SILVA I C, MESQUITA N, PAULA J. Lack of population structure in the fiddler crab Uca annulipes along an East African latitudinal gradient: genetic and morphometric evidence[J]. Marine Biology, 2010, 157: 1113-1126. DOI:10.1007/s00227-010-1393-9 |
| [28] |
杨明柳, 徐岩, 高霆炜, 等. 基于COⅠ基因片段的中国南部沿海近亲拟相手蟹的群体遗传多样性研究[J]. 海洋学报, 2021, 43(2): 105-115. |
| [29] |
BALLOUX F, LUGON-MOULIN N. The estimation of population differentiation with microsatellite markers[J]. Molecular Ecology, 2002, 11(2): 155-165. DOI:10.1046/j.0962-1083.2001.01436.x |
| [30] |
NI G, LI Q, KONG L F, et al. Comparative phylogeography in marginal seas of the northwestern Pacific[J]. Molecular Ecology, 2014, 23(3): 534-548. DOI:10.1111/mec.12620 |
| [31] |
PALUMBI S R. Genetic divergence, reproductive isolation, and marine speciation[J]. Annual Review of Ecology and Systematics, 1994, 25(1): 547-572. DOI:10.1146/annurev.es.25.110194.002555 |
| [32] |
JE LEE H, BOULDING E G. Spatial and temporal population genetic structure of four northeastern Pacific littorinid gastropods: the effect of mode of larval development on variation at one mitochondrial and two nuclear DNA markers[J]. Molecular Ecology, 2009, 18(10): 2165-2184. DOI:10.1111/j.1365-294X.2009.04169.x |
| [33] |
KRISHNAN T, KANNUPANDI T. Larval development of the mangrove crab Sesarma bidens (De Haan, 1853) in the laboratory (Brachyura: Grapsidae: Sesarminae)[J]. Mahasagar, 1987, 20(3): 171-181. |
| [34] |
RAGIONIERI L, CANNICCI S, SCHUBART C D, et al. Gene flow and demographic history of the mangrove crab Neosarmatium meinerti: a case study from the western Indian Ocean[J]. Estuarine, Coastal and Shelf Science, 2010, 86(2): 179-188. DOI:10.1016/j.ecss.2009.11.002 |
| [35] |
隋宥珍, 刘连为, 徐开达, 等. 基于线粒体Cytb基因的口虾蛄种群遗传结构研究[J]. 大连海洋大学学报, 2019, 34(3): 355-361. |
| [36] |
许瑞雯, 陈兴汉, 余祥勇. 基于线粒体cytb和d-loop基因序列的东南沿海可口革囊星虫遗传多样性分析[J]. 海洋渔业, 2022, 44(3): 288-302. |
| [37] |
彭敏, 肖珊, 陈晓汉, 等. 基于线粒体Cyt b基因的南海北部金钱鱼种群遗传结构分析[J]. 南方农业学报, 2021, 52(10): 2851-2860. |
| [38] |
彭敏, 肖珊, 洪传远, 等. 华南沿海长鳍篮子鱼不同地理群体的遗传多样性分析[J]. 水生态学杂志, 2022, 43(5): 127-133. |
| [39] |
SLATKIN M, HUDSON R R. Pairwise comparisons of mitochondrial DNA sequences in stable and exponentially growing populations[J]. Genetics, 1991, 129(2): 555-562. DOI:10.1093/genetics/129.2.555 |
| [40] |
RAY N, CURRAT M, EXCOFFIER L. Intra-deme molecular diversity in spatially expanding populations[J]. Molecular Biology and Evolution, 2003, 20(1): 76-86. DOI:10.1093/molbev/msg009 |
| [41] |
HEWITT G M. Genetic consequences of climatic oscillations in the Quaternary[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2004, 359(1442): 183-195. DOI:10.1098/rstb.2003.1388 |
| [42] |
董志国, 李晓英, 王普力, 等. 基于线粒体D-loop基因的中国海三疣梭子蟹遗传多样性与遗传分化研究[J]. 水产学报, 2013, 37(9): 1304-1312. |
| [43] |
SHAHDADI A, VON WYSCHETZKI K, LIU H C, et al. Molecular phylogeography reveals multiple Pleistocene divergence events in estuarine crabs from the tropical West Pacific[J]. PLoS One, 2022, 17(1): e0262122. DOI:10.1371/journal.pone.0262122 |



