2. 广西民族大学化学与生态工程学院, 广西南宁 530007
2. College of Chemistry and Engineering, Guangxi University for Nationalities, Nanning, Guangxi, 530007, China
制革行业是我国的重污染行业之一,在如今高标准的环保要求下,其生产废水的治理问题值得重视[1, 2]。制革废水具有高浓度、高盐量、高色度、悬浮物多以及水质波动大等特点[3],此外,在皮革加工过程中添加的助剂、中和剂、铬鞣剂、植物鞣剂、石灰、硫化钠、铵盐、酸、碱、蛋白酶等材料,使制革废水中的还原性物质增加,直接或间接导致其中的化学需氧量(COD)含量较高[4]。据报道,制革废水中COD含量为3 000-5 000 mg/L、氨氮含量为150-300 mg/L[5]。Feng等[6]采用电凝法处理制革废水,其COD、氨氮的去除率分别为68.0%和43.1%,氨氮的去除效果并不理想。高级氧化法处理污水虽然具有高效和迅速的优点,但是成本较高,经济性有待提高[7]。制革废水的处理方法还有混凝沉淀法、膜分离法等,虽然也取得不错的效果,但经济成本投入较大,同时,废水处理产生的二次污染问题,也限制了这些处理方法的大规模应用[8]。制革废水中含有大量的可溶性蛋白、脂肪等有机物和甲酸等低分子添加剂,m(BOD5)/m(CODcr)的值为0.3-0.6,可生化性较好,适合进行生物处理[9]。因此,面对目前日渐严苛的废水排放环保标准,生物处理方法作为一种成本低廉、不产生二次污染的废水处理方法[10],值得进一步探讨和优化。刘志刚等[11]利用微生物复合菌剂解决了黑臭河道水体污染问题,同时该微生物复合菌剂对COD和氨氮的去除具有一定的效果,表明可通过添加复合人工微生物组来提高废水处理效果。本研究拟在制革废水处理过程中投加复合人工微生物组,优化制革废水的处理效果,并通过高通量测序的方法,对其中的微生物群落多样性进行研究,为复合人工微生物组在制革废水处理中的实际应用提供参考。
1 材料与方法 1.1 材料 1.1.1 接种污泥及实验用水好氧污泥取自南宁市某污水处理厂,外加一定比例的水以及糖蜜作为碳源,尿素作为氮源,经过一定时间的驯化后,投入制革废水处理装置。制革废水取自台山某制革厂,初始COD含量为5 745.9 mg/L,氨氮含量为456.2 mg/L。
1.1.2 实验菌株复合人工微生物组所用菌株为实验室筛选出来的、对制革废水处理具有正向作用的菌株,包括阴沟肠杆菌(Enterobacter cloacae)、芽孢杆菌(Bacillus spp.)、假单胞菌(Pseudomonas spp.),复配后构建成人工微生物组,命名为GXAS1。每次均从平板上挑取单菌落,经过液体培养基培养至一定浓度后进行菌种投加。处理前期混合配比成100 mL的混合液,形成优势菌后,逐步减少人工微生物组的投加量,并根据每天监测的废水COD数值进行动态调整。
1.1.3 实验装置实验所用3个反应池总体积为15 L,其中2个为好氧反应池,总体积为10 L,1个为厌氧反应池,体积为5 L,实际进水总体积为13 L。使用蠕动泵每日定时定量流加进水,并且通过虹吸排水法进行排水,进水量和排水量根据每天监测的COD和氨氮数值进行调整,每日的进出水量约为500 mL。好氧池中采用内置曝气头的方式进行持续曝气供氧。
1.2 方法 1.2.1 实验方法采用相同运行参数和规模的两组厌氧/好氧(A/O)反应器进行对照实验。实验组额外添加调配好的复合人工微生物组,对照组则添加同等体积的清水。每天定时取样测定出水氨氮及COD含量。
从实验组末端出水反应池中取样,标记为样品1;按照同样方式从对照组中取样,标记为样品2。
1.2.2 分析方法制革废水处理实验中,氨氮使用纳氏试剂分光光度法测定,COD使用快速消解分光光度法测定。
活性污泥的微生物群落分析实验中,DNA使用Nanodrop进行定量分析,DNA提取质量通过1.2%琼脂糖凝胶电泳进行检测,利用高保真DNA聚合酶进行PCR扩增后,进行荧光定量分析,扩增引物为F: 5′-ACTCCTACGGGAGGCAGCA-3′,R: 5′-CGGACTACHVGGGTWTCTAAT-3′,采用Illumina公司的TruSeq Nano DNA LT Library Prep Kit制备测序文库。利用Illumina MiSeq/NovaSeq平台进行群落DNA片段双端测序分析。
DADA2序列去噪:利用qiime cutadapt trim-paired将序列的引物片段切除,并将未能匹配的引物序列剔除;接着利用qiime dada2 denoise-paired调用DADA2进行质控、去噪、拼接、去嵌合体等一系列操作。以上所涉及的步骤在每个文库中单独进行分析。在对各个数据库进行降噪处理后,整合ASVs特征序列和ASV表格,并剔除singletons ASVs。
对测序结果开展Alpha多样性分析,并在各个分类水平上进行物种组成分析。在Alpha多样性分析中,丰富度用Chao1指数表征,多样性用Shannon指数和Simpson指数表征[12]。本次测序相对于整体样本的覆盖程度用菌群覆盖度(Goods_coverage)指数表示。
2 结果与分析 2.1 水质变化经处理,添加了复合人工微生物组的实验组最终出水COD含量基本稳定在1 000 mg/L左右,氨氮测量值在2.4 mg/L左右,COD降解率约为82.60%,氨氮去除效率约为99.47%,效果显著。
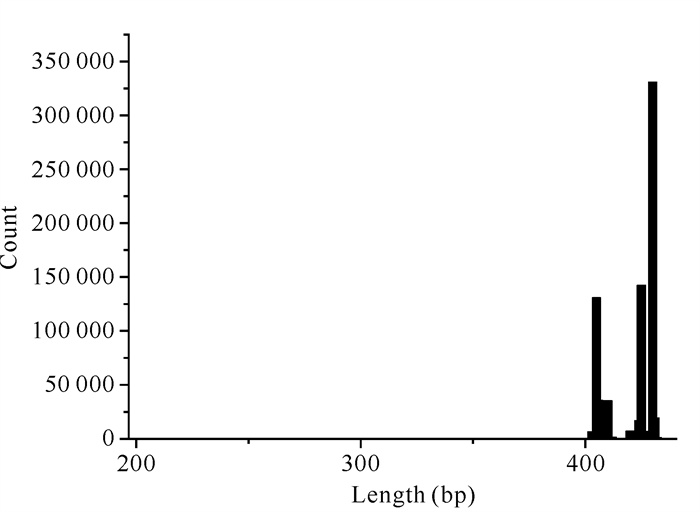
2.2 生物信息学分析 2.2.1 序列长度分布测序结果如图 1所示,序列长度基本上都集中在402-432 bp,平均长度418 bp,能满足后续的分析需求。
|
| 图 1 序列长度分布 Fig.1 Length distribution of the sequence |
2.2.2 DADA2序列去噪
对经过去噪处理后的各样本的测序量进行统计学处理,结果如表 1所示。处理结果满足后续的数据分析要求。
| 样品编号 Sample ID |
Input | Filtered | Denoised | Merged | Non-chimeric | Non-singleton |
| 1 | 97 793 | 85 527 | 82 397 | 73 349 | 57 303 | 56 582 |
| 2 | 83 060 | 74 398 | 71 775 | 65 884 | 54 194 | 53 576 |
2.3 Alpha多样性分析 2.3.1 Alpha多样性指数
Alpha多样性分析结果如表 2所示。Goods_coverage指数数值越高,表示覆盖程度越高[13]。2个样品的菌群覆盖度指数均大于0.98,表明本次测序的覆盖度高。Chao1指数越大,表明种群的丰富度越高[14]。从表 2可以看出,实验组微生物群落的丰富度高于对照组,说明添加的复合人工微生物能够在皮革废水处理系统中生长。Simpson指数值越大,说明群落多样性越低[15]。而Shannon指数相反,其值越高多样性越高。因此,综合对比实验组和对照组的Shannon指数和Simpson指数,可以反映出实验组微生物的多样性较高。
| 样品编号 Sample ID |
Chao1 | Simpson | Shannon | Goods_coverage |
| 1 | 1 881.89 | 0.956 735 | 7.267 66 | 0.988 129 |
| 2 | 1 486.75 | 0.962 624 | 7.131 28 | 0.992 116 |
2.3.2 稀疏曲线
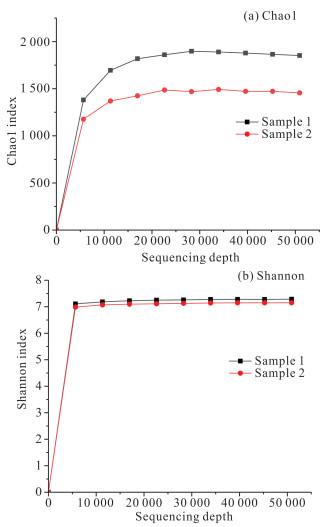
随着测序深度的增加,Chao1指数和Shannon指数均呈现先上升后平稳的趋势,后期Chao1指数趋于平缓,Shannon指数进入平台期,表明此测序深度有效,基本上已经完全覆盖样品中的所有微生物群落(图 2)。从图 2也可以较为直观地看出,实验组的微生物群落较丰富。
|
| 图 2 细菌群落稀疏曲线 Fig.2 Sparse curve of bacterial community |
2.4 物种组成分析
选取门水平上的物种组分进行对比,结果如图 3所示。在2组样品中占比前3的门类从高到低依次为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)。其中Proteobacteria在实验组和对照组样品中占比分别为44.7%、63.22%。Proteobacteria是城镇污水处理厂活性污泥系统和废水生物反应系统的主要种群,包含多种代谢种类,对废水中的碳、氮循环具有重要的作用,在降解有机物的同时,发挥脱氮除磷的作用[16]。Bacteroidetes在实验组中占比为28.95%,在对照组中占比为18.73%。复合人工微生物组的添加使Bacteroidetes比重明显高于对照组,而Bacteroidetes对废水中的有机物质具有很强的耐受性,可将复杂的有机物水解并降解为乳糖、乙酸、甲酸等[17],具有水解污泥絮体的作用,同时具有去除COD和氨氮的作用。因此从一定程度上反映出添加复合人工微生物组有助于皮革废水的处理。综合分析,投加复合人工微生物组对于3种优势菌门的地位没有影响,只是比重上有波动,且对COD和氨氮有去除作用的菌门占比增加。

|
| 图 3 细菌群落结构门水平上的组成 Fig.3 Bacterial community composition at phylum level |
选取纲水平上的物种组分进行对比。2组样品中比重在前3的纲均相同,分别为γ-变形菌纲(Gammaproteobacteria)、拟杆菌纲(Bacteroidia)和α-变形杆菌纲(Alphaproteobacteria),可以认为是它们共有的优势菌(图 4)。

|
| 图 4 细菌群落结构纲水平上的组成 Fig.4 Bacterial community composition at class level |
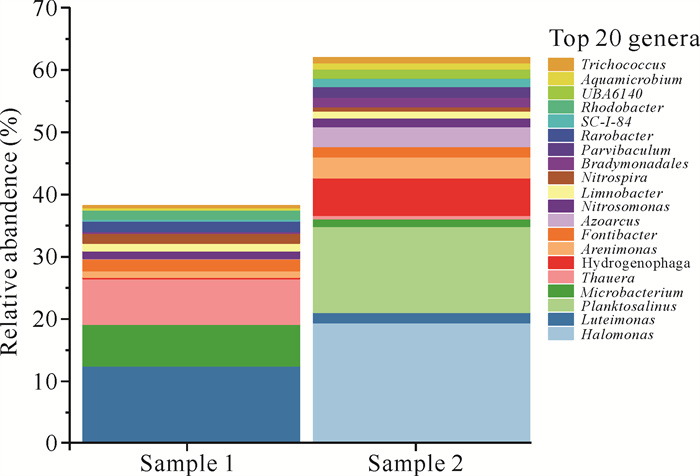
2组样品属水平丰度如图 5所示。对照组的优势属为盐单胞菌属(Halomonas)和湖水盐细菌(Planktosalinus),占比分别为19.11%和13.80%。对照组的优势属均为耐盐类菌属,嗜盐菌是一类能在高盐环境下进行正常生长代谢的细菌[18],其在对照组中占比高,反映出其能够适应制革废水的高盐含量环境,同时也能从侧面反映出制革废水属于高盐废水。实验组的优势属为藤黄色单胞菌属(Luteimonas)、陶厄氏菌属(Thauera)和微杆菌属(Microbacterium),占比分别为12.37%、7.13%和6.83%。Microbacterium是一类具有铬还原性的菌属,能降解毒性较高的Cr(Ⅵ),对废水中的Cr(Ⅵ)具有不错的降解能力[19]。Thauera在两个组中差异较大,在对照组中所占比重较小,不到1%。然而,根据前人研究,Thauera被认为是一类反硝化功能菌属,其降解污染物的能力非常广泛,甚至对于通常难以被生物分解的芳香族化合物也有很强的降解作用[20, 21]。由此在一定程度上说明,额外添加的复合人工微生物组使皮革废水处理体系中的Microbacterium、Thauera占比大幅增加,对反应体系中污染物的降解起到了强化的作用。
|
| 图 5 细菌群落结构属水平上的组成 Fig.5 Bacterial community composition at genus level |
3 结论
(1) 在制革废水的处理过程中投加复合人工微生物组,能够有效强化废水处理系统去除COD和氨氮的能力,并且对氨氮的处理效果显著,对制革废水中污染物的去除效果良好。
(2) 投加复合人工微生物组后,废水处理体系中微生物群落的多样性和丰富度均提高,但体系中的优势菌门并未发生变化,变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)依旧占据着主导地位。
(3) 复合人工微生物组的投加对于属水平上的物种组成影响较大,陶厄氏菌属(Thauera)成为制革废水处理体系中最主要的优势菌属,对COD和氨氮的降解起到了良好的作用,对制革废水处理体系起到了一定的强化作用。
| [1] |
唐行鹏, 孟宪礼, 侯成林, 等. 某皮革厂皮革废水处理工程案例分析[J]. 环境工程, 2016, 34(2): 63-68. |
| [2] |
胡静, 张晓宁. 制革废水脱氮处理技术的现状[J]. 西部皮革, 2016, 38(17): 33-35. DOI:10.3969/j.issn.1671-1602.2016.17.012 |
| [3] |
焦翔. 制革废水处理工艺运行优化研究[D]. 杭州: 浙江工业大学, 2017.
|
| [4] |
夏宏, 杨德敏. 制革废水及其处理现状综述[J]. 皮革与化工, 2014, 31(1): 25-29. DOI:10.3969/j.issn.1674-0939.2014.01.006 |
| [5] |
朱士彬. 制革废水及其处理工艺现状分析[J]. 魅力中国, 2020(35): 355. |
| [6] |
FENG J W, SUN Y B, ZHENG Z, et al. Treatment of tannery wastewater by electrocoagulation[J]. Journal of Environmental Sciences, 2007, 19(12): 1409-1415. DOI:10.1016/S1001-0742(07)60230-7 |
| [7] |
宋怡明, 徐少伟, 宋昊, 等. 高级氧化法污水处理技术综述[J]. 山东化工, 2019, 48(24): 211-213. DOI:10.3969/j.issn.1008-021X.2019.24.102 |
| [8] |
吴岩, 任相浩, 寇莹莹, 等. 复合微生物菌剂处理高浓度氨氮废水的强化作用[J]. 科学技术与工程, 2020, 20(25): 10544-10549. DOI:10.3969/j.issn.1671-1815.2020.25.062 |
| [9] |
张丽丽, 买文宁, 王晓慧. 制革废水处理技术的发展[J]. 工业用水与废水, 2004(5): 12-15. DOI:10.3969/j.issn.1009-2455.2004.05.004 |
| [10] |
梁波, 徐金球, 关杰, 等. 生物法处理印染废水的研究进展[J]. 化工环保, 2015, 35(3): 259-266. DOI:10.3969/j.issn.1006-1878.2015.03.008 |
| [11] |
刘志刚, 李东晓, 戴志东, 等. 基于复合微生物菌剂的黑臭河道治理[J]. 科学技术与工程, 2019, 19(1): 284-287. DOI:10.3969/j.issn.1671-1815.2019.01.043 |
| [12] |
LEGENDRE P, LEGENDRE L. Numerical ecology[M]. 2nd English edition. Amsterdam, the Netherlands: Elsevier Science B.V., 1998.
|
| [13] |
贺婧, 闫冰, 李俊生, 等. 秦岭中段北坡不同海拔土壤中细菌群落的分布特征及区域差异比较[J]. 环境科学研究, 2019, 32(8): 1374-1383. |
| [14] |
CHAO A, MA M C, YANG M C K. Stopping rules and estimation for recapture debugging with unequal failure rates[J]. Biometrika, 1993, 80(1): 193-201. DOI:10.1093/biomet/80.1.193 |
| [15] |
夏瑜, 何绪文, 文湘华. 微生物群落多样性数学表征方法及其在污水处理系统研究中的应用[J]. 微生物学通报, 2018, 45(8): 1778-1786. |
| [16] |
唐涛涛, 李江, 杨钊, 等. 污泥厌氧消化功能微生物群落结构的研究进展[J]. 化工进展, 2020, 39(1): 320-328. |
| [17] |
HILL V R, KAHLER A M, JOTHIKUMAR N, et al. Multistate evaluation of an ultrafiltration-based procedure for simultaneous recovery of enteric microbes in 100-liter tap water samples[J]. Applied Environmental Microbiology, 2007, 73(13): 4218-4225. |
| [18] |
向菲, 王弘宇, 姜宇. 嗜盐菌在高盐废水处理中的应用研究[J]. 山西建筑, 2012, 38(26): 139-140. |
| [19] |
王春勇, 张震斌, 崔岩山, 等. Cr(Ⅵ)还原菌Microbacterium sp.QH-2对铝氧化物吸附铬影响的研究[J]. 农业环境科学学报, 2021, 40(4): 801-805. |
| [20] |
毛跃建. 废水处理系统中重要功能类群Thauera属种群结构与功能的研究[D]. 上海: 上海交通大学, 2011.
|
| [21] |
LIU B B, ZHANG F, FENG X X, et al. Thauera and Azoarcus as functionally important genera in a denitrifying quinoline-removal bioreactor as revealed by microbial community structure comparison[J]. FEMS Microbiology Ecology, 2006, 55(2): 274-286. |



