2. 广西科学院, 广西海洋天然产物与组合生物合成化学重点实验室, 广西南宁 530007;
3. 南宁学院机电与质量技术工程学院, 广西南宁 530200
2. Guangxi Key Laboratory of Marine Natural Products and Combinatorial Biosynethesis Chemistry, Guangxi Academy of Sciences, Nanning, Guangxi, 530007, China;
3. College of Electromechanic and Quality Engineering, Nanning University, Nanning, Guangxi, 530200, China
海洋是地球上最大的水生生态系统,约占地球总表面积的71%。海洋中海流、波浪、潮汐和风暴潮等海水运动,可引起营养物质大规模的水平/垂直运输[1, 2]。在洋流的驱动下,体积小、扩散能力强的微生物遍布海洋的每个角落,承担着海洋生态系统中养分循环和能量流动[3, 4]。庄康等[5]指出,与陆地微生物相比,海洋微生物在物种类群、生理代谢通路及代谢产物等方面都更具新颖性和多样性。故利用可培养方法来获得海洋微生物资源,开展其生理生化特征与生态功能研究,对物种库与功能基因库的开发和应用有着重要的意义。目前,关于广西北部湾海域中可培养微生物的研究大多集中在海洋沉积物、海洋动植物及红树林生态系统[6, 7],对海水中可培养微生物的研究鲜有报道,究其原因可能是海水中微生物的丰度、潜在新物种数量及其代谢功能均较海洋其他来源的微生物低,未获得广泛关注。本研究选取的3处近岸港湾分别为广西北部湾茅尾海(002站位)、珍珠港(073站位)和北仑河口(067站位)。3处港湾的红树林生态系统生物群落丰富,加上大量的生蚝养殖,丰富了海水中的组成成分,加快了能量和物质循环速度,增强了微生物的生产力和有机质的代谢能力。此外,067站位位于敞开式海域,002站位和073站位属于半封闭内海,使得3处海域中海水的硅酸盐、铵盐及盐度等理化指标上存在较大差异。
本课题组致力于海洋沉积物、海洋动植物及红树林生态系统中可培养微生物的分离鉴定及功能菌株的挖掘[8-11],深知开展微生物次级代谢活性物质的分离、纯化、组成和结构解析等工作需要花费大量的精力、时间和费用,且往往会遇到次级代谢产物率低、重现性差和反复挖掘等问题[12]。此外,从微生物中分离到的化合物种类,远远多于其基因组中次级代谢产物生物合成基因簇的数目,即有为数不少的基因簇在实验室条件下表达量低或不表达,难以在传统研究中被发现[13, 14]。因此,通过次级代谢产物生物合成基因筛选这一种快速发现新型化合物的方法,能高效提升筛选速度。其中,PKS是一种多功能酶复合物,催化如阿维菌素、雷帕霉素、红霉素等聚酮类化合物的合成[15];NRPS则是一种非核糖体肽合成酶,催化如环孢菌素、博来霉素、达托霉素等多肽类化合物的合成[16]。Halo是一种具有修饰作用的酶,通过改变化合物的空间和电子效应,赋予微生物次级代谢产物生物活性,如黄素依赖型卤化酶[17]。本研究选取广西北部湾海域的3个站位,以其表层海水作为研究对象,通过4种分离培养基、分子鉴定和系统发育分析,获取海水中可培养细菌的多样性信息,并利用PKSⅠ型、PKSⅡ型、NRPS和Halo合成基因,对可培养海洋细菌的基因组DNA进行筛选,挖掘潜在合成活性代谢产物的能力,为深入研究表层海水来源的海洋细菌多样性及其功能提供前期基础。
1 材料与方法 1.1 材料 1.1.1 样品2021年8月中旬,采集广西北部湾海域中3处站位的海水样品,取样点见图 1。3个站位分别为茅尾海(002站位)、珍珠港(073站位)和北仑河口(067站位)。对每个站位采集3 L表层海水(0.5 m深度)样品。随船记录水温、盐度、pH值及溶解氧等环境参数,营养盐、化学需氧量(COD)、悬浮物及细菌总数等各指标均待收集水样后带回实验室测定,研究细菌多样性的水样置于50 mL无菌离心管中,4℃低温保藏备用。
|
| 图 1 北部湾海域取样点 Fig.1 Sampling sites in Beibu Gulf |
1.1.2 培养基
(1) 4种分离培养基:2216E固体培养基(青岛海博生物技术有限公司),改良高氏一号固体培养基(GN培养基,可溶性淀粉10.0 g,葡萄糖1.0 g,甘油10 mL,复合盐母液10 mL,琼脂14.0 g,去离子水1 000 mL),P7固体培养基(L-酪氨酸0.5 g,L-天门冬酰胺1.0 g,甘油10 mL,复合盐母液10 mL,琼脂14.0 g,去离子水1 000 mL,pH值7.2-7.4),M5固体培养基(海藻糖5.0 g,脯氨酸1.0 g,琼脂14.0 g,陈海水1 000 mL,pH值7.2-7.4)。复合盐母液配方为KNO3 1.0 g,NaCl 15.0 g,MgSO4·7H2O 0.5 g,K2HPO4 0.5 g,NH4NO3 0.1 g,FeSO4 0.01 g,去离子水10 mL。
(2) 纯化培养基:改良ISP2固体培养基(麦芽提取粉2.0 g,酵母粉2.0 g,葡萄糖2.0 g,去离子水1 000 mL,海盐25.0 g,琼脂14.0 g)。
(3) 含氨苄的LB固体培养基:酵母粉5.0 g,蛋白胨10.0 g,氯化钠5.0 g,氨苄青霉素100 mg,琼脂13.0 g,超纯水1 000 mL,pH值为7.2-7.4。
1.2 方法 1.2.1 海水参数测定作业现场采用多参数水质分析仪(JFE AAQ171,日本)测定水温、盐度、pH值及溶解氧;参照《海洋监测规范》(GB 17378.1-2007),对营养盐(氨氮、硝酸氮、亚硝酸氮、磷酸盐及硅酸盐)、化学需氧量、悬浮物浓度及细菌总数等指标进行测定。
1.2.2 菌株分离纯化用无菌水将样品稀释成10-1、10-2和10-3浓度的样液,取100 μL稀释后的样液涂布至4种固体培养基中,每个梯度两个平行,28℃培养5-7 d,挑取肉眼可见菌落进行纯化培养,记录其形态特征和菌落数,以30%(V∶V)甘油-ISP2混合液作为保护剂,将纯化好的菌株保藏于-80℃。
1.2.3 16S rRNA基因测序和系统发育分析用无菌牙签挑取少量培养好的菌苔,放置于装有无菌chelex-100树脂[18]的管子中进行研磨,100℃加热10 min后,5 000 r/min离心10 min,取上清液(细菌的DNA)为PCR模板,再根据Walsh等[19]的的方法对其进行PCR扩增。扩增和测序引物均为细菌通用引物27F和1492R,PCR反应条件参照李菲等[8]的方法设定。扩增产物经1%琼脂糖凝胶电泳检测合格后,委托生工生物工程(上海)股份有限公司进行测序分析。其中,潜在新物种的PCR扩增产物用Easy Pure Quick Gel Extraction Kit试剂盒进行切胶回收,将纯化好的DNA连接pEASY-T1克隆载体上,转化至Trans-T1感受态细胞中,涂布于含氨苄的LB固体培养基,37℃培养24 h后观察菌落情况,随机挑取5-6个菌落,用PCR法验证克隆的片段大小并送生工生物工程(上海)股份有限公司进行测序。序列经BioEdit Sequence Alignment Editor软件整理后,利用EzBioCloud数据库(https://www.ezbiocloud.net)进行在线比对[20];选取同源性最高的菌株序列作为参比对象,运用MEGA7.0软件,采用Neighbor-Joining法构建系统发育树,Boostrap 1 000次检测各分支的置信值,对各菌株的系统发育地位进行分析[21],并使用EvolView对系统发育树进行美化处理[22]。采用Excel软件计算Simpson、Shannon-Wiener和Pielou生物多样性指数[23],并用SPSS软件分析可培养细菌多样性与海水理化参数的Pearson相关性。
1.2.4 抗生素合成基因的扩增及检测对1.2.2节提取到的细菌DNA进行NRPS、PKS和Halo基因扩增。引物信息见表 1,PCR扩增参数参照文献[24, 25]的方法进行设定。取5 μL扩增产物进行1%琼脂糖凝胶电泳检测,用凝胶成像仪观察结果并拍照。
| 基因 Gene |
引物名称 Primers′s name |
引物序列(5′→3′) Primer′s sequence (5′→3′) |
目的片段长度(bp) Target fragment length (bp) |
| NRPS | A3F | GCSTACSYSATSTACACSTCSGG | 700-800 |
| A7R | SASGTCVCCSGTSCGGTAS | ||
| PKSⅠ | K1F | TSAAGTCSAACATCGGBCA | 1 200-1 500 |
| M6R | CGCAGGTTSCSGTACCAGTA | ||
| PKSⅡ | KSαF | TSGCSTGCTTGGAYGCSATC | 500-750 |
| KSβR | TGGAANCCGCCGAABCCTCT | ||
| Halo | B4-FW | TTCCCSCGSTACCASATCGGSGAG | 500-750 |
| B7-RV | GSGGGATSWMCCAGWACCASCC |
2 结果与分析 2.1 取样站位的海水理化性质
取样站位的环境特征与海水理化性质如表 2所示。3处取样站位海水的营养盐含量、盐度、化学需氧量和悬浮物浓度均存在较大差异,温度、pH值、溶解氧含量等指标差别不大。
| 站位 Station |
经纬度 Longitude and latitude |
硅酸盐(μmol/L) Silicate (μmol/L) |
磷酸盐(μmol/L) Phosphate (μmol/L) |
铵盐(μmol/L) Ammonium salt (μmol/L) |
亚硝酸盐(μmol/L) Nitrite (μmol/L) |
硝酸盐(μmol/L) Nitrate (μmol/L) |
温度(℃) Temperature (℃) |
| 002 | 108°32′53.52″ E 21°47′56.40″ N |
62.19 | 2.04 | 4.73 | 6.76 | 40.35 | 31.3 |
| 067 | 108° 05′ 26.00" E 21° 32′ 43.00" N |
74.58 | 2.12 | 34.65 | 3.56 | 13.02 | 32.5 |
| 073 | 108° 11′ 59.08" E 21° 34′ 11.78" N |
23.54 | 0.40 | 2.93 | 0.72 | 2.67 | 31.4 |
| 站位 Station |
盐度 Salinity |
pH值 pH value |
溶解氧含量(mg/L) Content of dissolved oxygen (mg/L) |
化学需氧量(mg/L) Chemical oxygen demand (mg/L) |
悬浮物浓度(mg/L) Suspended solids concen- tration (mg/L) |
细菌总数(cfu/mL) Total bacterial count (cfu/mL) |
|
| 002 | 15.486 | 7.415 | 5.22 | 2.36 | 1.67 | 115 000 | |
| 067 | 8.844 | 7.530 | 7.10 | 5.43 | 7.37 | 64 000 | |
| 073 | 30.352 | 8.000 | 5.13 | 0.95 | 8.60 | 460 000 | |
2.2 表层海水中可培养细菌
本研究采集了广西北部湾3处近岸海域的表层海水样品,共获得252株海洋细菌,隶属于4门7纲20目25科37属52种(图 2、图 3)。变形菌门(Proteobacteria,172株)、放线菌门(Actinobacteria,43株)、厚壁菌门(Firmicutes,22株)和拟杆菌门(Bacteroidetes,15株)分别占总分离菌株的68.25%、17.06%、8.73%和5.95%[图 2(a)]。在纲水平,γ-变形菌纲(Gammaproteobacteria,114株)细菌数最多,占总菌数的45.24%;其次为α-变形菌纲(Alphaproteobacteria,50株,19.84%)和放线菌纲(Actinomycetia,43株,17.06%)。此外,芽孢杆菌纲(Bacilli)、噬纤维菌纲(Cytophagia)、黄杆菌纲(Flavobacteria)和β-变形菌纲(Betaproteobacteria)分别包含22株、10株、5株和8株细菌[图 2(b)]。在属水平(图 3),假单胞菌属(Pseudomonas)菌株最多,分离出33株(3种);其次是链霉菌属(Streptomyces)、假交替单胞菌属(Pseudoalteromonas)和芽孢杆菌属(Bacillus),分别获得25株(4种)、21株(2种)和19株(4种)。此外,袁其鹏属(Qipengyuania)、噬冷菌属(Algoriphagus)、交替单胞菌属(Alteromonas)和盐单胞菌属(Halomonas)中的菌种数分别为3个、3个、3个和2个,其余29个菌属中均仅分离到1种细菌。根据细菌16S rRNA基因相似度低于98.65%属于不同物种的归类原则[26],本研究共分离培养出10株细菌的潜在新种(表 3),分别属黄杆菌纲、噬纤维菌纲、α-变形菌纲和γ-变形菌纲,其数目分别为4株、1株、2株和3株。
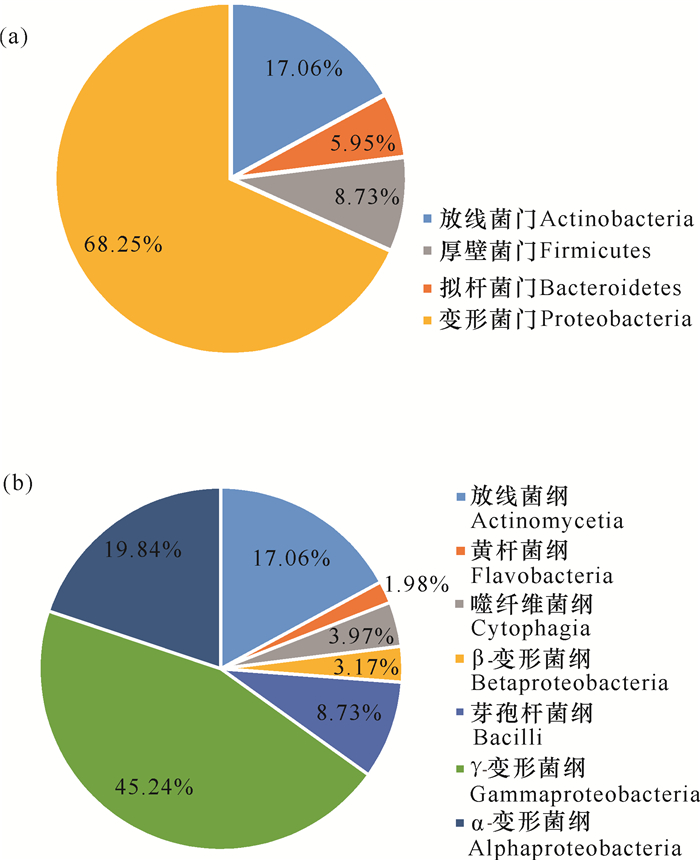
|
| 图 2 表层海水可培养细菌在门(a)和纲(b)水平的组成分布 Fig.2 Composition distribution of cultivable bacteria in surface seawater at phylum (a) and class (b) levels |
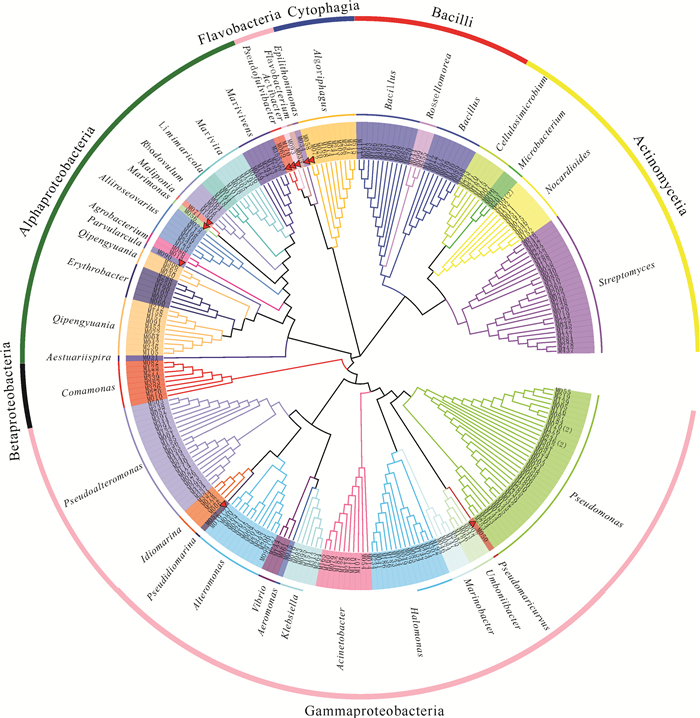
|
| 红色三角符号表示该菌为潜在的新分类单元 The red triangle symbol indicates that the bacterium is a potential new taxonomic unit 图 3 表层水体中海洋细菌的可培养细菌的系统发育树 Fig.3 Neighboring phylogenetic tree of cultivable bacteria from surface seawater based on 16S rRNA sequences |
| 菌株编号 Strain No. |
相似菌株 Similar strains |
相似度(%) Similarity (%) |
分离培养基 Isolation medium |
| M170 | Pseudofulvibacter geojedonensis YCS-9T | 93.17 | 2216E |
| M050 | Pseudomaricurvus alkylphenolicus KU41GT | 94.77 | 2216E |
| M069 | Pseudofulvibacter geojedonensis YCS-9T | 94.87 | GN |
| M056 | Marimonas lutisalis GH1-19T | 96.89 | GN |
| M093 | Actibacter haliotis W113T | 97.24 | P7 |
| M052 | Pseudidiomarina halophile BH195T | 97.32 | 2216E |
| M058 | Algoriphagus ornithinivorans IMSNU 14014T | 97.50 | 2216E |
| M071 | Maliponia aquimaris CECT 8898T | 97.60 | GN |
| M034 | Epilithonimonas mollis DSM 18016T | 98.13 | 2216E |
| M090 | Parvularcula lutaonensis CC-MMS-1T | 98.50 | 2216E |
对不同站位分离的细菌类群比较发现,002站位、067站位和073站位分别获得细菌76株(21属25种)、102株(23属31种)和74株(16属19种)(表 4)。其中,有8个属的细菌仅在002站位中分离得到,11个属的细菌仅在067站位中分离得到,4个属的细菌仅在073站位中分离获得,而有9个属的细菌在这3个站位均能分离得到(图 4)。对3处站位细菌多样性与环境参数进行Pearson相关性分析,结果显示,铵盐和溶解氧含量与细菌总数呈正相关性,存在显著性差异(P < 0.05)。细菌种类与硅酸盐、盐度也存在显著性差异,硅酸盐、磷酸盐和盐度在3处海域的细菌多样性上有显著性差异(表 5),而海水中的亚硝酸盐、硝酸盐、悬浮物及海水的pH值、温度、COD与3处海域的细菌多样性不相关(P>0.05)。
| 菌属 Genus |
002站位 Station 002 |
073站位 Station 073 |
067站位 Station 067 |
2216E | M5 | P7 | GN |
| 海源杆菌属Idiomarina | - | 4 | - | - | 3 | - | 1 |
| 假海源菌属Pseudidiomarina | - | 1 | - | 1 | - | - | - |
| 交替单胞菌属Alteromonas | - | 5 | 7 | 4 | 8 | - | - |
| 假交替单胞菌属Pseudoalteromonas | 10 | 2 | 9 | - | 9 | 4 | 8 |
| 气单胞菌属Aeromanas | 1 | - | - | - | - | 1 | - |
| 克雷伯氏菌属Klebsiella | 1 | 2 | 3 | 5 | - | - | 1 |
| 弧菌属Vibrio | - | - | 3 | - | - | 2 | 1 |
| Pseudomaricurvus | - | - | 1 | 1 | - | - | - |
| 伞形杆菌属Umboniibacter | - | - | 4 | 2 | 1 | 1 | - |
| 不动杆菌属Acinetobacter | 6 | 2 | 2 | 5 | - | - | 5 |
| 假单胞菌属Pseudomonas | 3 | 14 | 16 | 6 | 7 | 6 | 14 |
| 盐单胞菌属Halomonas | 8 | 4 | 2 | 3 | 6 | 2 | 3 |
| 海杆状菌属Marinobacter | - | - | 4 | - | 1 | 3 | - |
| 丛毛单胞菌属Comamonas | - | 8 | - | 6 | 2 | - | - |
| 海滨杆菌属Actibacter | - | - | 1 | - | - | 1 | - |
| 假黄棕杆菌属Pseudofulvibacter | 2 | - | - | 1 | 1 | - | - |
| 黄杆菌属Flavobacterium | - | - | 1 | 1 | - | - | - |
| Epilithonimonas | - | - | 1 | 1 | - | - | - |
| 嗜冷菌属Algoriphagus | 1 | - | 9 | 4 | 6 | - | - |
| 芽孢杆菌属Bacillus | 5 | 7 | 7 | 6 | 7 | 4 | 2 |
| Rossellomorea | 3 | - | - | 2 | 1 | - | - |
| 类诺卡氏菌属Nocardioides | 5 | 4 | - | 4 | - | 5 | - |
| 链霉菌属Streptomyces | 3 | 7 | 15 | 12 | 6 | - | 7 |
| 纤维菌属Cellulosimicrobium | - | 6 | - | - | 4 | 2 | - |
| 微杆菌属Microbacterium | 1 | 1 | 1 | - | - | - | 3 |
| 红杆菌属Erythrobacter | 3 | - | 3 | 3 | - | - | 3 |
| 袁其鹏属Qipengyuania | 3 | 5 | 5 | 4 | 4 | 5 | - |
| 滩螺菌属Aestuariispira | - | - | 1 | 1 | - | - | - |
| 短小盒菌属Parvularcula | - | - | 1 | 1 | - | - | - |
| 土壤杆菌属Agrobacterium | - | - | 2 | 2 | - | - | - |
| 小红卵菌属Rhodovulum | - | - | 4 | 1 | - | - | 3 |
| 玫瑰变色菌属Aliiroseovarius | 6 | - | - | 2 | 2 | 2 | - |
| Marivita | 4 | 2 | - | 1 | 5 | - | - |
| Limimaricola | 3 | - | - | 2 | - | 1 | - |
| Marivivens | 6 | - | - | 2 | 3 | - | 1 |
| Maliponia | 1 | - | - | - | 1 | - | - |
| Marimonas | 1 | - | - | - | 1 | - | - |
| 注:“-”表示菌种数为0 Note: "-" indicates that the number of strains is zero |
|||||||
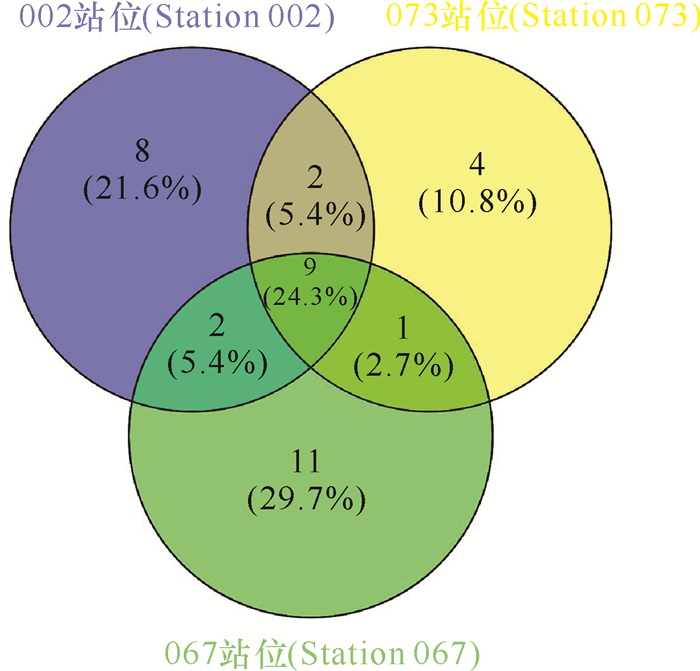
|
| 图 4 3处站位来源海洋细菌属级群类的维恩分析 Fig.4 Venn analysis of marine bacterial genera from three stations |
| 项目Item | r/P | 硅酸盐 Silicate |
磷酸盐 Phosphate |
铵盐 Ammonium salt |
盐度 Salinity |
溶解氧含量 Content of dissolved oxygen |
| 菌株数 Bacterial number |
r | 0.733 | 0.588 | 1.000 | -0.780 | 1.000 |
| P | 0.476 | 0.600 | 0.009 | 0.431 | 0.015 | |
| 菌属数 Genus′s number |
r | 0.999 | 0.971 | 0.755 | -1.000 | 0.748 |
| P | 0.029 | 0.153 | 0.456 | 0.016 | 0.462 | |
| Shannon-Wiener指数 Shannon-Wiener index |
r | 0.994 | 0.955 | 0.794 | -0.999 | 0.788 |
| P | 0.049 | 0.192 | 0.416 | 0.023 | 0.423 | |
| Simpon指数 Simpon index |
r | 0.995 | 0.996 | 0.651 | -0.985 | 0.643 |
| P | 0.044 | 0.049 | 0.549 | 0.109 | 0.555 | |
| Pielou指数 Pielou index |
r | 0.855 | 0.939 | 0.261 | -0.816 | 0.251 |
| P | 0.347 | 0.224 | 0.832 | 0.392 | 0.838 | |
| 注:r为Pearson相关性,P为显著性(双侧)。加粗表示差异性显著(P < 0.05) Note: r and P represent Pearson correlation and significance (bilateral) respectively. Bold font indicates significant difference (P < 0.05) |
||||||
2.3 培养基的分离效果
采用4种分离培养基对表层海水中海洋细菌进行培养,分离到细菌多样性情况如表 6所示。就多样性指数可知,2216E培养基的Simpon指数、Shannon-Wiener指数和Pielou指数均高于M5、P7和GN培养基。在纲水平,α-变形菌纲、γ-变形菌纲、放线菌纲和芽孢杆菌纲的细菌在4种培养基均能分离获得;2216E和M5培养基中均分离到β-变形菌纲、黄杆菌纲和噬纤维菌纲的细菌,P7培养基中分离到1株黄杆菌纲的细菌,GN则未分离到3个纲的细菌。在属水平(表 4),2216E、M5、P7和GN培养基均获得盐单胞菌属(Halomonas)、芽孢杆菌属(Bacillus)和假单胞菌属(Pseudomonas)的细菌,其中,优势类群假单胞菌属在GN培养基中菌株数目最多;作为本次分离培养的第二大优势类群,链霉菌属(Streptomyces)在2216E培养基中菌株数目最多,但在P7培养基中未分离到。此外,分别有7个、2个、2个与1个属的细菌仅在2216E、M5、P7及GN培养基中被培养。其中,2216E培养基中分离出6株细菌的潜在新种,GN和P7培养基中各培养出3株和1株(表 3)。
| 项目Item | 2216E | M5 | P7 | GN |
| 菌种数 Species′s number |
37 | 27 | 16 | 18 |
| 菌属数 Genus′s number |
27 | 20 | 14 | 13 |
| 菌株数 Bacterial number |
83 | 78 | 39 | 52 |
| Simpon指数 Simpon index |
0.965 | 0.951 | 0.917 | 0.929 |
| Shannon-Wiener指数 Shannon-Wiener index |
3.404 | 3.114 | 2.502 | 2.728 |
| Pielou指数 Pielou index |
0.770 | 0.715 | 0.683 | 0.690 |
2.4 抗生素合成基因的检测结果
为了进一步评价52种细菌是否存在次级代谢产物生物合成的潜能,对其进行PKS、NRPS和Halo基因的扩增。扩增和电泳检测结果(表 7)显示,22种细菌均至少扩增出1条目的条带,即其含有1种次级代谢产物合成基因。其中,PKSⅡ基因在15株细菌中检测到;Halo基因在13株细菌中检测到,3株细菌含PKSⅠ基因和2株细菌检测到NRPS基因,有10株细菌含2种或2种以上的生物合成基因簇。
| 供试菌株 Tested strains |
相似菌名 Name of similar bacteria |
抗生素生物合成基因 Antibiotic biosynthetic gene |
|||
| PKSⅠ | PKSⅡ | NRPS | Halo | ||
| M018 | Agrobacterium larrymoorei ATCC 51759T | - | + | - | - |
| M072 | Algoriphagus marincola DSM 16067T | - | + | - | - |
| M020 | Algoriphagus sanaruensis M8-2T | - | + | - | + |
| M010 | Comamonas aquatica NBRC 14918T | - | - | - | + |
| M073 | Erythrobacter tepidarius DSM 10594T | - | + | - | + |
| M041 | Halomonas sulfidaeris ATCC BAA-803T | - | - | - | + |
| M006 | Idiomarina aquatic SN-14T | - | - | + | - |
| M019 | Klebsiella variicola subsp.Tropica SB5531T | + | + | - | - |
| M071 | Maliponia aquimaris CECT 8898T | - | + | - | + |
| M090 | Parvularcula lutaonensis CC-MMS-1T | - | + | - | + |
| M170 | Pseudofulvibacter geojedonensis YCS-9T | - | - | - | + |
| M050 | Pseudomaricurvus alkylphenolicus KU41G T | - | - | - | + |
| M038 | Pseudomonas balearica DSM 6083T | - | + | - | + |
| M008 | Pseudomonas oryzae KCTC 32247T | - | + | + | - |
| M024 | Pseudomonas oryzihabitans NBRC 102199T | - | + | - | + |
| M030 | Qipengyuania citrea RE35F/1T | + | + | - | + |
| M022 | Qipengyuania flava SW-46T | - | + | - | - |
| M091 | Qipengyuania nanhaisediminis CGMCC 1.7715T | - | + | - | + |
| M045 | Streptomyces heilongjiangensis NEAU-W2T | - | - | - | + |
| M016 | Streptomyces koyangensis VK-A60T | + | - | - | - |
| M023 | Streptomyces violaceorectus NBRC 13102T | - | + | - | - |
| M061 | Vibrio astriarenae JCM 19233T | - | + | - | - |
| 注:字体加粗表示潜在新物种; “+”表示菌株中含有抗生素合成基因,“-”表示菌株中未检测到抗生素合成基因 Note: Bold font indicates the potential new taxon; "+"indicates the strains contain antibiotic synthetic genes, "-" indicates antibiotic synthetic genes are not detected |
|||||
3 讨论
从总体上看,该研究区域中海水可培养优势细菌类群与大多数前人研究报道[22, 27]较为一致,主要由γ-变形菌、α-变形菌、拟杆菌和放线菌等优势菌群组成。其中,γ-变形菌类群相对丰度最高(114株,45.24%),同时检测到海水中硝酸盐、磷酸盐、铵盐等无机盐浓度较高,两者是否存在关联,仍需要进一步去考证。此外,3处海域海水中的细菌群落组成存在明显差异。其中,细菌总数排序为珍珠港(073站位) < 茅尾海(002站位) < 北仑河口(067站位),细菌种类排序为北仑河口(067站位) >茅尾海(002站位)>珍珠港(073站位)。珍珠港(073站位)内潮流畅通、水质清澈, 随着养殖珍珠贝的数量减少,钢铁、火电和纸浆等工业快速发展,环境污染骤增,尤其是重金属类污染[28]。作为相同半封闭内海, 茅尾海(002站位)是广西沿海最大的养殖区,由于受到周边江水汇集和经济发展的影响,海水中大量的营养物质聚集,重金属污染加剧[29]。研究指出,重金属污染物可迅速地与悬浮物和沉积物结合而沉入海底,海水中重金属含量大大降低[30]。此外,海水的重金属含量与细菌数量的分布呈不同程度的相关性,一定浓度的重金属含量可对细菌的生长起到促进作用[30]。相比之下,敞开式浅水港湾北仑河口具有连片生长红树林,动植物种类组成和群落结构随海岸类型变化而呈多样化等特征,加速了海水运动,提升了营养物质反复置速率,使得该区域表层海水盐度低,铵盐、硅酸盐和溶解氧含量高,更利于多种微生物生存。此外,海水运动驱使下,表层和底层菌群趋于平衡,进而丰富细菌类群组成。
本研究采用4种营养类型与浓度差异较大的培养基,分别对北部湾3处海域表层海水进行细菌的分离纯化培养。结果表明,2216E培养基分离到的细菌种类和数量最多,其次是M5培养基,P7和GN培养基分离到的最少。其中,GN培养基主要以淀粉、葡萄糖和甘油等碳源为主,常用于分离培养放线菌及观察其形态特征;GN培养基中分离到的两大放线菌类群链霉菌和微杆菌,在广西北部湾已发现的海洋细菌类群中占最高比例,在土壤、水体和植物内都占据绝对优势[7, 9];GN培养基中未筛选到拟杆菌门类群,大多数拟杆菌可产生多种功能的胞外酶[31],而酶的合成与分泌需要大量的营养物质,尤其是氮源[32],故推测该类群难以在单一营养源的GN培养基中生长。相比较,2216E培养基更适合分离培养海水中的细菌,获得潜在新分类单元的菌株数高于其他培养基。因此,在分离海水中可培养细菌时,可通过营养类型多元化来培养更多的细菌资源。
在52种细菌中,有22种细菌检测出抗生素生物合成基因,总阳性率为42.31%。相对于红树植物和沉积物来源细菌,本研究海水来源细菌中检测到的抗生素生物合成基因率偏低,其可能原因是红树林生境中有机质较其他海洋区域的丰富[33],富含动植物残体的脂素、蛋白和纤维素等营养物质,促使其菌落组成多样化,可加快能量和物质循环速度,增强生产力和有机质的降解活性[34]。
4 结论广西北部湾海域表层海水中具有丰富的可培养细菌,具有较高的物种多样性,蕴藏着潜在新物种资源,且具有合成聚酮类、非核糖体肽类化合物及卤化酶的潜能,为后续开展拮抗菌筛选工作及其活性化合物的研究提供理论基础依据。
| [1] |
QU T D, GIRTON J B, WHITEHEAD J A. Deepwater overflow through Luzon Strait[J]. Journal of Geophysical Research, 2006, 111: C01002. |
| [2] |
WANG X W, LIU Z Y, PENG S Q. Impact of tidal mixing on water mass transformation and circulation in the South China Sea[J]. Journal of Physical Oceanography, 2017, 47: 419-432. DOI:10.1175/JPO-D-16-0171.1 |
| [3] |
SALAZAR G, CORNEJO-CASTILLO F M, BENITEZ-BARRIOS V, et al. Global diversity and biogeography of deep-sea pelagic prokaryotes[J]. The ISME Journal, 2016, 10: 596-608. DOI:10.1038/ismej.2015.137 |
| [4] |
LIU J W, YANG H M, ZHAO M X, et al. Spatial distribution patterns of benthic microbial communities along the Pearl Estuary, China[J]. Systematic and Applied Microbiology, 2014, 37: 578-589. DOI:10.1016/j.syapm.2014.10.005 |
| [5] |
庄康, 胡晓娟, 曹煜成, 等. 不同寡营养培养条件下南海水体细菌群落结构及其对碳源的利用特征[J]. 微生物学通报, 2020, 47(9): 2697-2710. |
| [6] |
李菲, 李喆, 胡文进, 等. 广西茅尾海红树林土壤放线菌多样性及功能酶活性研究[J]. 海洋学报, 2021, 43(2): 88-97. |
| [7] |
徐新亚, 杨宏, 宁小清, 等. 北部湾海洋微生物物种多样性与化学多样性研究进展[J]. 广西科学, 2020, 27(5): 433-450, 461. |
| [8] |
李菲, 高程海, 余炼, 等. 川蔓藻内生及根际细菌多样性与抑菌活性研究[J]. 广西植物, 2018, 38(7): 924-933. |
| [9] |
李菲, 李喆, 黄媛林, 等. 角果木树皮来源放线菌多样性及生物活性初探[J]. 中国抗生素杂志, 2021, 46(9): 845-853. DOI:10.3969/j.issn.1001-8689.2021.09.005 |
| [10] |
李菲, 黄庶识, 胡文进, 等. 半红树植物海芒果内生与根际细菌多样性及抗农用真菌活性研究[J]. 中国抗生素杂志, 2021, 46(5): 396-405. DOI:10.3969/j.issn.1001-8689.2021.05.006 |
| [11] |
黄媛林, 潘信利, 陆璐, 等. 一株红树林链霉菌所产抑菌活性化合物的分离及其生物合成基因簇的研究[J]. 中国抗生素杂志, 2021, 46(12): 1090-1101. DOI:10.3969/j.issn.1001-8689.2021.12.004 |
| [12] |
郑洁, 庹利, 李伟, 等. 江苏盐城沿海滩涂湿地放线菌多样性及功能基因的筛选[J]. 遵义医学院学报, 2019, 42(3): 282-290. DOI:10.3969/j.issn.1000-2715.2019.03.008 |
| [13] |
ZAZOPOULOS E, HUANG K, STAFFA A, et al. A genomics-guided approach for discovering and expressing cryptic metabolic pathways[J]. Nature Biotechnology, 2003, 21: 187-190. DOI:10.1038/nbt784 |
| [14] |
侯路宽, 李花月, 李文利. 隐性次级代谢产物生物合成基因簇的激活及天然产物定向发现[J]. 微生物学报, 2017, 57(11): 1722-1734. |
| [15] |
马敏, 唐敏, 洪葵. 四种红树植物根际土壤微生物Ⅰ型和Ⅱ型PKS基因的检测与多样性分析[J]. 微生物学通报, 2013, 40(7): 1231-1240. |
| [16] |
李红玲. 非核糖体肽合成酶结构研究进展[J]. 临床合理用药, 2013, 6(10上): 180-181. |
| [17] |
高鹏, 郗丽君, 朴玉华, 等. 不同生境放线菌的卤化酶基因分析及其对卤代产物筛选的意义[J]. 微生物学报, 2009, 49(10): 1367-1373. DOI:10.3321/j.issn:0001-6209.2009.10.008 |
| [18] |
周双清, 黄小龙, 黄东益, 等. Chelex-100快速提取放线菌DNA作为PCR扩增模板[J]. 生物技术通报, 2010(2): 123-125. |
| [19] |
WALSH P S, METZGER D A, HIGUCHI R. Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material[J]. Biotechniques, 1991, 10(4): 506-513. |
| [20] |
KIM K H, ROH S W, CHANG H W, et al. Nocardioides basaltis sp.nov.isolated from black beach sand[J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59: 42-47. DOI:10.1099/ijs.0.65785-0 |
| [21] |
KUMAR S, STECHER G, TAMURA K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [22] |
夏呈强, 李毅, 党延茹, 等. 中印度洋与南海西部表层海水细菌多样性[J]. 生物多样性, 2022, 30(1): 91-100. |
| [23] |
李菲, 李喆, 覃仙玲, 等. 山口红树林土壤芽孢杆菌多样性及纤维素酶活性筛选研究[J]. 广西科学, 2021, 28(5): 473-481. |
| [24] |
王海强, 安向向, 侯淑芬, 等. 39株内生放线菌次级代谢产物的合成潜能[J]. 河北大学学报(自然科学版), 2017, 37(3): 254-261. DOI:10.3969/j.issn.1000-1565.2017.03.006 |
| [25] |
王玉梅. 海洋链霉菌卤化酶基因筛选阳性菌株的活性物质研究[D]. 大连: 大连理工大学, 2014.
|
| [26] |
KIM M, OH H S, PARK S C, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64: 346-351. DOI:10.1099/ijs.0.059774-0 |
| [27] |
POMMIER T, CANBÄCK B, RIEMANN L, et al. Glo-bal patterns of diversity and community structure in marine bacterioplankton[J]. Molecular Ecology, 2007, 16: 867-880. |
| [28] |
吴祥庆, 黎小正, 兰柳春, 等. 广西防城珍珠港珍珠养殖区表层沉积物重金属污染评价[J]. 海洋通报, 2010, 29(5): 584-587. DOI:10.3969/j.issn.1001-6392.2010.05.019 |
| [29] |
雷富, 陈宪云, 许铭本, 等. 广西茅尾海海水和表层物中重金属污染的调查及评价[J]. 广西科学院学报, 2013, 29(3): 176-180, 185. DOI:10.3969/j.issn.1002-7378.2013.03.007 |
| [30] |
杨柳凤, 刘云霞, 李素霞, 等. 茅尾海细菌分布与重金属含量相关性分析[J]. 环境保护科学, 2018, 44(4): 109-113. |
| [31] |
QIN Q L, ZHANG X Y, WANG X M, et al. The complete genome of Zunongwangia profunda SM-A87 reveals its adaptation to the deep-sea environment and ecological role in sedimentary organic nitrogen degradation[J]. BMC Genomics, 2010, 11: 247. DOI:10.1186/1471-2164-11-247 |
| [32] |
FRANKENA J, VERSEVELD H W V, STOUTHAMER A H. Substrate and energy costs of the production of exocellular enzymes by Bacillus licheniformis[J]. Biotechnology and Bioengineering, 1988, 32: 803-812. DOI:10.1002/bit.260320612 |
| [33] |
孟昊, 薛智权, 唐杰, 等. 深圳福田红树林土壤可培养微生物和土壤酶活性研究[J]. 广东农业科学, 2013(1): 53-56. DOI:10.3969/j.issn.1004-874X.2013.01.019 |
| [34] |
赵雅慧, 张舒琳, 吴家法, 等. 山口红树林根际土壤可培养细菌多样性及其活性筛选[J]. 海洋学报, 2018, 40(8): 138-151. DOI:10.3969/j.issn.0253-4193.2018.08.014 |



