红树林是生长在热带和亚热带陆海交界处潮间带的木本植物群落,是陆地向海洋过渡的特殊生态系统,是维持近岸生物多样性和生态安全的关键屏障[1]。我国现有红树林面积相对于1973年减少41%[2],调查评估发现红树林减少的原因95%是由于人类活动的影响[3]。其中,沿海城市的填海造地、港口建设、围垦养殖等海岸工程是造成滨海湿地丧失和红树林生态系统退化的重要原因之一,也是最直接的原因[4]。国际学者主要关注海岸工程对湿地生物多样性及生态过程的影响,侧重于探索海岸工程引发的生态环境效应[5]。国内学者过去多局限于通过估算湿地面积、评估湿地功能退化等方法研究海岸工程对滨海湿地的影响[6],近年来海岸工程与湿地功能丧失之间的关系研究也逐渐受到关注,如有文献报道渤海湾、胶州湾、珠江口等地的围填海对近岸底栖生物造成不同程度的干扰,大型底栖动物、鱼类的丰度和多样性都有所下降[7-10]。不同生物的生理习性存在差异,对生态环境扰动的响应不同,使得量化海岸工程对生物资源的影响十分困难,特别是在红树林生态系统,国内外专门针对海岸工程影响红树林生物多样性和生物量的基础研究还较少。海岸工程疏浚和吹填的过程中产生大量悬浮泥沙,掩埋了红树林的呼吸根,导致红树死亡;泥沙导致水体混浊、溶解氧浓度降低,损害鱼类鳃部的滤水和呼吸功能从而导致鱼类死亡[10, 11];悬浮物造成水体透明度的下降,影响浮游植物的光合作用,进而影响以浮游生物为食的大型底栖动物的正常生长;另外,许多大型底栖动物幼体营浮游生活,悬浮物堵塞幼体的鳃部致其死亡。红树林是众多海洋动物的繁殖场、幼体栖息地,底栖动物幼体的死亡将影响红树林生物多样性的可持续发展[12]。
环境DNA不仅包含实时的物种信息,而且还包含过去数日内的物种信息[13]。因此环境DNA是一个极有价值的信息载体,目前已被应用于生物多样性监测[14, 15]、特定物种生物量估测、珍稀物种发现、入侵物种监控[16]、隐存种发现、群落系统发育重建、物种间食物链关系分析等领域[17-19]。相比传统的直接采样,环境DNA分析技术有着无创、高效、灵敏、经济等特点。红树林作为一个关注相对较少的生态系统,环境DNA的技术应用较少,广西红树林研究中心在国际国内率先应用环境DNA技术开展红树林的大型底栖动物多样性和物种生物量的研究,并在本论文中呈现初步应用的结果。本研究利用环境DNA技术结合高通量测序及荧光定量PCR,分析海岸工程影响下死亡红树林、严重退化红树林、尚存活红树林以及对照组红树林4种样地底栖动物多样性及优势物种生物量空间差异,旨在评估环境DNA技术在红树林底栖生物多样性和生物量研究中的可行性,并揭示海岸工程对红树林底栖生物资源的影响,为评估海岸工程的生态效应与红树林湿地生物资源保护提供理论数据及科学参考。
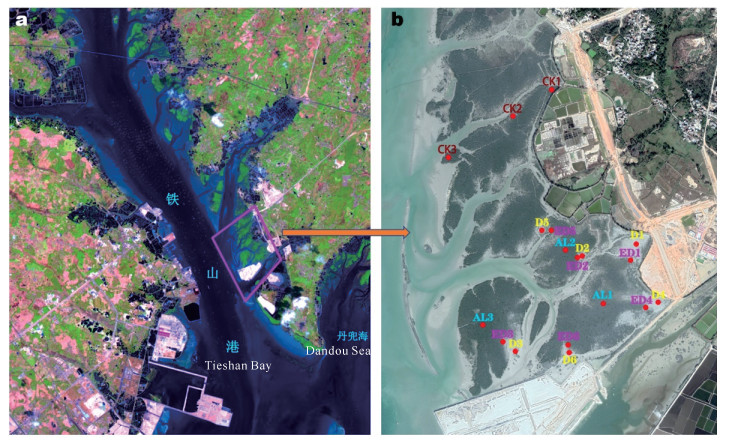
1 材料与方法 1.1 材料根据无人机航拍和实地勘察的海岸工程周边区域情况,设置采样点如图 1所示,植被覆盖度为0的死亡红树林样地(以下简称D样地) 6个(图 1中D1-D6),植被覆盖度≤30%的严重退化红树林样地(以下简称ED样地) 6个(图 1中ED1-ED6),尚存活红树林样地3个(以下简称AL样地) 3个(图 1中AL1-AL3),红树林对照区样地(以下简称CK样地) 3个(图 1中CK1-CK3),每个样地中设置10 m×10 m的采样样方,采样点经纬度见表 1。2020年3月6日及7日在上述18个采样样方中各采集3份沉积物样品,每份样品为采集于10棵树下1-5 cm表层的混合沉积物,约50 g左右,低温保存带回实验室后冻于-20℃冰箱。同时,按照《海洋监测规范》GB 17378.7-2007和《海洋调查规范》GB/T 12763.6-2007,进行大型底栖动物的采集:每个样地设置4个小样方,采用25 cm×25 cm定量样方框,深度0-30 cm,采集沉积物,过筛后带回实验室进行大型底栖动物的判读和生物量、优势物种的计算。该方法在后述中简称为传统“样方法”。
|
| 图 1 样品采集地 Fig.1 Location of sample collection |
|
样方 Quadrats |
编号 No. |
经度 Longitude (E) |
纬度 Latitude (N) |
| 死亡红树林1 Dead mangrove 1 |
D1 | 109°36′47.84319″ | 21°36′3.48874″ |
| 死亡红树林2 Dead mangrove 2 |
D2 | 109°36′33.99656″ | 21°35′59.25944″ |
| 死亡红树林3 Dead mangrove 3 |
D3 | 109°36′20.05336″ | 21°35′39.75441″ |
| 死亡红树林4 Dead mangrove 4 |
D4 | 109°36′50.99103″ | 21°35′50.41458″ |
| 死亡红树林5 Dead mangrove 5 |
D5 | 109°36′25.17102″ | 21°36′4.57021″ |
| 死亡红树林6 Dead mangrove 6 |
D6 | 109°36′32.02674″ | 21°35′39.44059″ |
| 严重退化红树林1 Severely degraded mangrove 1 |
ED1 | 109°36′45.52577″ | 21°35′58.78147″ |
| 严重退化红树林2 Severely degraded mangrove 2 |
ED2 | 109°36′33.12752″ | 21°35′59.16771″ |
| 严重退化红树林3 Severely degraded mangrove 3 |
ED3 | 109°36′17.37867″ | 21°35′41.02417″ |
| 严重退化红树林4 Severely degraded mangrove 4 |
ED4 | 109°36′48.86673″ | 21°35′49.79178″ |
| 严重退化红树林5 Severely degraded mangrove 5 |
ED5 | 109°36′26.72563″ | 21°36′4.58469″ |
| 严重退化红树林6 Severely degraded mangrove 6 |
ED6 | 109°36′31.74576″ | 21°35′40.33335″ |
| 尚存活红树林1 Surviving mangrove 1 |
AL1 | 109°36′39.28802″ | 21°35′50.08628″ |
| 尚存活红树林2 Surviving mangrove 2 |
AL2 | 109°36′30.13417″ | 21°36′0.05123″ |
| 尚存活红树林3 Surviving mangrove 3 |
AL3 | 109°36′13.31351″ | 21°35′44.25409″ |
| 对照红树林1 Control mangrove 1 |
CK1 | 109°36′25.83486″ | 21°36′34.16571″ |
| 对照红树林2 Control mangrove 2 |
CK2 | 109°36′17.28452″ | 21°36′28.30937″ |
| 对照红树林3 Control mangrove 3 |
CK3 | 109°36′3.32201″ | 21°36′19.03965″
|
1.2 方法 1.2.1 环境DNA提取
每份样品的沉积物用搅拌机混匀后冻干,称取0.3 g,使用DNeasy Power soil试剂盒(Qiangen公司)提取沉积物环境总DNA,提取方法参照产品说明书。使用Nanodrop One (Thermo Scientific公司)测定总DNA的浓度,分析A260/A280、A260/230数值,并电泳检验DNA质量。每份样品设置3个生物学重复。
1.2.2 优势物种选取将所采集的定量样品及时用75%酒精固定,待样品完全固定后,进行样品鉴定和称重,并计算大型底栖动物的栖息密度、样方平均生物量,根据生物量的排序选择本项工作中的优势物种。
1.2.3 荧光定量PCR特异性引物设计在Genbank中搜索优势物种的DNA条形码序列,使用Primer3Plus设计优势物种及内参的荧光定量PCR引物,经验证筛选后,得到本论文所用的引物。
1.2.4 相对生物量分析将每个样地提取的3份沉积物DNA混合,对优势物种及内参进行荧光定量PCR实验。实验使用RR42LR试剂盒(TAKARA公司),实验体系为10 μL SYBR ExTaq mix,2 μL DNA,0.4 μL正向引物,0.4 μL反向引物,7.2 μL超纯水,在Bio-Rad荧光定量PCR仪上进行扩增,运行程序如下:热启动95℃,30 s;循环扩增程序95℃,5 s,60℃,30 s,循环40次;熔解曲线程序。
1.2.5 数据处理和作图荧光定量PCR数据处理采用2-ΔΔCt法,本报告中数据处理和作图使用Excel和GraphPad Prism 7.0软件,数据统计采用单因素t检验。
1.2.6 高通量测序为更好地进行数据的统计分析,本项实验随机选取CK,AL,ED,D样地各3个样方,即12个样方进行高通量测序并研究调查区域生物多样性。每个样方取3份沉积物样本分别按1.2.1方法提取沉积物总环境DNA后等量混合,使用针对线粒体细胞色素氧化酶Ⅰ (COI)的通用引物mlCOlintF:5′-GGWACWGGWTGAACWGTWTAYCCYCC-3′和jgHCO2198:5′-TAIACYTCIGGRTGICCRAARA- AYCA-3′[20],进行PCR扩增。PCR扩增使用ABI 2720型PCR仪、TransStart Fastpfu DNA Polymerase (全式金公司)、20 μL反应体系,以尽可能使用低循环数扩增和保证每个样品扩增的循环数统一为原则,根据预实验的结果,选择35个循环进行PCR扩增。PCR产物切胶回收后经2%琼脂糖凝胶电泳检测后,送上海凌恩生物公司进行Illumina PE250文库构建和测序。
1.2.7 生物信息分析根据overlap关系,对Illumina PE250测序得到的PE reads进行拼接,同时对序列质量进行质控和过滤。区分样本后进行可操纵分类单元(Operational Taxonomic Units,以下简称OTU)聚类分析和物种分类学分析,使用Usearch (v10,http://drive5.com/uparse/)进行OTU的生物信息统计分析。采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,并分别在各个分类水平:domain(域)、kingdom(界)、phylum(门)、class(纲)、order(目)、family(科)、genus(属)、species(种)上统计各样本的群落组成。通过Excel绘制不同分类水平下的群落结构组份图,并基于bray-curtis算法分析样本间群落组成的层次聚类。
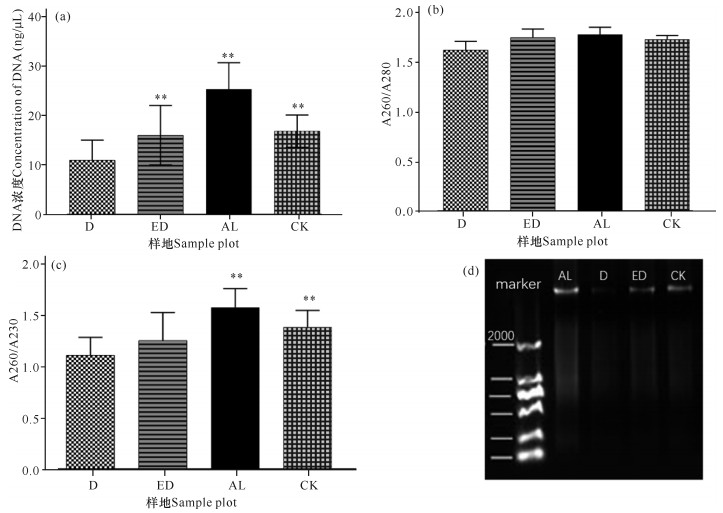
2 结果与分析 2.1 环境DNA提取提取18个样方,每个样方3个样本的沉积物总DNA后,对AL、ED、D和CK样地的DNA浓度、A260/A280和A260/A230数据进行统计分析。如图 2a所示,相同重量的冻干沉积物所提取的DNA浓度由低到高为D<ED<CK<AL样地,D样地沉积物提取的总DNA浓度最低,与其他3种样地的DNA浓度均存在显著性差异;D样地沉积物的总DNA浓度相对于AL样地下降56.68%。4种样地沉积物提取的总DNA的A260/A280均接近1.8,其中D样地略低,为1.66±0.09 (图 2b)。A260/A230的比值大小为D<ED<CK<AL样地,AL、CK样地的值与D样地有显著性差异(图 2c)。A260/A280和A260/A230的数值反应样品中是否存在碳水化合物、盐等,尤其是蛋白质的存在会大大降低A260/A230的比值,D样地提取环境总DNA的质量不高,分析认为与D样地中的高岭土含量较高有关(详见本刊李斌《高岭土悬浮物对榄根村红树林湿地底质环境的影响》)。图 2d展示了随机选择的4种样地沉积物样品提取的DNA电泳检验结果。
|
| **P<0.01表示差异极显著 **P<0.01 indicates extremely significant difference 图 2 沉积物提取的总环境DNA浓度及质量 Fig.2 Concentration and quality of total environmental DNA extracted from sediments |
2.2 优势物种选取
根据传统“样方法”的大型底栖动物调查结果,将该区域所有样方物种平均生物量进行排序。表 2所示为生物量排名前十的优势物种,其中6种为软体动物,4种为甲壳动物,这10种物种生物量占总生物量的比例达到92.34%(该部分结果详见本刊高霆炜等《海岸工程对北海铁山港红树林大底栖动物群落的影响》)。因为并非所有表中物种都能查到DNA barcode的信息,所以根据Genbank中DNA barcode序列的情况,选取扁平拟闭口蟹(16S rRNA,genbank No.: AB002128.1)、明秀大眼蟹(16S rRNA,genbank No.: LC097096.1)、珠带拟蟹守螺(18S rRNA,genbank No.: AM932846.1)和红树蚬(COI,genbank No.: MN849878)作为本实验的检测对象(4个物种在表 2中用加粗标记)。
|
物种名 Species |
生物量 Biomass (g/m 2) |
百分比 Percent (%) |
| 红树蚬 Gelonia erosa |
48.84 | 32.57 |
| 疏纹满月蛤 Lucina scarlatoi |
32.71 | 21.81 |
| 珠带拟蟹守螺 Cerithidea cingulata |
25.40 | 16.94 |
| 明秀大眼蟹 Macrophthalmus definitus |
10.02 | 6.68 |
| 古明圆蛤 Cycladicama cumingii |
5.28 | 3.52 |
| 团聚牡蛎 Saccostrea glomerata |
5.20 | 3.47 |
| 绒毛大眼蟹 Macrophthalmus tomentosus |
4.93 | 3.29 |
|
扁平拟闭口蟹 Paracleistostoma depressum |
2.43 | 1.62 |
| 青蛤 Cyclina sinensis |
2.01 | 1.34 |
| 优美鼓虾 Alpheus euphrosyne |
1.64 | 1.09 |
| 合计 Summation |
138.48 | 92.34 |
2.3 荧光定量PCR
经引物筛选,使用细菌16S rRNA和真菌ITS作为内参,检测上述4种优势物种相对生物量所用的引物如表 3所示。
|
物种 Species name |
引物序列 Primer sequences (5′→3′) |
|
内参1 Reference gene 1 |
AGAGTTTGATCCTGGCTCAG |
| GGTTACCTTGTTACGACTT | |
|
内参2 Reference gene 2 |
CTTGGTC ATTTAGAGGAAGTAA |
| GCTGCGTTCTT CATCGATGC | |
|
扁平拟闭口蟹 Paracleistostoma depressum |
AGTATGGGAGGGATTAGCTTT-
TCA |
| ATGCTACCTTTGCACGGTCA | |
|
明秀大眼蟹 Macrophthalmus definitus |
AGAGTTTGCGACCTCGATGT |
| CACGCCGGTTTGAACTCAAA |
|
|
珠带拟蟹守螺 Cerithidea cingulata |
GATTCCGGAGAGGGAGCATG |
| CACCAGACTTGCCCTCCAAT | |
|
红树蚬 Gelonia erosa |
ATGGAGTTGGCGTTACCTGG |
| CCAGTCCCAACACCACTCTC |
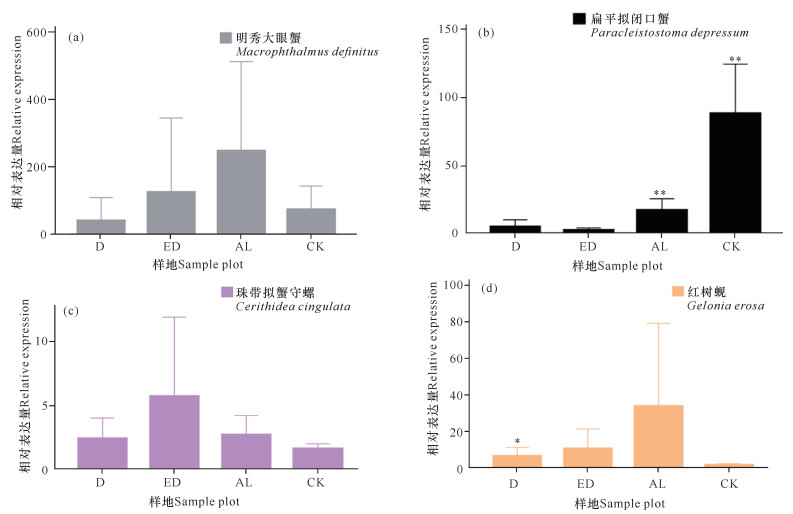
如图 3所示,两种甲壳动物DNA的相对表达量在D样地和ED样地中明显降低。由于方差较大,明秀大眼蟹16S rRNA的相对表达量在各样地间差异不显著,但相对表达量的平均值有明显差别,例如ED样地的平均表达量相当于AL样地的50.6%,D样地的表达量仅相当于AL样地的16.08%。扁平拟闭口蟹的16S rRNA的相对表达量在AL样地和CK样地这两种相对健康的红树林样地中与退化红树林中有显著性差异,ED样地和D样地中的相对表达量较AL样地中分别下降87.96%和72.06%。两种软体动物的DNA相对表达量显示出和甲壳动物不一样的趋势:珠带拟蟹守螺18S rRNA的相对表达量在ED样地最高,D样地和AL样地差异不大,提示该物种在海岸工程胁迫下生物量几乎没有改变;红树蚬的数据和珠带拟蟹守螺有相似处,红树蚬COI基因的相对表达量在AL样地、ED样地、D样地中均比CK样地高,其中AL样地最高。
|
| *P<0.05表示差异显著,**P<0.01表示差异极显著 *P<0.05 indicates significant difference, **P<0.01 indicates extremely significant difference 图 3 4个优势物种条形码基因的相对表达量 Fig.3 Relative expression of barcode gene from four dominated species |
2.4 高通量测序OTUs数量
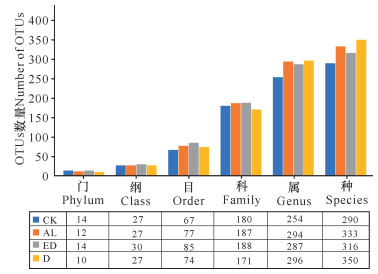
根据生物信息学比对结果,环境DNA中共得到4 651个OTUs样地,能比对到29个门、100个纲、315个目、955个科、2 419个属、4 046个种。将不属于本研究关注范围的细菌、真菌、浮游藻类的相关OTUs以及人、牛等物种造成的DNA污染剔除,并将读数 <100的OTUs也剔除,最后在门的水平上,CK样地鉴定出14个,AL样地鉴定出12个,ED样地鉴定出14个,D样地鉴定出10个;在纲的水平上,CK样地鉴定出27个,AL样地鉴定出27个,ED样地鉴定出30个,D样地鉴定出27个;在目的水平上,CK样地鉴定出67个,AL样地鉴定出77个,ED样地鉴定出85个,D样地鉴定出74个;在科的水平上,CK样地鉴定出180个,AL样地鉴定出187个,ED样地鉴定出188个,D样地鉴定出171个;在属的水平上,CK样地鉴定出254个,AL样地鉴定出294个,ED样地鉴定出287个,D样地鉴定出296个;在种的水平上,CK样地鉴定出290个,AL样地鉴定出333个,ED样地鉴定出316个,D样地鉴定出350个(图 4)。各个样地在各个分类水平上能鉴定出的OTUs数量并没有显著差异。
|
| 图 4 各样地不同分类水平下OTUs数量(读数≥100) Fig.4 Number of OTUs of different level of classification from different samples (reads≥100) |
2.5 底栖生物物种群落结构组成分析
根据分类学分析结果,可以得知一个或多个样本在各分类水平上的分类学比对情况。其结果包含了两个信息:样本中含有何种OTUs;样本中各分类水平上的OTUs序列数,即相对丰度。使用Excel对不同分类水平的群落结构按百分比作图。图 5a展示了简明分类组分图,分类单元有:藻类、真菌/细菌、无脊椎动物和脊椎动物。通过组分图可以直观地看出在各样地中,属于无脊椎动物OTUs的序列数最多,ED、D两个样地的藻类、真菌的占比相对于CK、AL样地较多,原因在于ED样地和D样地受到海岸工程的影响使其沉积物理化性质发生改变,造成藻类和真菌的快速繁殖。图 5b按门水平展示了各样地无脊椎动物的群落结构,相对丰度较高的门有节肢动物门(Arthropoda)、环节动物门(Annelida)、软体动物门(Mollusca)、刺胞动物门(Cnidaria),其中ED、D样地中环节动物门所占百分比明显比CK、AL样地多,刺胞动物也相对多,而CK和AL两种尚存活红树林样地以及对照红树林样地中的软体动物占比相对更多。在脊索动物水平上,D样地的鸟纲OTUs序列显著多,这一结果推测是因为D样地红树死亡,植被覆盖度几乎为0,所以鸟粪更易掉落到沉积物上而造成的,其余各样地脊索动物下属各纲的群落结构并没有明显的规律。辐鳍鱼纲(Actinopterygii)和海鞘纲(Ascidiacea)在CK和AL样地相对更多,CK2、AL2、ED1、ED3、D1样方出现了较多的属于哺乳动物纲的序列(图 5c)。究其原因,追溯到科的水平,这几个样地注释到了鼠科序列,鼠科的活动随机性较强,造成各样地的哺乳动物OTUs序列数没有规律性。ED2样地中两栖纲(Amphibia)的OTUs序列占比高达95.12%,追溯其原因,显示在科的水平该样地有69 050条序列注释到角花蟾科,占据ED2样地脊索动物OTUs总序列数的95.08%,推测因采样时刚好采集到该科物种的排泄物或者卵,造成该科物种序列数远远高于其他样地。因节肢动物门的序列在各样地中均占据较高比例,属于优势类群,因此绘制节肢动物门各纲的群落结构组分图(图 5d)。从图 5d中可以看出昆虫纲的序列在节肢动物门中占比最高,从35.43%(ED2)到93.11%(AL1);其次是软甲纲(Malacostraca),占比从0.88%(AL3)到51.05%(ED2)。
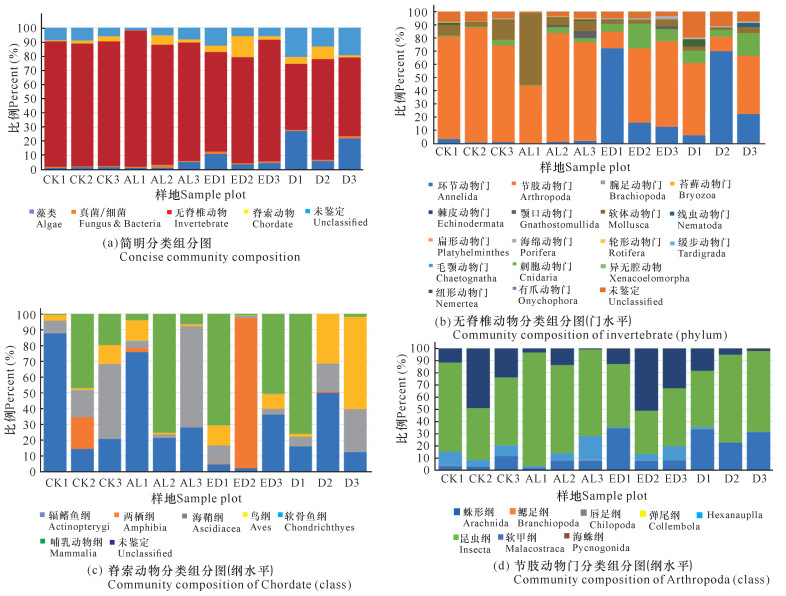
|
| 图 5 物种组成结构示意图 Fig.5 Schematic diagram of species composition structure |
2.6 样本聚类树
将每种样地3个样方门水平上的OTUs的序列数平均后,通过bray-curtis算法计算样本间基于群落组成(门水平)的层次聚类分析。如图 6所示,左边为聚类树,右边为群落结构组分。图 6的物种囊括了高通量测序得到的所有门,包括细菌、真菌、藻类等。从图 6中可以看出,CK和AL聚类在一起,相关性最近,随后是ED样地,D样地的群落结构相关性最远。
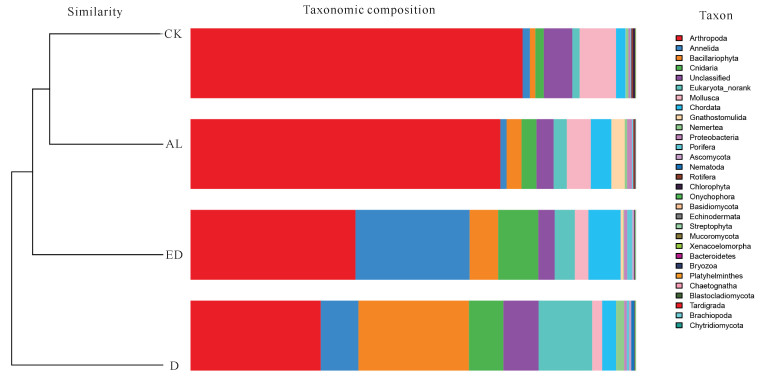
|
| 图 6 样本聚类树与群落结构组合分析 Fig.6 Combination analysis of sample clustering tree and taxonomic composition |
3 讨论 3.1 环境DNA技术研究生物量变化的初步探讨
环境DNA技术是一种新兴的技术,该技术主要应用于生物多样性的调查、入侵物种和珍稀物种的监测,也可用于水体环境中生物量的预测,例如鱼类产量的预测等。在红树林研究中,尚未有人使用此技术对底栖生物的生物量进行调查,本次研究是全球范围内首次在红树林中使用环境DNA技术开展生物量的研究。不同物种代谢能力不同,其释放DNA至环境中的速率也不同,因此环境DNA还不适合研究整个环境中所有动物生物量的变化。同种物种的不同个体向环境中释放DNA的速率相似,因此环境中某物种的DNA表达量可提示生物量的多少。本研究选择4种优势物种进行相对生物量的研究,结果显示退化红树林中,蟹类的相对生物量下降,提示海岸工程在疏浚和吹填施工过程产生了大量泥沙淤积,泥沙掩埋红树林的呼吸根,造成红树林的死亡,对蟹类的生境造成胁迫,蟹类的生物量受到影响。
将环境DNA中的相对生物量和传统调查结果(详见本刊高霆炜等《海岸工程对北海铁山港红树林大底栖动物群落的影响》)进行比较发现,两者整体变化趋势相符,说明环境DNA作为载体研究特定物种相对生物量的可行性和有效性。从数据比对中也可以看出环境DNA技术的优势:同期进行的传统“样方法”调查表明,红树蚬只出现在存活红树林中,甚至只在其中一个样地有发现,而通过荧光定量PCR可以看出多个样地中检出有红树蚬的基因。究其原因,传统“样方法”因采样困难费劳力,在10 m×10 m的样方中仅挖掘25 cm×25 cm的小样方,占样方面积的0.0625%;而环境DNA技术因采样方便,仅需数克沉积物,采样可以覆盖样方内的每棵树,因此研究结果更为精确。
明秀大眼蟹作为一个在红树林滩涂很活跃的蟹类物种,传统“样方法”的结果显示AL样地和CK样地中没有采集到该物种样本,但ED样地和D样地中生物量较高,该结果按照常理推断是不合理的。本研究的结果显示,AL样地和CK样地中明秀大眼蟹的生物量相对高于ED样地和D样地。两种调查方法的结果不同,推测原因除了传统“样方法”采样范围小外,也在于蟹类活动迅速,在传统调查中可能缺失。
3.2 环境DNA结合高通量测序全面展现退化红树林底栖动物多样性提取的环境DNA选择不同的引物可以分析特定类群的物种多样性及群落结构。本研究采用的是线粒体细胞色素氧化酶Ⅰ的通用引物,这对引物主要针对后生无脊椎动物设计,但高通量测序的结果显示样品中也包含了部分细菌、真菌、藻类、原生动物和脊椎动物。从生物信息学比对后得到的所有物种门类结果(图 5a)来看,D样地中的藻类丰度百分比高达20.24%,远高于ED样地中的6.85%、AL样地的3.00%和CK样地的1.84%,其中,D样地硅藻门的相对丰度尤其高,占总藻类的99.42%。
为避免干扰项影响,把生物信息分析结果中的藻类、真菌、细菌以及一些明显的干扰项(例如人、牛的DNA)去除,只对底栖动物的多样性和群落结构进行分析。从OTUs的数量来看,各样地能够注释到的各分类阶层类群的数量并没有显著差异。从优势类群来看,节肢动物门、环节动物门和软体动物门在各样地中占优势。从群落结构来看,退化红树林的环节动物门和刺胞动物门的相对丰度高于AL和CK样地,软体动物门的丰度则相对低于AL和CK样地。
如前述,传统“样方法”采样的随机性更强,而环境DNA方法因多点采样所以更能全面展现样地的生物多样性;环境DNA中能够鉴定出鱼类、鸟类以及哺乳动物的鼠类甚至人类的序列,说明环境DNA中不仅包括实时的物种信息,而且也包括一定时间内在样地中曾经出现的物种。由于生境、大小不同的物种采样方式/技术标准有区别,并且也鲜有研究者能够精通各种类群的形态分类学,传统的红树林生物多样性调查一般会分为浮游动植物、游泳动物、小型底栖和大型底栖动物四大类,而环境DNA技术采集的是环境样品,如水、沉积物甚至空气等,对样品采集和现场处理的要求不高,采集到的样本包含环境内所有的物种DNA在内,分析人员通过开放数据库也容易得到所有类群的分子条形码序列信息,通过生物信息学即可比对各类群的物种信息。综上所述,环境DNA技术对生物多样性的研究会更加全面。
3.3 环境DNA技术不足之处环境DNA技术也有其不足的地方。第一,以本论文为例,有一部分通过传统调查方法采集到的物种,在高通量数据中并没有发现,这是由于生物信息学注释依赖的是数据库中已有物种的分子条形码序列,而包括红树林在内的滨海湿地生态系统甚至海洋生态系统中的很多物种,并没有上传有效的分子条形码信息。第二,一对通用引物并不足以覆盖所有的生物类群,例如部分真菌、部分刺胞动物的COI基因中含有内含子,因此COI并不是鉴定真菌和此部分刺胞动物的最优条形码基因;而且,海绵和刺胞动物的进化速率比其他两侧对称的后生动物慢10-20倍,COI基因的变异不足以实现科以下级别的鉴定。第三,目前在生物多样性研究上,环境DNA技术主要应用于水体如淡水湖泊和海洋中,而较少用于土壤/沉积物的多样性研究,原因在于来源于水生动物的环境DNA在区域水体中呈现高度均匀分布状态,而沉积物中的环境DNA则随机性较大,在本实验中也发现各样地数据方差较大的现象。因此在后续实验中,需要进一步地自主扩充滨海湿地生物的DNA barcode数据库、设计针对不同类群的引物以及改进采样方式和生物信息学算法以去除干扰项,使得此项技术能够对红树林以及滨海湿地的生物多样性进行更加全面、准确的科学研究,并努力形成标准化方法使之推广应用。
4 结论环境DNA技术是一项新兴的技术,已经在各种生态系统的生物多样性研究中广泛开展,但是在红树林中的应用几乎是空白。广西红树林研究中心掌握了基于环境DNA和宏分子条形码技术的红树林大型底栖动物多样性研究方法,并一直在补充底栖物种的分子条形码数据以完善该技术方法。本实验首次在红树林中以环境DNA技术开展生物多样性研究和优势物种生物量的研究。定量结果表明,海岸工程的胁迫对蟹类有显著影响,对底栖贝类的影响不大;高通量测序得到的生物多样性和群落结构结果表明,海岸工程引起的退化红树林中底栖生物群落结构发生了改变。和传统调查方法结果的比对表明,环境DNA技术可以在红树林中研究生物多样性和相对生物量的变化,并且比传统调查方法的物种检出率高,覆盖度广,可以涵盖活动能力强的物种。环境DNA技术采样更加方便快捷,更适合于对固定样地开展生物多样性和优势物种演替的持续性研究和监测。
| [1] |
FRIESS D A. Mangrove forests[J]. Current Biology, 2016, 26(16): R746-R748. DOI:10.1016/j.cub.2016.04.004 |
| [2] |
JIA M M, WANG Z M, ZHANG Y Z, et al. Monitoring loss and recovery of mangrove forests during 42 years: The achievements of mangrove conservation in China[J]. International Journal of Applied Earth Observation and Geoinformation, 2018, 73: 535-545. DOI:10.1016/j.jag.2018.07.025 |
| [3] |
范航清. 红树林[M]. 南宁: 广西科学技术出版, 2018.
|
| [4] |
范航清, 王文卿. 中国红树林保育的若干重要问题[J]. 厦门大学学报: 自然科学版, 2017, 56(3): 323-330. |
| [5] |
HUANG F, LIN Y, ZHAO R, et al. Dissipation theory-based ecological protection and restoration scheme construction for reclamation projects and adjacent marine ecosystems[J]. International Journal of Environmental Research and Public Health, 2019, 16(21): 4303. DOI:10.3390/ijerph16214303 |
| [6] |
YANG Y, CHUI T F M, SHEN P P, et al. Modeling the temporal dynamics of intertidal benthic infauna biomass with environmental factors: Impact assessment of land reclamation[J]. Science of Total Environment, 2018, 618: 439-450. DOI:10.1016/j.scitotenv.2017.10.325 |
| [7] |
于杰, 陈作志, 徐姗楠. 围填海对珠江口南沙湿地资源与生物资源的影响[J]. 中国水产科学, 2016, 23(3): 661-671. |
| [8] |
崔磊, 吕颂辉, 董悦镭, 等. 围填海工程对淇澳岛附近水域环境因子与生物群落的影响[J]. 热带海洋学报, 2017, 36(2): 96-105. |
| [9] |
李晓静, 周政权, 陈琳琳, 等. 渤海湾曹妃甸围填海工程对大型底栖动物群落的影响[J]. 海洋与湖沼, 2017, 48(3): 617-627. |
| [10] |
李宝泉, 李晓静, 周政权, 等. 围填海及其对底栖生物群落的生态效应[J]. 广西科学, 2016, 23(4): 293-298. |
| [11] |
刘述锡, 孙淑艳, 于永海, 等. 围填海导致的生态问题和对策[J]. 海洋开发与管理, 2013(12): 63-66. DOI:10.3969/j.issn.1005-9857.2013.12.016 |
| [12] |
王初升, 黄发明, 于东升, 等. 红树林海岸围填海适宜性的评估[J]. 亚热带资源与环境学报, 2010, 15(1): 62-67. DOI:10.3969/j.issn.1673-7105.2010.01.010 |
| [13] |
THOMSEN P F, WILLERSLEV E. Environmental DNA-An emerging tool in conservation for monitoring past and present biodiversity[J]. Biological Conservation, 2015, 183: 4-18. DOI:10.1016/j.biocon.2014.11.019 |
| [14] |
BOHMANN K, EVANS A, GILBERT M T P, et al. Environmental DNA for wildlife biology and biodiversity monitoring[J]. Trends in Ecology & Evolution, 2014, 29(6): 358-367. |
| [15] |
DEINER K, WALSER J-C, MÄCHLER E, et al. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA[J]. Biological Conservation, 2015, 183: 53-63. DOI:10.1016/j.biocon.2014.11.018 |
| [16] |
BALASINGHAM K D, WALTER R P, MANDRAK N E, et al. Environmental DNA detection of rare and invasive fish species in two Great Lakes tributaries[J]. Molecular Ecology, 2018, 27(1): 112-127. DOI:10.1111/mec.14395 |
| [17] |
DRUMMOND A J, NEWCOMB R D, BUCKLEY T R, et al. Evaluating a multigene environmental DNA approach for biodiversity assessment[J]. Gigascience, 2015, 4: 46. DOI:10.1186/s13742-015-0086-1 |
| [18] |
PIAGGIO A J, ENGEMAN R M, HOPKEN M W, et al. Detecting an elusive invasive species: A diagnostic PCR to detect Burmese python in Florida waters and an assessment of persistence of environmental DNA[J]. Molecular Ecology Resources, 2014, 14(2): 374-380. DOI:10.1111/1755-0998.12180 |
| [19] |
TAKAHARA T, MINAMOTO T, YAMANAKA H, et al. Estimation of fish biomass using environmental DNA[J]. PLoS One, 2012, 7(4): e35868. DOI:10.1371/journal.pone.0035868 |
| [20] |
JEUNEN G-J, KNAPP M, SPENCER H G, et al. Environmental DNA (eDNA) metabarcoding reveals strong discrimination among diverse marine habitats connected by water movement[J]. Molecular Ecology Resources, 2019, 19(2): 426-438. |


