2. 广西壮族自治区渔业重大疫病防控与高效健康养殖产业技术工程研究中心, 广西南宁 530007;
3. 北部湾大学海洋学院, 广西北部湾海洋生物多样性养护重点实验室, 广西钦州 535011;
4. 河南师范大学生命科学学院, 河南新乡 453007;
5. 广西水产科学研究院, 广西水产遗传育种与健康养殖重点实验室, 广西南宁 530021
2. Guangxi Engineering Research Center for Fishery Major Diseases Control and Efficient Healthy Breeding Industrial Technology, Nanning, Guangxi, 530007, China;
3. Guangxi Key Laboratory of Beibu Gulf Marine Biodiversity Conservation, College of Marine Sciences, Beibu Gulf University, Qinzhou, Guangxi, 535011, China;
4. College of Life Science, Henan Normal University, Xinxiang, Henan, 453007, China;
5. Guangxi Key Laboratory of Aquatic Genetic Breeding and Healthy Breeding, Guangxi Academy of Fishery Science, Nanning, Guangxi, 530021, China
黑鲷Acanthopagrus schlegelii,属鲈形目Perciformes鲷科Sparidae棘鲷属Acanthopagrus, 是一种暖温性底层海水经济鱼类,广泛分布于我国沿海各个地区、朝鲜半岛、日本沿岸以及北太平洋西部。黑鲷生长快,抗病力强,同时兼具广盐性、广温性及营养价值高等优点,因此深受广大养殖户和消费者的欢迎。盐度是鱼类生态系统最重要的环境因子之一,直接影响着鱼类生长发育[1, 2]、繁殖[3]、生理和代谢活动[4, 5]。鱼类为响应盐度变化而改变体内生理代谢,从而进化出复杂的渗透压调控网络来适应盐度胁迫。目前有研究者指出盐度对黑鲷的生长[6, 7]、生理生化[8]和营养成分[9]等有一定的影响;此外,调控热休克蛋白和胰岛素生长因子I [10]、催乳激素及其受体[11, 12]、抗氧化[13]、水通道蛋白和精氨酸催产素受体[14]等功能基因对黑鲷适应盐度胁迫有着重要的调节作用。Na+-K+-Cl-协同转运蛋白1(NKCC1)属于SLC12基因家族的阳离子氯化物协同转运跨膜蛋白,能够介导Na+、K+和Cl-这3种离子以电中性方式协同穿过动物细胞质膜[15]。在急性盐度胁迫下,卵形鲳鲹NKCC1基因对盐度变化响应迅速,当转入淡水时,NKCC1基因在鳃和肾中的mRNA水平降低,而在肠中的表达相反[16]。在高渗盐度胁迫下,NKCC1基因的表达量在欧洲鳗鲡(Anguilla anguilla)[17]、底鳉(Fundulus heteroclitus)[18]和莫桑比克罗非鱼(Oreochromis mossambicus)[19]鳃组织中显著升高。此外,在卵形鲳鲹的鳃、肾和肠中,NKCC1基因的mRNA水平也显著增加。而转入淡水后,底鳉NKCC1基因在各个组织中的表达量显著降低[18]。因此,NKCC1基因在渗透压调节方面起到关键的作用。
目前尚未见有关黑鲷NKCC1基因在急性盐度胁迫中的相关报道。为探究黑鲷NKCC1基因在急性盐度胁迫中的表达特征,本研究克隆黑鲷NKCC1基因序列,分析其氨基酸同源性、进化关系及其在不同组织中的表达方式,阐明NKCC1基因在不同盐度下的mRNA表达特征,以期为解析黑鲷的渗透压调节机制奠定基础,并为黑鲷养殖的最适盐度范围提供理论参考。
1 材料与方法 1.1 材料从阳西县恒生水产养殖专业合作社购置500尾黑鲷幼鱼,体质量约为(30.1±3.2) g/尾,并置于300 L海水循环养殖缸中暂养一周,温度为(23.1±1.3)℃,溶氧量为6.5-7.0 mg/L。
试验所用TRIzol Reagent购自Invitrogen公司,2×Taq PCR Mastermix、PCR产物回收试剂盒、pMD18-T载体购自大连宝生物有限公司,SYBR Green,RT反转录试剂盒购自TOYOBO公司、大肠杆菌感受态DH5α购自天根生化科技(北京)有限公司。
1.2 方法暂养期间,随机挑选3尾鱼,采集其肝、脾、肾、心、脑、眼、鳃、肠、鳍条、皮肤、性腺和白肌等12个组织,液氮保存备用。
在盐度胁迫实验中,设置5,25,35共3个盐度实验组,每组50尾样品。黑鲷是河口性鱼类,在盐度15时生长速度较快,因此设置盐度15为对照组。将暂养的鱼随机转移到各组中,分别于第0,4,8,12,24,48,72 h随机在每组挑选3尾鱼采集鳃组织,使用液氮保存备用。
1.3 引物设计以黑鲷基因组中NKCC1基因的cDNA序列为模板,利用Primer Premier 5.0设计NKCC1基因的开放阅读框(Open Reading Frame, ORF)扩增引物和荧光定量PCR引物,以黑鲷18S RNA基因作为内参基因(表 1)。实验所有引物合成及测序均委托北京擎科生物有限公司完成。
| 引物Primer | 引物序列Primer sequence (5′-3′) | 用途Application |
| ASNKCC1-ORF-F1 | ATGTCAGCACCATCCTCCG | 基因扩增Gene amplification |
| ASNKCC1-ORF-R1 | AGTTGGGGTATCCAGTCA | 基因扩增Gene amplification |
| ASNKCC1-ORF-F2 | TCCTTACCTTTGTTATCG | 基因扩增Gene amplification |
| ASNKCC1-ORF-R2 | TTAAGAGTAGAAAGTGAG | 基因扩增Gene amplification |
| ASNKCC1-qPCR-F | AAGGTCGGTTCCGTGTG | 实时荧光定量PCR Real-time quantitative PCR |
| ASNKCC1-qPCR-R | CGTGTCGCCGTTCTGC | 实时荧光定量PCR Real-time quantitative PCR |
| β-actin-F | ACCCAGATCATGTTCGAGACC | 实时荧光定量PCR Real-time quantitative PCR |
| β-actin-R | ATGAGGTAGTCTGTGAGGTCG | 实时荧光定量PCR Real-time quantitative PCR |
1.4 黑鲷NKCC1基因全长cDNA的克隆
根据TRIzol Reagent试剂盒说明书提取黑鲷组织总RNA。利用1.2%琼脂糖凝胶电泳检测,使用紫外分光光度计测定总RNA的质量和浓度。根据PrimeScript RT Master Mix将提取的总RNA反转成cDNA。根据2×Taq PCR Master Mix试剂盒,以提取的cDNA为模板进行PCR,反应总体系为25 μL,上下游引物各1 μL,cDNA模板1 μL,反应程序为95℃ 3 min;95℃ 30 s,55℃ 30 s,72℃ 30 s,35个循环;72℃ 10 min。利用PCR产物试剂盒进行产物纯化回收,将片段连接至pMD18-T载体中,转化进入大肠杆菌DH5α感受态细胞中,摇菌3 h后,接入带氨苄抗性的LB平板中,挑取单克隆进行菌液PCR阳性检测。阳性检测结果送至北京擎科生物有限公司进行测序验证。
1.5 生物信息学分析利用NCBI ORF Finder在线工具(https://www.ncbi.nlm.nib.gov/orffinder)和基因组数据库(http://asia.ensembl.org/info/about/species.html)验证预测黑鲷NKCC1基因的开放阅读框(Open reading frame, ORF)。利用ProParam(http://web.expasy.org/cgi-bin/protparam/protparam)和NetNGlyc1.0(https://services.healthtech.dtu.dk/)分析蛋白质的理化性质。使用NCBI Protein BLAST(https://blast.ncbi.nlm.nib.gov/Blast.cgi)对黑鲷NKCC1进行结构分析。利用DNAMAN6.0和MEGA5.0软件对序列进行多重比较和进化树构建。
1.6 黑鲷NKCC1基因组织表达分析使用SYBR Premix Ex Taq试剂盒进行实时荧光定量PCR (RT-PCR)实验。样品的cDNA模板稀释至100 ng/μL,PCR反应总体系为12.5 μL,包括1 μL模板,6.5 μL SYBR Premix,上下游引物各0.5 μL,4 μL ddH2O。反应程序为95℃ 30 s;95℃ 15 s,55℃ 15 s,68℃ 30 s,45个循环;进行3次重复试验。
1.7 统计分析实时荧光定量PCR的试验数据采用△△CT法(2-△△CT)进行分析;数据统计采用SPSS 19进行单因素方差分析(AMOVA),并进行Duncan's多重比较分析各组间差异的显著性,显著水平为P<0.05。
2 结果与分析 2.1 黑鲷NKCC1基因克隆及序列分析黑鲷NKCC1基因ORF全长为3 504 bp,共编码1 167个氨基酸,预测蛋白分子量为127.19 kDa,理论等电点为5.75。利用NitNlyc1.0预测显示黑鲷NKCC1为跨膜蛋白。利用MEGA5.0对黑鲷NKCC1氨基酸序列分析,结果表明在51-1 167氨基酸残基间有一个典型的Na+-K+-Cl-共同转运蛋白SLC12A结构域,在不同物种中具有很高的保守性(图 1)。

|
| 图 1 黑鲷NKCC1的氨基酸序列对比和结构域分析 Fig.1 Amino acid sequences and domains analysis of NKCC1 in A.schlegelii |
2.2 同源性分析及进化树
黑鲷NKCC1氨基酸序列与其他物种的NKCC1同源性为66.1%-97.8%(表 2)。其中,与金头鲷的同源性最高,为97.8%;与斑马鱼的同源性最低,为66.1%。此外,与大黄鱼、鞍带石斑鱼和尖吻鲈的同源性均大于90%。
| 物种Species | 预测氨基酸的相似性Prediction of amino acid similarity (%) | 基因序列编号GenBank No. |
| 黑鲷A.schlegelii NKCC1 | 100.0 | —— |
| 金头鲷Sparus aurata NKCC1a | 97.8 | XP_030290529.1 |
| 金头鲷S.aurata NKCC1b | 73.3 | XP_030273833.1 |
| 斑马鱼Danio rerio NKCC1a | 79.6 | ENSDARP00000131505 |
| 斑马鱼D.rerio NKCC1b | 66.1 | ENSDARP00000140839 |
| 斑点雀鳝Lepisosteus oculatus NKCC1 | 77.7 | ENSLOCP00000010254 |
| 罗非鱼Oreochromis niloticus NKCC1b | 72.5 | ENSONIP00000017387 |
| 罗非鱼O.niloticus NKCC1a | 88.8 | ENSONIP00000014249 |
| 大黄鱼Larimichthys crocea NKCC1 | 92.0 | XP_019127704.1 |
| 欧洲鲈Dicentrarchus labrax NKCC1a | 93.7 | ENSDLAT00005051426.1 |
| 欧洲鲈D.labrax NKCC1b | 71.7 | ENSDLAT00005038362.1 |
| 鞍带石斑鱼Epinephelus lanceolatus | 92.2 | XP_033502841.1 |
| 尖吻鲈Lates calcarifer NKCC1 | 91.9 | XP_018529108.1 |
| 小鼠Mus musculus NKCC1 | 74.1 | NP_033220.2 |
| 人Homo sapiens NKCC1 | 72.8 | NP_001037.1 |
| 鸡Gallus gallus NKCC1 | 72.4 | XP_004949435.1 |
从系统进化树可以看出黑鲷NKCC1首先与金头鲷NKCC1a聚为一支(图 2),然后再与大黄鱼、鞍带石斑鱼、尖吻鲈NKCC1聚为一支,再与斑马鱼和斑点雀鳝NKCC聚为一支,而罗非鱼、金头鲷、欧洲鲈的NKCC1b则单独聚为一支。此外,小鼠、鸡和人等外源物种的NKCC1聚为一支。
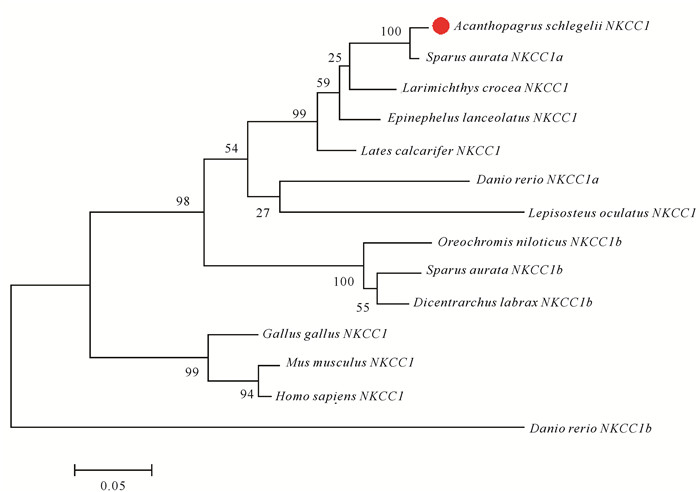
|
| 图 2 黑鲷NKCC1基因的系统进化树分析 Fig.2 Phylogenetic tree analysis of NKCC1 gene in A.schlegelii |
2.3 黑鲷NKCC1基因不同组织表达分析
黑鲷NKCC1基因在所检测的12个组织,即肝脏、肾脏、心、脑、眼、鳃、鳍条、脾脏、皮肤、肠、性腺和白肌中均有表达量(图 3)。其中,在肾脏、鳃和肠中表达量较高,显著高于其他组织(P<0.05);其次为肝脏、脑、脾脏、皮肤、性腺和白肌,表达量最低的组织为心、眼、鳍条,显著低于其他组织(P<0.05)。
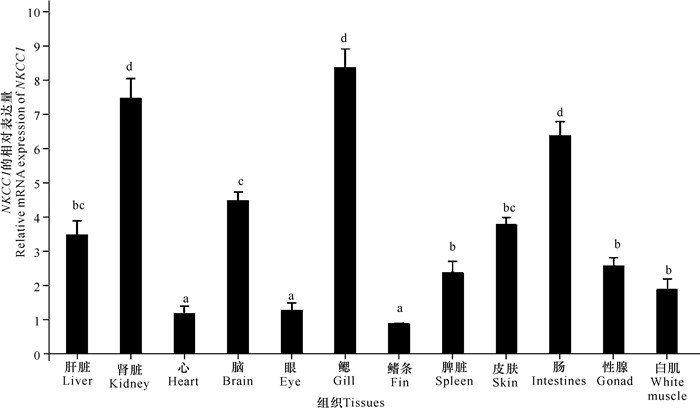
|
| 不同字母表示两组间具有显著性差异(P<0.05) Different letters indicate a significant difference between the two groups (P < 0.05) 图 3 黑鲷NKCC1基因的组织表达模式 Fig.3 Tissue expression patterns of NKCC1 genes in A.schlegelii |
2.4 不同盐度胁迫条件下黑鲷NKCC1基因的表达分析
不同盐度胁迫下NKCC1基因在黑鲷鳃和肾脏组织中的相对表达量如图 4所示。在鳃组织中(图 4a),在对照组(盐度15组)中,NKCC1基因在12 h后显著上升,在24 h后达到顶峰,随后开始逐渐下降;在盐度5组中,NKCC1基因在处理4 h后就显著上调,且其表达量在第4,8,12和24 h均显著高于对照组,而在72 h则显著低于对照组;在盐度25组中,NKCC1基因表达量随着时间推移而缓慢上升,至24 h达到顶峰,在24,48和72 h时的表达量均显著低于对照组;在盐度35组中,其表达变化与盐度25组类似。
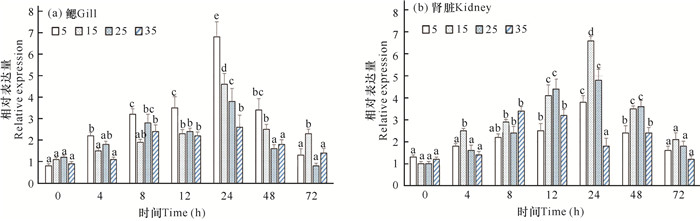
|
| 不同字母表示两组间具有显著性差异(P<0.05) Different letters indicate a significant difference between the two groups (P < 0.05) 图 4 急性盐度胁迫下黑鲷NKCC1基因在组织中的表达特征 Fig.4 Expression patterns of NKCC1 gene in tissues of A.schlegelii under acute salinity stress |
在肾脏组织中(图 4b),在对照组(盐度15组)中,NKCC1基因表达量随着时间推移而明显升高,至24 h后达到顶峰(相比于0 h提高6.6倍),随后慢慢下降,至72 h仍高于0 h;在盐度5组中,NKCC1基因的表达量随着时间推移而缓慢升高,至24 h达到顶峰,随后开始慢慢下降,至72 h后逐渐恢复到开始时的水平;在盐度25组中,NKCC1基因在第8 h开始出现明显上调(P<0.5),至24 h到达顶峰,随后开始下降;在盐度35组中,NKCC1基因表达量缓慢升高,至8 h到达顶峰,随后开始下降。
在鳃组织中(图 4a),在低盐度胁迫下,NKCC1基因的表达量对盐度变化反应十分迅速,在处理4 h后就会显著刺激NKCC1基因的表达量,且一直持续至48 h;而在高盐度胁迫下,NKCC1的表达量对盐度变化的响应在早期并不明显,至24 h后会出现显著抑制现象,一直持续至72 h。在肾脏组织中(图 4b),低盐度和高盐度胁迫下,NKCC1基因的表达量均会随着时间推移而出现先增高后降低的现象。此外,相比于对照组,低盐度和高盐度胁迫在都会在不同时间点抑制NKCC1基因的表达量。上述实验表明, NKCC1基因在维持黑鲷渗透压和离子平衡过程中起着重要的调节作用。
3 讨论NKCC是一类电中性跨膜转运蛋白,是溶质载体家族的成员之一,可以按Na+∶K+∶Cl-=1∶1∶2的比例进行离子跨膜转运[20, 21],从而参与调节极化细胞中的离子和水分运动。在脊椎动物和无脊椎动物中都发现有NKCC,其在维持细胞体积的稳态和电解质含量的过程中起关键作用[22]。在不同渗透压环境中,鱼类的NKCC会被激活,向体外分泌离子,从而维持渗透压平衡。在低盐度情况下,鱼类体内Na+、Cl-浓度低于外界环境;在高盐度情况下,鱼类体内的Na+、Cl-浓度高于外界,此时机体激活细胞膜上的NKCC蛋白,通过主动运输调节离子浓度,使机体内外的渗透压达到平衡[20, 21]。
许多研究报道NKCC蛋白参与鱼类渗透压的调节,例如在舩鱼(Fundulus heteroclitus)中,当进行淡水转移后,其NKCC1a表达量在每个组织中均被抑制; 而从微咸水转移至海水后,NKCC1a mRNA的表达增加[23]。在罗非鱼(Sarotherodon melanothern)中,不同盐度环境会显著影响鳃组织中NKCC1a mRNA的表达水平,在136‰盐度海水中其表达量会比在淡水中高4.9倍[24]。已有学者研究黑鲷在盐度胁迫下,为适应环境所起重要调节作用的相关基因[10-14],但尚未见有关于黑鲷NKCC1基因参与渗透压调节的相关报道。在本研究中,通过克隆获得黑鲷NKCC1基因,该基因cDNA的ORF全长为3 504 bp,编码1 167个氨基酸,理论蛋白分子量为127.19 kDa,理论等电点为5.75。蛋白结构域分析显示在51-1 167氨基酸残基间有一个典型的Na+-K+-Cl-共同转运蛋白SLC12A结构域,这与斑马鱼、金鲳、罗非鱼等物种的SLC12A结构域具有高度一致性,表明SLC12A结构域在不同物种中具有很高的保守性,同时也表明黑鲷NKCC1与其他物种的NKCC1一样,具有潜在的Na+-K+-Cl-离子共同转运功能。
组织表达分析表明NKCC1基因在黑鲷各组织中均有表达,且在肾脏、鳃和肠中表达量最高。在其他硬骨鱼类如舌齿鲈、罗非鱼和大西洋鲑的组织中,均发现有NKCC1基因的表达[25-27],而本研究的结果与其他鱼类中的研究类似。此外,肾脏、鳃和肠均为参与鱼类渗透压调节的重要组织,黑鲷NKCC1基因在上述组织中具有显著高于其他组织的表达量,暗示该基因在渗透压调节中发挥重要功能。
对不同盐度胁迫下NKCC1基因在鳃和肾脏中的时间表达特征分析表明,与肾脏组织相比,NKCC1基因在鳃组织中的渗透压调节作用更明显,且在低盐度胁迫下反应十分迅速。这一结果与大西洋鲑和攀鲈(Anabas teseuclineus)中的结果相似[28],说明黑鲷NKCC1基因主要发挥作用的器官是鳃组织。另外,相较于高盐度胁迫,黑鲷NKCC1基因对低盐度胁迫的响应更为明显迅速。
鱼类在海水中通过吞饮吸收水分,再通过肠道摄取,进而用鳃排泄。当转移至淡水环境中时,由于吞咽水的反射和水分扩散,鱼机体增加了肠内的水量。为停止反射、促进水分泌,鱼机体会提高NKCC1基因的表达,促进离子进入细胞以增加细胞液浓度。尽管黑鲷渗透调节能力较强,但仍不能在急性条件下适应淡水,因此也会激活NKCC1基因表达以适应低盐度环境。有学者认为NKCC1基因的快速响应受磷酸化和去磷酸化调节的影响,而触发NKCC1基因磷酸化的信号似乎是细胞内Cl-本身[29]。因此,推测在低盐度胁迫条件下,黑鲷鳃组织中的NKCC1基因的磷酸化因细胞内Cl-浓度的降低而激活,继而提高NKCC1基因的表达量,增加进入细胞内的离子量,从而促进水分泌,以维持细胞内的稳态。综上所述,本研究可为解析黑鲷NKCC1基因参与渗透压调节的功能机制和建立黑鲷最适盐度养殖模式提供了一定的技术理论,并为研究鱼类渗透压调节机制奠定理论基础。
4 结论本研究分析黑鲷NKCC1基因的分子特征及其在不同盐度胁迫环境下的表达特征,得出以下结论:(1)黑鲷NKCC1基因ORF全长为3 504 bp,共编码1 167个氨基酸,为跨膜蛋白,定位在细胞膜上,且含有一个典型的Na+-K+-Cl-共同转运蛋白SLC12A结构域; (2) NKCC1基因在黑鲷的渗透压调节组织中均有表达,其中,在肾脏、鳃和肠3个组织中的表达量显著高于其他组织(P<0.05);(3)与肾脏组织相比,NKCC1基因在鳃组织中发挥的渗透压调节作用更为明显,且在低盐度胁迫时发挥的作用更为迅速,推测是由于低盐度胁迫,黑鲷鳃组织NKCC1基因的磷酸化因细胞内Cl-浓度的降低而被激活,继而提高NKCC1基因的表达量,增加进入细胞内的离子量,从而促进水分泌,以维持细胞内的稳态。本研究成果有助于揭示黑鲷NKCC1参与渗透压调节的功能机制,并为鱼类渗透压调节研究提供一定的理论基础。
| [1] |
ALLEN P J, MITCHELL Z A, DEVRIES R J, et al. Salinity effects on Atlantic sturgeon (Acipenser oxyrinchus oxyrinchus Mitchill, 1815) growth and osmoregulation[J]. Journal of Applied Ichthyology, 2015, 30(6): 1229-1236. |
| [2] |
ANNI A I S, BIANCHINI A, JUNIOR V A S, et al. Salinity influence on growth, osmoregulation and energy turnover in juvenile pompano Trachinotus marginatus Cuvier 1832[J]. Aquaculture, 2016, 455: 63-72. DOI:10.1016/j.aquaculture.2016.01.010 |
| [3] |
PREZ-ROBLES J, DIAZ F, IBARRA-CASTRO L, et al. Effects of salinity on osmoregulation during the embryonic development of the bullseye puffer (Sphoeroides annulatus Jenyns 1842)[J]. Aquaculture Research, 2016, 47(3): 838-846. DOI:10.1111/are.12542 |
| [4] |
MUHAMMADAR A A, MAZLAN A G, SAMAT A, et al. Growth, survival and feed conversion of juvenile tiger grouper Epinephelus fuscoguttatus in different salinity regimes[J]. Aquaculture, Aquarium, Conservation & Legislation International Journal of the Bioflux Society, 2014, 7(4): 241-247. |
| [5] |
MOUTOU K A, PANAGIOTAKI P, MAMURIS Z. Effects of salinity on digestive protease activity in the euryhaline sparid Sparus aurata L.: A preliminary study[J]. Aquaculture Research, 2004, 35(9): 912-914. DOI:10.1111/j.1365-2109.2004.01068.x |
| [6] |
KODA K, TSUYUKI A, UMINO T, et al. Effect of rearing salinity on growth, body composition, and otolith Sr: Ca ratio in fingerling black seabream Acanthopagrus schlegelii[J]. Nippon Suisan Gakkaishi, 2016, 82(5): 720-726. DOI:10.2331/suisan.15-00074 |
| [7] |
王裕玉, 徐跑, 张志伟, 等. 不同养殖模式对黑鲷生长、血清生化指标及抗氧化性能的影响[J]. 江苏农业科学, 2020, 48(23): 155-160. |
| [8] |
MIN B H, SEO J S, PARK J J, et al. Gill Na+/K+-ATPase activity and expression in black sea bream Acanthopagrus schlegelii exposed to a hyposaline environment[J]. Korean Journal of Fisheries and Aquatic Sciences, 2015, 48(1): 64-70. DOI:10.5657/KFAS.2015.0064 |
| [9] |
贾超峰, 祝斐, 孟乾, 等. 低盐养殖对黑鲷营养成分的影响[J]. 食品工业科技, 2020, 41(1): 284-288, 294. |
| [10] |
邓利, 张为民, 林浩然. 盐度变化对黑鲷生长激素及其受体的影响[J]. 热带海洋学报, 2003, 22(6): 9-14. DOI:10.3969/j.issn.1009-5470.2003.06.002 |
| [11] |
CHANG Y J, MIN B H, CHOI C Y. Black porgy (Acanthopagrus schlegeli) prolactin cDNA sequence: mRNA expression and blood physiological responses during freshwater acclimation[J]. Comparative Biochemistry and Physiology b-Biochemistry & Molecular Biology, 2007, 147(1): 122-128. |
| [12] |
TOMY S, CHANG Y M, CHEN Y H, et al. Salinity effects on the expression of osmoregulatory genes in the euryhaline black porgy Acanthopagrus schlegeli[J]. General and Comparative Endocrinology, 2009, 161(1): 123-132. DOI:10.1016/j.ygcen.2008.12.003 |
| [13] |
AN K W, KIM N N, SHIN H S, et al. Profiles of antioxidant gene expression and physiological changes by thermal and hypoosmotic stresses in black porgy (Acanthopagrus schlegeli)[J]. Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology, 2010, 156(2): 262-268. |
| [14] |
AN K W, KIM N N, CHOI C Y. Cloning and expression of aquaporin 1 and arginine vasotocin receptor mRNA from the black porgy, Acanthopagrus schlegeli: Effect of freshwater acclimation[J]. Fish Physiology and Biochemistry, 2008, 34(2): 185-194. DOI:10.1007/s10695-007-9175-0 |
| [15] |
PAYNE J A, FORBUSH B. Molecular characterization of the epithelial Na-K-Cl cotransporter isoforms[J]. Current Opinion in Cell Biology, 1995, 7(4): 493-503. DOI:10.1016/0955-0674(95)80005-0 |
| [16] |
ZHAO C P, GUO H Y, ZHU K C, et al. Molecular characterization of Na+/K+/2Cl- cotransporter 1 alpha from Trachinotus ovatus (Linnaeus, 1758) and its expression responses to acute salinity stress[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2018, 223: 29-38. DOI:10.1016/j.cbpb.2018.05.003 |
| [17] |
CUTLER C P, CRAMB G. Two isoforms of the Na+/K+/2Cl- cotransporter are expressed in the European eel (Anguilla anguilla)[J]. Acta Biochimica Et Biophysica Sinica, 2002, 1566(1/2): 92-103. |
| [18] |
SCOTT G R, RICHARDS J G, FORBUSH B, et al. Changes in gene expression in gills of the euryhaline killifish Fundulus heteroclitus after abrupt salinity transfer[J]. American Journal of Physiology-Cell Physiology, 2004, 287(2): C300-C309. DOI:10.1152/ajpcell.00054.2004 |
| [19] |
INOKUCHI M, HIROI J, WATANABE S, et al. Gene expression and morphological localization of NHE3, NCC and NKCC1a in branchial mitochondria-rich cells of Mozambique tilapia (Oreochromis mossambicus) acclimated to a wide range of salinities[J]. Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology, 2008, 151(2): 151-158. |
| [20] |
SUVITAYAVAT W, DUNHAM P B, HAAS M, et al. Characterization of the proteins of the intestinal Na+-K+-2Cl- cotransporter[J]. American Journal of Physiology, 1994, 267(2): C375-C384. DOI:10.1152/ajpcell.1994.267.2.C375 |
| [21] |
TOWLE D W, RUSHTON M E, HEIDYSCH D, et al. Sodium/proton antiporter in the euryhaline crab carcinus maenas: Molecular cloning, expression and tissue distribution[J]. Journal of Experimental Biology, 1997, 200(Pt6): 1003-1014. |
| [22] |
PAYNE J A, FORBUSH B. Molecular characterization of the epithelial Na-K-Cl cotransporter isoforms[J]. Current Opinion in Cell Biology, 1995, 7(4): 493-503. DOI:10.1016/0955-0674(95)80005-0 |
| [23] |
SCOTT G R, RICHARDS J G, FORBUSH B, et al. Changes in gene expression in gills of the euryhaline killifish Fundulus heteroclitus after abrupt salinity transfer[J]. American Journal of Physiology-Cell Physiology, 2004, 287(2): C300-C309. DOI:10.1152/ajpcell.00054.2004 |
| [24] |
范武江, 李思发. 萨罗罗非鱼NKCC1α基因cDNA克隆及mRNA组织表达差异[J]. 动物学研究, 2010, 31(6): 601-609. |
| [25] |
LORIN-NEBEL C, BOULO V, BODINIER C, et al. The Na+/K+/2Cl-cotransporter in the sea bass Dicentrarchus labrax during ontogeny: Involvement in osmoregulation[J]. Journal of Experimental Biology, 2006, 209(24): 4908-4922. DOI:10.1242/jeb.02591 |
| [26] |
HIROI J, YASUMASU S, MCCORMICK S D, et al. Evidence for an apical Na-Cl cotransporter involved in ion uptake in a teleost fish[J]. Journal of Experimental Biology, 2008, 211(16): 2584-2599. DOI:10.1242/jeb.018663 |
| [27] |
SUNDH H, NILSEN T O, LINDSTR M J, et al. Development of intestinal ion-transporting mechanisms during smoltification and seawater acclimation in Atlantic salmon Salmo salar[J]. Journal of Fish Biology, 2014, 85(4): 1227-1252. DOI:10.1111/jfb.12531 |
| [28] |
HWANG P P, LEE T H, LIN L Y. Ion regulation in fish gills: Recent progress in the cellular and molecular mechanisms[J]. American Journal of Physiology-Regulatory, Integrative and Comparative Physiology, 2011, 301(1): R28-R47. DOI:10.1152/ajpregu.00047.2011 |
| [29] |
GAGNON K B, DELPIRE E. Molecular determinants of hyperosmotically activated NKCC1-ediated K+/K+ exchange[J]. Journal of Physiology, 2010, 588(18): 3385-3396. DOI:10.1113/jphysiol.2010.191932 |



