2. 河南师范大学生命科学学院, 河南新乡 453007;
3. 北部湾大学海洋学院, 广西北部湾海洋生物多样性养护重点实验室, 广西钦州 535011
2. College of Life Science, Henan Normal University, Xinxiang, Henan, 453007, China;
3. Guangxi Key Laboratory of Beibu Gulf Marine Biodiversity Conservation, College of Marine Sciences, Beibu Gulf University, Qinzhou, Guangxi, 535011, China
我国是水产养殖大国,但近年来水产病害的暴发和流行给水产养殖业造成了巨大的经济损失[1, 2]。其中,哈维氏弧菌(Vibrio harveyi)是海水养殖动物的主要致病菌之一,常见于广西等华南沿海地区,严重威胁着该地区水产养殖业的健康可持续发展[3-5]。目前主要采用抗生素等化学药物治理水产养殖中的弧菌病,但化学药物抗菌效果的持续时间短,且近年来抗生素的滥用加剧了病原菌耐药性的产生,带来了水产品有害药物残留等重大食品安全问题[6]。针对哈维氏弧菌的检测方法主要包括:基于细菌生理生化反应的检测方法、基于抗体的免疫学检测技术,以及聚合酶链式反应(PCR)等[7-9]。这些方法虽然能精确诊断哈维氏弧菌,但存在操作繁琐、检测耗时长、试剂保存条件苛刻等不足,从而难以满足在养殖现场快速、准确检测哈维氏弧菌的要求。弧菌病的防治一直是水生病害防控技术研究的重点,而发展成本低、耗时短、操作便捷、灵敏度高的致病菌快速检测技术,有助于及早发现、确定病原,进而制定精确的治疗方案来控制病原扩散,降低水产养殖业的经济损失[10]。
指数富集的配基系统进化(Systematic Evolution of Ligands by Exponential Enrichment, SELEX)是一种生物文库筛选技术,该技术使用容量高达1×1014-1×1015的随机寡核苷酸文库,在体外经过多轮筛选,最终获得能够特异性识别靶物质的单链寡核苷酸,即核酸适配体[11]。核酸适配体作为一种新型的识别分子,具有易筛选、易修饰、稳定性强、高特异性识别靶标分子等优点,目前已广受关注并发展成为一类新型的检测和治疗工具[12-16]。通常,筛选核酸适配体只需要6-30个SELEX循环,时间仅为3-8周,远低于制备生物抗体的时间(3-8个月),而且可避免生物抗体存在的成本高、易失活、保存条件苛刻、批次间的抗体质量存在差异等问题。另外,SELEX技术是在体外进行的化学筛选过程,因此靶标分子范围广范,包括金属离子、有机染料等简单体系,以及病毒、细胞、组织等复杂体系;特别是具有细胞毒性、无免疫原性或者弱免疫原性的靶标分子,使用SELEX技术可实现体外高特异性核酸适配体的筛选[11]。鉴于SELEX技术的优点,核酸适配体的研究已受到国内外学者的广泛关注,并在生物化学、生物医学、纳米材料、蛋白质科学等多学科得到迅速发展。但是,我国对核酸适配体应用于水产病害防治领域的研究尚显不足,待发展空间巨大。
本研究以哈维氏弧菌为靶标,利用SELEX技术筛选获得可高特异性、高亲和性识别哈维氏弧菌的核酸适配体,并对其性质进行系统性分析,包括核酸适配体的特异性、细胞毒性和靶位点的性质等,从而为水产病害的检测和防控提供新思路、新方法。
1 材料与方法 1.1 材料哈维氏弧菌(Vibrio harveyi)从广西北海市铁山港海域网箱养殖的发病卵形鲳鲹中分离得到,编号为GT-V.harveyi 1[17]。溶藻弧菌(Vibrio alginolyticus)从广西钦州湾网箱养殖的发病卵形鲳鲹中分离得到,编号为TOQZ01[18]。嗜水气单胞菌(Aeromonas hydrophila)和维氏气单胞菌(Aeromonas veronii)从广西南宁池塘养殖的发病草鱼中分离获得。
实验仪器:TS2倒置光学显微镜(日本Nikon公司),ACSAria Ⅱ流式细胞仪(美国BD公司), Nano Drop one超微量分光光度计(美国Thermo公司), Mastercycler nexus gradient PCR仪(德国Eppendorf公司), AC2-4S1生物安全柜(新加坡ESCO公司), Centrifuge 5417R离心机(德国Eppendorf公司), Gel Doc XR+凝胶成像系统(美国BIO-RAD公司), DK-8D水浴锅(博讯公司)。试剂材料:链酶亲和素标记的磁珠(美国Thermo公司);PCR纯化回收试剂盒、胶回收试剂盒、DL2000 Marker、DL50 bp Marker(日本TakaRa公司)。磷酸缓冲盐溶液(Phosphate Buffer Saline, PBS):2 mmol/L KH2PO4,10 mmol/L Na2HPO4,137 mmol/L NaCl,pH值为7.2。
1.2 方法 1.2.1 随机单链DNA文库(ssDNA Library)和引物的设计合成用于核酸适配体筛选的随机单链DNA文库(ssDNA Library)、引物和核酸适配体均委托北京奥科鼎盛生物科技有限公司合成。随机起始单链DNA文库中每条核酸单链均由中间50 bp的随机核苷酸序列(N50)和两端的固定核苷酸序列构成(5′-GACGCTTACTCAGGTGTGACTCG-N50-CGAAGGACGCAGAGAAGTCTC-3′)。上游引物(Forward Primer, FP): 5′-GACGCTTACTCAGGTGTGACTCG-3′,下游引物(Reverse Primer, RP):5′-GAGACTTCATCTGCGTCCTTCG-3′。其中,上游引物5′端标记着羟基荧光素(6-carboxy-fluorescein labeled FP, FAM-FP),下游引物5′端引物标记着生物素(Biotin labeled RP, biotin-RP)。
1.2.2 SELEX技术筛选特异性识别哈维氏弧菌的核酸适配体以羟基荧光素(FAM)标记的随机单链DNA文库与哈维氏弧菌的孵育结合为对照(Con),利用流式细胞术进行FAM荧光值检测。4 nmol的起始文库溶于1 000 μL PBS中,在92℃高温变性10 min,然后迅速插入到冰中复性10 min;起始文库与哈维氏弧菌在冰上孵育结合1 h;用PBS缓冲液离心清洗3次,随后将其在92℃水浴中处理5 min,离心分离得到与哈维氏弧菌结合的ssDNA;将分离得到的ssDNA作为模板进行PCR扩增。PCR程序:预变性94℃ 2 min,扩增20个循环(94℃ 1 min,60℃ 30 s,72℃ 1 min),终延伸72℃ 5 min。将100 μL链酶亲和素标记的纳米磁珠与PCR扩增所得的双链DNA在常温下孵育20 min,利用生物素与链酶亲和素的亲和作用,将双链DNA结合到磁珠表面;将离心管放在磁性分离器上除去上清,用1 mL PBS洗涤磁珠;然后在离心管中加入200 μL NaOH (200 mmol/L)溶液,室温反应15 min,利用磁性分离器分离回收上清;用PCR纯化回收试剂盒回收上清液中的正向ssDNA单链核酸,收集到含有ssDNA文库的溶液用于下一轮的筛选。为提高筛选得到的ssDNA文库的特异性和亲和力,在随后的筛选中,逐步缩短文库与哈维氏弧菌的结合时间,同时减少哈维氏弧菌和ssDNA文库的使用量,增加PBS缓冲液清洗靶标细胞的次数。
1.2.3 核酸适配体的序列分析对最终获得的核酸适配体库进行克隆、测序,确定各适配体的DNA序列。具体操作如下:将最终得到的核酸适配体库经PCR扩增成dsDNA;切胶纯化后连接到pMD18-T载体并转化到大肠杆菌DH5α中;菌液均匀涂布在LB固体培养基(氨苄抗性)上,倒置在37℃培养箱培养过夜;挑取100个单菌落并交由基因公司测序分析,得到核酸适配体序列。使用MFOLD(http://mfold.rna.albany.edu/?q=Mfold/DNA-Folding-Form)在线预测筛选得到核酸适配体的二级结构。
1.2.4 核酸适配体识别结合哈维氏弧菌的特异性分析使用流式细胞术分析核酸适配体与哈维氏弧菌的特异性结合。本研究中使用FAM对核酸适配体进行标记,羟基荧光素激发波长为495 nm,发射波长为535 nm。将FAM标记的核酸适配体与哈维氏弧菌孵育结合,然后使用流式细胞仪进行荧光分析。在相应波长范围内检测荧光信号,根据信号强弱判断核酸适配体与哈维氏弧菌的特异性识别结合情况。具体操作如下:FAM标记的核酸适配体(200 nmol/L)经过92℃恒温水浴变性、冰浴复性处理,然后与哈维氏弧菌4℃结合1 h,孵育结束后离心重复清洗3次,并混匀在500 μL PBS中,使用流式细胞仪进行荧光检测。对照组共4组,对照组1 (Con1)是FAM标记的非特异性起始库(200 nmol/L)和哈维氏弧菌的孵育结合;对照组2 (Con2)是FAM标记的核酸适配体(200 nmol/L)和溶藻弧菌的孵育结合;对照组3 (Con3)是FAM标记的核酸适配体(200 nmol/L)和嗜水气单胞菌的孵育结合;对照组4 (Con4)是FAM标记的核酸适配体(200 nmol/L)和维氏气单胞菌的孵育结合。每个反应均做3个重复。
1.2.5 核酸适配体的细胞毒性分析在96孔板中接入石斑鱼脾脏细胞(Grouper spleen cell line, GS),28℃培养18 h。然后将核酸适配体在细胞培养基中稀释至不同浓度(0, 100, 500, 1 000 nmol/L)。将不同浓度的核酸适配体分别与GS细胞于28℃条件下孵育48 h,对细胞进行光镜观察、拍照。然后在各孔中加入10 μL CCK-8溶液,室温孵育4 h后,利用酶标仪检测450 nm的吸光值来检测细胞活性,以未与核酸适配体孵育的正常GS细胞作为对照组,做3个平行实验。将测得的吸光值代入如下公式(1),计算各组的细胞存活率(Survival Rate, SR)[19]:
| $ {\rm{SR}}(\% ) = \mathit{O}{\mathit{D}_{450}}(实验组)/\mathit{O}{\mathit{D}_{450}}(对照组) \times 100\% $ | (1) |
将哈维氏弧菌用胰酶消化处理10 min,离心并使用500 μL PBS重复清洗3次,然后与FAM标记的核酸适配体(200 nmol/L)在4℃孵育1 h,然后离心重复清洗3次,并混匀在500 μL PBS中,使用流式细胞仪进行荧光检测,该组为实验组(Test)。对照组为FAM标记的200 nmol/L核酸适配体(Con)与未经胰酶消化处理的哈维氏弧菌孵育结合。每个反应均做3个重复。
1.2.7 数据统计方法应用Excel 2010及SPSS 26软件进行数据处理及统计,采用非参数T检验及卡方检验。P<0.05表示差异有统计学意义。
2 结果与分析 2.1 SELEX技术筛选哈维氏弧菌的特异性核酸适配体随着筛选轮数(共10轮)增加,ssDNA文库的特异性逐步升高(图 1),其中,第9轮的ssDNA文库对哈维氏弧菌的特异性结合丰度最高。因此对第10轮的ssDNA文库进行克隆测序,得到2条核酸适配体H10和H13 (表 1)。

|
| Con表示FAM标记的随机单链核酸ssDNA文库与哈维氏弧菌的结合;**P<0.01表示差异极显著, NS表示无显著性差异 Con represents the binding fluorescence of FAM-labeled random single stranded ssDNA library to V.harveyi; **P < 0.01 means extremely significant, and NS indicates no significant 图 1 基于SELEX技术筛选哈维氏弧菌的核酸适配体 Fig.1 Screening of aptamers for V.harveyi based on systematic evolution of ligands by exponential enrichment (SELEX)technology |
| 核酸适配体Aptamer | 核苷酸序列Nucleotide sequences |
| H10 | 5′-GACGCTTACTCAGGTGTGACTCGTTACTAC-CTCTGTAAGTCCGTCTTGCTTCTTTCTAAGAA-GTGGTGTGTCTCGAAGGACGCAGATGAAGTC-TC-3′ |
| H13 | 5′-GACGCTTACTCAGGTGTGACTCGTGTAATG-GGGGGGCTGGAAGGGGTTTGTGTTGGTTTGG-TCGGAAGTTCCACGAAGGACGCAGATGAAGTC-TC-3′ |
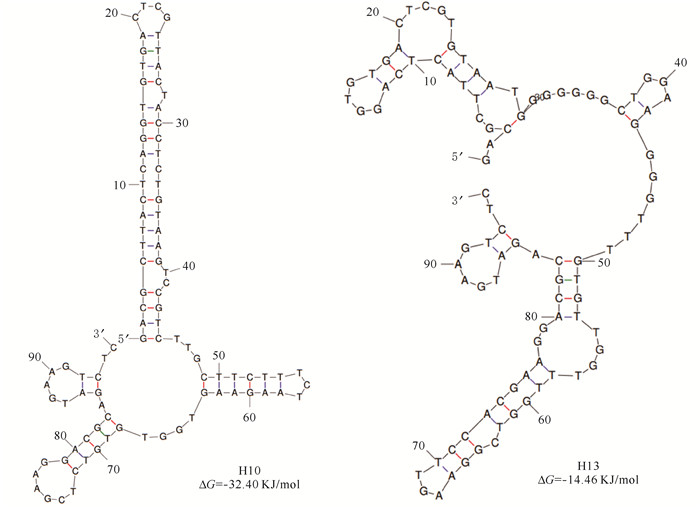
2.2 核酸适配体H10和H13的二级结构预测与分析
核酸适配体H10和H13的二级结构均能够形成独特复杂的茎环结构(图 2),且H10的吉布斯自由能(ΔG=-32.4 KJ/mol)低于H13 (ΔG=-14.46 KJ/mol),表明H10的结构比H13稳定。
|
| 图 2 核酸适配体H10和H13的二级结构预测 Fig.2 Secondary structures prediction of aptamers H10 and H13 |
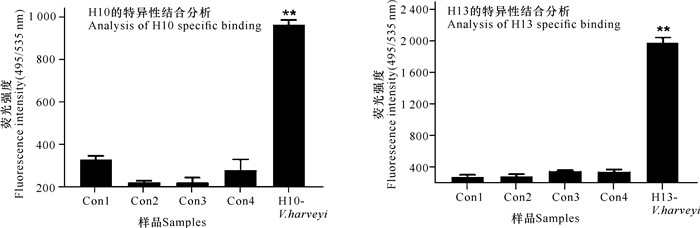
2.3 核酸适配体H10和H13的特异性分析
使用流式细胞术对筛选获得的核酸适配体H10和H13的特异性进行检测(图 3),结果显示,实验组中哈维氏弧菌分别与核酸适配体H10和H13孵育后所检测到的荧光值高,并显著高于4组对照组(Con1、Con2、Con3、Con4),表明核酸适配体H10和H13能够高特异性识别结合哈维氏弧菌。
|
| Con1、Con2、Con3和Con4表示FAM标记的非特异性起始库(200 nmol/L)分别与哈维氏弧菌、溶藻弧菌、嗜水气单胞菌、维氏气单胞菌的孵育结合; **P<0.01表示差异极显著 Con1, Con2, Con3, and Con4 denote the FAM-labeled non-specific initiatial library (200 nmol/L) bound with V.harveyi, V.alginolyticus, A.hydrophila and A.veronii, respectively.**P < 0.01 means extremely significant 图 3 流式细胞术检测核酸适配体H10和H13对哈维氏弧菌的识别 Fig.3 Detecting the recognition of aptamers H10 and H13 to V.harveyi by Flow cytometry |
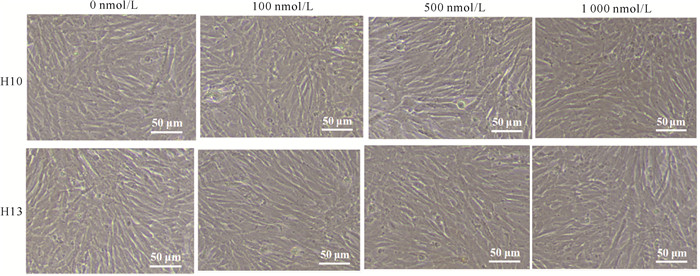
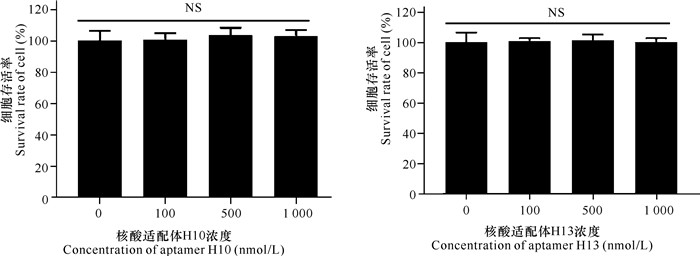
2.4 核酸适配体H10和H13的细胞毒性分析
核酸适配体H10和H13的细胞毒性光镜观察结果显示(图 4),不同浓度的核酸适配体与细胞孵育48 h后,与对照组(0 nmol/L)相比,细胞形态正常,无明显病变。当核酸适配体H10和H13的浓度为1 000 nmol/L时,实验组细胞的存活率均高于99%,并且实验组细胞存活率与对照组细胞无显著性差异(图 5),表明核酸适配体H10、H13无明显的细胞毒性,这与此前报道核酸适配体无细胞毒性的研究结果一致[10, 13, 16, 19]。
|
| 图 4 不同浓度的核酸适配体孵育细胞后的光镜结果 Fig.4 Light microscopy results after incubating with aptamers of different concentrations |
|
| NS表示组间差异不显著 NS indicates no significant 图 5 不同浓度的核酸适配体孵育细胞后的存活率 Fig.5 Survival rate of cells incubated with aptamers of different concentrations |
2.5 核酸适配体H10和H13的靶标性质分析
与对照组相比,实验组中胰酶消化处理后哈维氏弧菌的荧光值均显著降低(图 6),表明核酸适配体H10和H13对哈维氏弧菌表面的靶位点均为膜蛋白或膜蛋白相关结构。
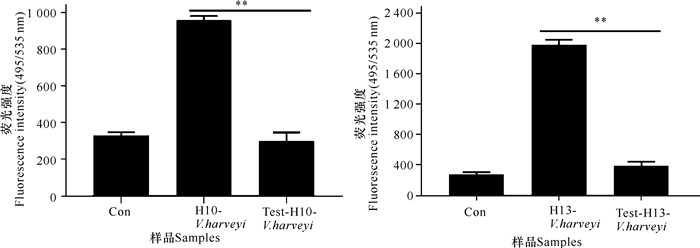
|
| Con表示FAM标记的非特异性起始库(200 nmol/L)和哈维氏弧菌的孵育结合;**P<0.01表示差异极显著 Con was FAM-labeled non-specific initial library (200 nmol/L) bound with V.harveyi; **P < 0.01 means extremely significant 图 6 核酸适配体H10和H13在哈维氏弧菌表面的靶标分子特性 Fig.6 Target molecule characterization of aptamers H10 and H13 on the surface of V.harveyi |
3 讨论
哈维氏弧菌是危害卵形鲳鲹、石斑鱼、对虾等大宗海水养殖动物的主要弧菌病原之一,其所导致的疾病具有高发、频发、死亡率高等特点,严重危害着我国海水养殖业的健康可持续发展[1-4, 17]。而针对哈维氏弧菌的快速检测诊断技术和抑菌技术等进行系统研究,并开发出具有市场发展潜力的快速检测试剂盒和抗病害功能产品,将有助于高效控制海水养殖中哈维氏弧菌的危害。
目前,已报道了一些针对水生病原微生物的特异性核酸适配体[11],包括溶藻弧菌(Vibrio alginolyticus)[20-22],创伤弧菌(Vibrio vulnificus)[23],沙门氏菌(Salmonella typhimurium)[24],卵形鲳鲹源神经坏死病毒(Trachinotus ovatus nervous necrosis virus)[25],病毒性出血性败血症病毒(Viral hemorrhagic septicemia virus)[26],中华鳖虹彩病毒(Soft-shelled turtle iridovirus)[14],赤点石斑鱼神经坏死症病毒(Red spotted grouper nervous necrosis virus)[27, 28],牙鲆弹状病毒(hirame rhabdovirus)[29],石斑鱼虹彩病毒(Singapore grouper iridovirus)[16, 19],草鱼呼肠孤病毒(Salmonella typhimurium)[30, 31]等。当前,SELEX技术尚未实现自动化和标准化,因此将不同物质作为靶标进行核酸适配体的筛选时,由于靶物质大小、性质等诸多差异,造成所需的SELEX技术筛选轮数和参数均显著不同,这就使得筛选高特异性识别靶物质的核酸适配体的难度显著提升。例如,Liang等[22]经过多达35轮的反复筛选,最终获得高特异性识别狂犬病病毒感染细胞的核酸适配体,而Yu等[16]仅通过11轮的SELEX筛选就获得了特异性识别石斑鱼虹彩病毒的核酸适配体。在本研究中,以哈维氏弧菌活菌为靶标进行核酸适配体的筛选,综合运用SELEX技术、磁性分离技术、流式细胞术等,经过9轮筛选获得能够高特异性、高亲和性识别哈维氏弧菌活菌的两条ssDNA核酸适配体H10和H13,随后二级结构预测分析表明H10和H13均能够形成独特复杂的茎环结构,而且核酸适配体H10和H13对哈维氏弧菌均具有较好的特异性结合能力(图 3),并且对细胞无毒副作用(图 4)。进一步研究发现,核酸适配体H10和H13对哈维氏弧菌表面的靶位点均为膜蛋白或膜蛋白相关结构(图 6)。据报道,核酸适配体能够在二级结构的基础上,通过氢键、G四联体、范德华力、疏水作用力、碱基堆积等折叠形成更复杂的三级结构,进而与靶物质发生特异性识别和结合[11]。目前,大规模应用核酸适配体的限制条件之一是合成成本较高。然而,考虑到原代核酸适配体中两端的引物序列等核苷酸序列或结构域对于其特异性是必需的,因此在未来研究中,有必要对核酸适配体的高特异性识别靶物质的核苷酸序列等必需序列进行分析鉴定,这对于优化核酸适配体结构、降低其合成成本、研发基于核酸适配体的市场化功能产品具有重要意义[32]。
核酸适配体作为新型的核酸分子探针,具有特异性高、亲和性强、无免疫原性、稳定性高、高温导致的结构变性具有可逆性、易于合成和化学修饰、批次间质量稳定等诸多优点,因此核酸适配体已成为广受关注的新型检测和治疗工具,并广泛应用于病原检测、疾病诊断、靶向药物开发和细胞功能分析等生命科学各领域的研究中[11, 33-37]。例如,利用核酸适配体能够高特异性识别病原微生物的特性,构建高灵敏的检测技术,用于病原快速检测和疾病精确诊断[11]。Li等[38]利用高特异性识别结合石斑鱼虹彩病毒感染细胞的核酸适配体Q3构建出新型的酶联核酸适配体吸附检测技术(Aptamer-based Enzyme-Linked Apta-Sorbent Assay, ELASA),成功检测出样品中石斑鱼虹彩病毒,灵敏度可媲美PCR技术。Yu等[39]利用特异性识别溶藻弧菌的核酸适配体VA2构建了VA2-ELASA技术,该技术可用于致病性溶藻弧菌的快速检测,具有特异性强、灵敏度高的特点。Zhou等[40]以石斑鱼神经坏死病毒的衣壳蛋白(Coat Protein, CP)为靶标,筛选获得特异性识别CP蛋白的核酸适配体A10,并建立了适配体-衣壳蛋白-适配体的“三明治”夹心酶联吸附检测技术,能够用于鱼类神经坏死病毒的精确快速检测。余庆等[41]利用核酸适配体Q5构建出核酸适配体Q5荧光分子检测探针(Aptamer Q5-based fluorescent molecular probe, Q5-AFMP),并证实Q5-AFMP能够高特异性、高灵敏性识别石斑鱼虹彩病毒,有望开发出实用的病毒快速检测功能产品。此外,基于核酸适配体开发的高灵敏生物传感器,已成功应用于金黄色葡萄球菌、副溶血弧菌和沙门氏菌等多种致病菌的快速精确检测[34]。基于本研究筛选得到核酸适配体H10和H13,具有高特异性和高亲和性识别结合哈维氏弧菌的特性,因此,未来可利用核酸适配体H10和H13开展哈维氏弧菌快速检测诊断技术的研究,以期实现对哈维氏弧菌病的快速诊断、实时监控和有效预防。
4 结论本研究以重要的水生病原哈维氏弧菌为靶标,运用SELEX技术筛选获得ssDNA核酸适配体H10和H13,这两种核酸适配体在识别哈维氏弧菌过程中显现出高特异性、高亲和性,并且细胞无毒性等优点。该成果有望用于构建成本低、快速便捷、灵敏度高的哈维氏弧菌检测技术,并在水生病原快速检测技术研发领域展现出良好的应用前景。
| [1] |
李鹏飞, 余庆, 覃仙玲, 等. 广西北部湾海水养殖业现状与病害防控技术体系研究展望[J]. 广西科学, 2018, 25(1): 15-25. |
| [2] |
AUSTIN B. Vibrios as causal agents of zoonoses[J]. Veterinary Microbiology, 2010, 140(3/4): 310-317. |
| [3] |
闫茂仓, 陈少波, 单乐州, 等. 海水养殖动物致病弧菌的研究进展[J]. 水产科学, 2009, 28(8): 475-481. DOI:10.3969/j.issn.1003-1111.2009.08.014 |
| [4] |
崔婧, 范雪亭, 刘文竹, 等. 华南地区海水养殖鱼类主要弧菌病原的分离与鉴定[J]. 海南大学学报: 自然科学版, 2014, 32(3): 244-251. DOI:10.3969/j.issn.1004-1729.2014.03.010 |
| [5] |
LIU C H, CHEN J C. Effect of ammonia on the immuneresponse of white shrimp Litopenaeus vannamei and its susceptibility to Vibrio alginolyticus[J]. Fish & Shellfish Immunology, 2004, 16(3): 321-334. |
| [6] |
曾德乾, 冯娟, 徐力文, 等. 海水养殖鱼哈维弧菌分离株的耐药谱型分析[J]. 中国水产科学, 2015, 22(1): 129-138. |
| [7] |
高晓建, 姚东瑞, 孙晶晶, 等. 4株长牡蛎致病性哈维氏弧菌鉴定及其毒力基因检测[J]. 海洋湖沼通报, 2015(3): 87-96. |
| [8] |
SITHIGORNGUL P, RUKPRATANPORN S, PECH-ARABURANIN N, et al. A simple and rapid immunochromatographic test strip for detection of pathogenic isolates of Vibrio harveyi[J]. Journal of Microbiological Methods, 2007, 71(3): 256-264. DOI:10.1016/j.mimet.2007.09.012 |
| [9] |
PHIANPHAK W, RENGPIPAT S, RUKPRATAN-PORN S, et al. Production of monoclonal antibodies for detection of Vibrio harveyi[J]. Diseases of Aquatic Organisms, 2005, 63(2/3): 161-168. |
| [10] |
李鹏飞, 余庆, 罗永巨, 等. 广西水产疫病防控技术体系建设与水产养殖业高质化生态发展展望[J]. 广西科学院学报, 2019, 35(3): 161-165. |
| [11] |
YU Q, LIU M Z, WEI S N, et al. Research progress and prospects for the use of aptamers in aquaculture biosecurity[J]. Aquaculture, 2021, 534: 736257. DOI:10.1016/j.aquaculture.2020.736257 |
| [12] |
LIU M Z, XIAO H H, WU S T, et al. Aptamer-based high-throughput screening model for medicinal plant drugs against SGIV[J]. Journal of Fish Diseases, 2020, 43(11): 1479-1482. DOI:10.1111/jfd.13254 |
| [13] |
ZHOU J H, ROSSI J J. Therapeutic potential of apta-mer-siRNA conjugates for treatment of HIV-1[J]. BioDrugs, 2012, 26(6): 393-400. DOI:10.1007/BF03261896 |
| [14] |
LI P F, ZHOU L L, YU Y P, et al. Characterization of DNA aptamers generated against the soft-shelled turtle iridovirus with antiviral effects[J]. BMC Veterinary Research, 2015, 11(1): 245. DOI:10.1186/s12917-015-0559-6 |
| [15] |
SYED M A, PERVAIZ S. Advances in aptamers[J]. Oligonucleotides, 2010, 20(5): 215-224. DOI:10.1089/oli.2010.0234 |
| [16] |
YU Q, LIU M Z, XIAO H H, et al. Selection and characterization of aptamers for specific detection of iridovirus disease in cultured hybrid grouper (Epinephelus fuscoguttatus♀×E.lanceolatus♂)[J]. Chinese Journal of Analytical Chemistry, 2020, 48(5): 650-661. DOI:10.1016/S1872-2040(20)60021-4 |
| [17] |
YU Q, LIU M Z, LI F, et al. Identification and characterization of marine pathogenic vibrios in cultured golden pompano (Trachinotus ovatus) in Guangxi, China[J]. Annals of Marine Science, 2018, 2(1): 16-19. |
| [18] |
余庆, 李菲, 王一兵, 等. 广西北部湾大宗海水养殖鱼类卵形鲳鲹感染溶藻弧菌及其致病性研究[J]. 广西科学, 2018, 25(1): 68-73. |
| [19] |
LI P F, YAN Y, WEI S N, et al. Isolation and characterization of a new class of DNA aptamers specific binding to Singapore grouper iridovirus (SGIV) with antiviral activities[J]. Virus Research, 2014, 188: 146-154. DOI:10.1016/j.virusres.2014.04.010 |
| [20] |
YU Q, LIU M Z, SU H F, et al. Selection and characterization of ssDNA aptamers specifically recognizing pathogenic Vibrio alginolyticus[J]. Journal of Fish Diseases, 2019, 42(6): 851-858. DOI:10.1111/jfd.12985 |
| [21] |
ZHENG J, TANG X M, WU R X, et al. Identification and characteristics of aptamers against inactivated Vibrio alginolyticus[J]. LWT-Food Science and Technology, 2015, 64(2): 1138-1142. DOI:10.1016/j.lwt.2015.07.021 |
| [22] |
LIANG H, HU G, ZHANG T, et al. Isolation of ssDNA aptamers that inhibit rabies virus[J]. International Immunopharmacology, 2012, 14(3): 341-347. DOI:10.1016/j.intimp.2012.06.019 |
| [23] |
YAN W L, GU L D, LIU S, et al. Identification of a highly specific DNA aptamer for Vibrio vulnificus using systematic evolution of ligands by exponential enrichment coupled with asymmetric PCR[J]. Journal of Fish Diseases, 2018, 41(12): 1821-1829. DOI:10.1111/jfd.12891 |
| [24] |
PARK H C, BAIG I A, LEE S C, et al. Development of ssDNA aptamers for the sensitive detection of Salmonella typhimurium and Salmonella enteritidis[J]. Applied Biochemistry and Biotechnology, 2014, 174(2): 793-802. DOI:10.1007/s12010-014-1103-z |
| [25] |
YU Q, LIU M Z, WEI S N, et al. Characterization of ssDNA aptamers specifically directed against Trachinotus ovatus NNV (GTONNV)-infected cells with antiviral activities[J]. Journal of General Virology, 2019, 100(3): 380-391. |
| [26] |
PUNNARAK P, SANTOS M D, HWANG S D, et al. RNA aptamers inhibit the growth of the fish pathogen viral hemorrhagic septicemia virus (VHSV)[J]. Marine Biotechnology, 2012, 14(6): 752-761. DOI:10.1007/s10126-012-9448-1 |
| [27] |
ZHOU L L, WANG S W, YU Q, et al. Characterization of novel aptamers specifically directed to red-spotted grouper nervous necrosis virus (RGNNV)-infected cells for mediating targeted siRNA delivery[J]. Frontiers in Microbiology, 2020, 11: 660. DOI:10.3389/FMICB.2020.00660 |
| [28] |
ZHOU L L, LI P F, YANG M, et al. Generation and characterization of novel DNA aptamers against coat protein of grouper nervous necrosis virus (GNNV) with antiviral activities and delivery potential in grouper cells[J]. Antiviral Research, 2016, 129: 104-114. DOI:10.1016/j.antiviral.2016.02.009 |
| [29] |
HWANG S D, MIDORIKAWA N, PUNNARAK P, et al. Inhibition of hirame rhabdovirus growth by RNA aptamers[J]. Journal of Fish Diseases, 2012, 35(12): 927-934. DOI:10.1111/jfd.12000 |
| [30] |
YU Q, LIU M Z, WU S T, et al. Generation and characterization of aptamers against grass carp reovirus infection for the development of rapid detection assay[J]. Journal of Fish Diseases, 2020, 44(1): 33-44. |
| [31] |
余庆, 刘明珠, 肖贺贺, 等. 特异性识别草鱼呼肠孤病毒感染细胞的ssDNA核酸适配体筛选与鉴定[J]. 南方农业学报, 2020, 51(12): 3057-3065. |
| [32] |
LI P F, YU Q, ZHOU L L, et al. Probing and characterizing the high specific sequences of ssDNA aptamer against SGIV-infected cells[J]. Virus Research, 2018, 246: 46-54. DOI:10.1016/j.virusres.2018.01.006 |
| [33] |
YU Q, LIU M Z, WU S T, et al. Specific aptamer-based probe for analyzing biomarker MCP entry into Singapore grouper iridovirus-infected host cells via clathrin-mediated endocytosis[J]. Frontiers in Microbiology, 2020, 11: 1206. DOI:10.3389/fmicb.2020.01206 |
| [34] |
DUAN N, WU S J, DAI S L, et al. Advances in aptasensors for the detection of food contaminants[J]. Analyst, 2016, 141(13): 3942-3961. DOI:10.1039/C6AN00952B |
| [35] |
PARASHAR A. Aptamers in therapeutics[J]. Journal of Clinical and Diagnostic Research, 2016, 10(6): BE01-6. DOI:10.7860/JCDR/2016/18712.7922 |
| [36] |
SUN H, ZU Y. A highlight of recent advances in apta-mer technology and its application[J]. Molecules, 2015, 20(7): 11959-11980. DOI:10.3390/molecules200711959 |
| [37] |
YU Q, LIU M, WEI S, et al. Identification of major capsid protein as a potential biomarker of grouper iridovirus-infected cells using aptamers selected by SELEX[J]. Frontiers in Microbiology, 2019, 10: 2684. DOI:10.3389/fmicb.2019.02684 |
| [38] |
LI P, ZHOU L, WEI J, et al. Development and characterization of aptamer-based enzyme-linked apta-sorbent assay for the detection of Singapore grouper iridovirus infection[J]. Journal of Applied Microbiology, 2016, 121(3): 634-643. DOI:10.1111/jam.13161 |
| [39] |
YU Q, LIU M, SU H, et al. Selection and characterization of ssDNA-aptamers specifically recognized pathogenic Vibrio alginolyticus[J]. Journal of Fish Diseases, 2019, 42(6): 851-858. DOI:10.1111/jfd.12985 |
| [40] |
ZHOU L, LI P, NI S, et al. Rapid and sensitive detection of redspotted grouper nervous necrosis virus (RGNNV) infection by aptamer-coat protein-aptamer sandwich enzyme-linked apta-sorbent assay (ELASA)[J]. Journal of Fish Diseases, 2017, 40(12): 1831-1838. |
| [41] |
余庆, 刘明珠, 肖贺贺, 等. 基于核酸适配体Q5的石斑鱼虹彩病毒快速检测技术[J]. 广西科学院学报, 2019, 35(3): 193-199. |



