2. 广西职业技术学院, 广西南宁 530226
2. Guangxi Vocational and Technical College, Nanning, Guangxi, 530226, China
酿酒酵母(Saccharomyces cerevisiae)是重要的工业生产菌株,在工业应用中会面临各种各样的环境压力。高糖环境是酿酒酵母在乙醇发酵过程中常遇到的一种压力条件,筛选耐高糖酿酒酵母菌株,研究酿酒酵母耐高糖机制,了解耐高糖性状与基因表达、调控的关系,对选育、改造优良菌株和提高工业生产效率意义重大。Tup1基因是一个普遍抑制因子,负责调节酿酒酵母在不同环境下的转录表达,有研究发现敲除Tup1基因后,明显解抑制的基因不少于300个[1]。由4个Tup1和1个Ssn6亚基组成的Tup1-Ssn6复合体是最早被识别的共阻遏复合体之一[2, 3]。酿酒酵母Tup1蛋白由713个氨基酸组成,从功能上分为3个蛋白-蛋白互作区域,不同区域参与不同基因的转录调控[4]。Lin等[5]利用基因缺失研究Mig1、Tup1和Ssn6对酵母葡萄糖抑制作用的影响,发现Tup1基因与其他两个基因不同,缺失后对酵母解除葡萄糖抑制麦芽糖利用的能力有负作用。Han等[6]研究发现调节糖酵解和糖原异生转录所需的3, 5-二磷酸磷脂酰肌醇激酶受Tup1基因抑制。丛建国等[7]利用生物信息学研究Tup1基因网络的非线性行为特征,基因缺失突变芯片数据显示,Tup1对13号染色体上的基因是激活而不是抑制作用。Rizzo等[8]利用MNase-ChIP-chip法对酿酒酵母野生菌株和Tup1 Δ菌株的核小体进行测序分析,首次获得Tup1蛋白在染色质上的结合位置。最新研究发现, Tup1基因参与白色念珠菌毒性相关基因的表达调控[9],Rania等[10]发现Tup1基因表达量与新设计合成的抗白色念珠菌药物SR的活性之间存在正相关性,为今后抑制白色念珠菌及其他依赖Tup1基因的真菌感染药物设计提供参考。在前期研究中,本研究团队对筛选获得的耐高糖酿酒酵母突变菌株进行转录组测序分析[11],PheNetic网络分析结果显示, Tup1基因是突变菌株突变性状的主要诱发因子之一,在互作网络中直接或间接影响众多其他基因。本研究通过测定Tup1基因缺失菌株在高糖培养基中的生长和发酵情况,更准确地了解Tup1基因对酿酒酵母耐高糖性状的影响,为后续研究酿酒酵母耐高糖机制和酿酒酵母菌株改造提供理论依据。
1 材料与方法 1.1 材料 1.1.1 菌种和质粒Saccharomyces cerevisiae UV02_HG(以下简称UV02_HG)为本研究团队以甘蔗糖厂废弃物中筛选的酿酒酵母菌株为出发菌株紫外诱变获得。Saccharomyces cerevisiae UV02_HGΔTup1 (以下简称UV02_HGΔTup1)为缺失Tup1基因的UV02_HG菌株。pUG6和pSH65质粒保存于国家非粮生物质能源工程技术研究中心。
1.1.2 培养基酵母膏胨葡萄糖(YPD)培养基:胰蛋白胨2%,酵母提取物1%,葡萄糖2%,固体培养基添加2%琼脂粉;G418在液体培养基中的终浓度为200 μg/mL,在固体培养基中的终浓度为300 μg/mL;Zeocin的使用浓度为50 μg/mL。
葡萄糖梯度培养基:胰蛋白胨2%,酵母提取物1%,葡萄糖浓度梯度分别为20%(YP20)、30%(YP30)、40%(YP40)。
产孢培养基:酵母粉0.25%,葡萄糖0.1%,KCl 0.18%,NaAc 0.82%,琼脂粉2%。
1.1.3 酶与试剂PCR试剂、DNA marker、限制性内切酶为TaKaRa公司产品;Yeast extract,Trytone为OXOID产品;抗生素、SDS、EDTA、This·HCl、山梨醇购自上海生工生物工程有限公司;TTC为Sigma公司产品;Tris饱和酚为Solarbio公司产品;蜗牛酶购自上海源叶生物科技有限公司。
1.1.4 主要仪器设备超净工作台型号为苏州净化SW-CJ,PCR仪型号为Biometra Tpersonal P,分光光度计型号为Bechman coulter DU800,电转化仪型号为BTX ECM630,显微镜型号为Nikon 80i,高效气相色谱仪型号为Agilent 6890N,离心机型号为HETTICH Mimiko 200R,凝胶成像系统为Biometra。
1.2 方法 1.2.1 Tup1基因敲除酿酒酵母单倍体(MATa型和MATα型)细胞制备参照文献[12],阳性重组子PCR验证参照文献[13],单倍体细胞杂交复倍及抗性消除参照文献[14, 15]。敲除组件扩增引物名称和序列见表 1(小写碱基是目的基因读码框两侧同源序列,大写碱基是pUG6质粒上loxP两端序列)。基因缺失菌株PCR验证引物见表 2。
| 引物名称 Name of primers |
引物序列(5′-3′) Sequence of primers (5′-3′) |
| Tup1 Kan-F | ttcacgccaagttacctttcgcacaccactactctttttctatt- gCCAGCTGAAGCTTCGTACGC |
| Tup1 Kan-R | cacaggaaaaggaggggaagggatgaatggtgaggaaa- gtaactgGCATAGGCCACTAGTGGATCTG |
| 引物名称 Name of primers |
引物序列(5′-3′) Sequence of primers (5′-3′) |
| Tup1-A | CCTCTATCTGTCCTTCTG |
| Tup1-D | GAACCGCAATATTCAGAAAC |
| Kan-B | GGATGTATGGGCTAAATG |
| Kan-C | TCGACATCATCTGCCCAGATG |
1.2.2 不同浓度葡萄糖固体培养基上的生长情况
计算活化的新鲜菌液细胞浓度,每株取约1×109个细胞离心收集菌体,用无菌水重悬,使细胞初始浓度为1×109 cells/mL,再将浓度分别稀释至1×108,1×107,1×106,1×105,1×104 cells/mL,每个浓度吸取2 μL菌悬液,分别点到不同浓度葡萄糖固体培养基上,30℃培养2 d,观测菌株生长情况。
1.2.3 测定YPD培养基中的生长曲线计算活化的新鲜菌液细胞浓度,每株取约1.5×107个细胞转接至5 mL YPD培养基中,摇床培养(30℃,180 r/min),每隔4 h取样,用细胞计数法测定各菌株的生长曲线。
1.2.4 测定YP40培养基中的生长和发酵曲线计算活化的新鲜菌液细胞浓度,每株取约1.5×108个细胞转接至50 mL YP40培养基中,摇床培养(30℃,200 r/min),每隔4 h取样,用细胞计数法测定菌株的生长曲线,气相色谱测定乙醇发酵曲线。
1.2.5 测定细胞相对呼吸强度参照文献[16],用2, 3, 5-三苯基氯化四唑(TTC)法测定细胞的相对呼吸强度。按照1%(V/V)的接种量将活化的新鲜菌液接入5 mL YPD或YP40培养基中,30℃,180 r/min培养至对数生长期时离心收集菌体,转接至10 mL YPD或YP40培养基中,调整菌液浓度至OD600=1.0,向菌液中加入TTC(终浓度为0.5 mg/mL),30℃,180 r/min培养6 h后取5 mL离心收集菌体,用1 mL 95%乙醇重悬,剧烈震荡(约10 min)使细胞破壁,12 000 r/min离心10 min,测定上清液在480 nm下的吸光值,以出发菌株的呼吸强度为100%,根据测定结果计算每株菌的相对呼吸强度。
2 结果与分析 2.1 基因缺失菌株PCR验证以筛选的阳性重组子DNA为模板,用Tup1 -A、Kan-B、Kan-C、Tup1 -D引物进行PCR扩增验证,如图 1所示,两对引物在MATa型和MATα型单倍体DNA中分别扩增到大小约为300 bp的片段,片段送测序公司测序后证实目的基因片段缺失成功。

|
| M.DL2000 DNA marker;1.MATa型,Tup1-A和Kan-B引物扩增产物;2.MATa型,Kan-C和Tup1-D引物扩增产物;3.MATα型,Tup1-A和Kan-B引物扩增产物;4.MATα型,Kan-C和Tup1 -D引物扩增产物 M.DL2000 DNA marker, 1.MATa type, product amplified by primers Tup1-A and Kan-B, 2.MATa type, product amplified by primers Kan-C and Tup1-D, 3.MATα type, product amplified by primers Tup1-A and Kan-B, 4.MATα type, product amplified by primers Kan-C and Tup1 -D 图 1 PCR验证基因缺失 Fig.1 PCR verification of gene missing |
2.2 基因缺失菌株性状测定
通过PCR验证的单倍体基因缺失菌株,复倍和消抗后,进行性状测定。
2.2.1 不同浓度葡萄糖固体培养基上的生长情况如图 2所示,对不同浓度梯度细胞在固体培养基平板上生长形成的菌落大小、菌落数量进行分析,发现基因缺失菌株UV02_HGΔTup1在YPD培养基上的生长情况不如出发菌株UV02_HG,在YP20、YP30和YP40培养基上的生长情况优于出发菌株。说明Tup1基因缺失后,酿酒酵母细胞在YPD培养基上的生长受到抑制,但对高糖环境的适应能力却得到增强。
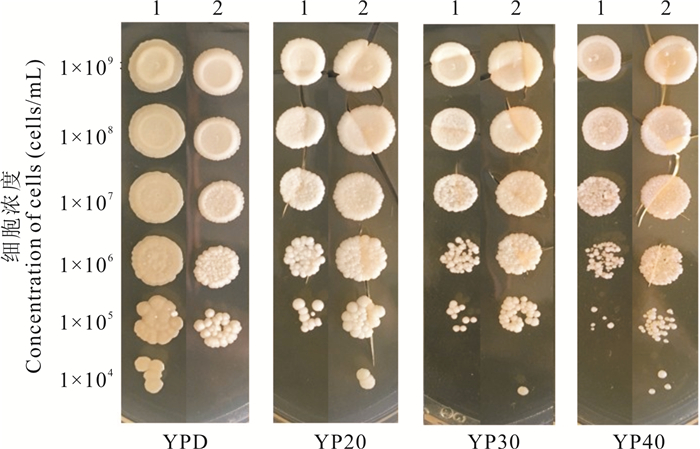
|
| 1.菌株UV02_HG,2.菌株UV02_HGΔTup1 1.Strain UV02_HG, 2.Strain UV02_HGΔTup1 图 2 菌株UV02_HG和UV02_HGΔTup1在不同浓度葡萄糖培养基上的生长情况 Fig.2 Growth situation of strains UV02_HG and UV02_HGΔTup1 in solid medium under different glucose concentration |
2.2.2 在YPD中的生长曲线
如图 3所示,在YPD培养基上,基因缺失菌株UV02_HGΔTup1的生长速率和最大细胞数都比出发菌株UV02_HG低,这与在YPD固体平板上的生长情况一致,说明Tup1基因的缺失影响了酵母细胞的正常生长。
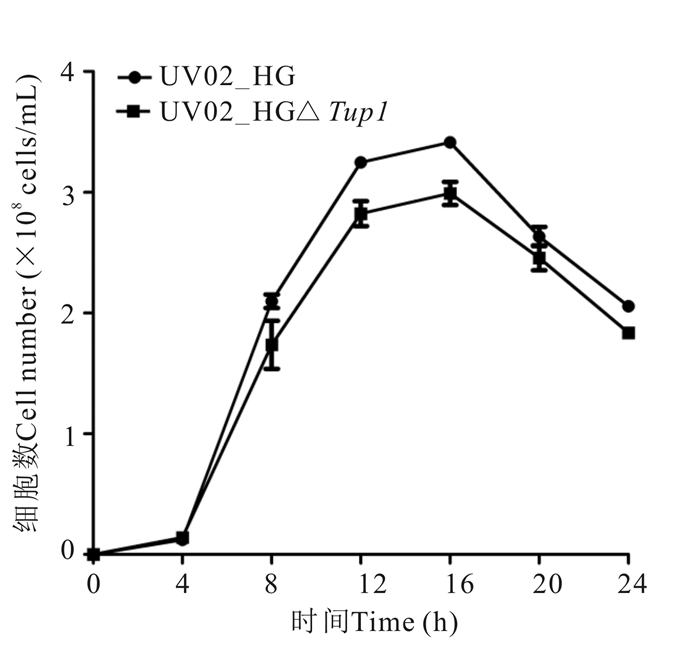
|
| 图 3 菌株UV02_HG和UV02_HGΔTup1在YPD培养基中的生长曲线 Fig.3 Growth curves of strains UV02_HG and UV02_HGΔTup1 in medium of YPD |
2.2.3 在YP40中的生长和发酵曲线
在YP40培养基中,基因缺失菌株UV02_HGΔTup1前期生长速率高于出发菌株,28 h后生长速率降低,最大细胞数为7.43×108 cells/mL,略小于出发菌株的最大细胞数7.60×108 cells/mL(图 4)。基因缺失菌株在YP40培养基中乙醇发酵能力也略优于出发菌株,最高乙醇产率平均达15.30%(图 5)。
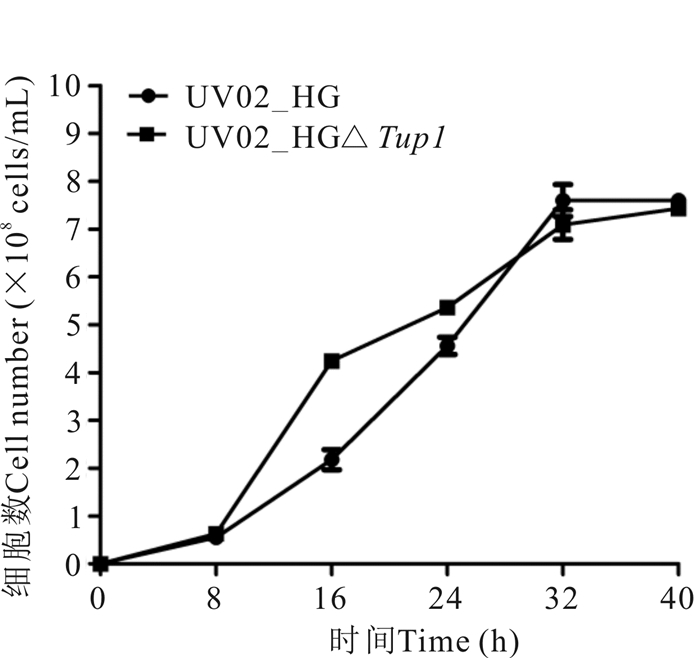
|
| 图 4 菌株UV02_HG和UV02_HGΔTup1在YP40培养基中的生长曲线 Fig.4 Growth curves of strains UV02_HG and UV02_HGΔTup1 in medium of YP40 |
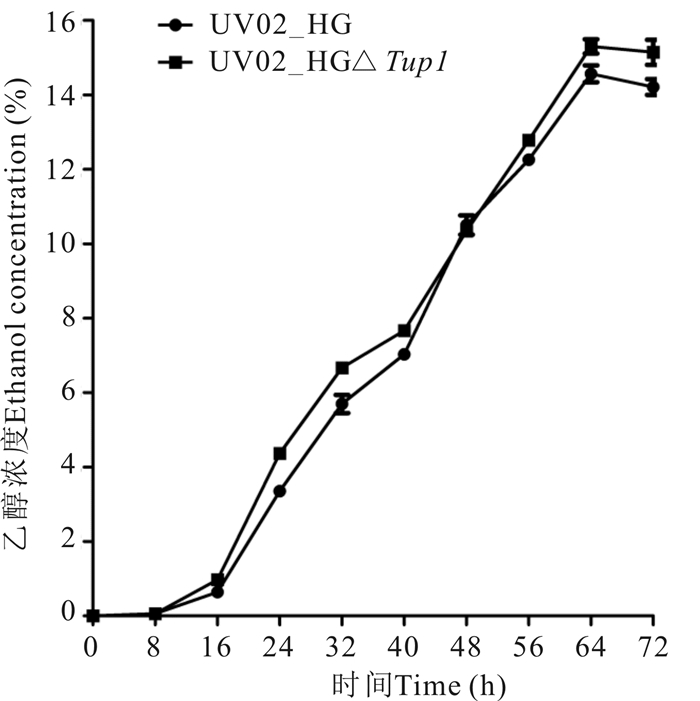
|
| 图 5 UV02_HG和UV02_HGΔTup1在YP40培养基中的乙醇发酵曲线 Fig.5 Ethanol fermentation curves of strains UV02_HG and UV02_HGΔTup1 in medium of YP40 |
2.2.4 相对呼吸强度检测
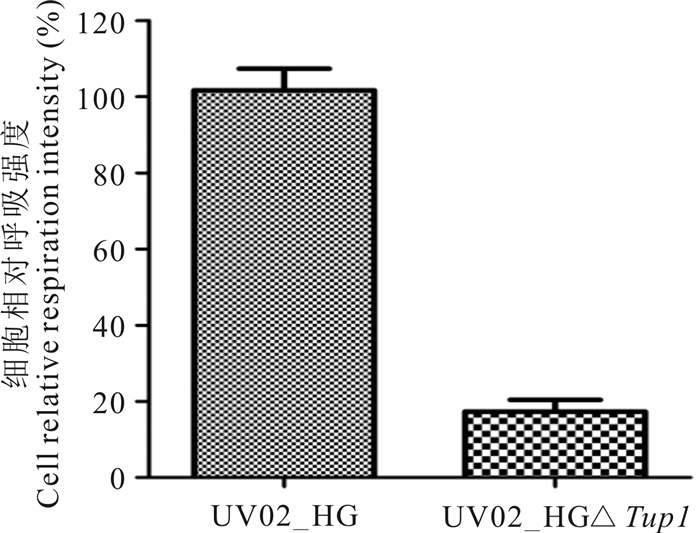
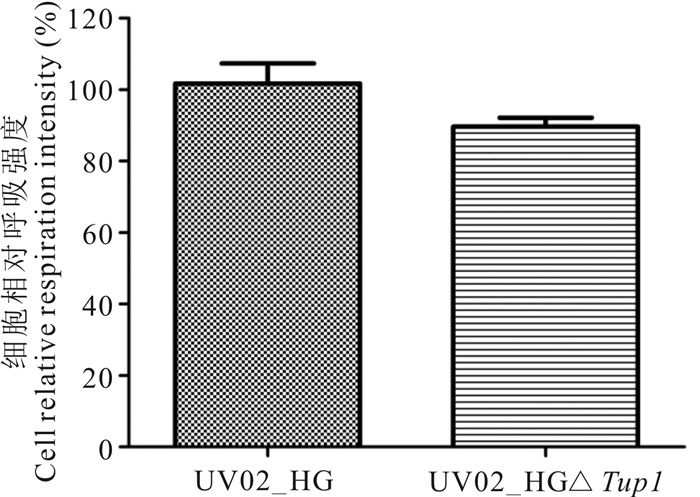
用TTC法测定在YPD和YP40培养基中处于对数生长期的两菌株的呼吸强度,测定结果如图 6和7所示。在YPD培养基中,菌株UV02_HGΔTup1的呼吸强度是UV02_HG的20.0%,在YP40培养基中升高至88.2%。
|
| 图 6 菌株UV02_HG和UV02_HGΔTup1在YPD培养基中的相对呼吸强度 Fig.6 Relative respiration intensity of strains UV02_HG and UV02_HGΔTup1 in medium of YPD |
|
| 图 7 菌株UV02_HG和UV02_HGΔTup1在YP40培养基中的相对呼吸强度 Fig.7 Relative respiration intensity of strains UV02_HG and UV02_HGΔTup1 in medium of YP40 |
3 讨论 3.1 Tup1基因对酿酒酵母耐高糖性状的影响
Tup1基因缺失后,菌株在YP40培养基中28 h前生长速率较快,28 h后有所降低,发酵能力整体提高,这与呼吸强度降低是相符的,乙醇发酵后期,无氧环境更有利于乙醇产量提高[17],说明生长后期菌株更偏向厌氧发酵。但该结果与之前研究中突变菌株生长和发酵能力提高,呼吸强度降低,Tup1基因表达量却上调的结果[11]相比,存在矛盾。Green等[18]通过微列阵技术分析了野生酿酒酵母菌株和Tup1基因保守序列区5个定点突变体的基因表达情况,发现5个突变体解抑制的对象不同,表达上调的基因数量不等;又通过Suc2、Spi1、Hsp12这3个基因在Tup1 Δ及5个定点突变体中表达量的对比,发现Tup1 Δ菌株比点突变的突变株解抑制作用更强。可见,酵母细胞自身表达调控是一项牵一发而动全身的精细工程,前期研究中的突变菌株是经紫外诱变筛选获得,Tup1基因表达量上调这一因素的影响可能被其他基因突变的作用抵消或覆盖,本研究只对Tup1基因进行缺失,影响因素相对单一,所以菌株性状的改变与Tup1基因的缺失有直接关系,可见Tup1基因缺失对酿酒酵母耐高糖性状有正向作用。
3.2 Tup1基因对酿酒酵母呼吸强度的影响转录因子Tup1与Cyc8(Ssn6)形成复合体,正常生长条件下参与酿酒酵母超过300个基因的转录调控,涉及多种代谢途径,包括孢子形成及减数分裂、蛋白转运、高渗应激、葡萄糖代谢、淀粉及氧的利用、DNA修复等[19, 20]。Wang等[21]研究发现,Cyc8-Tup1复合体和Dbp2联合作用抑制一系列基因表达,GO富集分析显示这些基因主要集中在糖质新生、细胞呼吸和对碳源的选择利用上。本文中菌株UV02_HGΔTup1在YPD培养基中的呼吸强度严重降低,影响了细胞的正常生长,而在高糖环境下其呼吸强度明显提高,可能是由于在高渗压力下的应激反应中,Tup1基因的缺失诱发其他调控因子对细胞呼吸进行补偿。接下来的工作中可以利用转录组测序和蛋白互作等方法深入研究Tup1基因对酿酒酵母呼吸途径的调节,呼吸强度变化与酿酒酵母耐高糖性状的具体关系,以及在Tup1缺失的情况下高渗应激反应如何补偿酵母细胞呼吸强度。
4 结论Tup1基因缺失显著降低正常生长条件下酿酒酵母细胞的呼吸强度。在高糖环境下,Tup1基因缺失细胞呼吸强度有所恢复,菌株生长速率和发酵乙醇产率优于出发菌株,由此可以推断,Tup1基因参与调控细胞呼吸,高糖压力下Tup1基因缺失可能诱发其他呼吸补偿途径来调控细胞的高糖应激反应。
| [1] |
GREEN S R, JOHNSON A D. Promoter-dependent roles for the Srb10 cyclin-dependent kinase and the Hda1 deacetylase in Tup1-mediated repression in Saccharomyces cerevisiae[J]. Molecular Biology of the Cell, 2004, 15(9): 4191-4202. DOI:10.1091/mbc.e04-05-0412 |
| [2] |
TANAKA N, MUKAI Y. Yeast Cyc8p and Tup1p proteins function as coactivators for transcription of Stp1/2p-dependent amino acid transporter genes[J]. Biochemical and Biophysical Research Communications, 2015, 468(1/2): 32-38. |
| [3] |
VARANASI U S, KLIS M, MIKESELL P B, et al. The Cyc8(Ssn6)-Tup1 corepressor complex is composed of one Cyc8 and four Tup1 subunits[J]. Molecular and Cellular Biology, 1996, 16(12): 6707-6714. DOI:10.1128/MCB.16.12.6707 |
| [4] |
ZHANG Z, VARANASI U, TRUMBLY R J. Functional dissection of the global repressor Tup1 in yeast:Dominant role of the C-terminal repression domain[J]. Genetics, 2002, 161(3): 957-969. |
| [5] |
LIN X, ZHANG C Y, BAI X W, et al. Effects of MIG1, TUP1 and SSN6 deletion on maltose metabolism and leavening ability of baker's yeast in lean dough[J]. Microbial Cell Factories, 2014, 13(1): 93. DOI:10.1186/s12934-014-0093-4 |
| [6] |
HAN B K, EMR S D. The phosphatidylinositol 3, 5-bisphosphate(PI(3, 5)P2)-dependent Tup1 conversion (PIPTC) regulates metabolic reprogramming from glycolysis to gluconeogenesis[J]. The Journal of Biological Chemistry, 2013, 288(28): 20633-20645. DOI:10.1074/jbc.M113.452813 |
| [7] |
丛建国, 王志伟, 任德华, 等. Tup1突变对转录抑制局部网络中基因表达的非线性影响[J]. 生物信息学, 2009, 7(4): 272-276. |
| [8] |
RIZZO J M, MIECZKOWSKI P A, BUCK M J, et al. Tup1 stabilizes promoter nucleosome positioning and occupancy at transcriptionally plastic genes[J]. Nucleic Acids Research, 2011, 39(20): 8803-8819. DOI:10.1093/nar/gkr557 |
| [9] |
SOPHIA R, ENRICO G, SELENE M, et al. Ahr1 and Tup1 contribute to the transcriptional control of virulence-associated gene in Candida albicans[J]. Molecular Biology and Physiology, 2020, 11(2): e00206-20. |
| [10] |
RANIA H, SAMEH S M S, ABRAR I A, et al.Design and synthesis of new drug inhibitors of Candida albicans hyphae and biofilm formation by upregulating the expression of TUP1 transcription repressor gene[J].European Journal of Pharmaceutical Sciences, 2020, 148: 105327.DOI: https://doi.org/10.1016/j.ejps.2020.105327.
|
| [11] |
CHEN Y, LU Z L, CHEN D, et al. Transcriptomic analysis and driver mutant prioritization for differentially expressed genes from a Saccharomyces cerevisiae strain with high glucose tolerance generated by UV irradiation[J]. RSC Advances, 2017, 7: 38784-38797. DOI:10.1039/C7RA06146C |
| [12] |
肖冬光, 刘青, 李静, 等. 酿酒酵母单倍体制备方法的优化[J]. 酿酒科技, 2004, 4: 21-22. |
| [13] |
宋浩雷, 郭晓贤, 杨月梅, 等. 酿酒酵母ADH3基因敲除[J]. 工业微生物, 2006, 36(4): 28-32. |
| [14] |
罗贞贞, 陈小玲, 练梅华, 等. 酿酒酵母Mbp1缺陷型菌株的构建及其乙醇发酵特性[J]. 微生物学通报, 2015, 42(11): 2108-2114. |
| [15] |
王艳尊, 雷娟娟, 江贤章, 等. 酿酒酵母adh2和adh6双基因缺失突变株的构建[J]. 微生物学通报, 2009, 36(2): 211-216. |
| [16] |
韦缘, 张穗生, 陈东, 等. 两株高糖分利用能力的酿酒酵母呼吸突变体选育[J]. 生物技术通报, 2012, 7: 158-162. |
| [17] |
ANTONIUS J A M, DEREK A A, ELEONORA B, et al. Alcoholic fermentation of carbon sources in biomass hydrolysates by Saccharomyces cerevisiae:Current status[J]. Antonie Van Leeuwenhoek, 2006, 90(4): 391-418. DOI:10.1007/s10482-006-9085-7 |
| [18] |
GREEN S R, JOHNSON A D. Genome-wide analysis of the functions of a conserved surface on the corepressor Tup1[J]. Molecular Biology of the Cell, 2005, 16(6): 2605-2613. DOI:10.1091/mbc.e05-02-0126 |
| [19] |
TANIA M M, SHARON D. Transcriptional repression by Tup1-Ssn6[J]. Biochemistry and Cell Biology, 2006, 84(4): 437-443. DOI:10.1139/o06-073 |
| [20] |
SMITH R L, JOHNSON A D. Turning genes off by Ssn6-Tup1:A conserved system of transcriptional repression in eukaryotes[J]. Trends in Biochemical Sciences, 2000, 25(7): 325-330. DOI:10.1016/S0968-0004(00)01592-9 |
| [21] |
WANG S W, ZHING X, PETE E P, et al. Metabolic adaptation to uutrients involves coregulation of gene expression by the RNA helicase Dbp2 and the Cyc8 corepressor in Saccharomyces cerevisiae[J]. Genes Genomes Genetics, 2017, 7(7): 2235-2247. |



