已有研究显示,看似均一的微生物群体,其细胞间却在遗传、生理、生化或者个体行为上存在很大的差异[1],这种遗传上或表观上的异质性是微生物细胞能够适应并生存于逆境,或持久生存并给人带来疾病的基础[2, 3]。该异质性对诸如抗生素或者杀菌剂抗性的研究、工业发酵的生产力和稳定性、食物防腐的有效性,以及病原微生物的致病潜力等都有非常重要的影响[4-8]。传统上对微生物的认识多数是通过研究其群体样品而得到的统计平均信息,但对微生物的深入研究、认识微生物细胞与环境的相互作用,仅靠传统的群体分析难以获得突破性进展,而技术的进步为探测分析单个细胞甚至单分子提供了可能[9]。单细胞分析技术的发展在认识细胞的异质性、拓展细胞生物学研究深度等方面发挥了重要的作用[10],更有不少报道综述有关单细胞分析在微生物领域的应用及其分析方法[3, 10-14]。基于此,本文简单回顾基于单细胞的代谢过程分析方法,并重点回顾拉曼镊子在微生物发酵领域的一些应用进展,分析其在应用中所存在的问题,认为拉曼镊子在克服自身不足、融合新的分析技术与数据处理方法后,将是了解细胞异质性、认知复杂生物过程的有力手段。
1 基于单细胞的代谢过程分析单细胞代谢分析可以提供细胞群体内单个细胞代谢异质性的信息。分析过程包含两方面:单个细胞的分离与单个细胞的分析。目前的分析技术虽然已经可以从单个细胞中测量出数百种代谢物,但样品的制备仍然是单细胞代谢分析中的一个难题[15]。
细胞的分离(即样品的制备)是进行单细胞分析的前提,其目的是实现单个细胞的独立,便于后续分析。传统的分离方法是梯度稀释,新发展的技术方法主要有:(1)流式技术以及在此基础上衍生的各种方法,包括流式细胞仪、微流控[16, 17]与液滴微流控[18]等;(2)能够可逆和非动态地定位单个或多个单细胞的永久固定方法,包括3D微腔,0维、1维和2维禁锢等;(3)单个或多个单细胞被固定并根据需要释放的动态定位方法,通常使用的是外部刺激法,包括机械微操作、光学微操作(光镊)、散射力(辐射压力)、光电镊子、电泳微操作等[11]。
根据基本属性,单细胞的分析技术有两大类:一类是能够对细胞实行动态、无损分析的技术,包括无须标记的微光谱化学分析(如拉曼光谱、共振拉曼、表面增强拉曼、相干拉曼和红外显微光谱等分子振动光谱方法,以及X射线等)、电化学分析以及荧光介导的微分析(如需要荧光染色的流式细胞仪、荧光显微镜、荧光寿命成像、荧光相关光谱等);另一类是非动态的、破坏性的分析技术,即质谱和核磁共振等[11],具体的方法和应用参见Ishii等[12]、Rubakhin等[15]、Fritzsch等[19]、Zenobi[20]和Hodzic[21]的综述。不断出现的新技术和新方法,大大地促进了单细胞分析的发展,提高了人们对生物细胞异质性的认识。然而,很多技术方法的样品制备过程比较复杂,且需要较长的分析时间,限制了其发展应用。因此,可以快速提供细胞信息、实时显示微生物细胞即时生理反应且又简便灵敏的分析方法,是未来重点发展的方向。其中,拉曼镊子技术因具有非接触、无须标记、无损等优点,且具有对单个细胞实施分离和动态分析的能力,在生物医学领域得到了广泛的应用。
2 拉曼镊子在微生物发酵中的应用拉曼光谱是一种分子振动光谱,其效应来源于光的非弹性散射,直接反映物质分子振动/旋转振动状态[22, 23],在应用于生物材料研究时具有以下独特优势:一是水分子的拉曼效应很弱,基本上不影响生物细胞的拉曼分析;二是样品无须特别处理,对样品没有破坏性;三是实时、快速;四是样品需要量极少,可以在单细胞甚至亚细胞水平检测,因此其在生物医学领域有着广泛的应用[24-28]。在微生物发酵领域,拉曼光谱主要应用在发酵进程的总体监控方面[29-36],在单细胞水平开展的研究较少。单个细胞的拉曼光谱包含核酸、蛋白质、多糖和脂类等细胞物质的固有特性信息,是细胞基因型、表型和生理状态的表现[37-40]。单细胞拉曼光谱为细胞分析提供了一种便利的、无损的定位手段,可以在亚微米尺度(单细胞水平)探测细胞的生理状态[24],分析化学因子对细胞的影响[41, 42]。
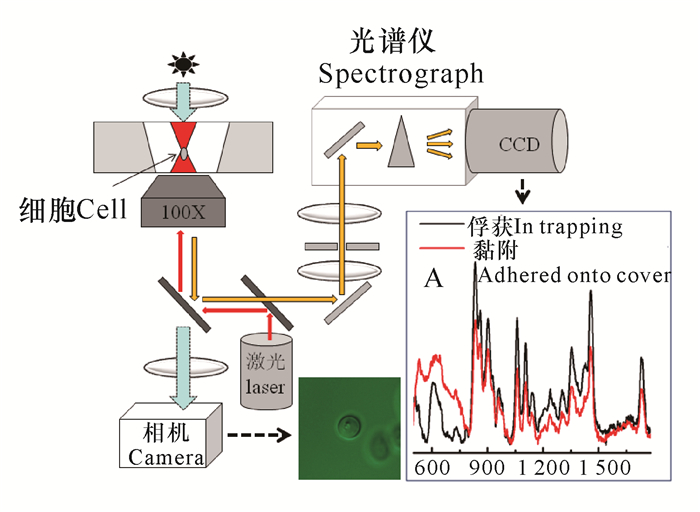
拉曼镊子(Raman tweezers,也称Laser tweezers Raman Spectroscopy)是激光镊子与显微拉曼的结合,融合了无接触光学操控和拉曼分析的优势,可在接近自然的生理状态下研究单个细胞或细胞器,或判别不同类型、不同状态的细胞[37, 43-45]。与一般的显微拉曼相比,拉曼镊子有以下优点:(1)光镊可以将单个活细胞固定在焦点附近进行长时间监测(图 1);(2)细胞处于光束焦点位置,优化了散射光的收集光路,获得更高的信噪比(图 1插图A),得到的是被俘获细胞生化组分光谱信息的叠加;(3)常规的共焦拉曼光谱采集前需要预处理微生物细胞,费时费力且对细胞的生理状态带来未知的影响,而拉曼镊子直接将样品细胞悬浮在无菌水或者生理溶液中,省时省力且能保持细胞正常的生理状态。
Chan[37]、Redding等[43]和Snook等[48]较为详细地回顾了拉曼镊子的应用,但比较宽泛。因此,本部分结合笔者小组在微生物发酵领域所做的一些探索,重点回顾拉曼镊子在微生物发酵领域的一些应用。
2.1 发酵过程单个细胞的实时监测实时无损无标记生理分析是拉曼镊子的优势之一,因而其可以长时间对同一个细胞进行实时监测,获知微生物细胞实时的生理变化。拉曼镊子主要监测有特殊产物的细胞或者是有明显指纹峰的物质,甚至定量分析单个细胞产特定物质的生理过程,比如乙醇发酵[39]或者物理(化学)胁迫下细胞的动态生化变化[49]。Peng等[39]通过实时分析单个酵母细胞的发酵过程,发现酵母细胞启动好氧发酵3 min内就能产生乙醇,而且在高浓度底物下,发酵起始阶段胞内的乙醇浓度高于胞外(胞内外实时乙醇浓度比为1.5左右,图 2),胞内累积的乙醇随着胞外底物浓度的降低而释放。Singh等[49]实时观察单个酵母细胞在热处理和高渗透溶液下的应激反应,观测到迟滞期的酵母细胞转入含10%葡萄糖的培养液后33 min开始生成甘油和乙醇,在39 min生成速率达到峰值,单个细胞生成约300 amol的甘油和700 amol乙醇。
当拉曼镊子系统中使用双光镊或者多光镊俘获细胞时,可以在相同的生理条件下记录多个微生物单细胞的实时光谱,了解细胞间的生长动力学和异质性[50]。由于拉曼光谱的信号比较弱,要获得较强的拉曼信号,需要较强的激光激发,因此实时分析主要应用在较短时间内的监测。
2.2 发酵过程大量单个细胞的分析要较好地反映完整的群体发酵过程,需要分析大量的单个细胞,但是拉曼镊子难以对多个单细胞实施长时间的实时监测。因此,不同的学者应用拉曼镊子针对不同的发酵(培养)阶段随机选择一批细胞进行分析,从拉曼光谱的角度展示发酵过程单个细胞胞内物质的变化动态,既可以获知发酵的总体进程,又可以获知发酵过程不同阶段或同一阶段细胞间的差异,发现蛋白质二级结构的改变,该技术比常规的分子生物学手段更加简便快捷。其中涉及的主要光谱分析手段一是直接读取蛋白质、脂类、核酸或者目标产物的特征峰信号强度,二是把细胞光谱信息作为一个数据整体,结合化学计量学,判别细胞所处的生理状态,通过影响因子来分析引起差异的原因。
2.2.1 特殊产物发酵微生物细胞通常会在胞内累积聚酯、色素等物质,由于这类物质通常有比较特殊的拉曼特征峰,是拉曼镊子研究的便利对象。
2.2.1.1 聚酯聚β-羟基丁酸(PHB)是一种生物塑料[51-53]。已有针对PHB合成的研究主要是基于群体细胞水平在宏观层面上探索,比如优化发酵环境[54, 55],应用显微拉曼进行定量检测[56]以及分析其含量的异质性[57]。笔者所在的小组[58-61]应用拉曼镊子分析C. necator H16菌株PHB累积过程,并跟踪胞内核酸、蛋白质的代谢动态。Tao等[59]的实验结果显示迟滞期与对数生长前期是胞内生物大分子代谢的活跃时期,而PHB在发酵后不久即开始产生,在对数初期开始大量累积,表明该菌株启动PHB合成的因素并非营养因子的匮乏(图 3)。
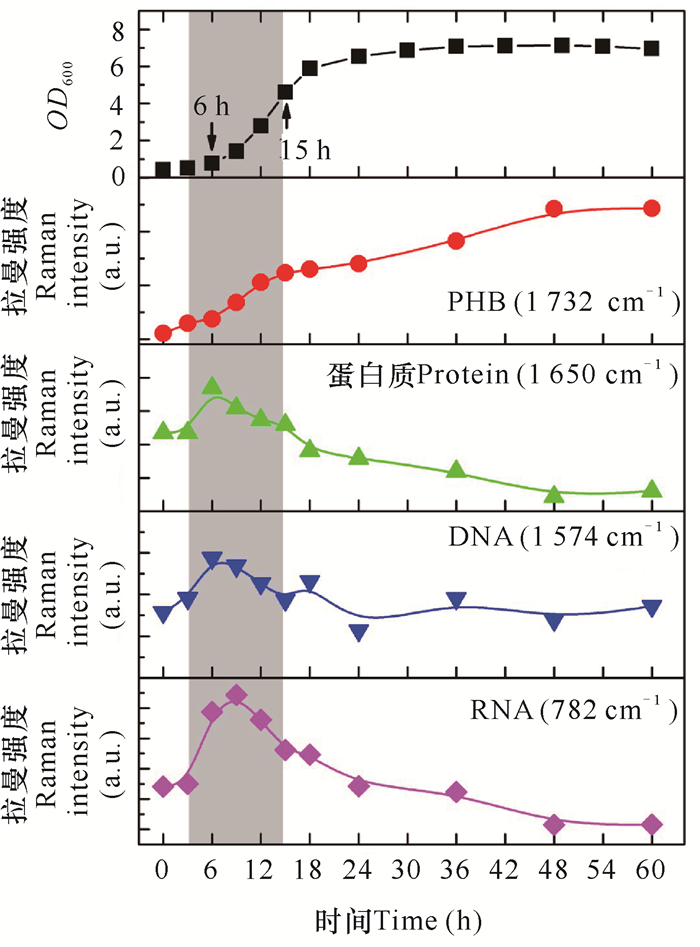
|
| 图 3 C.necator H16菌株的生长曲线及胞内RNA (782 cm-1)、DNA (1 574 cm-1)、蛋白质(1 650 cm-1)、PHB (1 732 cm-1)等物质的特征峰强度随发酵进程的变化动态 Fig.3 Growth curve of C.necator H16 strain and characteristic peak intensity change of intracellular RNA(782 cm-1), DNA(1 574 cm-1), protein (1 650 cm-1), and PHB (1 732 cm-1) with the fermentation process |
单个细胞主要生物大分子特征拉曼峰分析显示,RNA是PHB合成过程代谢中最为活跃的生物大分子。在较高碳浓度下,细胞核酸物质长时间维持在较高的水平;即使仅提供有限的碳源,H16细胞还是将部分碳源用于合成PHB,而不是将所有的碳源完全用于细胞生长[58]。在不同的氮源下,PHB快速合成期也是胞内生物大分子的活跃期,氮源主要影响RNA和蛋白质的代谢进而影响PHB产物的合成[60]。
不同碳氮比条件下的PHB发酵分析显示,在高氮环境下细胞的RNA含量(782 cm-1峰)和蛋白质含量(1 656 cm-1峰)一直处于较高的水平,两者呈线性正相关关系。但蛋白质含量(1 656 cm-1峰)与PHB含量(1 732 cm-1峰)呈负相关关系,胞内活跃且含量高的RNA有可能加强蛋白质的合成,进而削弱PHB的合成。在高碳氮比环境下,核酸以及蛋白质的合成受阻,导致碳源的代谢途径从三羧酸循环转向PHB合成路径[61]。
2.2.1.2 类胡萝卜素微生物可以产生各种色素。其中生物来源的类胡萝卜素是一类重要的天然色素,比化学合成的类胡萝卜素具有更低的毒性和更高的生物利用度。类胡萝卜素是异戊二烯类化合物,具有多个共轭双键结构,在1 157,1 524 cm-1附近各有一个典型的、强烈的特征峰。Tao等[40]采用拉曼镊子对圆红冬孢酵母的类胡萝卜素进行实时定量监测发现,类胡萝卜素和脂质的积累主要发生在指数期的后期和静止阶段,即当细胞生长受到营养限制时;同时,类胡萝卜素浓度随核酸浓度的变化而变化,在培养的第一阶段增加,在培养的最后阶段减少。袁玉峰等[62]利用拉曼镊子对红酵母合成类胡萝卜素进行分析,考查氮源和碳源对类胡萝卜素产量的影响,发现适宜红酵母细胞生长和类胡萝卜素合成的氮源和碳源分别是酵母粉+胰蛋白胨、葡萄糖。王雪等[63, 64]用拉曼镊子分析红法夫酵母内虾青素含量,优化了红法夫酵母生产虾青素的条件。孙美娟等[65]应用拉曼镊子对圆红冬孢酵母合成油脂和类胡萝卜素进行定量分析,考察不同碳氮比、碳磷比以及碳硫比对圆红冬孢酵母油脂和类胡萝卜合成能力的影响,结果表明随着碳氮比、碳磷比和碳硫比的升高,圆红冬孢酵母细胞内油脂含量均逐步增加,而类胡萝卜素的含量均显著降低。
2.2.1.3 脂肪酸脂类物质也是学者们研究的重点对象,微藻因能够合成和储存可以转化为生物柴油的脂类(如脂肪酸和三酰甘油TAG)而重新引起学者的重大关注。Wu等[66]应用拉曼镊子对产油微藻的脂质谱进行直接的、无须标记的在体定量分析,测定微藻内组成脂质的不饱和度和转变温度,为微藻的脂质浓度提供相对快速的监测。Wang等[67]采用类似的技术在单细胞水平定量表征了微藻产油过程,发现传统群体层面细胞油脂分析方法难以揭示的规律,即单个细胞水平的TAG含量与脂类不饱和度呈显著的负相关关系。单细胞TAG定量技术直接跳过细胞培养环节,因此能够分析未培养的微生物。
2.2.1.4 葡萄球菌黄素陶站华等[68]分析单个金黄色葡萄球菌细胞内葡萄球菌黄素相对含量,并考察吲哚浓度及培养时间对色素含量的影响。其群体和单细胞水平的光谱数据均表明,吲哚可剂量依赖性地抑制葡萄球菌黄素的合成;在分批培养中,群体中大多数细胞的色素含量在对数生长中期同步达到最大值,各个时间点的群体内部细胞间色素含量的异质性较小。用1 523 cm-1特征峰强度表征胞内葡萄球菌黄素的相对含量,显示在22—37℃时,温度对葡萄球菌黄素合成的影响不大;中性pH最有利于葡萄球菌黄素的合成;在一定浓度范围内(0.2—5.0 g/L),葡萄糖可剂量依赖性地促进葡萄球菌黄素的合成,黄酮则可剂量依赖性地抑制葡萄球菌黄素的合成[69]。
上述研究显示,拉曼镊子能够在单细胞水平可靠地分析微生物细胞特殊产物的含量,既可以监测有特殊拉曼峰的产物发酵动态,又可以同时获知发酵过程胞内主要生物大分子的变化动态。
2.2.2 重组蛋白发酵重组蛋白质发酵是将携带重组质粒的工程菌培养至一定密度,经诱导后获得目的蛋白的一种手段。该技术过量表达目的基因,从而获得大量廉价的目的蛋白,因而单个细胞的拉曼信号可能会有明显的不同,可通过拉曼光谱方法监测其发酵进程,从而省去蛋白提取、电泳等繁杂的环节,为分析细胞的发酵过程开辟新的可能性。Xie等[70]最先在大肠杆菌和毕赤酵母两种不同的表达系统中监测斑马鱼β生长抑素的表达,发现单个大肠杆菌或者毕赤酵母相关蛋白峰的信号强度随着发酵时间的增加而增强,与凝胶电泳和蛋白印迹分析结果相符。Chan等[71]监测髓鞘少突胶质细胞糖蛋白在大肠杆菌中的表达,发现该蛋白质表达主要发生在第2小时内,相对于第1小时表达的蛋白质增加470%,而第3小时相对第2小时增加230%,表明3 h内蛋白质的表达开始平稳。
卢明倩等[72]研究甲酸脱氢酶(FDH)重组蛋白在大肠杆菌细胞中的表达水平,FDH的特征峰1 004,1 355,1 455和1 667 cm-1随着IPTG诱导时间的延长而增强,峰信号强度的增加值所反映的FDH表达量增多与SDS-PAGE电泳分析结果一致。
周冰等[73]分析大肠杆菌所表达的蚕豆Vicia faba L. 14-3-3b可溶性蛋白与包涵体蛋白的结构差异,以及不同温度下两种蛋白在重组菌中的表达水平,结果表明:16℃下诱导的14-3-3b可溶性蛋白特征峰强度明显强于28℃下诱导的,而包涵体蛋白的特征峰强度则相反。黄庶识等[74]实时分析不同温度下上述两种蛋白的动态表达水平时发现:28℃培养条件下,重组菌蛋白质过量表达并以形成包涵体为主;16℃条件下以可溶性蛋白为主,且在该温度下表达的蛋白质能正确折叠,有利于细胞形成稳定的可溶性蛋白。
上述文献表明,拉曼镊子对单个活细胞中蛋白质表达等生物过程的实时、无损和定量监测具有足够的敏感性。
2.2.3 生物乙醇发酵通过追踪单个酵母细胞特征拉曼峰信号强度的变化,可有效地分析葡萄糖、乙醇等物质的变化,以及胞内核酸、脂类及蛋白质等生物大分子含量及蛋白质结构的变化。
2.2.3.1 不同发酵过程下的动态分析彭立新等[75]监测活性干酵母活化与生长过程:酿酒活性干酵母复水活化后,核酸类物质迅速增加,RNA在第6小时达到最大值;蛋白质和脂类物质从第6小时开始快速增加,在第9小时达到最大值,而后呈下降趋势;胞内乙醇则是在第9小时开始出现,在第9—12小时期间大量生成。
李自达等[76]分析500 L发酵罐木薯淀粉浓醪乙醇发酵过程中酵母细胞内物质的变化,结果显示酵母细胞为适应浓醪发酵环境会调整细胞的生理状态和胞内组分:随着乙醇浓度的升高,酵母细胞累积蛋白质和脂类物质,蛋白质二级结构逐渐以无规则卷曲为主;在发酵后期,部分酵母细胞在胞内累积大量的嘌呤类物质。
pH值是影响酵母乙醇发酵的重要因子。覃赵军等[77]从拉曼光谱学角度对不同初始pH值的乙醇发酵过程进行分析,结果显示发酵后期,在pH值为3.0的条件下,胞内脂类和蛋白质的拉曼信号最强,表明在低pH值环境下部分底物被转化为胞内储藏物质;主成分分析显示,pH值对酵母细胞生理状态的影响始于发酵初始阶段;1 440和1 600 cm-1峰一直是影响主成分PC1、PC2和PC3分值的主要特征峰,说明pH条件可能影响酵母细胞的脂类物质合成和呼吸代谢,进而影响底物的代谢方向和产物的合成。
覃赵军等[78]分析不同氮源对酵母乙醇发酵的影响及其可能的分子机制。在有机氮源下,酵母细胞进入新的发酵环境后立即快速合成RNA,并且含量显著高于无机氮源下的,其RNA拉曼峰的最大平均强度是初始平均强度的1.9—2.1倍(无机氮源的为1.2—1.4倍);无机氮源下脂类物质和DNA的含量高于有机氮源。有机氮源缩短酵母的迟滞期,促进胞内RNA快速大量合成,促进与氮源代谢、乙醇发酵相关基因的快速转录和表达,进而促进乙醇发酵。
覃赵军等[79]分析高渗环境对酵母乙醇发酵代谢的影响。高渗透压会显著影响782,1 301,1 602,1 657 cm-1峰所表征物质的合成时间和强度,进而影响代谢方向,而耐高渗菌株能适应高渗环境,调整胞内组分含量,实现高产发酵。不同渗透压下影响第一主成分的主要特征峰是1 302—1 306 cm-1和1 443 cm-1等源自脂类物质的拉曼峰,说明渗透压影响胞内脂类物质的合成。
在逆境发酵中,不管是普通菌株还是耐乙醇的菌株,RNA物质往往在发酵前期就开始大量合成,而蛋白质、脂类物质则是在乙醇大量合成的时候才开始大量合成;蛋白质、脂类物质峰的升高与环境中乙醇浓度有关,与菌株关联性不大,可能是酵母菌株应对逆境的手段(结果待发表)。
2.2.3.2 蛋白质结构的变化拉曼镊子除可以监测胞内物质的总量变化外,还可以监测发酵过程酵母细胞蛋白质二级结构的变化。
赖钧灼等[80]在监测以葡萄糖为底物的乙醇发酵动态过程中发现:在发酵前期,细胞1 662 cm-1峰(表征蛋白质酰胺Ⅰ的无规则卷曲)与1 652 cm-1峰(表征蛋白质酰胺Ⅰ的α螺旋)的强度之比(I1662/I1652)小于1,7 h后大于1,9—18 h基本维持在同一水平,而后呈现上升趋势,说明发酵初期胞内蛋白的二级结构以有序的α螺旋为主,随着发酵的进行,酵母细胞蛋白质以无规则卷曲为主。在李自达等[76]研究的高浓度底物酵母发酵后期,也出现了类似的现象。而在以酵母粉为氮源的不同发酵阶段,部分细胞的蛋白质酰胺Ⅰ的二级结构以β折叠为主,也有部分细胞以α螺旋为主,还有一部分细胞则是两者基本均衡,而以其他物质为氮源的细胞则是以α螺旋占绝对主导地位[78]。出现上述现象的原因有可能是在较高浓度的乙醇环境下,酵母细胞为适应胞内外乙醇环境的变化而改变蛋白质的二级结构。
2.2.4 发酵过程中细胞的异质性针对大量单个细胞的分析可以获知群体细胞的生理状态以及细胞间的差异。
在浓醪乙醇发酵中,胞内蛋白质和脂类物质(特征峰1 301,1 440,1 654 cm-1)在发酵前、中期的细胞间差异较小,而发酵后期(32,44 h)这两类物质的含量显著增加,胞间差异明显增大;核酸类物质,在发酵前、中期,782 cm-1与1 094 cm-1的信号峰强度呈离散分布,但进入发酵后期后,核酸含量增加且在细胞间的分布接近正态分布;嘌呤物质(特征峰1 480 cm-1)在发酵的前、中期胞内含量很少,胞间差异不大,而在发酵的后期迅速增加,细胞间差异大且呈两极分化,含量较少的细胞占多数,含量很高的细胞占少数[76]。
在PHB发酵中,直方统计分析显示,发酵前期细胞间胞内PHB含量差异较大,发酵中期相对均匀、胞间差异小,但发酵后期胞间差异显著[59]。根据奇异值分布可知菌体的代谢方向、产物的差别自发酵开始就不一致,并且随着发酵的进行差异越来越大[60]。
3 存在的问题 3.1 荧光背景荧光对拉曼监测有非常严重的干扰[81],拉曼镊子也存在同样的问题,特别是在溶液或者细胞有自发荧光的时候,拉曼信号通常湮灭于荧光中。Cormack等[82]发现使用拉盖尔-高斯(LG)和Holey高斯激光时,可以减少拉曼系统中光学元件的背景荧光。另外,也可以使用长波长的激光(比如1 064 nm)来激发拉曼信号[83],或者通过数据处理来消除荧光的影响[84]。其他荧光背景消除方法还有表面增强拉曼光谱技术、傅立叶变换拉曼光谱技术,或者加入荧光淬灭剂,但是后者可能会影响细胞的活性。
3.2 激光损伤激光长时间俘获同一个细胞可能会对细胞产生伤害,特别是在激光功率比较大的时候。激光光损伤主要包括光化学损伤(例如氧化、DNA损伤、细胞代谢)和光热(热致)损伤,损伤的程度与使用的波长、细胞类型有关。由于拉曼镊子的细胞处于溶液中,因此减小了光热损伤,对细胞的损失主要是光化学损伤。Singh等[49]使用能量较低的长波长近红外激光(1 064 nm)用于俘获细胞,而用短波长的激光(532 nm或者785 nm)激发细胞的拉曼信号;或者是用同一束激光俘获与激发细胞,在激发拉曼的时候用较高功率而在俘获的时候用较低功率,以减少激光对被俘细胞的伤害。
3.3 分析效率单个光镊的分析效率比较低,特别是长时间监测细胞的实时动态时。为提高分析效率,陆续发展了双光镊[50]或者多光镊的实验系统[85, 86],可以在相同的条件下同步监测多个细胞。对于大量单个细胞的非实时在线分析,黄超等[87]、毛丽华等[88]将拉曼镊子与微流控结合;Casabella等[89]发展了自动分析单个细胞的拉曼镊子系统,实现单个细胞光谱信息的自动采集;而Pilát等[90]将拉曼镊子与微流控系统结合,发展了自动分析和在无菌条件下分选微生物细胞的方法。除对实验系统的提升之外,Zhang等[91]在多光镊拉曼镊子的基础上发展了一种基于压缩传感的单次采集多焦拉曼光谱技术,将常规拉曼光谱的速度提高2—3个数量级,并有望应用于监测大尺寸生物系统的快速动力学研究。同时,Zhang等[92]还发展了一种压缩感知框架下的分层稀疏方法,结合组内稀疏性和组内选择策略进行光谱重建,有望在长时间同步监测多个细胞的生物过程中得到广泛应用。
3.4 信号增强相对而言,拉曼镊子在一定程度上提高了信噪比,获得了更好的信号,但信号弱依然是其需要克服的弱点。为解决该问题,表面增强拉曼散射(SERS)、共振拉曼光谱(RRS)、相干反斯托克斯拉曼光谱(CARS)以及受激拉曼光谱(SRS)相继发展,将拉曼信号增强几个数量级。Huai等[93]将光镊与SERS结合应用于蛤蚌毒素的快速检测,Ramser等[94-96]将光镊与共振拉曼结合应用于功能血红细胞和血红蛋白的分析,以及细菌表达神经珠蛋白,但这些技术方法很少应用到发酵分析领域。
3.5 数据处理与挖掘拉曼镊子得到包含核酸、蛋白质、多糖和脂类物质等反映细胞固有特性的信息,是所有光谱信息的叠加,如何把关键的信息提取出来是拉曼光谱分析的难点[97]。由于同一个拉曼谱带有可能来源于几个不同的分子或基团,单变量分析严重限制了详细信息的获得。如果把整个拉曼信息看成数学数据,则可以应用常规的化学计量学方法来提取光谱特征信息,从而判别细胞的生理状态。常用的方法有主成分分析(PCA)和奇异值分解,两者都是一种简化数据集的技术。通过PCA并分析主要PC的载荷与相关特征峰的相关性,从中可以获知不同发酵阶段微生物细胞胞内主要生物大分子的合成特点[58-61, 76, 77, 79]。Iwasaki等[98]通过拉曼显微镜可视化分子特异性信息,用多元曲线分辨率分析从拉曼光谱中提取隐藏的关键信息,首次在微藻Euglena gracilis蜡酯发酵中发现两种不同形式的肉豆蔻酸酯的积累。此外,中科院青岛过程与能源所提出“拉曼组”(Ramanome)概念(指特定状态下一个细胞群体的单细胞拉曼光谱集合),并以莱茵衣藻、微拟球藻等为模式,基于拉曼组技术,建立可同时定量单个细胞中淀粉、蛋白质、甘油三酯含量和脂质不饱和度的方法[99-101]。
3.6 与其他技术的结合从前面的论述可以看出,拉曼镊子最适宜用于实时监测的是目标明确的特征拉曼峰,但一般的细胞光谱是整个细胞物质的叠加,影响了拉曼镊子实时监测的应用。Wang等[102],Xu等[103]将拉曼光谱与稳定同位素标记(SIP)结合(同位素的掺入改变了所掺入分子键的拉曼带),通过氘化代谢底物推导物质循环过程,获知物质代谢动态。Lu等[104]将拉曼镊子与人工智能技术结合,实现微生物单细胞水平的快速准确鉴定。
拉曼镊子得到的主要信息是底物、产物和核酸、蛋白质、脂类等生物大分子的信息,如果与单细胞测序、基因表达分析或者组学分析相结合,广泛涵盖单个细胞中的大多数成分,则可深入了解细胞异质性的机制。
此外,目前的研究还没有完全阐明细胞异质性对生物性能的影响,但所有研究的最终目标是将其应用到实际生产中,提高发酵性能。对拉曼镊子来说,在相对短的时间内获取大量单个细胞的信息用于系统生物学的建模[105],还需要提高系统的灵敏度和检测数量。
4 展望微生物发酵是个复杂的过程,是大量单个细胞综合表现的结果,但细胞间存在明显的异质性,通过单细胞分析了解微生物细胞的异质性,有利于发酵环境的优化,从而减少细胞间的异质性,提高发酵性能。拉曼镊子融合了拉曼光谱和光学操控的优点,无破坏、无须标记,可以监测单个细胞的实时生理状态,表征微生物细胞的生理功能,还可以作为一种工具来分析大群体中相对较小的单细胞群体(数千个细胞),以获得样本异质性的统计信息,并在单细胞水平上研究细胞动力学。本文重点回顾了拉曼镊子在监测乙醇发酵、细胞色素、聚酯及脂肪酸发酵以及重组蛋白表达等领域的应用进展,显示拉曼镊子实时监测发酵过程中物质变化的能力以及从单个细胞水平上研究发酵过程的潜能。由于细胞代谢快速、胞内成分多样,以及拉曼技术还存在信号较弱、分析效率不高以及光损伤等问题,拉曼技术不能完全、迅速和无损地监测发酵过程多个单细胞的代谢物。即便如此,已有的研究仍从分子光谱和单细胞角度为微生物发酵提供了新的认识。因此,如能克服自身不足,融合新的分析技术和数据处理手段,拉曼镊子将在微生物单细胞分析领域发挥更大作用。将拉曼分析、微流控分选与组学分析结合起来,则可同时获知更全面的信息,无疑将推进生物单细胞分析及对其异质性的认识,提高对复杂生物系统的认知。
| [1] |
KOUTSOUMANIS K P, ASPRIDOU Z. Individual cell heterogeneity in predictive food microbiology:Challenges in predicting a "noisy" world[J]. International Journal of Food Microbiology, 2017, 240: 3-10. DOI:10.1016/j.ijfoodmicro.2016.06.021 |
| [2] |
SHAPIRO H M. Microbial analysis at the single-cell level:Tasks and techniques[J]. Journal of Microbiological Methods, 2000, 42(1): 3-16. DOI:10.1016/S0167-7012(00)00167-6 |
| [3] |
BREHM-STECHER B F, JOHNSON E A. Single-cell microbiology:Tools, technologies, and applications[J]. Microbiology and Molecular Biology Reviews, 2004, 68(3): 538-559. DOI:10.1128/MMBR.68.3.538-559.2004 |
| [4] |
DHAR N, MCKINNEY J D. Microbial phenotypic heterogeneity and antibiotic tolerance[J]. Current Opinion in Microbiology, 2007, 10(1): 30-38. DOI:10.1016/j.mib.2006.12.007 |
| [5] |
LIDSTROM M E, KONOPKA M C. The role of physiological heterogeneity in microbial population behavior[J]. Nature Chemical Biology, 2010, 6(10): 705-712. DOI:10.1038/nchembio.436 |
| [6] |
MAGDANOVA L A, GOLYASNAYA N V. Heterogeneity as an adaptive trait of microbial populations[J]. Microbiology, 2013, 82(1): 1-10. DOI:10.1134/S0026261713010074 |
| [7] |
DELVIGNE F, GOFFIN P. Microbial heterogeneity affects bioprocess robustness:Dynamic single-cell analysis contributes to understanding of microbial populations[J]. Biotechnology Journal, 2014, 9(1): 61-72. DOI:10.1002/biot.201300119 |
| [8] |
HEINS A L, WEUSTER-BOTZ D. Population heterogeneity in microbial bioprocesses:Origin, analysis, mechanisms, and future perspectives[J]. Bioprocess and Biosystems Engineering, 2018, 41(7): 889-916. DOI:10.1007/s00449-018-1922-3 |
| [9] |
ANSELMETTI D. Single cell analysis:Technologies and applications[M]. Hanover, Germany: Wiley-VCH Verlag GmbH & Co.KGaA, 2009: 284.
|
| [10] |
JONES S. Single-cell microbiology[J]. Nature Biotechnology, 2016, 34(11): 1077. DOI:10.1038/nbt.3728 |
| [11] |
VASDEKIS A E, STEPHANOPOULOS G. Review of methods to probe single cell metabolism and bioenergetics[J]. Metabolic Engineering, 2015, 27: 115-135. DOI:10.1016/j.ymben.2014.09.007 |
| [12] |
ISHⅡ S, TAGO K, SENOO K. Single-cell analysis and isolation for microbiology and biotechnology:Methods and applications[J]. Applied Microbiology and Biotechnology, 2010, 86(5): 1281-1292. DOI:10.1007/s00253-010-2524-4 |
| [13] |
陈子熙, 陈磊, 张卫文. 单细胞尺度下的微生物学研究:意义与方法[J]. 微生物学报, 2017, 57(6): 920-931. |
| [14] |
刘聪, 谢伟, 何林, 等. 单细胞拉曼光谱在微生物研究中的应用[J]. 微生物学报, 2020, 60(6): 1051-1062. |
| [15] |
RUBAKHIN S S, LANNI E J, SWEEDLER J V. Progress toward single cell metabolomics[J]. Current Opinion in Biotechnology, 2013, 24(1): 95-104. DOI:10.1016/j.copbio.2012.10.021 |
| [16] |
CHAO T C, ROS A. Microfluidic single-cell analysis of intracellular compounds[J]. Journal of the Royal Society Interface, 2008, 5(Suppl 2): S139-S150. |
| [17] |
GAO D, JIN F, ZHOU M, et al. Recent advances in single cell manipulation and biochemical analysis on microfluidics[J]. The Analyst, 2019, 144(3): 766-781. DOI:10.1039/C8AN01186A |
| [18] |
WORONOFF G, NGHE P, BAUDRY J, et al. Meta-bolic cost of rapid adaptation of single yeast cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(20): 10660-10666. DOI:10.1073/pnas.1913767117 |
| [19] |
FRITZSCH F S, DUSNY C, FRICK O, et al. Single-cell analysis in biotechnology, systems biology, and biocatalysis[J]. Annual Review of Chemical and Biomolecular Engineering, 2012, 3: 129-155. DOI:10.1146/annurev-chembioeng-062011-081056 |
| [20] |
ZENOBI R. Single-cell metabolomics:Analytical and biological perspectives[J]. Science, 2013, 342(6163): 1243259. DOI:10.1126/science.1243259 |
| [21] |
HODZIC E. Single-cell analysis:Advances and future perspectives[J]. Bosnian Journal of Basic Medical Sciences, 2016, 16(4): 313-314. |
| [22] |
CORDERO E, LATKA I, MATTHAUS C, et al. In-vivo Raman spectroscopy:From basics to applications[J]. Journal of Biomedical Optics, 2018, 23(7): 071210. |
| [23] |
JONES R R, HOOPER D C, ZHANG L W, et al. Raman techniques:Fundamentals and frontiers[J]. Nanoscale Research Letters, 2019, 14(1): 231. DOI:10.1186/s11671-019-3039-2 |
| [24] |
HUSER T, CHAN J. Raman spectroscopy for physiological investigations of tissues and cells[J]. Advanced Drug Delivery Reviews, 2015, 89: 57-70. DOI:10.1016/j.addr.2015.06.011 |
| [25] |
SMITH R, WRIGHT K L, ASHTON L. Raman spectroscopy:An evolving technique for live cell studies[J]. The Analyst, 2016, 141(12): 3590-3600. |
| [26] |
BUCKLEY K, RYDER A G. Applications of Raman spectroscopy in biopharmaceutical manufacturing:A short review[J]. Applied Spectroscopy, 2017, 71(6): 1085-1116. DOI:10.1177/0003702817703270 |
| [27] |
WIERCIGROCH E, SZAFRANIEC E, CZAMARA K, et al. Raman and infrared spectroscopy of carbohydrates:A review[J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2017, 185: 317-335. DOI:10.1016/j.saa.2017.05.045 |
| [28] |
EMBER K J I, HOEVE M A, MCAUGHTRIE S L, et al. Raman spectroscopy and regenerative medicine:A review[J]. NPJ Regenerative Medicine, 2017, 2: 12. DOI:10.1038/s41536-017-0014-3 |
| [29] |
PICARD A, DANIEL I, MONTAGNAC G, et al. In situ monitoring by quantitative Raman spectroscopy of alcoholic fermentation by Saccharomyces cerevisiae under high pressure[J]. Extremophiles:Life under Extreme Conditions, 2007, 11(3): 445-452. DOI:10.1007/s00792-006-0054-x |
| [30] |
SHIH C J, SMITH E A. Determination of glucose and ethanol after enzymatic hydrolysis and fermentation of biomass using Raman spectroscopy[J]. Analytica Chimica Acta, 2009, 653(2): 200-206. DOI:10.1016/j.aca.2009.09.012 |
| [31] |
AVILA T C, POPPI R J, LUNARDI I, et al. Raman spectroscopy and chemometrics for on-line control of glucose fermentation by Saccharomyces cerevisiae[J]. Biotechnology Progress, 2012, 28(6): 1598-1604. DOI:10.1002/btpr.1615 |
| [32] |
UYSAL R S, SOYKUT E A, BOYACI I H, et al. Monitoring multiple components in vinegar fermentation using Raman spectroscopy[J]. Food Chemistry, 2013, 141(4): 4333-4343. DOI:10.1016/j.foodchem.2013.06.122 |
| [33] |
GRAY S R, PERETTI S W, LAMB H H. Real-time monitoring of high-gravity corn mash fermentation using in situ Raman spectroscopy[J]. Biotechnology and Bioengineering, 2013, 110(6): 1654-1662. DOI:10.1002/bit.24849 |
| [34] |
IVERSEN J A, BERG R W, AHRING B K. Quantitative monitoring of yeast fermentation using Raman spectroscopy[J]. Analytical and Bioanalytical Chemistry, 2014, 406(20): 4911-4919. DOI:10.1007/s00216-014-7897-2 |
| [35] |
LEGNER R, WIRTZ A, KOZA T, et al. Application of green analytical chemistry to a green chemistry process:Magnetic resonance and Raman spectroscopic process monitoring of continuous ethanolic fermentation[J]. Biotechnology and Bioengineering, 2019, 116(11): 2874-2883. DOI:10.1002/bit.27112 |
| [36] |
JIANG H, XU W D, DING Y H, et al. Quantitative analysis of yeast fermentation process using Raman spectroscopy:Comparison of CARS and VCPA for variable selection[J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2020, 228: 117781. DOI:10.1016/j.saa.2019.117781 |
| [37] |
CHAN J W. Recent advances in laser tweezers Raman spectroscopy (LTRS) for label-free analysis of single cells[J]. Journal of Biophotonics, 2013, 6(1): 36-48. |
| [38] |
PUPPELS G J, DE MUL F F, OTTO C, et al. Studying single living cells and chromosomes by confocal Raman microspectroscopy[J]. Nature, 1990, 347(6290): 301-303. DOI:10.1038/347301a0 |
| [39] |
PENG L, WANG G, LIAO W, et al. Intracellular ethanol accumulation in yeast cells during aerobic fermentation:A Raman spectroscopic exploration[J]. Letters in Applied Microbiology, 2010, 51(6): 632-638. DOI:10.1111/j.1472-765X.2010.02941.x |
| [40] |
TAO Z H, WANG G W, XU X D, et al. Monitoring and rapid quantification of total carotenoids in Rhodotorula glutinis cells using laser tweezers Raman spectroscopy[J]. FEMS Microbiology Letters, 2011, 314(1): 42-48. DOI:10.1111/j.1574-6968.2010.02139.x |
| [41] |
MIAO Z B, ZHANG P F, ZHANG Y, et al. Single-cell analysis reveals the effects of glutaraldehyde and formaldehyde on individual Nosema bombycis spores[J]. The Analyst, 2019, 144(9): 3136-3143. DOI:10.1039/C8AN02425A |
| [42] |
ZHANG Y, MIAO Z B, HUANG X H, et al. Laser tweezers Raman spectroscopy (LTRS) to detect effects of chlorine dioxide on individual Nosema bombycis spores[J]. Applied Spectroscopy, 2019, 73(7): 774-780. DOI:10.1177/0003702818817522 |
| [43] |
REDDING B, SCHWAB M, PAN Y L. Raman spec-troscopy of optically trapped single biological micro-particles[J]. Sensors, 2015, 15(8): 19021-19046. DOI:10.3390/s150819021 |
| [44] |
XIE C A, DINNO M A, LI Y Q. Near-infrared Raman spectroscopy of single optically trapped biological cells[J]. Optics Letters, 2002, 27(4): 249-251. DOI:10.1364/OL.27.000249 |
| [45] |
XIE C A, GOODMAN C, DINNO M, et al. Real-time Raman spectroscopy of optically trapped living cells and organelles[J]. Optics Express, 2004, 12(25): 6208-6214. DOI:10.1364/OPEX.12.006208 |
| [46] |
NAVAS-MORENO M, CHAN J W. Laser tweezers Raman microspectroscopy of single cells and biological particles[J]. Methods in Molecular Biology, 2018, 1745: 219-257. |
| [47] |
KONG L B, ZHANG P F, WANG G W, et al. Characterization of bacterial spore germination using phase-contrast and fluorescence microscopy, Raman spectroscopy and optical tweezers[J]. Nature Protocols, 2011, 6(5): 625-639. DOI:10.1038/nprot.2011.307 |
| [48] |
SNOOK R D, HARVEY T J, CORREIA FARIA E, et al. Raman tweezers and their application to the study of singly trapped eukaryotic cells[J]. Integrative Biology:Quantitative Biosciences from Nano to Macro, 2009, 1(1): 43-52. DOI:10.1039/B815253E |
| [49] |
SINGH G P, CREELY C M, VOLPE G, et al. Real-time detection of hyperosmotic stress response in optically trapped single yeast cells using Raman microspectroscopy[J]. Analytical Chemistry, 2005, 77(8): 2564-2568. DOI:10.1021/ac048359j |
| [50] |
LI Y, WANG G W, YAO H L, et al. Dual-trap Raman tweezers for probing dynamics and heterogeneity of interacting microbial cells[J]. Journal of Biomedical Optics, 2010, 15(6): 067008. DOI:10.1117/1.3526357 |
| [51] |
LEE S Y. Bacterial polyhydroxyalkanoates[J]. Bio-technology and Bioengineering, 1996, 49(1): 1-14. |
| [52] |
EGGERS J, STEINBUCHEL A. Poly(3-hydroxybu-tyrate) degradation in Ralstonia eutropha H16 is mediated stereoselectively to (S)-3-hydroxybutyryl coenzyme A (CoA) via crotonyl-CoA[J]. Journal of Bacteriology, 2013, 195(14): 3213-3223. DOI:10.1128/JB.00358-13 |
| [53] |
BATCHA A F, PRASAD D M, KHAN M R, et al. Biosynthesis of poly(3-hydroxybutyrate) (PHB) by Cupriavidus necator H16 from jatropha oil as carbon source[J]. Bioprocess and Biosystems Engineering, 2014, 37(5): 943-951. DOI:10.1007/s00449-013-1066-4 |
| [54] |
JARUTE G, KAINZ A, SCHROLL G, et al. On-line determination of the intracellular poly (beta-hydroxybutyric acid) content in transformed Escherichia coli and glucose during PHB production using stopped-flow attenuated total reflection FT-IR spectrometry[J]. Analytical Chemistry, 2004, 76(21): 6353-6358. DOI:10.1021/ac049803l |
| [55] |
KHANNA S, SRIVASTAVA A K. On-line characterization of physiological state in poly (beta-hydroxybutyrate) production by Wautersia eutropha[J]. Applied Biochemistry and Biotechnology, 2009, 157(2): 237-243. DOI:10.1007/s12010-008-8395-9 |
| [56] |
GELDER J D, WILLEMSE-ERIX D, SCHOLTES M J, et al. Monitoring poly (3-hydroxybutyrate) production in Cupriavidus necator DSM 428(H16) with Raman spectroscopy[J]. Analytical Chemistry, 2008, 80(6): 2155-2160. DOI:10.1021/ac702185d |
| [57] |
HERMELINK A, STAMMLER M, NAUMANN D. Observation of content and heterogeneity of poly-beta-hydroxybutyric acid (PHB) in Legionella bozemanii by vibrational spectroscopy[J]. The Analyst, 2011, 136(6): 1129-1133. DOI:10.1039/c0an00526f |
| [58] |
覃赵军, 彭立新, 竺利波, 等. 碳源浓度影响微生物PHB合成代谢的单细胞拉曼光谱分析[J]. 中国激光, 2015, 42(3): 0315003. |
| [59] |
TAO Z H, PENG L X, ZHANG P, et al. Probing the Kinetic Anabolism of Poly-Beta-Hydroxybutyrate in Cupriavidus necator H16 using single-cell Raman spectroscopy[J]. Sensors, 2016, 16(8): 1257. DOI:10.3390/s16081257 |
| [60] |
覃赵军, 陶站华, 廖威, 等. 氮源影响PHB合成代谢的拉曼光谱分析[J]. 光学学报, 2016, 36(4): 0417001. |
| [61] |
TAO Z H, ZHANG P F, QIN Z J, et al. Poly(3-hydroxybutyrate) anabolism in Cupriavidus necator cultivated at various carbon-to-nitrogen ratios:Insights from single-cell Raman spectroscopy[J]. Journal of Biomedical Optics, 2016, 21(9): 97005. DOI:10.1117/1.JBO.21.9.097005 |
| [62] |
袁玉峰, 陶站华, 刘军贤, 等. 拉曼镊子分析红酵母合成类胡萝卜素[J]. 光谱学与光谱分析, 2011, 31(4): 1001-1005. |
| [63] |
王雪, 孙美娟, 刘军贤, 等. 光镊拉曼光谱法分析红法夫酵母内虾青素含量[J]. 光谱学与光谱分析, 2012, 32(9): 2433-2437. |
| [64] |
王雪, 孙美娟, 刘军贤, 等. 激光镊子拉曼光谱法优化红法夫酵母合成虾青素条件[J]. 光散射学报, 2013, 25(2): 152-157. |
| [65] |
孙美娟, 蒋玉凌, 来爱华, 等. 激光镊子拉曼光谱技术分析圆红冬孢酵母生成油脂和类胡萝卜素[J]. 激光与光电子学进展, 2013, 50(3): 174-179. |
| [66] |
WU H, VOLPONI J V, OLIVER A E, et al. In vivo lipidomics using single-cell Raman spectroscopy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(9): 3809-3814. DOI:10.1073/pnas.1009043108 |
| [67] |
WANG T T, JI Y T, WANG Y, et al. Quantitative dynamics of triacylglycerol accumulation in microalgae populations at single-cell resolution revealed by Raman microspectroscopy[J]. Biotechnology for Biofuels, 2014, 7(1): 58. DOI:10.1186/1754-6834-7-58 |
| [68] |
陶站华, 刘军贤, 师德强, 等. 基于单细胞拉曼光谱技术的葡萄球菌黄素生物合成分析[J]. 分析化学, 2016, 44(3): 456-461. |
| [69] |
陶站华, 柯珂, 师德强, 等. 利用光镊拉曼光谱研究环境因素对葡萄球菌黄素生物合成的影响[J]. 激光与光电子学进展, 2017, 54(12): 477-482. |
| [70] |
XIE C Y, NGUYEN N, ZHU Y, et al. Detection of the recombinant proteins in single transgenic microbial cell using laser tweezers and Raman spectroscopy[J]. Analytical Chemistry, 2007, 79(24): 9269-9275. DOI:10.1021/ac0710329 |
| [71] |
CHAN J W, WINHOLD H, CORZETT M H, et al. Monitoring dynamic protein expression in living E.coli bacterial cells by laser tweezers Raman spectroscopy[J]. Cytometry Part A:The Journal of the International Society for Analytical Cytology, 2007, 71(7): 468-474. |
| [72] |
卢明倩, 董蓉, 温顺华, 等. 单细胞激光拉曼光谱检测重组大肠杆菌细胞表达甲酸脱氢酶[J]. 分析化学, 2012, 40(12): 1845-1851. |
| [73] |
周冰, 卢明倩, 赵丽伟, 等. 激光镊子拉曼光谱检测重组大肠杆菌表达蚕豆14-3-3b可溶性蛋白与包涵体蛋白[J]. 分析化学, 2013, 41(12): 1789-1794. |
| [74] |
黄庶识, 卢明倩, 李冰, 等. 重组大肠杆菌表达可溶性蛋白和包涵体过程的拉曼光谱实时分析[J]. 中国激光, 2014, 41(12): 251-259. |
| [75] |
彭立新, 王桂文, 廖威, 等. 光镊拉曼光谱分析酿酒活性干酵母的活化与生长[J]. 微生物学通报, 2009, 36(8): 1137-1142. |
| [76] |
李自达, 赖钧灼, 威廖, 等. 浓醪乙醇发酵的单细胞拉曼光谱表征[J]. 光学学报, 2012, 32(3): 0317001. |
| [77] |
覃赵军, 赖钧灼, 刘斌, 等. 不同初始pH值的乙醇发酵过程拉曼光谱分析[J]. 中国激光, 2013, 40(2): 0215001. |
| [78] |
覃赵军, 赖钧灼, 彭立新, 等. 拉曼光谱分析有机氮源促进乙醇发酵的机制[J]. 分析化学, 2014, 42(10): 1471-1477. |
| [79] |
覃赵军, 彭立新, 王晓春, 等. 单细胞拉曼光谱分析高渗环境对酵母乙醇发酵代谢的影响[J]. 光子学报, 2016, 45(2): 0217002. |
| [80] |
赖钧灼, 李自达, 刘斌, 等. 基于单细胞拉曼光谱的酵母乙醇发酵动态过程研究[J]. 中国酿造, 2010(6): 48-52. |
| [81] |
GOLDRICK S, LOVETT D, MONTAGUE G, et al. Influence of incident wavelength and detector material selection on fluorescence in the application of Raman spectroscopy to a fungal fermentation process[J]. Bioengineering, 2018, 5(4): 79. DOI:10.3390/bioengineering5040079 |
| [82] |
CORMACK I G, MAZILU M, DHOLAKIA K, et al. Fluorescence suppression within Raman spectroscopy using annular beam excitation[J]. Applied Physics Letters, 2007, 91(2): 023903. DOI:10.1063/1.2756311 |
| [83] |
GALLIMORE P J, DAVIDSON N M, KALBERER M, et al. 1064 nm dispersive Raman microspectroscopy and optical trapping of pharmaceutical aerosols[J]. Analytical chemistry, 2018, 90(15): 8838-8844. DOI:10.1021/acs.analchem.8b00817 |
| [84] |
高国明, 李雪, 覃宗定, 等. 消除拉曼光谱荧光背景的新方法及其应用[J]. 光学学报, 2013, 33(2): 0230002-1-023002-9. |
| [85] |
LIU R, TAYLOR D S, MATTHEWS D L, et al. Parallel analysis of individual biological cells using multifocal laser tweezers Raman spectroscopy[J]. Applied Spectroscopy, 2010, 64(11): 1308-1310. DOI:10.1366/000370210793334972 |
| [86] |
ZHANG P F, KONG L B, SETLOW P, et al. Multiple-trap laser tweezers Raman spectroscopy for simultaneous monitoring of the biological dynamics of multiple individual cells[J]. Optics Letters, 2010, 35(20): 3321-3323. DOI:10.1364/OL.35.003321 |
| [87] |
黄超, 王强, 姚辉璐, 等. 微流控芯片-拉曼光镊的红细胞光谱检测技术[J]. 分析化学, 2007, 35(10): 1410-1414. |
| [88] |
毛丽华, 刘军贤, 陈萍, 等. 拉曼光镊流式细胞仪的构建及其在地中海贫血分析中的应用[J]. 分析化学, 2010, 38(11): 1578-1582. |
| [89] |
CASABELLA S, SCULLY P, GODDARD N, et al. Automated analysis of single cells using laser tweezers Raman spectroscopy[J]. The Analyst, 2016, 141(2): 689-696. DOI:10.1039/C5AN01851J |
| [90] |
PILÁT Z, JEŽEK J, KAŇKA J, et al.Raman tweezers in microfluidic systems for analysis and sorting of living cells[C].San Francisco: Proceedings of the Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues Ⅻ, International Society for Optics and Photonics, 2014: 89471M.1-89471M.9.
|
| [91] |
ZHANG P F, WANG G W, ZHANG X J, et al. Single-acquisition 2-D multifocal Raman spectroscopy using compressive sensing[J]. Analytical Chemistry, 2020, 92(1): 1326-1332. DOI:10.1021/acs.analchem.9b04495 |
| [92] |
ZHANG P F, WANG G W, HUANG S S. Parallel micro-Raman spectroscopy of multiple cells in a single acquisition using hierarchical sparsity[J]. The Analyst, 2020, 145(18): 6032-6037. DOI:10.1039/D0AN01081B |
| [93] |
HUAI Q Y, GAO C L, MIAO J L, et al. Fast detection of saxitoxin using laser tweezers surface enhanced Raman spectroscopy[J]. Analytical Methods, 2013, 5(23): 6870-6873. DOI:10.1039/c3ay41504j |
| [94] |
RAMSER K, LOGG K, GOKSOR M, et al. Resonance Raman spectroscopy of optically trapped functional erythrocytes[J]. Journal of Biomedical Optics, 2004, 9(3): 593-601. DOI:10.1117/1.1689336 |
| [95] |
RAMSER K, WENSELEERS W, DEWILDE S, et al. The combination of resonance Raman spectroscopy, optical tweezers and microfluidic systems applied to the study of various heme-containing single cells[J]. Spectroscopy, 2008, 22(4): 287-295. DOI:10.1155/2008/463191 |
| [96] |
RAMSER K, WENSELEERS W, DEWILDE S, et al. Micro-resonance Raman study of optically trapped Escherichia coli cells overexpressing human neuroglobin[J]. Journal of Biomedical Optics, 2007, 12(4): 044009. DOI:10.1117/1.2753478 |
| [97] |
DE GELDER J, DE GUSSEM K, VANDENABEELE P, et al. Methods for extracting biochemical information from bacterial Raman spectra:Focus on a group of structurally similar biomolecules-fatty acids[J]. Analytica Chimica Acta, 2007, 603(2): 167-175. DOI:10.1016/j.aca.2007.09.049 |
| [98] |
IWASAKI K, KANEKO A, TANAKA Y, et al. Visualizing wax ester fermentation in single Euglena gracilis cells by Raman microspectroscopy and multivariate curve resolution analysis[J]. Biotechnology for Biofuels, 2019, 12(1): 128. DOI:10.1186/s13068-019-1471-2 |
| [99] |
HE Y H, ZHANG P, HUANG S, et al. Label-free, simultaneous quantification of starch, protein and triacylglycerol in single microalgal cells[J]. Biotechnology for Biofuels, 2017, 10(1): 275. DOI:10.1186/s13068-017-0967-x |
| [100] |
TENG L, WANG X, WANG X J, et al. Label-free, rapid and quantitative phenotyping of stress response in E.coli via ramanome[J]. Scientific Reports, 2016, 6: 34359. DOI:10.1038/srep34359 |
| [101] |
HE Y H, WANG X X, MA B, et al. Ramanome technology platform for label-free screening and sorting of microbial cell factories at single-cell resolution[J]. Biotechnology Advances, 2019, 37(6): 107388. DOI:10.1016/j.biotechadv.2019.04.010 |
| [102] |
WANG Y, SONG Y Z, TAO Y F, et al. Reverse and multiple stable isotope probing to study bacterial metabolism and interactions at the single cell level[J]. Analytical Chemistry, 2016, 88(19): 9443-9450. DOI:10.1021/acs.analchem.6b01602 |
| [103] |
XU J B, ZHU D, IBRAHIM A D, et al. Raman deuterium isotope probing reveals microbial metabolism at the single-cell level[J]. Analytical Chemistry, 2017, 89(24): 13305-13312. DOI:10.1021/acs.analchem.7b03461 |
| [104] |
LU W, CHEN X, WANG L, et al. Combination of an artificial intelligence approach and laser tweezers Raman spectroscopy for microbial identification[J]. Analytical Chemistry, 2020, 92(9): 6288-6296. DOI:10.1021/acs.analchem.9b04946 |
| [105] |
GONZLEZ-CABALEIRO R, MITCHELL A M, SMITH W, et al. Heterogeneity in pure microbial systems:Experimental measurements and modeling[J]. Frontiers in Microbiology, 2017, 8: 1813. DOI:10.3389/fmicb.2017.01813 |