2. 广西水产科学研究院, 广西南宁 530021
2. Guangxi Academy of Fishery Science, Nanning, Guangxi, 530021, China
罗非鱼具有食性广、环境适应性和抗病能力强等特点,是主要的水产养殖种类之一。池塘养殖是我国淡水养殖业最重要的养殖模式。2018年我国池塘养殖面积为266.68万hm2,占全国淡水养殖面积的51.82%,同年我国淡水养殖产量为2 959.84万t,池塘养殖产量为2 210.97万t,占全国淡水养殖产量的74.70%[1]。近年来,稻鱼综合种养生态养殖技术得到广泛应用,主要是由于该技术能够有效利用稻鱼互利共生的关系,改善养殖生态条件,对提高水产养殖业的经济社会效益具有重要作用。
动物的肠道菌群组成与其生活的环境因子、生理状态和发育阶段等因素有着密切联系[2-4]。水生动物生活在水环境中,其消化道短,肠道菌群的相对丰度容易受到外界环境的影响,进而也会影响养殖动物健康及其肠道微生物组成[5-6]。肠道微生物在鱼类的生长发育过程中起着非常重要的作用,既能参与机体自身营养物质的消化吸收,同时也可调控机体的防御功能,从而促进鱼类的健康发育。李存玉等[7]在池塘和工厂化两种不同养殖条件下养殖牙鲆,发现牙鲆的肠道微生物菌群结构差别很大,菌群丰度与多样性差异明显,不同养殖环境对牙鲆肠道微生物的影响不同;Li等[8]采用16S rRNA克隆文库方法,对实验室养殖条件下的南美白对虾肠道细菌进行多样性研究,发现南美白对虾肠道的16S rRNA克隆文库中126个克隆子分属2个不同的细菌类群:变形菌门和厚壁菌门;佟延南等[9]研究发现,罗非鱼肠道内乳球菌属随养殖周期的推移呈现下降趋势,芽孢杆菌属、假单胞菌属和分枝杆菌属呈先增后减的变化趋势,梭菌属和邻单胞菌属在养殖前期所占比例最高。之前的研究主要集中在养殖环境和饲料添加对鱼类肠道微生物的影响方面,对其肠道微生物分布情况的研究比较匮乏[10]。因此,本实验采用16S rRNA高通量测序技术,对池塘和稻田两种不同养殖环境下,罗非鱼肠道菌群组成变化进行研究,为不同养殖方式下罗非鱼肠道微生物生理学研究提供基础数据和参考。
1 材料与方法 1.1 实验动物及养殖实验鱼分为稻田组(DT组)和池塘组(CT组)两组,分别养殖于隆安县那桐镇定江村定典屯水稻研究所实验基地和国家级罗非鱼良种场(广西壮族自治区水产科学研究院武鸣基地),养殖鱼苗规格均为150—200 g,同一天投放,投放密度为3 000尾/hm2,每天投喂罗非鱼2次,上午、下午各一次。
1.2 取样方法在基地每隔30 d检测养殖水体的水温、溶氧(DO)、pH值和氨氮(纳氏试剂比色法)。
养殖202 d后,每组随机选取健康无病、体格相近的实验鱼3尾(分别记为稻田组DT1、DT2、DT3,池塘组CT1、CT2、CT3),经测量,体长为(20.0±3.5) cm,体质量为(400±26) g。使用间氨基苯甲酸乙酯甲磺酸盐(MS-222)对其深度麻醉,用70%乙醇擦拭罗非鱼体表,解剖罗非鱼后取肠道微生物置于2 mL冻存管中,随即投入液氮中速冻,后置-80℃冰箱保存备用。
1.3 PCR扩增及基因文库构建通过16S rRNA高通量测序检测其肠道微生物。
1.3.1 基因组DNA的提取和PCR扩增采用CTAB方法[11]对样本基因组DNA进行提取,并利用琼脂糖凝胶电泳检DNA纯度和浓度,取适量的样本DNA于离心管中,使用无菌水稀释至1 ng/μL。以稀释后的基因组DNA为模板,根据测序区域的选择,使用带Barcode的特异引物(表 1),New England Biolabs公司的PhusionⓇ High-Fidelity PCR Master Mix with GC Buffer和高效高保真酶进行PCR,确保扩增效率和准确性。
| 区域 Region |
引物名称 Primer name |
引物序列(5′→3′) Primer sequence (5′→3′) |
| V4 | 515F | GTGCCAGCMGCCGCGGTAA |
| 806R | GGACTACHVGGGTWTCTAAT | |
| V3-V4 | 341F | CCTAYGGGRBGCASCAG |
| 806R | GGACTACNNGGGTATCTAAT | |
| 注:下划线部分为Barcode序列 Note:Barcode sequence is underlined |
||
1.3.2 PCR产物的混样和纯化
根据浓度进行将上述PCR产物等量混样,充分混匀后使用1×TAE浓度2%的琼脂糖凝胶电泳纯化PCR混合产物,再用Thermo Scientific公司的GeneJET胶回收试剂盒回收产物。
1.3.3 基因文库构建使用Thermofisher公司的Ion Plus Fragment Library Kit 48 rxns建库试剂盒,参照试剂盒说明书进行基因文库的构建。
1.4 测序分析 1.4.1 数据处理所有测序分析均由生工生物公司完成。Barcode序列是Miseq测序序列中的一种,主要是对reads中的数据进行拆分,此外还有一种接头序列[12]。在对测序的数据进行处理时,需要将引物接头序列去除。此外,存在于PE reads之间的overlap关系可以将成对的reads[13]整合成一条序列,通过Barcode标签可将相应的序列识别出来,进而得到各种各样的样本数据。为了确保样本数据的有效性,需对这些样本数据的质量进行控制和过滤,从而得到实验最终所需的样本数据[14]。稀释曲线采用Mothur v1.30进行分析。
1.4.2 OUTs聚类和物种注释利用Uparse软件[15]对所有样品的全部Clean Reads进行聚类,默认以97%的一致性(Identity)将序列聚类成为Operational Taxonomic Units(OTUs),利用R语言工具制作Rank Abundance曲线图,同时依据其算法原则,筛选OTUs中出现频数最高的序列作为代表序列。
用Mothur方法与SILVA132[16]的SSUrRNA数据库[17]对OTUs序列进行物种注释分析(设定阈值为0.8—1.0),获得分类学信息并分别在门(Phylum)和属(Genus)水平统计各样本的群落组成。使用MUSCLE[18]软件进行快速多序列比对,得到所有OTUs序列的系统发生关系。最后以样品中数据量最少的为标准,进行均一化处理。
2 结果与分析 2.1 水质变化情况稻田和池塘水质参数比较如图 1所示。结果显示,养殖水质参数均在正常范围内,两种环境下,溶氧、水温、pH值差异不大,氨氮参数有显著差异。

|
| 图 1 稻田和池塘溶氧、水温、pH值、氨氮参数比较(P < 0.05) Fig.1 Comparison of dissolved oxygen, water temperature, pH, ammonia nitrogen parameters between rice field and pond (P < 0.05) |
2.2 肠道菌群α多样性分析结果
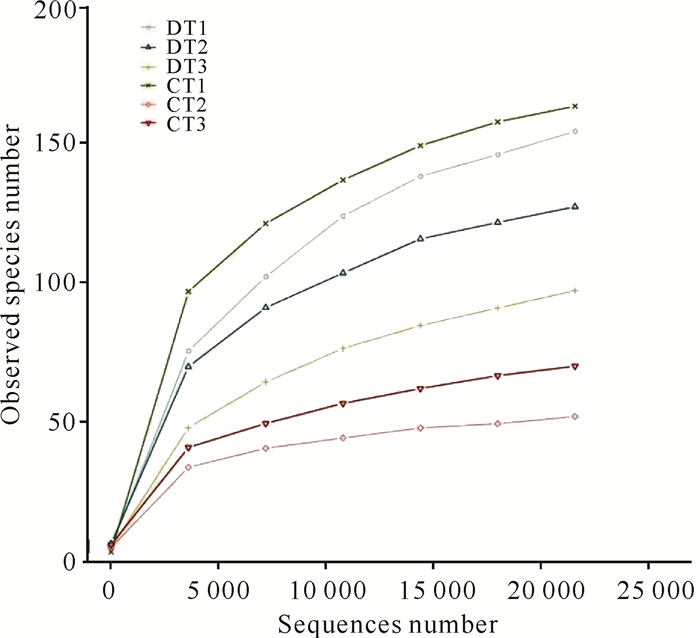
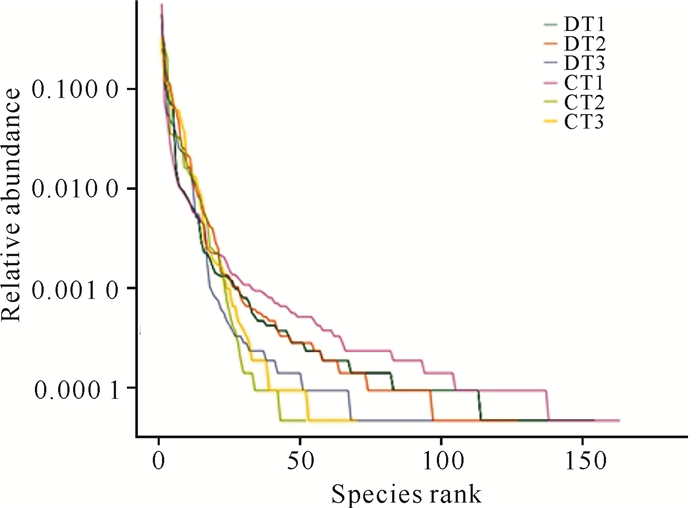
实验样本稀释曲线斜率随测序通量的增大而逐渐降低,最终趋于平稳(图 2)。Rank abundance曲线反映出两组罗非鱼的肠道内物种分布均匀(图 3)。综上所述,随着测序深度的增加,测序量已饱和且测序数据量合理,各样本中几乎所有细菌序列均被检测出。2个实验组样品的菌群α多样性因养殖环境的不同而存在差异。
|
| 图 2 不同实验组罗非鱼肠道内容物样本的稀释曲线 Fig.2 Dilution curves of tilapia intestinal contents samples from two experimental groups |
|
| 图 3 Rank abundance曲线 Fig.3 Curve of Rank abundance |
2.3 肠道菌群结构 2.3.1 门水平下的肠道菌群结构
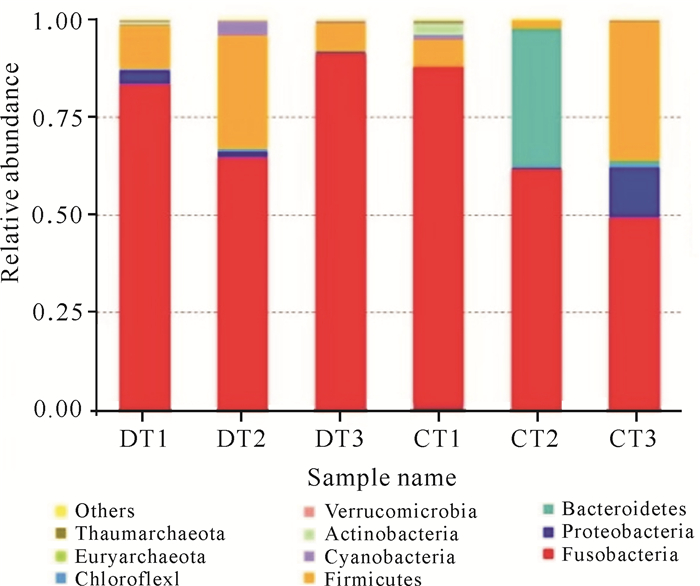
实验结果表明,梭杆菌门(Fusobacteria)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)在稻田组和池塘组罗非鱼肠道菌群中相对丰度均较高,蓝菌门(Cyanobacteria)在稻田组的相对丰度较高,拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)在池塘组的相对丰度较高(图 4)。表明不同养殖环境下罗非鱼肠道菌群种类相似,菌群的相对丰度有差异。变形菌门、拟杆菌门、梭杆菌门、厚壁菌门以及放线菌门的相对丰度都发生不同程度的变化。其中池塘组的罗非鱼肠道梭杆菌门相对丰度低于稻田组,变形菌门相对丰度高于稻田组,其他优势菌门之间也存在差异。
|
| 图 4 门水平下不同实验组罗非鱼肠道菌群的相对丰度 Fig.4 Relative abundance of tilapia intestinal flora at the phyla level in different test groups |
2.3.2 属水平下的肠道菌群结构
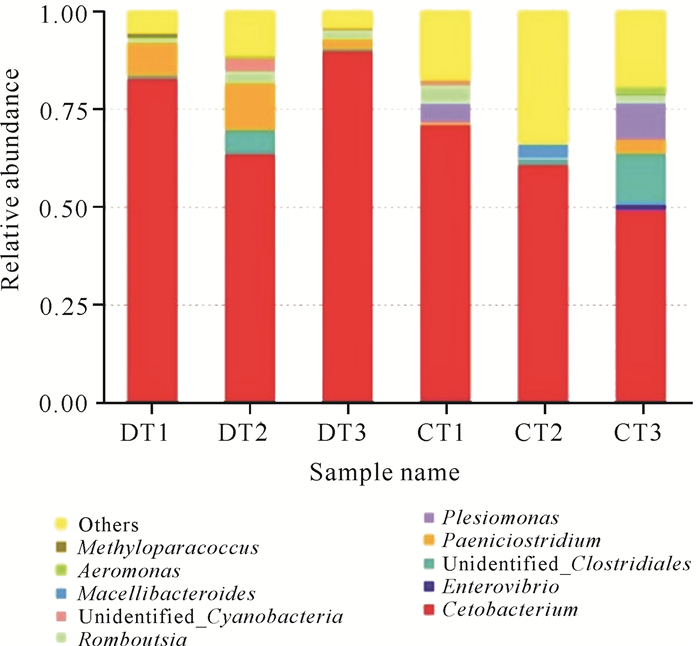
研究发现,Cetobacterium,Paeniclostrdium和Romboutsia等菌属在稻田组罗非鱼肠道菌群相对丰度较高,池塘组罗非鱼肠道菌群相对丰度较高的菌属为Cetobacterium,Enterovibrio和Plesiomonas等;Cetobacterium在稻田组和池塘组的相对丰度最高,均占50%以上;Paeniclostrdium占稻田组的相对丰度高于池塘组,Plesiomonas在池塘组中相对丰度较高,但在稻田组中所占比例较小(图 5)。
|
| 图 5 属水平下不同实验组罗非鱼肠道菌群的相对丰度 Fig.5 Relative abundance of tilapia gut flora at the genus level in different test groups |
3 讨论 3.1 水质参数的影响
本研究结果显示,养殖水质参数均在正常范围内,两种环境下,溶氧、水温、pH值差异不大,氨氮参数有显著差异,这可能是因为集约化、高密度养殖以及饲料的过度使用,造成水体中氨氮含量升高,引起水体富营养化,对环境产生污染,从而影响罗非鱼的生长发育,导致罗非鱼肠道微生物的结构组成和多样性发生变化。张皓[5]通过高通量测序技术调查了南美白对虾、鲤科鱼养殖池塘水体、底泥、肠道微生物群落结构和多样性的变化发现,水体中的氨氮、化学需氧量及总氮对水体中细菌种群影响最大,对水生动物肠道微生物种群结构也产生相互影响。这说明水体中的氨氮会影响养殖水体中细菌种群的数量,进而影响肠道菌群的组成和多样性。
3.2 肠道菌群结构微生物具有调控养殖环境的能力,对水产养殖系统的健康至关重要[19]。肠道微生物菌群结构、功能与罗非鱼的营养代谢、免疫防御、胃肠道发育等生理过程密切相关,是维持机体肠道内环境稳定的重要因素,在罗非鱼健康养殖过程中发挥着重要作用[20]。根据肠道菌群α多样性分析结果可看出,本实验测序数据量充足,池塘养殖和稻田养殖的罗非鱼肠道中物种均匀度高,物种丰富。两个环境组的样品菌群多样性因养殖环境不同存在差异,说明不同养殖环境下,罗非鱼的肠道微生物多样性有明显变化。
随着二代测序技术和生物信息学分析的应用,大量鱼类肠道菌群逐渐被发现,主要包括变形菌门(Proteobacteria)、梭杆菌门(Fusobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、梭状芽孢杆菌门(Clostridia)、杆菌门(Bacilli)和疣微菌门(Verrucomicrobia)[21-23]。其中,这些优势菌群在不同鱼类的肠道菌群中占90%以上,且对鱼类肠道功能起着不可替代的影响和作用[24]。本实验对不同养殖环境下罗非鱼肠道菌群变化情况进行比较分析发现,变形菌门、厚壁菌门和拟杆菌门在不同养殖环境条件下的罗非鱼肠道菌群中相对丰度较高,蓝细菌门在稻田组肠道菌群相对丰度较高,梭杆菌门和放线菌门在池塘组肠道菌群相对丰度较高。表明不同养殖环境下罗非鱼肠道菌群分布情况存在一定差异,这可能是由于鱼类的肠道微生物与水体环境密切相关,容易受到食物和环境变化等影响[2]。梭杆菌门、变形菌门[25-27]、厚壁菌门[28]和拟杆菌门[29]是罗非鱼肠道中的优势菌群,这一结果与鲤鱼、虹鳟[30]及金鲳[31]肠道菌群多样性规律一致。Lyons等[32]和Ye等[33]研究发现,优势菌群与宿主的健康和营养息息相关,优势菌群会影响宿主自身微生物的组成、群落结构以及菌落结构,而肠道菌群结构的失衡也有可能会导致宿主疾病的产生,但其在不同鱼类肠道中所发挥的作用有待进一步证实。
属水平下,不同养殖环境的罗非鱼肠道菌群相对菌属分布情况存在差异,稻田组罗非鱼肠道菌群相对丰度较高的菌属是Cetobacterium,Paeniclostrdium和Romboutsia等,而池塘组相对丰度较高菌属是Cetobacterium,Enterovibrio和Plesiomonas等。大量研究发现,Cetobacterium是淡水鱼类微生物群的组成部分[34],例如Tsuchiya等[35]和Van等[36]研究发现,Cetobacterium和Plesiomonas在各养殖阶段罗非鱼肠道菌群数量均占2.00%,是罗非鱼肠道菌群的重要组成成分,这与本实验结果相符。本研究还发现,池塘组和稻田组罗非鱼含有大量的未知菌属,还需要后续的深入研究。
鱼类肠道微生物的数量、群落结构与环境、饵料和发育等因素密切相关,具体表现为养殖水体中的微生物、盐度、水温,鱼类摄食的饵料、药物及其生理状态和发育阶段对鱼类消化道菌群结构均有影响[21, 37]。该结论可为罗非鱼肠道益生菌的筛选提供科学依据,并倡导罗非鱼养殖生产过程中微生物制剂的科学使用,减少药残对生态环境的污染,改善罗非鱼的生长性能及品质。
| [1] |
农业农村部渔业渔政管理局编制. 中国渔业统计年鉴[M]. 北京: 中国农业出版社, 2019.
|
| [2] |
RUNG W, KLAN A, MAIBUNK S, et al. Characterization of intestinal bacteria in wild and domesticated adult black tiger shrimp (Penaeus monodon)[J]. PloS One, 2014, 9(3): e91853. DOI:10.1371/journal.pone.0091853 |
| [3] |
ELENI M, ANDREW T, ELENI N, et al. Gut microbial communities associated with the molting stages of the giant freshwater prawn Macrobrachium rosenbergii[J]. Aquaculture, 2016, 463: 181-188. DOI:10.1016/j.aquaculture.2016.05.045 |
| [4] |
LEY R E, PERERSON D A, GORDON J I. Ecological and evolutionary forces shaping microbial diversity in the human intestine[J]. Cell, 2006, 124(4): 837-848. DOI:10.1016/j.cell.2006.02.017 |
| [5] |
张皓.养殖环境微生物群落的动态变化及与水环境的互作[D].苏州: 苏州大学, 2015.
|
| [6] |
尹军霞, 沈文英, 郦萍. 水温对南美白对虾肠道菌群影响的研究[J]. 海洋科学, 2004, 28(5): 33-36. DOI:10.3969/j.issn.1000-3096.2004.05.007 |
| [7] |
李存玉, 徐永江, 柳学周, 等. 池塘和工厂化养殖牙鲆肠道菌群结构的比较分析[J]. 水产学报, 2015, 39(2): 245-255. |
| [8] |
LI K, ZHENG T L, TIAN Y, et al. Bacterial community structure in intestine of the white shrimp, Litopenaeus vannamei[J]. Acta Microbiologica Sinica, 2007, 47(4): 649-653. |
| [9] |
佟延南, 李芳远, 李忠琴, 等. 不同养殖阶段罗非鱼肠道微生物多样性的动态分析[J]. 南方农业学报, 2018, 49(7): 1415-1422. DOI:10.3969/j.issn.2095-1191.2018.07.24 |
| [10] |
孙振丽, 宣引明, 张皓, 等. 南美白对虾养殖环境及其肠道细菌多样性分析[J]. 中国水产科学, 2016, 23(3): 594-605. |
| [11] |
左欣欣, 陈泽宇, 马腾, 等. 从动物组织中提取总RNA的CTAB法[J]. 中国西部科技, 2011, 10(2): 35-36, 76. DOI:10.3969/j.issn.1671-6396.2011.02.018 |
| [12] |
MARTIN M. Cut adapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet Journal, 2011, 17(1): 10-12. DOI:10.14806/ej.17.1.200 |
| [13] |
ROGNES T, FLOURI T, NICHOLS B, et al. VSEAR-CH:A versatile open source tool for metagenomics[J]. Peer J, 2016, 4(10): e2584. |
| [14] |
HAAS B J, GEVERS D, EARL A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110 |
| [15] |
EDGAR R C. UPARSE:Highly accurate OTU seque-nces from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [16] |
WANG Q, GARRITY G M, TIEDJE J M, et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [17] |
QUAST C, PRUESSE E, YILMAZ P, et al. The SILVA ribosomal RNA gene database project:Improved data processing and web-based tools[J]. Nucleic Acids Research, 2012, 41(D1): 590-596. DOI:10.1093/nar/gks1219 |
| [18] |
EDGAR R C. MUSCLE:Multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research, 2004, 32(5): 1792-1797. DOI:10.1093/nar/gkh340 |
| [19] |
MARTINEZ L R, EMERENCIANO M, MIRANDA A, et al. Microbial-based systems for aquaculture of fish and shrimp:An updated review[J]. Reviews in Aquaculture, 2015, 7(2): 131-148. |
| [20] |
陈孝煊, 吴志新, 周文豪. 鱼类消化道菌群的作用与影响因素研究进展[J]. 华中农业大学学报, 2005, 24(5): 523-528. DOI:10.3321/j.issn:1000-2421.2005.05.024 |
| [21] |
赵翔刚, 罗衡, 刘其根, 等. 稻田养殖沙塘鳢对稻田水体及底泥微生物群落结构及多样性的影响[J]. 淡水渔业, 2017, 47(4): 8-14. DOI:10.3969/j.issn.1000-6907.2017.04.003 |
| [22] |
陈明强, 李有宁, 邓正华, 等. 恩诺沙星曝露下企鹅珍珠贝肠道微生物多样性及优势菌变化规律[J]. 南方农业学报, 2019, 50(4): 851-859. DOI:10.3969/j.issn.2095-1191.2019.04.24 |
| [23] |
陈琼, 李贵阳, 罗坤, 等. 凡纳滨对虾(Litopenaeus vannamei)亲虾繁殖期水体微生物多样性[J]. 海洋与湖沼, 2017, 48(1): 130-138. |
| [24] |
ZHENG Y F, YU M, LIU Y, et al. Comparison of cultivable bacterial communities associated with Pacific white shrimp (Litopenaeus vannamei) larvae at different health statuses and growth stages[J]. Aquaculture, 2016, 451: 163-169. DOI:10.1016/j.aquaculture.2015.09.020 |
| [25] |
DEMERGASSO C, CASAMAYOR E O, CHONG G, et al. Distribution of prokaryotic genetic diversity in athalassohaline lakes of the Atacama Desert, Northern Chile[J]. FEMS Microbiology Ecology, 2004, 48(1): 57-69. DOI:10.1016/j.femsec.2003.12.013 |
| [26] |
STEVENS H, STUBNER M, SIMON M, et al. Phylogeny of Proteobacteria and Bacteroidetes from oxic habitats of a tidal flat ecosystem[J]. FEMS Microbiology Ecology, 2005, 54(3): 351-365. DOI:10.1016/j.femsec.2005.04.008 |
| [27] |
WU L Y, WEN C Q, QIN Y J, et al. Phasing amplicon sequencing on Illumina Miseq for robust environmental microbial community analysis[J]. BMC Microbiology, 2015, 15(1): 125. |
| [28] |
KOMAROFF A L. The microbiome and risk for obesity and diabetes[J]. JAMA, 2017, 317(4): 355-356. DOI:10.1001/jama.2016.20099 |
| [29] |
COTTRELL M T, KIRCHMAN D L. Natural assemblages of marine proteobacteria and members of the Cytophaga-Flavobacter cluster consuming low-and high-molecular-weight dissolved organic matter[J]. Applied and Environmental Microbiology, 2000, 66(4): 1692-1697. DOI:10.1128/AEM.66.4.1692-1697.2000 |
| [30] |
SPANGGAARD B, HUBER I, NIELSEN J, et al. The microflora of rainbow trout intestine:A comparison of traditional and molecular identification[J]. Aquaculture, 2000, 182(1/2): 1-15. |
| [31] |
CHEN B, GAO L L, PAN Q. Woody forages effect the intestinal bacteria diversity of golden pompano Trachinotus ovatus[J]. AMB Express, 2018, 8(1): 29. DOI:10.1186/s13568-018-0550-2 |
| [32] |
LYONS P P, TURNBULL J F, DAWSON K A, et al. Exploring the microbial diversity of the distal intestinal lumen and mucosa of farmed rainbow trout Oncorhynchus mykiss (Walbaum) using next generation sequencing (NGS)[J]. Aquaculture Research, 2017, 48(1): 77-91. DOI:10.1111/are.12863 |
| [33] |
YE L, AMBERG J, CHAPMAN D, et al. Fish gut microbiota analysis differentiates physiology and behavior of invasive Asian carp and indigenous American fish[J]. The ISME Journal, 2014, 8(3): 541-551. DOI:10.1038/ismej.2013.181 |
| [34] |
RAM C, CORONADO J, SILVA A, et al. Cetobacterium is a major component of the microbiome of giant amazonian fish (Arapaima gigas) in Ecuador[J]. Animals, 2018, 8(11): 189. DOI:10.3390/ani8110189 |
| [35] |
TSUCHIYA C, SAKATA T, Sugita H. Novel ecological niche of Cetobacterium somerae, an anaerobic bacterium in the intestinal tracts of freshwater fish[J]. Letters in Applied Microbiology, 2008, 46(1): 43-48. |
| [36] |
VAN M A, DUTILH B E, NEVELING K, et al. Pyrosequencing of 16S rRNA gene amplicons to study the microbiota in the gastrointestinal tract of carp (Cyprinus carpio L.)[J]. AMB Express, 2011, 1(1): 41. DOI:10.1186/2191-0855-1-41 |
| [37] |
ZHANG M L, SUN Y H, CHEN K, et al. Characterization of the intestinal microbiota in Pacific white shrimp, Litopenaeus vannamei, fed diets with different lipid sources[J]. Aquaculture, 2014, 434: 449-455. DOI:10.1016/j.aquaculture.2014.09.008 |



