2. 广西壮族自治区海洋研究所, 广西海洋生物技术重点实验室, 广西北海 536000;
3. 广西民族大学海洋与生物技术学院, 广西南宁 530006
2. Guangxi Key Laboratory for Marine Biotechnology, Guangxi Institute of Oceanography, Beihai, Guangxi, 536000, China;
3. School of Marine Sciences and Biotechnology, Guangxi University for Nationalities, Nanning, Guangxi, 530006, China
广西是我国重要的海水养殖省份之一,其长达1 600 km的海岸线及优质的水环境为广西沿海水产增养殖业的发展奠定了坚实的基础[1-3]。“十二五”期间,广西北部湾海域的海水养殖业迅猛发展,海水养殖面积达到550 km2[1, 4]。然而自2008年以来,广西北部湾经济区上升为国家战略,随着周边社会经济的发展,以及城市化、工业化进程加快,给优越的广西海域生态环境带来了前所未有的巨大压力[5]。在工业污染及人类活动的影响下,广西近岸海域富营养化趋势明显,有害藻华频繁暴发,发生的次数、规模、持续时间和有害藻种类均呈现明显上升趋势,对广西海水养殖业造成巨大的破坏,严重影响着广西海水养殖业的健康可持续发展[6]。例如,2014年和2015年冬季,广西近岸海域的棕囊藻赤潮暴发,影响区覆盖了防城港附近海域、钦州湾和北海附近海域,严重破坏了海洋生态环境[7]。
形成有害藻华的微藻种类多种多样,它们对海洋生物产生危害的途径大致有几种,主要包括改变水体理化环境导致水体恶化,或分泌黏液附着在海水动物的鳃组织造成严重损伤、造成水体缺氧等。部分有害藻华会产生毒素,这些毒素不仅导致海产品的污染及死亡,还可能通过食物链危及人类健康[7-8]。相对于珠江口及长江口等海域,广西近海的赤潮发生次数较少,其规模与危害也较小,然而广西北部湾海域有害藻华的研究工作却相对滞后,尤其是关于广西海水养殖区域的有害藻华种类及其危害,尚缺乏系统的研究。由于不同的藻类引起的藻华危害养殖生物的途径有差异,故对养殖区有害藻华的种类进行准确地分类学鉴定是藻华防控的基础工作。传统的海洋微藻鉴定工作主要借助显微镜观察,不仅繁琐耗时,还需要较高的专业水平。近年来,随着分子生物学的发展,分子生物学技术成为物种鉴定的经济快捷精确的手段。尤其是18S rDNA和ITS基因已被广泛应用于分子系统学研究,解决了许多疑难海洋藻类的分类鉴定问题[9]。
为了对北部湾养殖区有害藻华的种类进行精准鉴定。本研究在北部湾对虾养殖区的藻华水样中分离得到一株原甲藻并进行单克隆培养,采用传统的显微镜观察法结合18S rDNA序列分析对其进行种类鉴定,拟为了解该原甲藻的分类学地位及其与其他种或株系间的亲缘关系,并为今后北部湾海水养殖区域有害藻华的监控和防治提供基础资料。
1 材料与方法 1.1 主要试剂和仪器试剂:鲁格氏碘液,福尔马林溶液,乙醇,丙酮,植物DNA提取试剂盒(康为),琼脂糖,TEB缓冲溶液。
仪器:倒置显微镜(型号:Ts2, Nikon),扫描电子显微镜(型号:S-3400 N,Hitachi),离心机(型号:3-18KS,Sigma),PCR仪(型号:BOEN-25697B,上海伯恩),电泳仪(型号:170-3940,Bio-Rad)。
1.2 采样和藻种分离培养2018年5月在广西钦州的北部湾对虾养殖区采集藻华水样,其中一瓶加入鲁格试剂固定,用于藻密度计数以及形态观察。另一瓶不加固定剂,用于分离藻种:在倒置显微镜下,用毛细管分离法从水样中挑选活力较好的细胞于96孔板中进行单克隆培养。待细胞分裂到一定数量后,转移到具塞玻璃管中,用f/2培养基在22 ℃、光暗比为12 h:12 h的条件下培养。分离获得的藻种保存于广西科学院,编号为BBGA-9003。
1.3 藻体形态观察 1.3.1 光学显微镜观察吸取适量用鲁格试剂固定的水样滴于载玻片上,在倒置显微镜下观察藻体形态并拍照。
1.3.2 扫描电子显微镜观察取20 mL处于指数生长期的藻液,用体积分数为1%的鲁格氏碘液和体积分数为4%的福尔马林固定1 h,用10 μm筛绢过滤浓缩,用超纯水洗涤3~4次,再依次用体积分数分别为15%、30%、50%、70%、80%、90%及100%的乙醇梯度脱水,每个浓度脱水10 min,最后1个梯度重复脱水2次,最后以体积分数100%丙酮置换30 min。在扫描电子显微镜下观察并拍照。
1.4 基因组DNA的提取及18S rDNA的克隆取约50 mL新鲜藻体于4 ℃、5 000 r/min下离心5 min,弃上清,收集藻体用于提取DNA。总DNA提取按照植物DNA提取试剂盒(康为)的使用说明进行,以提取的总DNA为模板进行PCR克隆核糖体18S rDNA片段。PCR引物为18S-F:5′-GCTTGTCTCAAAGATTAAGCCATGC-3′和18S-R:5′- CACCTACGGAAACCTTGTTACGAC-3′,由华大基因公司合成。PCR条件:94 ℃预变性3 min;然后94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,共30个循环;最后72 ℃延伸10 min。
1.5 PCR产物电泳分析采用琼脂糖凝胶电泳法对PCR产物进行检测,缓冲液为TBE。PCR产物切胶回收并交由华大基因公司测序。
1.6 数据分析使用NCBI对18S rDNA基因序列进行比对。在MEGA7软件中运用邻接法(Neighbor-Joining method)构建分子系统树并计算各序列间的遗传距离。
2 结果与分析 2.1 藻密度、藻体形态特征及种类鉴定计数结果表明,广西北部湾对虾养殖区藻华水样中藻密度为5.5×105 cells·L-1,是水样中的唯一优势种。
分离水样获得编号为BBGA-9003的藻种,显微观察发现,该藻细胞形态多变,大多细胞类似心形或卵形,少数近似三角形,细胞顶端较宽,后部较窄,长和宽分别为(16.55±1.42) μm和(13.84±1.86) μm,顶端有2条鞭毛(图 1a)。在相差显微镜下,可以看到该藻壳面上分布有稀疏的刺丝胞孔,细胞前端中央的鞭毛孔周围呈“V”字形,具有约1 μm长的顶刺(图 1b)。以上特征与原甲藻属(Prorocentrum)形态特征符合,故可初步判断BBGA-9003为原甲藻属。
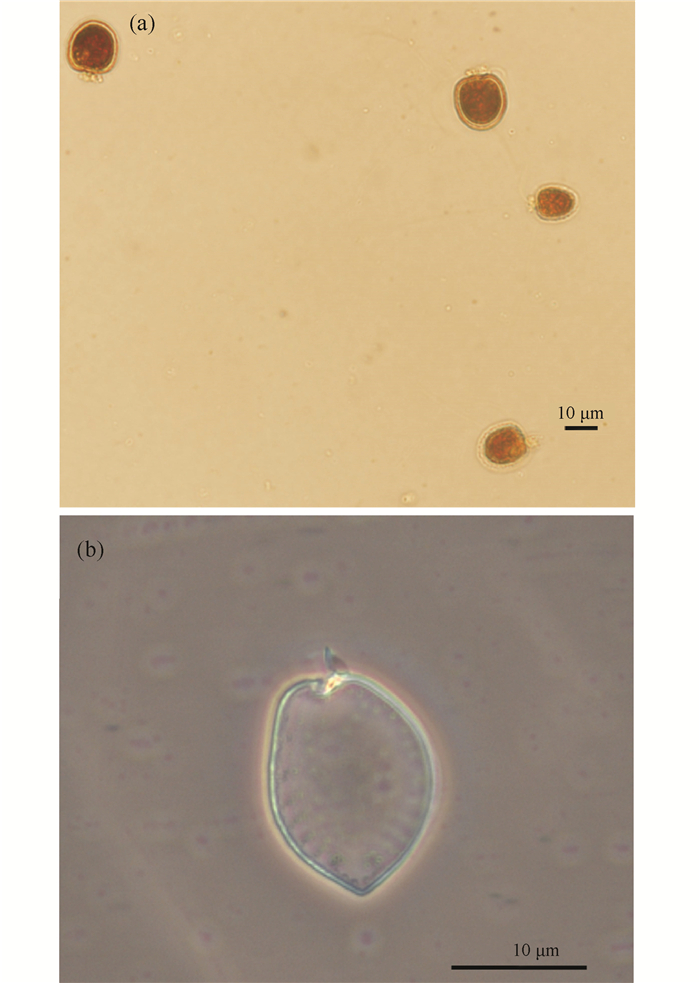
|
| (a)明视野观察; (b)相差显微镜观察 (a)Observed by bright-field microscope; (b)Observed by phase-contrast microscopy 图 1 BBGA-9003在显微镜下的结构 Fig.1 BBGA-9003 structure under the microscope |
电镜观察发现细胞正面观也呈心形或卵形(图 2a),腹面观为凸透镜或椭圆形(图 2b)。藻体由左右两壳板组成,壳面覆盖许多小刺,刺丝胞孔稀疏,细胞表面呈现微小原甲藻典型的规则花纹,因此可以确认该藻为微小原甲藻(Prorocentrum minimum)。

|
| (a)正面观Anterior view; (b)腹面观Side view 图 2 BBGA-9003藻种的扫描电镜结果(图中白色箭头所指处为刺丝胞孔) Fig.2 Scanning electron microscopy results of BBGA-9003 algae species (The white arrows shows the position of thorn holes) |
2.2 18S rDNA序列的克隆与测序
琼脂糖凝胶电泳显示,PCR产物大小约1 800 bp。经测定,BBGA-9003藻株18S rDNA部分序列的长度为1 220 bp。
2.3 序列比对将BBGA-9003藻株18S rDNA序列在NCBI中进行BLAST同源性分析,结果显示该序列与GenBank中56条原甲藻属的序列具有很高的同源性(96%~100%),这些序列来源包括微小原甲藻(P.minimum)、心形原甲藻(P.cordatum)、东海原甲藻(P.donghaiense)、具齿原甲藻(P.dentatum)、P.shikokuense、海洋原甲藻(P.micans)。纤细原甲藻(P.gracile)、三角棘原甲藻(P.triestinum)、墨西哥原甲藻(P.mexicanum)、慢原甲藻(P.rhathymum)和P.koreanum等。
采用邻接法,将从GenBank下载同源性靠前的16条序列(表 1)与本研究的序列一起构建系统发育树,结果见图 3。结果表明,BBGA-9003和来自不同海域的9个不同株系的微小原甲藻以及2株心形原甲藻聚在同一大分支上,但是又进化为独立的一小分支。而原甲藻属的其他种,包括东海原甲藻(DQ336054.1)、P.shikokuense(AB781324.1)和具齿原甲藻(AY803742.1)则在另一大分支上。

|
| 图 3 用邻接法基于原甲藻属基因序列构建的系统发育树 Fig.3 Phylogenetic tree based on DNA sequences of Prorocentrum species using adjacency method |
| 种名 Species |
株系 Strains |
来源地 Origins |
GenBank登录号 Accesstion No. |
覆盖率 Query cover (%) |
相似度 Ident (%) |
| P.cordatum | D5 | Chesapeake Bay, USA | MH976700.1 | 100 | 99.39 |
| P.cordatum | PmK | The Adriatic Sea, Italy | MH976699.1 | 100 | 99.39 |
| P.minimum | D-127 | Tongyeong, Korea | JX402086.1 | 100 | 99.39 |
| P.minimum | CCMP 1392 | Long Island Sound, USA | DQ336060.1 | 100 | 99.39 |
| P.minimum | — | — | AY803740.1 | 100 | 99.39 |
| P.minimum | PmiPrMu21 | Geoje, Korea | AY421791.1 | 100 | 99.39 |
| P.minimum | PMDH01 | The East China sea | DQ028763.1 | 100 | 99.39 |
| P.minimum | — | — | AJ415520.1 | 100 | 99.39 |
| P.minimum | PmS1 | Mediterranean Sea, Sète | Y16238.1 | 100 | 99.39 |
| P.shikokuense | — | — | AB781324.1 | 100 | 98.90 |
| P.donghaiense | — | The East China sea | DQ336054.1 | 100 | 98.90 |
| P.dentatum | CCMP 1517 | — | AY803742.1 | 100 | 98.90 |
| P.minimum | CCMP696 | Long Island Sound, USA | FJ587221.1 | 98 | 99.38 |
| P.cordatum | BAL | The Baltic Sea | MH976698.1 | 100 | 98.78 |
| P.donghaiense | - | - | AJ841810.1 | 100 | 98.78 |
| P.donghaiense | MABT-1 | The East China sea | KF032443.1 | 100 | 98.66 |
采用MEGA 7软件建立这17条序列间的遗传距离矩阵结果见表 2。结果表明,BBGA-9003与下载的所有9条微小原甲藻的序列亲缘关系最近,遗传距离均为0.002 8,这些微小原甲藻分别来自韩国统营湾(JX402086.1)及巨济海域(AY421791.1)、美国长岛湾(DQ336060.1和FJ587221.1)、中国东海株(DQ028763.1)以及地中海塞特港(Y16238.1)等海域,意味着北部湾BBDA-9003株极大可能属于微小原甲藻。此外,该藻与美国切萨皮克湾(MH976700.1)和意大利亚得里亚海(MH976699.1)的2株心形原甲藻遗传距离也为0.002 8,而与其他原甲藻属,如东海原甲藻、具齿原甲藻以及来自波罗的海的心形原甲藻(MH976698.1)的遗传距离较大。系统发育树上微小原甲藻以及东海原甲藻种内遗传距离均小于它们与其他种间的遗传距离,说明利用18S rDNA的序列分析也可确定BBGA-9003为微小原甲藻。
| No. | 种名 Species |
登录号 Registration No. |
1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| 1 | P.cordatum | MH976700.1 | |||||||||||||||||
| 2 | P.cordatum | MH976699.1 | 0.000 0 | ||||||||||||||||
| 3 | P.dentatum | AY803742.1 | 0.005 6 | 0.005 6 | |||||||||||||||
| 4 | P.donghaiense | KF032443.1 | 0.007 0 | 0.007 0 | 0.001 4 | ||||||||||||||
| 5 | P.donghaiense | AJ841810.1 | 0.007 0 | 0.007 0 | 0.001 4 | 0.002 8 | |||||||||||||
| 6 | P.donghaiense | DQ336054.1 | 0.005 6 | 0.005 6 | 0.000 0 | 0.001 4 | 0.001 4 | ||||||||||||
| 7 | P.minimum | AJ415520.1 | 0.000 0 | 0.000 0 | 0.005 6 | 0.007 0 | 0.007 0 | 0.005 6 | |||||||||||
| 8 | P.minimum | Y16238.1 | 0.000 0 | 0.000 0 | 0.005 6 | 0.007 0 | 0.007 0 | 0.005 6 | 0.000 0 | ||||||||||
| 9 | P.minimum | AY803740.1 | 0.000 0 | 0.000 0 | 0.005 6 | 0.007 0 | 0.007 0 | 0.005 6 | 0.000 0 | 0.000 0 | |||||||||
| 10 | P.minimum | AY421791.1 | 0.000 0 | 0.000 0 | 0.005 6 | 0.007 0 | 0.007 0 | 0.005 6 | 0.000 0 | 0.000 0 | 0.000 0 | ||||||||
| 11 | P.minimum | FJ587221.1 | 0.000 0 | 0.000 0 | 0.005 6 | 0.007 0 | 0.007 0 | 0.005 6 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | |||||||
| 12 | P.minimum | DQ336060.1 | 0.000 0 | 0.000 0 | 0.005 6 | 0.007 0 | 0.007 0 | 0.005 6 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | ||||||
| 13 | P.minimum | JX402086.1 | 0.000 0 | 0.000 0 | 0.005 6 | 0.007 0 | 0.007 0 | 0.005 6 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | |||||
| 14 | P.minimum | DQ028763.1 | 0.000 0 | 0.000 0 | 0.005 6 | 0.007 0 | 0.007 0 | 0.005 6 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | 0.000 0 | ||||
| 15 | P.shikokuense | AB781324.1 | 0.005 6 | 0.005 6 | 0.000 0 | 0.001 4 | 0.001 4 | 0.000 0 | 0.005 6 | 0.005 6 | 0.005 6 | 0.005 6 | 0.005 6 | 0.005 6 | 0.005 6 | 0.005 6 | |||
| 16 | BBGA-9003 | — | 0.002 8 | 0.002 8 | 0.008 4 | 0.009 8 | 0.009 8 | 0.008 4 | 0.002 8 | 0.002 8 | 0.002 8 | 0.002 8 | 0.002 8 | 0.002 8 | 0.002 8 | 0.002 8 | 0.008 4 | ||
| 17 | P.cordatum | MH976698.1 | 0.007 0 | 0.007 0 | 0.001 4 | 0.002 8 | 0.000 0 | 0.001 4 | 0.007 0 | 0.007 0 | 0.007 0 | 0.007 0 | 0.007 0 | 0.007 0 | 0.007 0 | 0.007 0 | 0.001 4 | 0.009 8 | |
综上,形态学观察及18S rDNA序列分析结果均表明,广西钦州北部湾对虾养殖区藻华水样中分离获得的藻种BBGA-9003为微小原甲藻。
3 讨论原甲藻属种类繁多且广泛分布于全球各海域,是有害藻华发生频率较高的藻类之一。目前,在我国能引起藻华的原甲藻主要有东海原甲藻、海洋原甲藻、微小原甲藻、尖叶原甲藻、利玛原甲藻以及反曲原甲藻等[10-11]。其中,东海原甲藻自2000年开始,常年在长江口及临近海域暴发大规模的藻华,成为我国近海有害藻华的第一优势种[12]。由于形态及生理上的相似,原甲藻与卵甲藻属(Exuviaella)被合并到一个属[13]。后来随着分子生物学的发展,人们发现了两者之间的区别,才再次恢复卵甲藻属的地位[14]。然而,由于原甲藻具有形态多变的特点,其种类鉴定存在较大的困难。近年来,分子生物学技术被成功应用于物种的精确便捷鉴定。例如,我国大亚湾的慢原甲藻曾被认为是墨西哥原甲藻,最终通过18S rDNA序列分析鉴定才被确定为慢原甲藻[15];而海洋原甲藻与纤细原甲藻之间也极易混淆,龙超等[11]通过18S rDNA序列分析克服了北部湾海洋原甲藻种类鉴定中的困难。此外,微小原甲藻也曾被认为是微型卵甲藻(E.minima)、三角原甲藻(P.triangulatum)、E.mariaelebouriae、P.cordiformis及P.mariae-lebouriae的同种异名[16]。
对有害藻华的原因种进行准确的分类鉴定是开展相关防治工作的基础。随着分子生物学的发展,获取藻类的基因片段,利用NCBI的BLAST分析功能可以从遗传的角度去寻找物种的进化发生史,已成为越来越普遍的手段[17]。本研究中,虽然光学显微镜下观测到的BBGA-9003藻种的形态学特征符合原甲藻属的典型外观描述,但是仍不能准确鉴定其种类,需要电子显微镜观察才能确定其种类。然而使用电子显微镜鉴定海洋海洋微藻种类时,其样品处理不仅繁琐耗时,还需要较高的专业水平。通过分子手段分析,系统发育树和遗传距离矩阵均证实了BBGA-9003与微小原甲藻和心形原甲藻具有最近的亲缘关系,且这些株系来自世界各海域。1999年以前,微小原甲藻和心形原甲藻作为2种不同的种类存在,它们之间的唯一区别是心形原甲藻没有顶刺。直到在电子显微镜下发现了心形原甲藻顶刺的存在,两者才被证实是同种异名[18]。综上所述,18S rDNA能准确快速鉴定BBGA-9003藻种属于微小原甲藻。
微小原甲藻是一种能够产生腹泻性贝毒的藻类[19],该藻藻华的发生对水产养殖业具有巨大的威胁。微小原甲藻曾在我国渤海湾和胶州湾引发赤潮,导致鱼虾和贝类的大量死亡,造成严重经济损失[10]。海水养殖是广西北部湾经济的重要支撑,其健康可持续发展对于对广西经济社会发展起着极其重要的作用,因此加强养殖区有害藻华的监测刻不容缓,而有害藻华原因种的鉴定是其监测与防治工作的基础和关键。目前有记录的、在广西北部湾海域曾引起藻华的种类主要有微囊藻、夜光藻、中肋骨条藻、束毛藻以及球形棕囊藻[5]。2018年5月在广西钦州的北部湾对虾养殖区的微小原甲藻藻华为首次记录。微小原甲藻藻华的出现意味着北部湾藻华种类多样性的增加。未来研究中,我们将利用鱼类细胞系和斑马鱼模式生物,分别在细胞和组织水平继续深入研究微小原甲藻对鱼类细胞的毒性作用和致毒致死机制,以期为微小原甲藻的高效防控技术提供数据支持、奠定理论基础[20-24]。
4 结论本研究首先通过光学显微镜及电子显微镜,初步确定引起北部湾养殖区养殖区藻华的原因种为微小原甲藻,接着通过18S rDNA序列分析进一步确认该种为微小原甲藻。本研究不仅进一步证实了18S rDNA序列分析方法在海洋微藻种类鉴定中的可靠性,而且为广西北部湾养殖区有害藻华的赤潮预警与防治提供基础性数据。
| [1] |
李鹏飞, 余庆, 覃仙玲, 等. 广西北部湾海水养殖业现状与病害防控技术体系研究展望[J]. 广西科学, 2018, 25(1): 15-25. |
| [2] |
张少峰, 张春华, 邢素坤. 广西海洋经济发展现状与对策分析[J]. 海洋开发与管理, 2015, 32(4): 103-106. DOI:10.3969/j.issn.1005-9857.2015.04.025 |
| [3] |
李坚明. "十三五"广西水产养殖业发展战略研究[J]. 中国渔业经济, 2016, 34(4): 25-31. DOI:10.3969/j.issn.1009-590X.2016.04.004 |
| [4] |
罗春业, 廖愚. 广西大力发展"九化渔业"[J]. 中国水产, 2015(1): 20-22. DOI:10.3969/j.issn.1002-6681.2015.01.006 |
| [5] |
彭在清, 李天深, 蓝文陆. 广西海域赤潮研究[M]. 北京: 海洋出版社, 2017.
|
| [6] |
罗金福, 李天深, 蓝文陆. 北部湾海域赤潮演变趋势及防控思路[J]. 环境保护, 2016, 44(20): 40-42. |
| [7] |
于仁成, 刘东艳. 我国近海藻华灾害现状、演变趋势与应对策略[J]. 中国科学院院刊, 2016, 31(10): 1167-1174. |
| [8] |
BRUCE K L, LETERME S C, ELLIS A V, et al. Approaches for the detection of harmful algal blooms using oligonucleotide interactions[J]. Analytical and Bioanalytical Chemistry, 2015, 407(1): 95-116. |
| [9] |
曲凌云, 吕颂辉, 高春蕾, 等. 棕囊藻渤海株核糖体18S rDNA和ITS基因结构序列分析[J]. 海洋科学进展, 2008, 26(2): 200-206. DOI:10.3969/j.issn.1671-6647.2008.02.010 |
| [10] |
韩笑天, 郑立, 俞志明, 等. 赤潮生物原甲藻Prorocentrum分子识别和系统发育学研究[J]. 高技术通讯, 2006, 16(8): 864-869. DOI:10.3321/j.issn:1002-0470.2006.08.017 |
| [11] |
龙超, 陈波, 何碧娟, 等. 北部湾海洋原甲藻的形态特征及其系统进化分析[J]. 热带亚热带植物学报, 2013, 21(4): 332-338. DOI:10.3969/j.issn.1005-3395.2013.04.006 |
| [12] |
ZHANG C S, WANG J T, ZHU D D, et al. The preliminary analysis of nutrients in harmful algal blooms in the East China Sea in the spring and summer of 2005[J]. Acta Oceanologica Sinica, 2008, 30(2): 153-159. |
| [13] |
DODGE J D. The Prorocentrales (Dinophyceae).Ⅱ.revision of the taxonomy within the genus Prorocentrum[J]. Botanical Journal of the Linnean Society, 2008, 71(2): 103-125. |
| [14] |
McLACHLAN J L, BOALCH G T, JAHN R. Reinstatement of the genus Exuviaella (Dinophyceae, Prorocentrophycidae) and an assessment of Prorocentrum lima[J]. Phycologia, 1997, 36(1): 38-46. DOI:10.2216/i0031-8884-36-1-38.1 |
| [15] |
梁计林, 何伟宏, 龙超, 等. 慢原甲藻——南海热带附生甲藻新记录[J]. 热带亚热带植物学报, 2011, 19(1): 40-44. DOI:10.3969/j.issn.1005-3395.2011.01.005 |
| [16] |
LIN S J, ZHANG H, JIAO N Z. Potential utility of mitochondrial cytochrome b and its mRNA editing in resolving closely related dinoflagellates:a case study of Prorocentrum (Dinophyceae)[J]. Journal of Phycology, 2006, 42(3): 646-654. DOI:10.1111/j.1529-8817.2006.00229.x |
| [17] |
覃仙玲, 赖俊翔, 陈波, 等. 棕囊藻北部湾株的18S rDNA分子鉴定[J]. 热带亚热带植物学报, 2016, 24(2): 176-181. |
| [18] |
VELIKOVA V N, LARSEN J. The Prorocentrum cordatum/Prorocentrum minimum taxonomic problem[J]. Grana, 1999, 38(2/3): 108-112. |
| [19] |
GRZEBYK D, DENARDOU A, BERLAND B, et al. Evidence of a new toxin in the red tide dinoflagellate Prorocentrum minimum[J]. Journal of Plankton Research, 1997, 19(8): 1111-1124. DOI:10.1093/plankt/19.8.1111 |
| [20] |
LI P, ZHOU L, NI S, et al. Establishment and characterization of a novel cell line from the brain of golden pompano (Trachinotus ovatus)[J]. In Vitro Cellular & Developmental Biology-Animal, 2016, 52(4): 410-418. |
| [21] |
余庆, 李菲, 王一兵, 等. 广西北部湾大宗海水养殖鱼类卵形鲳鲹感染溶藻弧菌及其致病性的研究[J]. 广西科学, 2018, 25(1): 68-73. |
| [22] |
余庆, 李菲, 覃仙玲, 等. 广西卵形鲳鲹小脑来源细胞系的建立及特征分析[J]. 广西科学, 2018, 25(1): 74-79. |
| [23] |
LI P, ZHOU L, WEI S, et al. Establishment and characterization of a cell line from the head kidney of golden pompano (Trachinotus ovatus), and its application in toxicology and virus susceptibility[J]. Journal of Fish Biology, 2017, 90(5): 1944-1959. DOI:10.1111/jfb.13277 |
| [24] |
ZHOU L, LI P, LIU J, et al. Establishment and characterization of a mid-kidney cell line derived from golden pompano Trachinotus ovatus, a new cell model for virus pathogenesis and toxicology studies[J]. In Vitro Cellular & Developmental Biology - Animal, 2017, 53(4): 320-327. |



