2. 广西大学, 广西南宁 530000;
3. 广西科学院, 广西南宁 530007
2. Guangxi University, Nanning, Guangxi, 530000, China;
3. Guangxi Academy of Sciences, Nanning, Guangxi, 530007, China
【研究意义】作为一种应用越来越广泛的核酸定量技术, 荧光定量PCR可在PCR扩增的同时加入荧光染料或基团, 通过对荧光信号的实时监测对PCR扩增产物进行实时准确定量。荧光定量PCR分为绝对定量和相对定量, 绝对定量多用于对单个样品进行准确定量[1], 相对定量因比较简单、准确且高效, 应用相对更广泛, 但必须用表达相对持续、稳定的内参基因来校准目的基因的相对表达量, 实现数据的均一化处理。【前人研究进展】管家基因是最先也是最常作为内参的基因, 但不同物种或同一物种的不同组织、不同发育阶段及病毒感染、污染物胁迫等不同环境下的管家基因表达并不稳定, 必须利用BestKeeper[2]、geNorm[3]、NormFinder[4]、RefFinder[5]等软件或在线分析工具对其进行基因表达稳定性分析, 从而筛选出最合适的内参基因。【本研究切入点】海洋污染产生后, 早在生物体产生严重损害前, 生物标志物尤其是基因表达变化就早已产生[6], 该现象进一步开发利用后可用于海洋污染监测, 且将成为环境毒理学研究重点。红树蚬(Polymesoda erosa)属双壳纲、帘蛤目、蚬科, 主要栖息于河口咸、淡水滩涂, 尤其潮间带红树林中分布较多[7-9], 最近几年研究发现红树蚬具较好的海洋污染监测指示物种的潜力[10-12]。鳃作为海洋生物呼吸器官, 是新陈代谢最旺盛器官; 肝胰腺是动物体内重要的解毒器官, 在降低或消除有毒化学物质危害方面发挥重要而复杂作用[13]。环境污染物在海洋生物体内各组织器官内的累积具选择性, 而鳃和肝胰往往是各海洋生物有毒物质积累的主要位点[14-17], 因此本研究选择鳃及肝胰腺作为靶组织。【拟解决的关键问题】通过研究18S rRNA、β-actin、GAPDH、α-tubulin这4个管家基因在六溴环十二烷(HBCD)胁迫后红树蚬鳃及肝胰腺组织内的表达情况, 筛选出相对稳定的内参基因, 为研究红树蚬在环境污染条件下各基因的表达提供方法及依据。
1 材料与方法 1.1 实验动物的驯化及胁迫红树蚬采自广西北海市草头村潮间带红树林下滩涂, 选择大小、重量相近的个体用于实验。胁迫实验前26℃实验室条件下先驯养两周, 所用海水取自广西海洋研究所养殖基地, 为脱脂棉二次过滤的自然海水, 用水调节盐度至15‰, 充分曝气后使用。每天按0.5 L/只的量彻底换水, 并按30 mg/只的量投喂螺旋藻干粉。
胁迫前随机将红树蚬分成3组, 彻底换水并停投喂。第二天两个胁迫组加入六溴环十二烷(HBCD)(0.05%二甲基亚砜DMSO助溶)至终浓度为0.86 μg/L、8.6 μg/L, 空白组加入等体积0.05%的DMSO[18-19]。胁迫处理24 h后每个处理组取4个雌性个体的鳃或肝胰腺混合作为一个样品, 每组取3个样品, 用液氮速冻后于-80℃冻存备用。荧光定量过程中每个样品又设定3个技术性重复。
所用药品和试剂有Sigma公司的HBCD、DMSO, 及宝生物工程(大连)有限公司的RNAiso Plus提取试剂、EASY Dilution试剂、cDNA逆转录试剂盒、荧光定量PCR试剂盒等。
1.2 总RNA的提取和cDNA的合成取100 g的组织样品用液氮研磨至粉末状, 加入2 000 μL RNA iso plus, 匀浆后室温静置, 然后将上清液转移到新EP管中, 氯仿抽提后异, 丙醇沉淀, 75%酒精清洗, 用RNase free water溶解RNA。最后用蛋白核酸分析仪测定其OD260/280, 并用琼脂凝胶电泳的方法检测RNA的完整性。
取1~2 μg的总RNA, 加入gDNA Eraser后42℃反应2 min去除基因组DNA, 然后加入PrimeScript RT Enzyme Mix I、RT Primer Mix、5×PrimeScript Buffer、RNase Free Water先37℃反应15 min, 然后85℃反应5 s反转录合成cDNA。
1.3 荧光定量引物的设计及qRT-PCR扩增从红树蚬转录组文库中经功能注释后挑选出其GAPDH、β-actin、α-tubulin、18S rRNA序列, 并用Primer 5软件设计其引物序列(表 1), 于上海英潍捷基公司合成。
| 表 1 qRT-PCR引物序列及产物大小信息 Table 1 The information of primers sequences and products lengths for qRT-PCR |
荧光定量PCR扩增使用大连宝生物公司的SYBR® Premix Ex TaqTM Ⅱ(Tli RNaseH Plus)试剂盒(RR820A), 冰上配制反应体系:SYBR® Premix Ex Taq Ⅱ(2×)12.5 μL, 正反向引物(至终浓度0.4 mmol/L)各1 μL, 稀释后的cDNA模板2 μL, 加超纯水至总体系25 μL。qRT-PCR反应条件如下:95℃预变性30 s, 95℃变性5 s, 60℃退火30 s, 40个循环。扩增在美国Bio-Rad公司的Minioption型荧光定量PCR仪上进行。
1.4 数据处理与分析根据管家基因18S rRNA、β-actin、GAPDH、α-tubulin在红树蚬鳃及肝胰腺中的Ct值, 用BestKeeper、geNorm、NormFinder软件对各个管家基因进行表达稳定性分析, 且在使用geNorm、NormFinder前需将原始Ct值转化为标准化量值Q(具体方法见下文描述), 从而筛选出实验条件下最合适的内参基因。
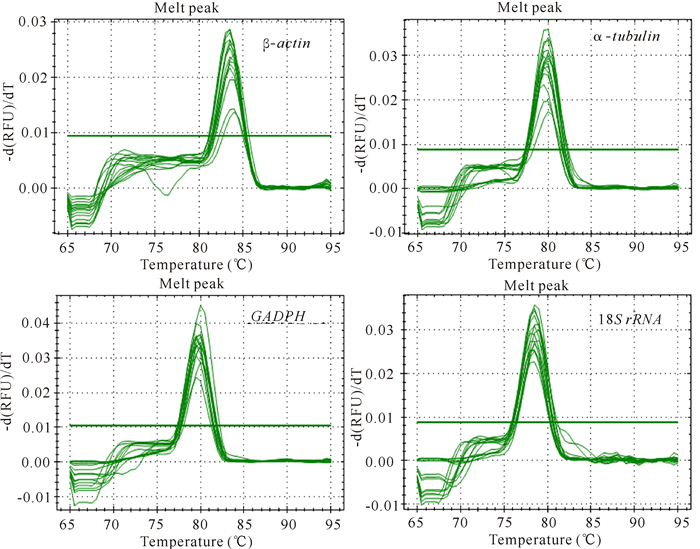
2 结果与分析 2.1 qRT-PCR扩增对各候选内参梯度稀释后的标准品作出标准曲线, 从而得出各引物的扩增效率及相关系数(表 2)。结果显示, 4个候选内参基因的扩增效率为94.4%~105.2%, 相关系数范围为0.990~0.996, 符合荧光定量PCR的基本扩增要求。各引物熔解曲线(图 1)中各内参均只有单一信号峰, 且出峰温度和各内参引物的Tm值温度相符, 说明荧光定量PCR扩增产物的特异性较好, 并无引物二聚体或非特异性扩增。
| 表 2 红树蚬各内参基因定量扩增效率 Table 2 Amplification efficiency of reference genes for Polymesoda erosa |
|
图 1 红树蚬各内参基因qRT-PCR扩增的熔解曲线 Fig.1 The melting curve of qRT-PCR of reference genes for Polymesoda erosa |
BestKeeper是Pfaffl等[2]于2003年首次提出的一种基于Excel的宏运算。BestKeeper软件以各基因在各样品中Ct值为输入初始数据, 并需根据各基因的标准曲线提供扩增效率E(2.0左右数值), 根据所得各基因的标准差(std dev)、变异系数(CV)及相关系数(r)来确定出合适的内参基因。标准差(std dev)及变异系数(CV)越高, 表示基因在样品间的重复性越差, 基因在不同样品之间的稳定性越低, 而相关系数(r)越大, 表示此基因与其他基因的相关性越大, 越适合协同作为内参, 并由此分析各候选内参基因的表达稳定性。故最佳内参及最佳内参组合一般为标准差(std dev)及变异系数(CV)较低, 相关系数(r)较高的候选内参基因及组合。
软件分析结果(表 3)表明, 在鳃及肝胰腺中α-tubulin的稳定性略高于β-actin, 其稳定性排序为α-tubulin>β-actin > GAPDH>18S rRNA, β-actin、α-tubulin适合单独或协同作为内参。
| 表 3 BestKeeper分析不同浓度HBCD处理下红树蚬内参基因稳定性 Table 3 Gene expression stability of reference genes of Polymesoda erosa under different concentrations of HBCD as calculated by BestKeeper |
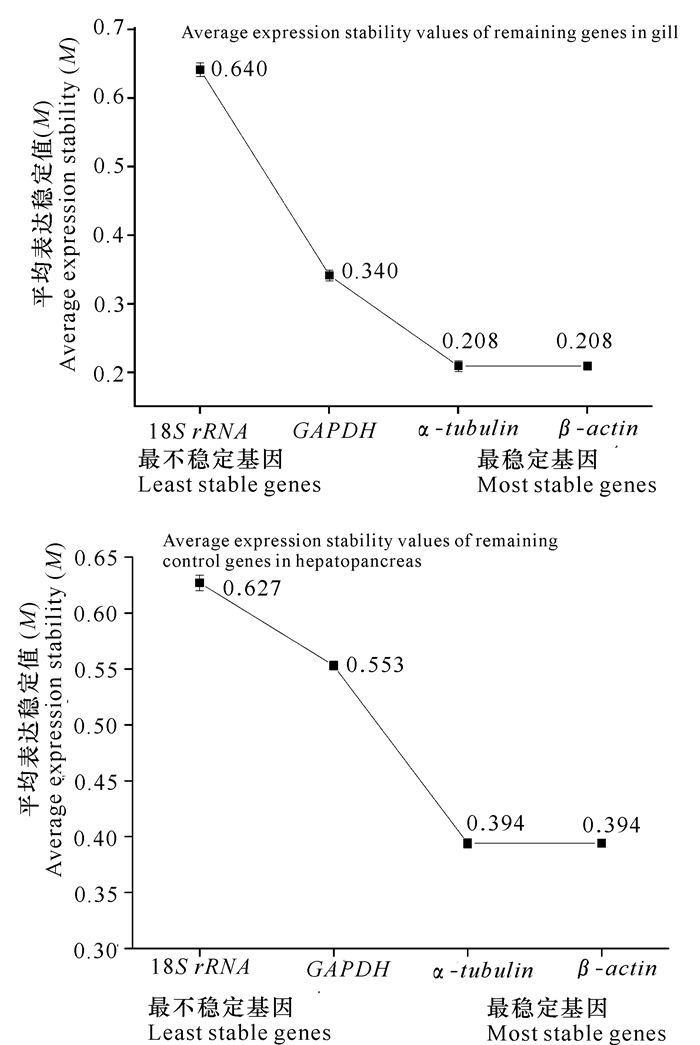
geNorm是Vandesompele等[3]于2002年开发的一款基于excel的用于筛选内参稳定性的软件。geNorm会筛选出2个以上的候选基因作为内参, 以便用于目的基因表达定量时结果更准确和可靠[20], 故geNorm在用于研究目的基因异常细微的差异表达时尤其重要[21]。与BestKeeper以各基因在各样品中的Ct值为初始数据不同, geNorm是将Ct值转化为标准化量值Q, 并以此作为初始数据。具体转化方法:Q=E△Cq, 其中E为基因扩增效率由标准曲线得到, △Cq=Cqmin-Cqsample(Cqmin为所有样品中最低的Cq值, 故△Cq为负数)。并以此Q值作为geNorm软件的初始输入数据, 而非原始的Ct值。geNorm以各基因在各样品间的表达稳定度平均值M来分析各基因的表达稳定性。M值越大, 表示该基因在样品间的稳定性越差。
软件分析所得表达稳定值及排序如图 2所示。由此可知, 鳃、肝胰腺中各候选内参基因稳定性排序均为β-actin=α-tubulin>GAPDH>18S rRNA, M值越小越适合做内参, M值最高的为最差内参基因。故不同浓度HBCD处理下红树蚬鳃及肝胰腺中表达最稳定的一对基因均为β-actin和α-tubulin。
|
图 2 geNorm分析不同浓度HBCD处理下红树蚬内参基因稳定值(M) Fig.2 Gene expression stability values(M) of reference genes of Polymesoda erosa under different concentrations of HBCD as calculated by geNorm |
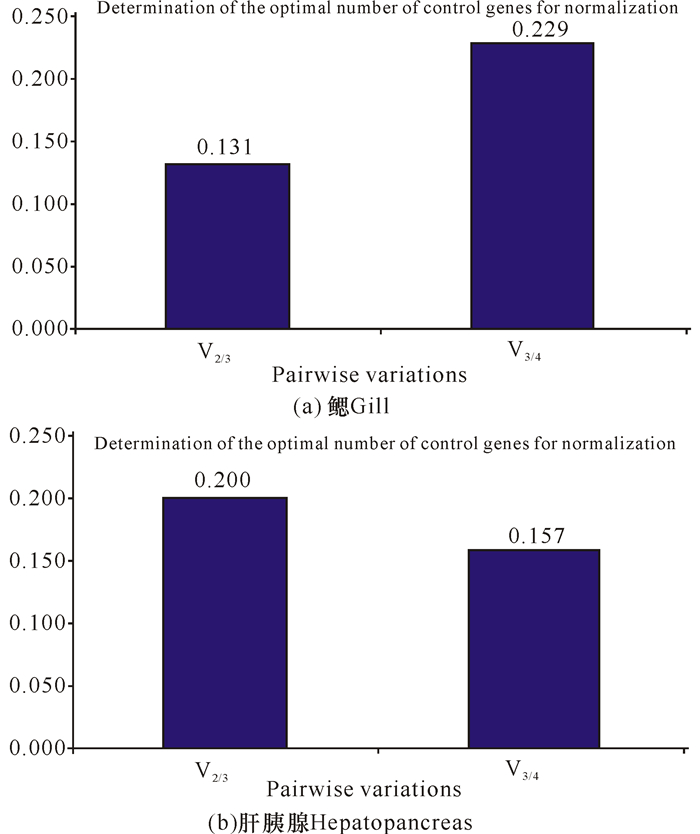
标准化因子的配对差异分析(Vn/n+1)结果如图 3所示。软件默认Vn/n+1值为0.2, 小于0.2的V值所对应的n值为最佳内参个数。由此可知实验的最佳内参基因的个数为2, 故不同浓度HBCD处理下鳃及肝胰腺中表达最稳定的两个基因β-actin/α-tubulin适合单独或协同作为内参。
|
图 3 geNorm对不同浓度HBCD处理下红树蚬内参基因的配对差异分析 Fig.3 Pairwise variations V of reference genes of Polymesoda erosa under different concentrations of HBCD as calculated by geNorm |
NormFinder是由Andersen等[4]于2004年首次提出的, 是一款基于Excel的用于比较不同基因在各样品中表达稳定性的软件。与BestKeeper以各基因在各样品中的Ct值为初始数据不同, NormFinder则是利用标准曲线将Ct值转化为标准化量值Q, 并以此作为初始数据。具体转化方法为:将各基因在各样品中的Ct值作为y, 代入各基因标准曲线的线性表达式y=kx+b中, 以计算出此Ct值下各基因在各样品中的分子拷贝数对数x(X=(y-b)/k), 即所谓的标准化量值Q。并以此Q值作为NormFinder软件的初始输入数据, 而非原始的Ct值。NormFinder以各基因在各样品间的随机稳定性值(Arbitrary stability value)来判断各基因在各样品间的表达稳定性, 并据此筛选出最佳内参基因。随机稳定值(Stability value)越低, 表示该基因在样品间的稳定性越好。故随机稳定值越小越适合做内参, 最佳内参及最佳内参组合一般为随机稳定值最小的候选内参基因[22]。
软件分析结果如表 4所示:在鳃及肝胰腺中β-actin的稳定性略高于α-tubulin, 其稳定性排序为β-actin>α-tubulin>GAPDH>18S rRNA, 所以最佳内参为β-actin和α-tubulin基因。
| 表 4 NormFinder分析不同浓度HBCD处理下红树蚬内参基因稳定性 Table 4 Gene expression stability of the candidate reference genes of Polymesoda erosa under different concentrations of HBCD as calculated by NormFinder |
另外内参分析软件除了BestKeeper、geNorm、NormFinder以外, 比较常用的还有RefFinder, 这是Xie等[5]在geNorm、NormFinder、BestKeeper等软件以及Delta Ct法基础上, 整合成的一个在线分析工具, 可不用数据转化而直接利用Ct值进行内参分析, 可作为上述4种分析方法的综合, 避免了单个分析方法的片面性。
综上所述, BestKeeper、geNorm和NormFinder软件分析结果基本一致, 即在HBCD胁迫下β-actin与α-tubulin的稳定性相当排第一位, 适合单独或协同作为内参。
3 讨论荧光定量PCR中所有数据必须经均一化处理后才可信并具实际意义, 因为RNA在提取和纯化过程中, 即使RNA的样品大小及质量完全相同, 也很难保证RNA的产量及质量完全相同, 而且因为反转录合成cDNA的效率存在很大差异, 为了真正反应目的基因表达的微小差异, 必须用内参基因进行校准和数据均一化[23-24]。一个合格的内参基因必须满足以下条件:(1)表达水平不受实验因素或外界环境条件的影响; (2)在生物有机体及组织发育的各生理阶段表达水平差异极小。符合所有条件的只有管家基因—管家基因的定义就是在细胞生存过程中必须稳定、持续表达的基因, 也是最先作为内参基因的基因[25]。
β-actin、GAPDH、18S rRNA、α-tubulin作为常见的几种管家基因, 表达水平相对较恒定、持续, 被较多用于相对荧光定量中目的基因数据的均一化, 即内部控制或内部参照。但最近几年, 越来越多的研究发现, 某些管家基因的表达也并不稳定, 直接拿来做内参基因不科学, 比如β-actin在很多研究中常被用做双壳贝类荧光定量内参基因, 但在三角帆蚌(Hyriopsis cumingii)3个生物矿化过程中[26]、虾夷扇贝(Patinopecten yessoensis)成年各组织或胚胎/幼体阶段[27]、紫贻贝(Mytilus edulis)配子发生的不同阶段[28]内参基因表达分析中作为管家基因的β-actin稳定性却最差。
国内外越来越多研究均发现, 双壳贝类不同物种间或同一物种不同组织、发育阶段及环境处理条件下适合用做基因表达分析的内参基因各不相同。Morga等[29]用geNorm及NormFinder比较了欧洲扁牡蛎(Ostrea edulis)受病原体BO侵染后血细胞中5个候选内参稳定性进行分析, 并确认GAPDH、EF1-α组合为最佳内参; Bai等[26]用BestKeeper、geNorm及NormFinder比较了三角帆蚌季节性壳生长、壳愈合、珍珠囊发育3个生物矿化过程中7个管家基因的稳定性, 并确认Ubi(泛素蛋白)、Rpl18(核糖体蛋白L18)、EF1-α(延伸因子)为最佳内参组合; Feng等[27]对虾夷扇贝中12个候选内参进行分析, 发现成年各组织样中HEL1(RNA解螺旋酶)、UBQ(泛素蛋白)、RpL16(核糖体蛋白L16)组合稳定性最好; 而不用胚胎/幼体阶段, CB(细胞色素b)、CC(细胞色素c)、His3.3(组氨酸)、GAPDH为最佳内参组合; Cubero-Leon等[28]评定了紫贻贝中6个候选内参稳定性, 发现在早期及性成熟等配子发生的不同阶段18S rRNA、EF1α最为稳定, 而在雌激素胁迫下18S rRNA、28S rRNA最为稳定; Mauriz等[30]对不同繁殖阶段的大海扇蛤(Pecten maximus)中7个内参稳定性进行分析, 卵巢中Ndufa7、Rpsa、EF1α较稳定, 睾丸中18S rRNA、Ndufa7、GAPDH较稳定, 适合协同作为内参; Llera-Herrera等[31]用geNorm及NormFinder比较了雌雄同体的小狮爪海扇蛤(Nodipecten subnodosus)的两性腺区域中5个候选内参的稳定性, 并确定18S rRNA稳定性最好; Du等[32]用geNorm对比了发育中注射孢疹病毒OsHV-1和正常生长的太平洋牡蛎(Crassostrea gigas)Pacific oyster幼体间14个候选内参基因稳定性, 并确定RL7(核糖体蛋白L7)、RS18(核糖体蛋白S18)稳定性最好; 王琦等[33]研究发现β-actin在不同组织和胚胎发育不同阶段表达最稳定, 18S rRNA在性腺发育不同时期表达最稳定; 刘颖等[34]研究发现5个候选内参中在栉孔扇贝(Chlamys farreri)不同发育阶段和雌激素暴露下EF-1α的表达均最为稳定; 鲍相渤等[35]研究发现虾夷扇贝在饥饿胁迫下外套膜、肝胰脏、鳃、肾、血淋巴、闭壳肌等各组织中最佳内参基因各不相同, 致病菌感染前后及水环境升温前后不同时间段血样中最佳内参分别为GAPDH、α-tubulin组合及GAPDH、cytb组合。
4 结论综上所述, 红树蚬经不同浓度HBCD胁迫后, 经BestKeeper、geNorm、NormFinder等软件进行候选内参基因稳定性分析结果基本一致, 即β-actin与α-tubulin的稳定性最好, 可单独或协同作为内参用于荧光定量PCR。从而为污染物胁迫下红树蚬目的基因mRNA定量表达分析提供数据均一化依据, 并为进一步开展分子毒理研究、开发分子生物标志物及进行海洋污染预警奠定了基础。
| [1] |
李丽, 赵成萍, 李宏, 等. 质粒制备绝对定量PCR标准曲线方法的建立[J]. 农业生物技术学报, 2011, 19(6): 1157-1162. LI L, ZHAO C P, LI H, et al. Establishment of the plasmid standard curve generation method for absolute quantification PCR[J]. Journal of Agricultural Biotechnology, 2011, 19(6): 1157-1162. DOI:10.3969/j.issn.1674-7968.2011.06.024 |
| [2] |
PFAFFL M W. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Research, 2001, 29(9): e45. DOI:10.1093/nar/29.9.e45 |
| [3] |
VANDESOMPELE J, DE P K, PATTYN F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): 1-12. |
| [4] |
ANDERSEN C L, JENSEN J L, ORNTOFT T F. Normalization of real-time quantitative reverse transcription-PCR data:A model- based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64(15): 5245-5250. |
| [5] |
XIE F L, SUN G L, STILLER J W, et al. Genome-wide functional analysis of the cotton transcriptome by creating an integrated EST database[J]. PLoS ONE, 2011, 6(11): e26980. |
| [6] |
BODIN N, BURGEOT T, STANISIERE J Y, et al. Seasonal variations of a battery of biomarkers and physiological indices for the mussel Mytilus galloprovincialis transplanted into the northwest Mediterranean Sea[J]. Comparative Biochemistry and Physiology C-toxicology & Pharmacology, 2004, 138(4): 411-427. |
| [7] |
蔡英亚, 黄翔鹄, 吴洞科. 红树蚬的生态观察[J]. 热带海洋, 1995, 4(1): 94-98. CAI Y Y, HUANG X H, WU D K. Studies on the ecology of Polymesoda Erosa (solander)[J]. Tropic Oceanology, 1995, 4(1): 94-98. |
| [8] |
赖廷和, 何斌源. 广西红树林区大型底栖动物种类多样性研究[J]. 广西科学, 1998, 5(3): 166-172. LAI T H, HE B Y. Studies on the macrobenthos species diversity for Guangxi mangrove areas[J]. Guangxi Sciences, 1998, 5(3): 166-172. |
| [9] |
周浩郎, 张俊杰, 邢永泽, 等. 广西红树蚬的分布特征及影响因素分析[J]. 广西科学, 2014, 21(2): 147-152. ZHOU H L, ZHANG J J, XING Y Z, et al. Characteristics of distribution and the influential factors of mangrove clam, Polymesoda erosa (Solander 1768), in Guangxi[J]. Guangxi Sciences, 2014, 21(2): 147-152. |
| [10] |
DSIKOWITZKY L, NORDHAUS I, JENNERJAHN T C, et al. Anthropogenic organic contaminants in water, sediments and benthic organisms of the mangrove-fringed Segara Anakan Lagoon, Java, Indonesia[J]. Marine Pollution Bulletin, 2011, 62(4): 851-862. DOI:10.1016/j.marpolbul.2011.02.023 |
| [11] |
赖廷和, 何斌源, 范航清, 等. 重金属Cd胁迫对红树蚬的抗氧化酶、消化酶活性和MDA含量的影响[J]. 生态学报, 2011, 31(11): 3044-3053. LAI T H, HE B Y, FAN H Q, et al. Effects of cadmium stress on the activities of antioxidant enzymes, digestive enzymes and the membrane lipid peroxidation of the mangrove mud clam Geloina coaxans (Gmelin)[J]. Acta Ecologica Sinica, 2011, 31(11): 3044-3053. |
| [12] |
武文丽, 杨明柳, 吴斌, 等. 露空对红树蚬生物标志物的影响[J]. 广西科学, 2014, 21(2): 153-157, 163. WU W L, YANG M L, WU B, et al. The effect of air exposure on biomarkers of mangrove mud clam (Polymesoda erosa)[J]. Guangxi Sciences, 2014, 21(2): 153-157, 163. |
| [13] |
GIBSON R, BARKER P. The decapod hepatopancreas[J]. Oceanogr Mar Biol Ann Rev, 1979, 17(17): 285-346. |
| [14] |
MCLEESE D W, METCALFE C D. Toxicity of creosote to larval and adult lobsters and Crangon and its accumulation in lobster hepatopancreas[J]. Bulletin of Environmental Contamination and Toxicology, 1979, 22(6): 796-799. |
| [15] |
卢彤岩, 杨雨辉, 徐连伟, 等. 达氟沙星对施氏鲟的急性毒性及组织残留检测[J]. 中国水产科学, 2004, 11(6): 542-547. LU T Y, YANG Y H, XU L W, et al. Acute toxicity of danofloxacin in Amur sturgeon and the body residue[J]. Journal of Fishery Sciences of China, 2004, 11(6): 542-547. DOI:10.3321/j.issn:1005-8737.2004.06.010 |
| [16] |
吕晓华, 古燕, 宋艳. 壬基酚对红鲫、草鱼和鲢鱼的毒性及组织蓄积研究[J]. 卫生研究, 2012, 41(5): 785-789. LV X H, GU Y, SONG Y. Toxicity and tissue accumulation of nonylphenol in Carassius auratus red variety, Grass Carp and Sliver Carp[J]. Journal of Hygiene Research, 2012, 41(5): 785-789. |
| [17] |
许星鸿, 阎斌伦, 徐加涛, 等. 重金属镉胁迫对日本蟳抗氧化酶活力、丙二醛含量及组织蓄积的影响[J]. 水产科学, 2014, 33(9): 551-555. XU X H, YAN B L, XU J T, et al. Effects of cadmium stress on activities of antioxidant enzymes, malonaldehyde content and cadmium accumulation in asian swimming crab Charybdis japonica[J]. Fisheries Science, 2014, 33(9): 551-555. DOI:10.3969/j.issn.1003-1111.2014.09.003 |
| [18] |
ZHANG H, PAN L Q, TAO Y X, et al. Identification and expression of differentially expressed genes in clam Venerupis philippinarum in response to environmental pollutant hexabromocyclododecane (HBCD)[J]. Journal of Experimental Marine Biology and Ecology, 2013, 445(3): 166-173. |
| [19] |
ZHANG H, PAN L Q, TAO Y X. Antioxidant re- sponses in clam Venerupis philippinarum exposed to environmental pollutant hexabromocyclododecane[J]. Environmental Science and Pollution Research, 2014, 21(13): 8206-8215. DOI:10.1007/s11356-014-2801-3 |
| [20] |
ETSCHMANN B, WILCKEN B, STOEVESAND K, et al. Selection of reference genes for quantitative real-time PCR analysis in canine mammary tumors using the geNorm algorithm[J]. Veterinary Pathology, 2006, 43(6): 934-942. DOI:10.1354/vp.43-6-934 |
| [21] |
张艳君, 朱志峰, 陆融, 等. 基因表达转录分析中内参基因的选择[J]. 生物化学与生物物理进展, 2007, 3(5): 546-550. ZHANG Y J, ZHU Z F, LU R, et al. Selection of control genes in transcription analysis of gene expression[J]. Progress in Biochemistry and Biophysics, 2007, 3(5): 546-550. DOI:10.3321/j.issn:1000-3282.2007.05.015 |
| [22] |
马飞.极大节旋藻实时荧光定量PCR内参基因的选择[D].青岛: 中国海洋大学, 2012. MA F.Reference genes for Arthrospira maxima when using quantitative real-time PCR[D].Qingdao: Ocean University of China, 2012. |
| [23] |
BUSTIN S A, BENES V, GARSON J A, et al. The MIQE guidelines:Minimum information for publication of quantitative real-time PCR experiments[J]. Clinical Chemistry, 2009, 55(4): 611-622. |
| [24] |
MALLONA I, LISCHEWSKI S, WEISS J, et al. Validation of reference genes for quantitative real-time PCR during leaf and flower development in Petunia hybrida[J]. BMC Plant Biology, 2010, 10: 4. DOI:10.1186/1471-2229-10-4 |
| [25] |
车志群, 莫祖英, 孙仁杰, 等. 荧光定量PCR方法及分子标志物在海洋污染监测中的应用进展[J]. 生态科学, 2015, 34(5): 233-240. CHE Z Q, MO Z Y, SUN R J, et al. Review on application of methods of realtime fluorescent quantitative PCR and molecular marker in marine pollution monitoring[J]. Ecological Science, 2015, 34(5): 233-240. |
| [26] |
BAI Z Y, LIN J Y, MA K Y, et al. Identification of housekeeping genes suitable for gene expression analysis in the pearl mussel, Hyriopsis cumingii, during biomineralization[J]. Molecular Genetics and Genomics, 2014, 289(4): 717-725. DOI:10.1007/s00438-014-0837-1 |
| [27] |
FENG L Y, YU Q, LI X, et al. Identification of reference genes for qRT-PCR analysis in Yesso Scallop patinopecten yessoensis[J]. PLoS ONE, 2013, 8(9): e75609. |
| [28] |
CUBERO-LEON E, CIOCAN C M, MINIER C, et al. Reference gene selection for qPCR in mussel, Mytilus edulis, during gametoge nesis and exogenous estrogen exposure[J]. Environmental Science and Pollution Research, 2012, 19(7): 2728-2733. |
| [29] |
MORGA B, ARZUL I, FAURY N, et al. Identification of genes from flat oyster Ostrea edulis as suitable housekeeping genes for quantitative real time PCR[J]. Fish & Shellfish Immunology, 2010, 29(6): 937-945. |
| [30] |
MAURIZ O, MANEIRO V, PEREZ-PARALLE M, et al. Selection of reference genes for quantitative RT-PCR studies on the gonad of the bivalve mollusc Pecten maximus L[J]. Aquaculture, 2012, 370-371(2): 158-165. |
| [31] |
LLERA-HERRERA R, GARCIA-GASCA A, HUVET A, et al. Identification of a tubulin-α gene specifically expressed in testis and adductor muscle during stable reference gene selection in the hermaphrodite gonad of the lion's paw scallop Nodipecten subnodosus[J]. Marine Genomics, 2012, 6: 33-44. |
| [32] |
DU Y S, ZHANG L L, XU F, et al. Validation of housekeeping genes as internal controls for studying gene expression during Pacific oyster (Crassostrea gigas) development by quantitative real-time PCR[J]. Fish & Shellfish Immunology, 2013, 34(3): 939-945. |
| [33] |
王琦, 何毛贤. 合浦珠母贝实时定量PCR内参基因的稳定性比较[J]. 南方水产科学, 2013, 9(6): 33-40. WANG Q, HE M X. Stability comparison of reference genes of Pinctada fucata by real-time qPCR[J]. South China Fisheries Science, 2013, 9(6): 33-40. DOI:10.3969/j.issn.2095-0780.2013.06.006 |
| [34] |
刘颖, 王双耀, 安立会, 等. 栉孔扇贝内参基因稳定性研究[J]. 生态毒理学报, 2013, 8(4): 616-622. LIU Y, WANG S Y, AN L H, et al. Study on the expression stability of reference genes in Chlamys farreri[J]. Asian Journal of Ecotoxicology, 2013, 8(4): 616-622. |
| [35] |
鲍相渤, 刘卫东, 姜冰, 等. 内参基因在虾夷扇贝定量PCR中表达稳定性的比较[J]. 水产科学, 2011, 30(10): 603-608. BAO X B, LIU W D, JIANG B, et al. Expression stability of reference genes for quantitative PCR in Japanese Scallop Patinopecten yessoensis[J]. Fisheries Science, 2011, 30(10): 603-608. DOI:10.3969/j.issn.1003-1111.2011.10.003 |
 2018, Vol. 34
2018, Vol. 34 


