2. 广西大学亚热带农业生物资源保护与利用国家重点实验室,广西南宁 530004;
3. 广西大学信息网络中心,广西南宁 530004;
4. 钦州学院电子与信息工程学院,广西钦州 535000
2. State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources, Guangxi University, Nanning, Guangxi, 530004, China;
3. Information Network Center, Guangxi University, Nanning, Guangxi, 530004, China;
4. School of Electronics and Information Engineering, Qinzhou University, Qinzhou, Guangxi, 535000, China
【研究意义】下一代测序 (Next Generation Sequencing,NGS)[1]是第二代DNA测序技术,其主要平台有Illumina、Polonator、SOLiD测序仪等。这些平台都使用循环芯片的测序方法,对充满DNA样本的芯片进行重复的DNA聚合酶反应和荧光序列读取反应。与传统的测序方法相比,NGS具有高通量、成本低、效率高的优点,但得到的序列通常包含大量的错误,需要利用计算机对序列进行辅助修正,其修正过程是先检测出序列中错误的碱基,然后通过特定的算法对其进行修正。计算机纠错处理的核心是设计一个好的纠错算法,以提高序列修正的性能和质量。因此,本研究对现有的纠错工具进行比较,给相关研究提供参考依据;设计与实现并行分布式纠错算法,解决大规模基因数据分析处理中计算机内存消耗大和运算时间长的问题。【前人研究进展】目前的错误修正算法主要基于以下3种方法实现[2-3]:通过进行K-mer[4]统计的基于K-spectrum方法、基于后缀树的方法和基于MSA[5]的方法。其中,基于K-spectrum方法的工具主要有Sga[6]、Racer[7]、Musket[8]和Lighter[9]等,基于后缀树方法的工具主要有SHREC[10]、HiTec[11]等,基于MSA方法的工具主要有ECHO[12]、Coral[13]等。
基于K-spectrum的错误修正算法简单易懂,修正效果较好,且易于在各种平台上实现。其中心思想是首先把每个短序列划分为长度为k的K-mers,然后将所有的K-mers进行排列并计数。当一个K-mer出现的次数达到一定的阈值N时,则认为该K-mer是solid的,否则认为该K-mer是非solid的,即该序列含有错误碱基,接着用solid序列去修正含有错误碱基的序列。基于K-spectrum方法的4种主要纠错工具各具特点:Sga是2012年提出的一个基因组装工具,同时使用FM-index和BWT算法对序列进行压缩存储。其自带Map功能,淘汰了不能匹配的数据,所以修正性能较好。Racer是2013年提出的一个纠错工具,因为Racer把序列存放到hash表中,所以程序对数据处理占用内存较小且运行速度较快。Musket是一个专为Illumina测序产生的序列进行纠错的工具,主要使用双端纠错、单端推进和选举法进行纠错。Lighter通过采样算法获得一系列尽可能与参考基因相似的K-mers,然后从中找出属于solid的K-mers存放到Bloom filter。Lighter通过对K-mers的采样处理加快了程序对K-mers的统计操作,进而缩短程序的运行时间。【本研究切入点】由于目前的纠错软件主要是在单机上运行的,如要处理大规模的基因数据,则要求用高性能计算机来实现。Hadoop[14]的HDFS系统可以快速地访问存储在各计算节点的数据,非常适用于开发处理海量数据集的应用程序,且HDFS系统具有极高的容错性,用户可以将Hadoop部署在多台性能一般的计算机集群上组成分布式系统,所以在Hadoop上实现分布式并行纠错算法有利于解决数据的存储和运算的优化问题,且在生物计算领域,越来越多的问题已通过Hadoop平台得到解决,如基因表达双聚类和重叠社区算法等。【拟解决的关键问题】对现有基于K-spectrum的纠错工具进行比较,找出性能较好的纠错工具,然后在借鉴前人的纠错算法的基础上,在Hadoop平台上实现分布式并行纠错算法,降低NGS错误修正算法对计算机内存的消耗, 提高其计算性能。
1 基于K-spectrum方法的常用纠错工具性能分析 1.1 数据来源本研究采用的实验数据Staphylococcus aureus (SA)、Rhodobacter sphaeroides (RS)、Human Chromosome 14(HC14)、Bombus impatiens (BI) 来自GAGE网站 (http://gage.cbcb.umd.edu/data/index.html),对应的参考基因组数据来自NCBI,实验数据的大小依次为242 MB、436 MB、9.6 GB、92 GB。
1.2 数据预处理在进行实验前,首先需要删除含A、T、C、G以外的其它任何字母的基因序列。本实验使用的基因数据都是双链的,每一个基因数据都有两个Fastq文件,一个对应序列的左链,一个对应序列的右链。在处理一个文件的增删时,必须要同时对另一个文件进行对应的增删操作,以保证序列是paired-end的。接着便可使用纠错工具对序列进行纠错处理,并用BWA工具把序列映射到参考基因组上,得到每个序列在参考基因组上的起始位置。
1.3 Gain综合分析通过对原序列、纠错工具生成的序列和参考基因组中对应的碱基进行比对,对修正后原序列中错误碱基的各种修改状态分别进行统计,其修改状态包括修改正确、修改错误、没有进行修改。并利用统计结果计算出衡量纠错工具性能的信息增益Gain。Gain数据的计算公式如下:
| $ {\rm{Gain = }}\frac{{{\rm{TP}} - {\rm{FP}}}}{{{\rm{TP}} + {\rm{FN}}}} $ | (1) |
其中:TP表示纠错工具改对的碱基个数, FP表示纠错工具改错的碱基个数, FN表示纠错工具没有对错误碱基进行修改的碱基个数。
1.4 性能分析结果Gain综合分析结果如图 1所示,Lighter对小规模数据的错误修正效果较好,但在处理大规模数据时效果较差。从总体上看,无论是在处理小规模数据还是大规模数据上,Sga和Racer都表现出了较好的修正效果。
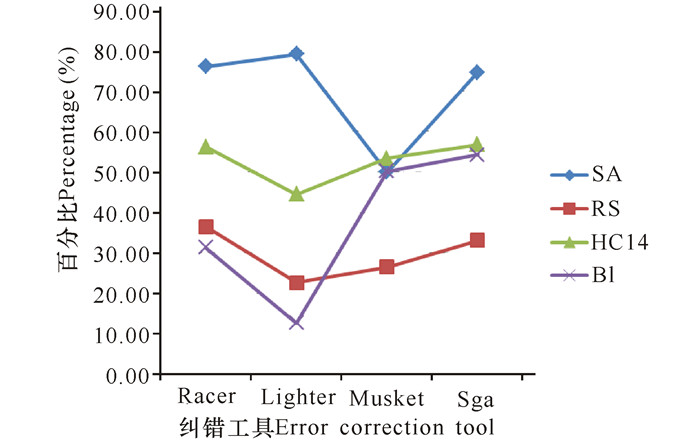
|
图 1 SA、RS、HC14、BI的Gain信息 Fig.1 Gain of SA, RS, HC14 and BI |
图 2和图 3分别是Lighter、Racer、Sga和Musket处理SA、RS、HC14、BI数据时,计算机的内存峰值和运行时间比较。由图 2和图 3分析可知,对于相同的数据,Lighter对内存和运行时间的消耗较少。说明Lighter采用的采样操作算法对提高程序的运行时间和内存的性能是有效的。Sga在处理每一个数据时,消耗的时间总是远远大于其他纠错工具。但从实验结果上看,大多数的纠错工具都存在内存占用大的问题。为了解决该问题,可以利用分布式计算平台把普通的计算机有效地联合起来,对大规模数据进行分布式并行处理。
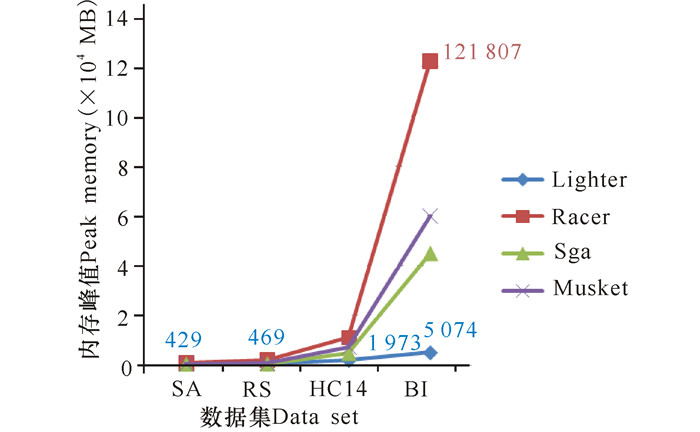
|
图 2 4种算法的内存峰值比较 Fig.2 Peak memory comparison of 4 algorithms |

|
图 3 4种算法的运行时间比较 Fig.3 Running time comparison of 4 algorithms |
本研究的Hadoop平台搭建在广西大学亚热带农业生物资源保护与利用国家重点实验室生物信息学团队的Openstack[15]平台上。其中,Openstack平台搭建在5台曙光物理节点上,每个节点的主要配置为6核处理器,32 GB内存和300 GB硬盘。
程序的设计思想:把预处理后的文件只取基因序列交给Hadoop的任务机制,Hadoop通过判断任务的数据量大小来决定该任务需要分配多少个节点进行处理。然后将每个节点的数据交给Map函数,Map函数并行地处理该文件,并将序列数据以 < key, value>的形式输出,value中存放序列数据,key不赋值。文件读取完毕后,Map函数会调用Correct函数进行纠错处理,其过程为程序按设定的K-mer长度,遍历所有的序列,分析每一步生成的K-mer是否在Bloom Filter中,如果存在则说明此K-mer是正确的,否则说明遍历到的碱基是错误的,把错误的碱基用A、T、C、G进行遍历替换。定义一个记录得分的变量score,如果替换碱基后的K-mer在Bloom Filter中,则score加1。找到一个得分值达到所检查K-mers数量的一半以上,且分值最大的碱基进行替换,否则不进行修改。把数据放到Hadoop平台上进行纠错的伪代码如下:
protected void map (LongWritable key, Text value, Context context) {
String line=value.toString (); //从HDFS上读取一行数据
String fixed=corrector.correct (line); //修正序列
text.set (fixed);
context.write (NullWritable.get (), text); //写入HDFS
}
public String correct (String seq) {
……
for (int i=k-1; i < sequence.length; i++) { //默认不修正首段
if (!lastIsSolid (i)) { //判断序列是否在已构建的Bloom Filter中
for (int i=0; i < DNA.length; i++) { //DNA[]中存放ATCG四个元素
……
for (j=curIndex-k + 1; j < =curIndex; j++) { //定义K的长度为17
for (p=0; p < k; p++) { //截取curIndex-k+1~curIndex间的kmer
if (p + j >=sequence.length) break;
kmer[p]=sequence[p + j];
}
if (p < k) break; //不能组成完整的kmer
…….
kmer[pos--]=DNA[i]; //在kmer的pos位置替换DNA[i]
if (isExist (kmer)) score++; //如kmer在已构建的Bloom Filter中得分+1
}
}
//如所检查的kmers数量一半以上是soild的则进行碱基替换
sequence[i]=score >=checkCount / 2? DNA[rIndex] :sequence[curIndex];
}
}
return new String (this.sequence); //返回修改后的序列
}
2.2算法验证
为了验证基于Hadoop平台的分布式并行纠错算法 (Parallel algorithm) 的可行性,本研究利用SA、RS和HC14数据对程序进行测试,并与程序的串行单机运行方式 (Serial algorithm)、Lighter和Racer做比较。其中本研究提出的算法的单机方式运行在广西大学计算机与电子信息学院的惠普服务器上,其配置为32核处理器,64 GB内存和1 TB硬盘。实验得到的运行时间比较情况如图 4所示,内存的比较情况如图 5所示。

|
图 4 4种算法的内存峰值比较 Fig.4 Peak memory comparison of 4 algorithms |
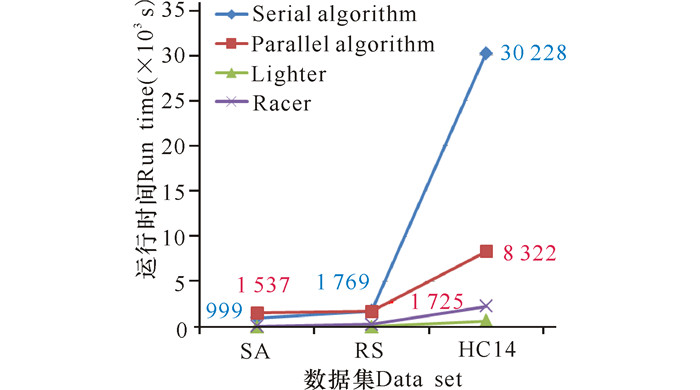
|
图 5 4种算法的运行时间比较 Fig.5 Running time comparison of 4 algorithms |
通过对不同程序在处理相同数据时的运行时间和内存峰值进行比较,发现基于Hadoop平台的分布式并行纠错程序在内存的消耗方面相比其它程序少,且随实验数据规模的增大,其内存的变化非常平稳。由于程序设计得还不够完善,所以在运行时间上分布式算法的时间要比Lighter和Racer算法的运行时间长。与算法的单机运行方式比较时,发现当数据集小于RS时,单机运行方式较快,其主要原因是分布式并行算法在进行MapReduce运算时消耗的时间较多。但当数据集大于RS时,分布式并行算法运行的时间比单机串行算法的运行时间减少了72.5%。从整体上来看,Hadoop分布式并行纠错算法能更有效地解决现有基于K-spectrum的NGS错误修正算法对大规模基因数据处理时,在内存和运行时间上的瓶颈问题。
3 结论基于K-spectrum的NGS错误修正算法直观易懂、错误修正效果较好,但在处理规模较大的数据时消耗大量内存。现有的基于K-spectrum的常用纠错工具中,Sga和Racer的纠错效果较好,Lighter的纠错效果较差,但Lighter的采样算法使数据存储空间大大减少,降低了对计算机内存的需求。本研究基于Hadoop的HDFS系统与MapReduce编程模式,提出了Hadoop分布式并行纠错算法,并与其串行程序、Racer和Lighter进行比较,发现该算法降低了对计算机单机的内存消耗,并在一定程度上提高了纠错算法的运算性能,说明该算法是可行的。
| [1] |
SHANKS M E, DOWNES S M, COPLEY R R, et al. Next-generation sequencing (NGS) as a diagnostic tool for retinal degeneration reveals a much higher detection rate in early-onset disease[J]. European Journal of Human Genetics, 2013, 21(3): 274-280. DOI:10.1038/ejhg.2012.172 |
| [2] |
YANG X, CHOCKALINGAM S P, ALURU S. A survey of error-correction methods for next-generation sequencing[J]. Briefings in Bioinformatics, 2013, 14(1): 56-66. DOI:10.1093/bib/bbs015 |
| [3] |
江育娥, 黄伟, 林劼. 下一代测序纠错方法综述[J]. 北京工业大学学报, 2016, 42(3): 377-386. JIANG Y E, HUANG W, LIN J. Error correction in preprocessing of next-generation sequencing[J]. Journal of Beijing University of Technology, 2016, 42(3): 377-386. |
| [4] |
CHOR B, HORN D, GOLDMAN N, et al. Genomic DNA k-mer spectra:Models and modalities[J]. Genome Biology, 2009, 10: R108. DOI:10.1186/gb-2009-10-10-r108 |
| [5] |
DIERINGER D, SCHLÖ TTERERER C. Microsatel-lite analyser (MSA):A platform independent analysis tool for large microsatellite data sets[J]. Molecular Ecology Notes, 2003, 3(1): 167-169. DOI:10.1046/j.1471-8286.2003.00351.x |
| [6] |
GONNELLA G, KURTZ S. Readjoiner:A fast and memory efficient string graph-based sequence assembler[J]. Bmc Bioinformatics, 2012, 13: 82. DOI:10.1186/1471-2105-13-82 |
| [7] |
ILIE L, MOLNAR M. RACER:Rapid and accurate correction of errors in reads[J]. Bioinformatics, 2013, 29(19): 2490-2493. DOI:10.1093/bioinformatics/btt407 |
| [8] |
LIU Y C, SCHRÖDER J, SCHMIDT B. Musket:A multistage k-mer spectrum-based error corrector for illumina sequence data[J]. Bioinformatics, 2013, 29(3): 308-315. DOI:10.1093/bioinformatics/bts690 |
| [9] |
LI S, FLOREA L, LANGMEAD B. Lighter:Fast and memory-efficient sequencing error correction without counting[J]. Genome Biology, 2014, 15(1): R1. DOI:10.1186/gb-2014-15-1-r1 |
| [10] |
SCHRÖDER J, SCHRÖDER H, PUGLISI S J, et al. SHREC:A short-read error correction method[J]. Bioinformatics, 2009, 25(17): 2157-2163. DOI:10.1093/bioinformatics/btp379 |
| [11] |
ILIE L, FAZAYELI F, ILIE S. HiTEC:Accurate error correction in high-throughput sequencing data[J]. Bioinformatics, 2011, 27(3): 295-302. DOI:10.1093/bioinformatics/btq653 |
| [12] |
KAO W C, CHAN A H, SONG Y S. ECHO:A reference-free short-read error correction algorithm[J]. Genome Research, 2011, 21(7): 1181-1192. DOI:10.1101/gr.111351.110 |
| [13] |
SALMELA L, SCHRÖDER J. Correcting errors in short reads by multiple alignments[J]. Bioinformatics, 2011, 27(11): 1455-1461. DOI:10.1093/bioinformatics/btr170 |
| [14] |
WHITE T, CUTTING D. Hadoop:The definitive guide[J]. O'reilly Media Inc Gravenstein Highway North, 2010, 215(11): 1-4. |
| [15] |
CORRADI A, FANELLI M, FOSCHINI L. VM consolidation:A real case based on open stack cloud[J]. Future Generation Computer Systems, 2014, 32: 118-127. DOI:10.1016/j.future.2012.05.012 |
 2017, Vol. 33
2017, Vol. 33 


