2. 上海辰山植物园华东野生濒危资源植物保育中心, 上海 201602
2. Conservation Centre for Wild Endangered Plant of East China, Shanghai Chenshan Botanical Garden, Shanghai, 201602, China
福建省地处我国的东南沿海地区,地理位置较为独特,横跨南亚热带和中亚热带,热量丰富,雨量充沛,蕨类植物资源较为丰富[1]。1991年版《福建植物志:第1卷》(修订本)记载蕨类植物有45科374种(含变种)[2]。近些年来,对于福建蕨类资源的后续调查陆续有一些新记录科如水韭科(Isoetaceae)[3]、新记录属如长片蕨属(Abrodictyum)[4]和新记录种如假镰羽短肠蕨(Allantodia petrii)[5]被发现,但燕尾蕨属(Cheiropleuria)在福建尚无分布记录。燕尾蕨属传统上被认为是一个孤立的类群置于燕尾蕨科(Cheiropleuriaceae)[6],近年来,随着蕨类植物分子系统学的深入研究,发现燕尾蕨属与双扇蕨科(Dipteridaceae)具有亲缘关系而将其置于双扇蕨科[7-8]。中国产燕尾蕨属植物有两种,分别是产于华南的全缘燕尾蕨(Cheiropleuria integrifolia)和仅产于海南的燕尾蕨(C.bicuspis)[9]。2023年1月,作者在福建木兰溪源省级自然保护区境内发现一种蕨类植物——木兰溪源蕨类标本1号,从其形态上初步鉴定为全缘燕尾蕨。
在经典分类学研究中,专业人员通过植物形态特征比对和实践经验进行植物鉴定分类,但大部分植物形态性状受环境影响巨大,具有极大的不稳定性。尤其是蕨类植物,因其没有形态结构复杂的生殖器官,仅可通过羽片、茎、根等孢子体器官及其附属物进行分类研究。DNA条形码(DNA barcoding) 是一段特定的DNA序列,作为一种快速、准确的分子鉴定技术,在多数植物的物种鉴定中展现出独特优势[10-11]。最早的蕨类分子系统学研究是Stein[12]利用叶绿体限制性内切酶对蕨类植物进行研究,而Hasebe等[13]最早利用DNA测序方法对蕨类植物进行研究。目前分子生物学技术在被子植物分类系统中已被广泛应用,与之相比,蕨类植物领域分子系统学研究的深度和广度仍相对不足。
蕨类分子系统学研究所用的基因片段涉及植物的3个基因组(核基因组、叶绿体基因组和线粒体基因组)。不同的基因组因其结构和功能上的差异,进化速率有所不同。总的来说,核基因组进化最快,进化速率约为叶绿体基因组的2倍;线粒体基因组进化最慢,进化速率约是叶绿体基因组的1/3。目前在蕨类分子系统学研究中应用的基因片段主要来自叶绿体基因组[14]。在大多数被子植物中,叶绿体基因组表现为经典的四分体结构,该结构包括一个大单拷贝区(LSC区)、一个小单拷贝区(SSC区)及两个反向重复区(IRa和IRb区),约100个基因(包括蛋白质编码基因、rRNA基因和tRNA基因),大小为115-165 kb[15]。叶绿体基因组的长度变异主要是由两个反向重复区引起的,与核基因组相比,叶绿体基因组具有碱基数少、拷贝数多、结构简单等特点[16]。叶绿体基因组具有相对独立的进化历史,两个反向重复区作为一个共同的遗传单位进行进化,这对解释植物的进化历史非常有用,在揭示物种起源、生物进化及物种之间亲缘关系等方面具有重要作用[17]。此外,叶绿体基因组在进化过程中很少发生基因重排事件[18],其受核基因遗传重组的影响较小,主要是母系遗传,少数是父系遗传,这对解释植物复杂的系统发育十分重要[19]。叶绿体基因组中的一些片段基因,可作为分子标记,在研究系统发育方面具有较大意义[20]。其中rbcL基因位于叶绿体基因组的LSC区,长度约为1 400 bp,编码1,5-二磷酸核酮糖羧化酶/氧化酶大亚基,是目前蕨类植物分子系统学研究中应用最广泛且最重要的分子标记[21]。本研究对木兰溪源蕨类标本1号进行叶绿体全基因组提取、测序、拼接、注释和特征分析,并进行形态和分子水平鉴定,以确认木兰溪源蕨类标本1号的具体种属。
1 材料与方法 1.1 材料凭证标本:木兰溪源蕨类标本1号采集于福建木兰溪源省级自然保护区。地理位置:北纬25°38′50″、东经118°35′49″,海拔800 m,采集人:陈炳华、陈彬等,采集时间:2023-01-19,采集号:CBH04635,馆藏号:FNU0041370。
生境:生于常绿阔叶林下,土生,海拔800 m。以鹅掌柴(Heptapleurum heptaphyllum)、栲(Castanopsis fargesii)、黧蒴锥(Castanopsis fissa)、百日青(Podocarpus neriifolius)、杉木(Cunninghamia lanceolata)、小果山龙眼(Helicia cochinchinensis)、薄叶润楠(Machilus leptophylla)为主要的乔木层植物;灌木层植物有草珊瑚(Sarcandra glabra)、冠盖藤(Pileostegia viburnoides)、杜鹃(Rhododendron simsii)、秀丽四照花(Cornus hongkongensis subsp.elegans)、密花树(Myrsine seguinii)、莲座紫金牛(Ardisia primulifolia)等;藤本植物有当归藤(Embelia parviflora);草本层植物有福建细辛(Asarum fukienense)、五节芒(Miscanthus floridulus)、十字薹草(Carex cruciata)、华山姜(Alpinia oblongifolia)等,并有多种蕨类植物伴生,如深绿卷柏(Selaginella doederleinii)、狗脊(Woodwardia japonica)、金毛狗(Cibotium barometz)、光里白(Diplopterygium laevissimum)、乌毛蕨(Blechnopsis orientalis)、镰羽瘤足蕨(Plagiogyria falcata)、瘤足蕨(Plagiogyria adnata)、倒挂铁角蕨(Asplenium normale)、缩羽复叶耳蕨(Arachniodes japonica)等。
1.2 方法 1.2.1 形态学鉴定对木兰溪源蕨类标本1号各器官的构造进行细致观察。测量其株高、叶片长宽及裂片之间的夹角等,在显微镜下观察其异形叶上的孢子形态,根据植物典型特征比对后开展鉴定。
1.2.2 基因组的提取、测序、拼接、注释使用DNeasy Plant Mini Kit试剂盒(QIAGEN GmbH)从木兰溪源蕨类标本1号的新鲜叶片中提取总DNA,使用1%琼脂糖凝胶电泳检测总DNA的质量,将样品送到北京贝瑞和康生物技术有限公司进行测序,使用Illumina NovaSeq 6000平台进行基因组测序。利用GetOrganelle (https://github.com/Kinggerm/GetOrganelle)软件组装完整的叶绿体基因组[22],再通过Bandage (http://rrwick.github.io/Bandage/)对最终装配图进行可视化[23],以评估其完整性,使用CPGAVAS2[24](在线地址:47.96.249.172:16019/analyzer/annotate)和Plastid Genome Annoator[25]进行基因注释,运用BLASTX[26]对蛋白编码序列的注释进行验证,使用tRNAscan-SE 2.0[27]验证tRNA基因的注释,用Geneious Prime v11.0.14.1 (https://www.geneious.com/)手动校正个别基因。最后使用OGDRAW (https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)绘制叶绿体基因组图谱。经拼接和注释后,采用Geneious Prime v11.0.14.1软件分析叶绿体基因组的特征,包括基因组全长、4个区域长度和GC含量、基因类型和数量、基因拷贝情况等。
1.2.3 木兰溪源蕨类标本1号系统位置分析利用美国国家生物技术信息中心(NCBI)库中的罗伞蕨属(Matonia)、显子蕨属(Phanerosorus)、燕尾蕨属、双扇蕨属(Dipteris)、芒萁属(Dicranopteris)的rbcL基因序列构建系统进化树,以芒萁属的芒萁(D.pedata)作为外类群,详见表 1。同时利用Geneious Prime v11.0.14.1软件从木兰溪源蕨类标本1号的叶绿体基因组中提取rbcL基因序列。
| 属 Genes |
种 Species |
GenBank登录号 GenBank accession |
| Matonia | M.foxworthyi | AF012266.1 |
| M.pectinata | AF012267.1 | |
| Phanerosorus | P.major | AF012268.1 |
| P.sarmentosus | AF012269.1 | |
| Cheiropleuria | C.bicuspis | AB042571.1 |
| C.integrifolia | AB042569.1 | |
| Dipteris | D.conjugata | EF588692.1 |
| D.lobbiana | AB042561.1 | |
| Dicranopteris | D.pedata | DQ838070.1 |
利用PhyloSuite[28]软件对序列进行mafft比对。最大似然法(ML)[29]分析的最佳模型由IQ-TREE 2[30]中的ModelFinder[31]确定,并以最优模型在IQ-TREE 2软件构建最大似然系统发育树。贝叶斯法(BI)[32]分析的最优核苷酸替代模型可通过Phylosuite软件确定,并以最优核苷酸替代模型在MrBayes 3.2[32]软件构建贝叶斯系统发育树。分析结果上传至ITOL在线软件(https://itol.embl.de/)完成可视化与美化。
2 结果与分析 2.1 形态特征及生境描述全缘燕尾蕨Cheiropleuria integrifolia (D.C.Eaton ex Hooker) M.Kato Y.Yatabe, Sahashi & N.Murakami, Blumea.46:522.2001 [图 1: (a)、(b)]。

|
| (a) and (b) are the adaxial and abaxial surfaces of the leaves of Cheiropleuria integrifolia (photoed by Xu Yechun); (c)-(f) habitat, plant, leaves, and sporophylls of Fern Specimen No.1 from Mulanxi Source. 图 1 全缘燕尾蕨与木兰溪源蕨类标本1号形态特征比较 Fig. 1 Comparison of morphological characteristics between Cheiropleuria integrifolia and Fern Specimen No.1 from Mulanxi Source |
形态:该种在福建省分布的形态特征与《中国植物志(第六卷第二分册)》[33]中描述有些差异,主要表现为植株普遍更高大,不育叶片卵形、全缘,未见浅裂,仅叶柄就长达40 cm,主脉3-5条,自基部呈掌状向顶部放射状伸展,叶片分叉较少[图 1: (c)-(f)]。
2.2 木兰溪源蕨类标本1号系统位置分析基于rbcL基因序列构建的系统进化树表明,罗伞蕨属(Matonia foxworthyi、M.pectinata)和显子蕨属(Phanerosorus major、P.sarmentosus)聚为一支(BP/PP: 100/1.00),燕尾蕨属(Cheiropleuria bicuspis、C.integrifolia)和双扇蕨属(Dipteris conjugata、D.lobbiana)聚为一支(BP/PP: 100/1.00),燕尾蕨属和双扇蕨属亲缘关系最近,木兰溪源蕨类标本1号rbcL基因序列与全缘燕尾蕨(Cheiropleuria integrifolia)聚为一支(BP/PP: 96/1.00),且均为强支持,再结合形态特征,可判定该物种为全缘燕尾蕨(Cheiropleuria integrifolia)(图 2)。

|
| Data on the branches are the Bootstrap Probabilities (BP) and the Bayesian Posterior Probabilities (PP) respectively. 图 2 基于rbcL基因序列的木兰溪蕨类标本1号的系统进化分析 Fig. 2 Phylogenetic analysis of Fern Specimen No.1 from Mulanxi Source based on rbcL |
2.3 全缘燕尾蕨及同科相关分类群叶绿体基因组基本特征分析
为组装全缘燕尾蕨叶绿体基因组,本研究采用Illumina NovaSeq 6000平台对全缘燕尾蕨进行全基因组测序。原始下机数据共获得3 530.37万条reads (约10.59 Gb碱基),超出预设需求数据量(33.33万条reads,10 Gb)。数据质量评估显示,Q20与Q30分别为98.26%和94.74%,表明98.26%的碱基识别错误率≤1%(Q20),94.74%的碱基识别错误率≤0.1%(Q30),符合后续基因组组装与分析要求。
全缘燕尾蕨叶绿体基因组(GenBank登录号:OR911355)为典型的四分体结构,呈双链环状结构,叶绿体基因组全长为153 682 bp,LSC、SSC、IRa和IRb各区长度分别为79 025、19 669、27 494、27 494 bp。全缘燕尾蕨叶绿体基因组LSC区、SSC区的GC含量分别为45.58%和44.85%,IR区GC含量为46.49%。从整体上分析,IR区的GC含量略高于其他两个区域。从AT、GC偏斜值来看,全缘燕尾蕨基因组双链上碱基组成的不对称性较低(表 2、图 3)。
| 区域 Region |
碱基长度/bp Base length/bp |
T含量/% T content/% |
C含量/% C content/% |
A含量/% A content/% |
G含量/% G content/% |
GC含量/% GC content/% |
AT偏斜值 AT skew |
GC偏斜值 GC skew |
| LSC | 79 025 | 27.67 | 23.44 | 26.75 | 22.14 | 45.58 | -0.016 9 | -0.028 5 |
| SSC | 19 669 | 27.28 | 23.41 | 27.87 | 21.44 | 44.85 | 0.010 8 | -0.043 8 |
| IRa | 27 494 | 25.81 | 22.67 | 27.70 | 23.82 | 46.49 | 0.035 2 | 0.024 7 |
| IRb | 27 494 | 27.70 | 23.82 | 25.81 | 22.67 | 46.49 | -0.035 2 | -0.024 7 |
| Note: ATskew=(A-T)/(A+T); GCskew=(G-C)/(G+C). | ||||||||
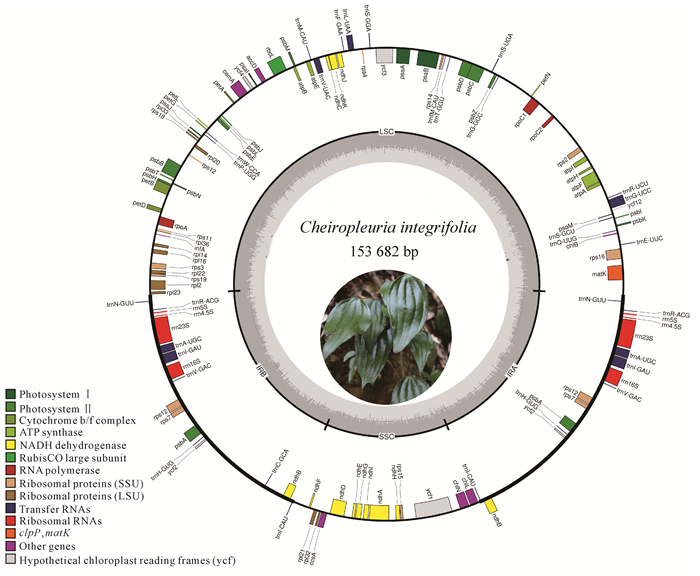
|
| 图 3 全缘燕尾蕨叶绿体基因组图谱 Fig. 3 Chloroplast genome graph of Cheiropleuria integrifolia |
经基因注释分析(表 3),全缘燕尾蕨叶绿体基因组共包含124个基因,包括31个tRNA基因,8个rRNA基因和85个蛋白质编码基因。内含子在基因表达调控中发挥着重要作用[34],全缘燕尾蕨叶绿体基因组中具有内含子的基因数量为15个,基因petB、atpF、ndhA、rpoC1、rpl2、rps16、trnA-UGC、trnG-UCC、trnI-GAU、trnL-UAA、trnV-UAC和ycf1各含有1个内含子,而rps12、matK和ycf3含有2个内含子。根据基因的功能可分为以下4个大类。第1类:48个与光合作用相关的基因,包括5个光合系统Ⅰ基因、15个光合系统Ⅱ基因、12个NADH脱氢酶基因、6个细胞色素b/f复合物基因、6个ATP合酶基因、1个二磷酸核酮糖羧化酶基因和3个叶绿素还原酶基因。第2类:除rRNA基因和tRNA基因外,有26个与自我复制相关的基因,包括10个核糖体蛋白大亚基基因、13个核糖体蛋白小亚基基因和3个RNA聚合酶基因。第3类:5个其他编码蛋白质的基因。第4类:6个功能未知的基因。
| 基因分类 Category |
基因分组 Gene group |
基因名称 Gene name |
| Photosynthesis | Subunits of photosystemⅠ | psaA,psaB,psaI,psaJ,psaM |
| Subunits of photosystemⅡ | psbA(2),psbB,psbC,psbD,psbE,psbH,psbI,psbJ,sbK,psbL,psbM,psbN,psbT,psbZ | |
| Subunits of NADH dehydrogenase | ndhA*,ndhB(2),ndhC,ndhD,ndhE,ndhF,ndhG,ndhH,ndhI,ndhJ,ndhK | |
| Subunits of cytochrome b/f complex | petA,petB*,petD,petG,petL,petN | |
| Subunits of ATP synthase | atpA,atpB,atpE,atpF*,atpH,atpI | |
| Large subunit of rubisco | rbcL | |
| Subunits of photochlorophyllide reductase | chlB,chlL,chlN | |
| Self-replication | Subunits of RNA polymerase | rpoA,rpoC1*,rpoC2 |
| Proteins of large ribosomal subunit | rpl14,rpl16,rpl2*,rpl20,rpl21,rpl22,rpl23,rpl32,rpl33,rpl36 | |
| Proteins of small ribosomal subunit | rps11,rps12**(2),rps14,rps15,rps16*,rps18,rps19,rps2,rps3,rps4,rps7(2) | |
| Transfer RNAs | trnA-UGC*(2),trnC-GCA,trnE-UUC,trnF-GAA,trnG-GCC,trnG-UCC*,trnH-GUG(2),trnI-CAU(2),trnI-GAU*(2),trnL-UAA*,trnM-CAU,trnN-GUU(2),trnP-UGG,trnQ-UUG,trnR-ACG(2),trnR-UCU,trnS-GCU,trnS-GGA,trnS-UGA,trnT-GGU,trnV-GAC(2),trnV-UAC*,trnW-CCA,trnfM-CAU | |
| Ribosomal RNAs | rrn16S(2),rrn23S(2),rrn4.5S(2),rrn5S(2) | |
| Other genes | Maturase | matK** |
| Envelope membrane protein | cemA | |
| C-type cytochrome synthesis gene | ccsA | |
| Acetyl-CoA carboxylase | accD | |
| Translation initiation factor | infA | |
| Genes of unknown function | Conserved hypothetical chloroplast ORF | ycf1*,ycf12,ycf2(2),ycf3**,ycf4 |
| Note: the number in parentheses indicates the copy number of that gene; *indicates one intron inclusive; ** indicates two intron inclusive. | ||
目前双扇蕨科共包含11个物种,截至2024年5月1日,在NCBI数据库中仅公布了燕尾蕨属燕尾蕨、双扇蕨属双扇蕨和喜马拉雅双扇蕨(Dipteris wallichii) 3个物种叶绿体全基因组数据,且其中部分数据未经验证,故笔者仅对其进行简单分析。经分析发现,4种植物的质体长度在123 674 bp (双扇蕨)到161 342 bp (喜马拉雅双扇蕨)之间,对于LCS区和SSC区,这些物种之间的长度变化不明显,GC含量在这些质体中基本上是保守的(表 4)。IR区的结构极大地影响整个基因组的结构完整性。在所有比较的物种中,双扇蕨IR区显著减少,而其LCS区和SSC区又基本不变,考虑是由IR区的部分基因丢失导致的,因此其质体的大小减小。
| 物种 Species |
登录号 Accession No. |
长度/bp Length/bp |
GC含量/% GC content/% |
|||
| 总计Total | LSC | SSC | IR | |||
| Cheiropleuria integrifolia | OR911355 | 153 682 | 79 025 | 19 669 | 27 494 | 45.81 |
| Cheiropleuria bicuspis* | OM419348.1 | 160 237 | 84 438 | 21 115 | 27 348 | 45.83 |
| Dipteris conjugata | KP136829.1 | 123 674 | 81 224 | 21 084 | 10 683 | 45.78 |
| Dipteris wallichii* | OM419347.1 | 161 342 | 84 885 | 21 161 | 27 648 | 44.84 |
| Note: species with * indicate that the whole chloroplast genome data for that species is unverified in the NCBI database. | ||||||
3 讨论 3.1 全缘燕尾蕨的分布及形态特征
全缘燕尾蕨在国内分布于四川(合江)、贵州(荔波)、湖南(宜章莽山)、浙江(平阳、苍南)、广西、广东、海南和台湾;在国外分布于巴布亚新几内亚、印度尼西亚、马来西亚、泰国、越南和日本南部[35]。说明该种的分布存在连续性,尽管全缘燕尾蕨已在我国8个省级行政区有分布报道,但野外分布点极少。如在四川的分布仅基于1977年在合江采集的一份存于中国科学院成都生物研究所标本馆的标本(CDBI0005301!)。浙江则把该种纳入省级重点保护野生植物管理,且截至目前未再发现该种,福建也许是该种在国内的最北分布点。
笔者在福建木兰溪源省级自然保护区境内发现的这种全缘燕尾蕨,与《中国植物志(第六卷第二分册)》[33]中的描述有些差异,主要表现为植株普遍更高大,仅叶柄就长达40 cm,主脉3-5在叶片基部,叶片分叉较少。植物形态可塑性是适应异质环境的一个重要特征[36],可塑性的响应包括两方面:一方面是环境条件对植物生长和生理特性的必然影响,另一方面是为增强生物体在环境中成功生存的适应调节。正是这些适应的可塑性响应使植物能够在一系列多变的环境中维持其功能和适合度,从而影响物种生态幅和对自然选择的响应。因此研究可塑性的响应具有生态学和进化学意义[37-38],这在后续研究中可进行深入了解。
3.2 燕尾蕨属叶绿体基因组的系统发育本研究使用最大似然法以及贝叶斯法构建系统发育树并进行聚类分析。聚类结果表明该物种与燕尾蕨聚为一小支,燕尾蕨属与双扇蕨属聚为一支,再结合其形态特征,可鉴定该物种为全缘燕尾蕨,为福建省蕨类植物科、属和种的新记录。但考虑到燕尾蕨属乃至双扇蕨科内物种的nrITS (non-redundant Internal Transcribed Spacer)、叶绿体全基因组数据不足,无法从其他层面构建系统发育树,随着后续研究的深入,可做进一步验证。该研究结果为双扇蕨科各属植物的起源及亲缘关系的进一步确定提供了相关证据,为后续双扇蕨科分子系统发育研究奠定了基础。
3.3 全缘燕尾蕨及同科相关分类群叶绿体基因组特征植物叶绿体基因大小一般为115-165 kb,包含约100个基因,主要包含光合作用、叶绿体基因组转录和表达的相关基因等[15]。本研究组装的全缘燕尾蕨叶绿体基因组全序列长度是153 682 bp(其中LSC区长度和GC含量分别为79 025 bp、45.85%),其叶绿体基因组共包含124个基因,包括31个tRNA基因、8个rRNA基因和85个蛋白质编码基因,但因为燕尾蕨属乃至双扇蕨科内物种的叶绿体全基因组数据不足,且其中在NCBI上已公布的数据又多为未经验证的,故无法准确对该属植物的叶绿体基因组特征进行对比、总结、概括,相信随着研究的深入,将会弥补这一块的空白。
| [1] |
付厚华, 马良, 韦宏金, 等. 福建省蕨类植物分布新记录(Ⅱ)[J]. 植物资源与环境学报, 2022, 31(6): 93-95. DOI:10.3969/j.issn.1674-7895.2022.06.12 |
| [2] |
福建省科学技术委员会《福建植物志》编写组. 福建植物志: 第一卷[M]. 修订本. 福州: 福建科学技术出版社, 1991.
|
| [3] |
李燕飞, 丁鑫, 耿贺群, 等. 珍稀濒危石松类植物东方水韭的再次发现[J]. 厦门大学学报(自然科学版), 2013, 52(3): 411-413. |
| [4] |
林晟, 梁晖, 杨成梓, 等. 长片蕨属: 福建膜蕨科新记录属[J]. 亚热带植物科学, 2020, 49(3): 203-204. DOI:10.3969/j.issn.1009-7791.2020.03.009 |
| [5] |
温桂梅, 张凤生, 林宇豪, 等. 福建省新记录植物(Ⅶ)[J]. 福建师范大学学报(自然科学版), 2020, 36(3): 45-51. |
| [6] |
秦仁昌. 中国蕨类植物科属系统排列和历史来源[J]. 植物分类学报, 1978, 16(4): 16-37. |
| [7] |
KRAMER K U, GREEN P S, KUBITZKI K. The families and genera of vascular plants.Ⅴ. 1: pteridophytes and gymnosperms[M]. Berlin: Springer, 1990.
|
| [8] |
PPG I. A community-derived classification for extant lycophytes and ferns[J]. Journal of Systematics and Evolution, 2016, 54(6): 563-603. DOI:10.1111/jse.12229 |
| [9] |
严岳鸿, 苑虎, 何祖霞, 等. 中国燕尾蕨属(Cheiropleuria C. Presl)植物修订[J]. 武汉植物学研究, 2010, 28(5): 550-553. |
| [10] |
LI D Z, LIU J Q, CHEN Z D, et al. Plant DNA barcoding in China[J]. Journal of Systematics and Evolution, 2011, 49(3): 165-168. DOI:10.1111/j.1759-6831.2011.00137.x |
| [11] |
LI M, CAO H, BUT P P H, et al. Identification of herbal medicinal materials using DNA barcodes[J]. Journal of Systematics and Evolution, 2011, 49(3): 271-283. DOI:10.1111/j.1759-6831.2011.00132.x |
| [12] |
STEIN D B. Nucleic acid comparisons as a tool in understanding species interrelationships and phylogeny[J]. Proceedings of the Royal Society of Edinburgh Section B Biological Sciences, 1985, 86: 283-288. |
| [13] |
HASEBE M, ITO M, KOFUJI R, et al. Phylogenetic relationships of ferns deduced from rbcL gene sequence[J]. Journal of Molecular Evolution, 1993, 37(5): 476-482. DOI:10.1007/BF00160428 |
| [14] |
WOLFE K H, LI W H, SHARP P M. Rates of nucleotide substitution vary greatly among plant mitochondrial, chloroplast, and nuclear DNAs[J]. Proceedings of the National Academy of Sciences of the United States of America, 1987, 84(24): 9054-9058. |
| [15] |
WANG J, KAN S L, LIAO X Z, et al. Plant organellar genomes: much done, much more to do[J]. Trends in Plant Science, 2024, 29(7): 754-769. DOI:10.1016/j.tplants.2023.12.014 |
| [16] |
RAVI V, KHURANA J P, TYAGI A K, et al. An update on chloroplast genomes[J]. Plant Systematics and Evolution, 2008, 271(1/2): 101-122. |
| [17] |
尹明华, 余锾媛, 肖心怡, 等. 江西铅山红芽芋叶绿体基因组特征及系统发育分析[J]. 生物技术通报, 2023, 39(6): 233-247. |
| [18] |
GASTONY G J, YATSKIEVYCH G. Maternal inheritance of the chloroplast and mitochondrial genomes in cheilanthoid ferns[J]. American Journal of Botany, 1992, 79(6): 716-722. DOI:10.1002/j.1537-2197.1992.tb14613.x |
| [19] |
黄瑶, 李朝銮, 马诚, 等. 叶绿体DNA及其在植物系统学研究中的应用[J]. 植物学通报, 1994, 11(2): 11-25. |
| [20] |
RITLAND K, CLEGG M T. Evolutionary analysis of plant DNA sequences[J]. The American Naturalist, 1987, 130: S74-S100. DOI:10.1086/284693 |
| [21] |
刘红梅, 张宪春, 曾辉. DNA序列在蕨类分子系统学研究中的应用[J]. 植物学报, 2009, 44(2): 143-158. DOI:10.3969/j.issn.1674-3466.2009.02.002 |
| [22] |
JIN J J, YU W B, YANG J B, et al. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes[J]. Genome Biology, 2020, 21: 241. DOI:10.1186/s13059-020-02154-5 |
| [23] |
WICK R R, SCHULTZ M B, ZOBEL J, et al. Bandage: interactive visualization of de novo genome assemblies[J]. Bioinformatics, 2015, 31(20): 3350-3352. DOI:10.1093/bioinformatics/btv383 |
| [24] |
SHI L C, CHEN H M, JIANG M, et al. CPGAVAS2, an integrated plastome sequence annotator and analyzer[J]. Nucleic Acids Research, 2019, 47(W1): W65-W73. DOI:10.1093/nar/gkz345 |
| [25] |
QU X J, MOORE M J, LI D Z, et al. PGA: a software package for rapid, accurate, and flexible batch annotation of plastomes[J]. Plant Methods, 2019, 15: 50. DOI:10.1186/s13007-019-0435-7 |
| [26] |
HUSON D H, XIE C. A poor man's BLASTX: high-throughput metagenomic protein database search using PAUDA[J]. Bioinformatics, 2014, 30(1): 38-39. DOI:10.1093/bioinformatics/btt254 |
| [27] |
CHAN P P, LIN B Y, MAK A J, et al. tRNAscan-SE 2.0:improved detection and functional classification of transfer RNA genes[J]. Nucleic Acids Research, 2021, 49(16): 9077-9096. DOI:10.1093/nar/gkab688 |
| [28] |
ZHANG D, GAO F, JAKOVLIC I, et al. PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Molecular Ecology Resources, 2020, 20(1): 348-355. DOI:10.1111/1755-0998.13096 |
| [29] |
NGUYEN L T, SCHMIDT H A, VON HAESELER A, et al. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Molecular Biology and Evolution, 2015, 32(1): 268-274. DOI:10.1093/molbev/msu300 |
| [30] |
MINH B Q, SCHMIDT H A, CHERNOMOR O, et al. IQ-TREE 2: new models and efficient methods for phylogenetic inference in the genomic era[J]. Molecular Biology and Evolution, 2020, 37(5): 1530-1534. DOI:10.1093/molbev/msaa015 |
| [31] |
KALYAANAMOORTHY S, MINH B Q, WONG T K F, et al. ModelFinder: fast model selection for accurate phylogenetic estimates[J]. Nature Methods, 2017, 14(6): 587-589. DOI:10.1038/nmeth.4285 |
| [32] |
RONQUIST F, TESLENKO M, VAN DER MARK P, et al. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology, 2012, 61(3): 539-542. DOI:10.1093/sysbio/sys029 |
| [33] |
中国科学院中国植物志编辑委员会. 中国植物志: 第六卷第二分册[M]. 北京: 科学出版社, 2000: 5.
|
| [34] |
FU H, KIM S Y, PARK W D. A potato Sus3 sucrose synthase gene contains a context-dependent 3' element and a leader intron with both positive and negative tissue-specific effects[J]. The Plant Cell, 1995, 7(9): 1395-1403. |
| [35] |
喻智勇, 陈文红, 王玉琴, 等. 云南省蕨类植物两个新纪录属: 燕尾蕨属和黑心蕨属[J]. 林业调查规划, 2024, 49(2): 140-144. |
| [36] |
SULTAN S E. Phenotypic plasticity for plant development, function and life history[J]. Trends in Plant Science, 2000, 5(12): 537-542. DOI:10.1016/S1360-1385(00)01797-0 |
| [37] |
VAN KLEUNEN M, FISCHER M. Adaptive evolution of plastic foraging responses in a clonal plant[J]. Ecology, 2001, 82(12): 3309. DOI:10.1890/0012-9658(2001)082[3309:AEOPFR]2.0.CO;2 |
| [38] |
DORN L A, HAMMOND PYLE E, SCHMITT J. Plasticity to light cues and resources in Arabidopsis thaliana: testing for adaptive value and costs[J]. Evolution, 2000, 54(6): 1982. |



