2. 广西师范大学生命科学学院, 广西桂林 541006;
3. 广西雅长兰科植物国家级自然保护区管理中心, 广西百色 533209
2. College of Life Sciences, Guangxi Normal University, Guilin, Guangxi, 541006, China;
3. Yachang Orchid National Nature Reserve Management Center of Guangxi, Baise, Guangxi, 533209, China
兜兰是兰科(Orchidaceae)兜兰属(Paphiopedilum)植物的统称,以其花唇瓣呈兜状闻名,又因酷似旧时欧洲女子拖鞋,亦被称为“拖鞋兰”和“仙履兰”。兜兰属共包括109个物种,其中有27个分布在中国[1]。该属花形奇特,并且在生长习性(地生型、石上附生型和树上附生型)、开花时间(春季开花型、夏季开花型、秋冬季开花型和四季开花型等)和开花类型(单花、双花或多花)等方面呈现丰富的多样性[2]。因此,兜兰是具有高观赏价值和经济价值的花卉植物。然而,由于过度采集和非法贸易,大多数野生兜兰的栖息地人为破坏严重,因此其种群和个体数量都在不断减少,濒临灭绝。
同色兜兰(P.concolor)是兜兰属中罕见的四季开花型物种,曾自然分布于广西、云南和贵州的部分地区[3],但近期的全国专项调查显示仅在广西西南地区一带见其自然分布[4]。带叶兜兰(P.hirsutissimum)是春季开花型兜兰,花期在4-5月,主要分布在广西西部至北部、贵州西南部和云南东南部一带[5]。在2021年版《国家重点保护野生植物名录》中,同色兜兰和带叶兜兰分别被列为国家一级和二级重点保护野生植物。对于濒危植物而言,了解它们的遗传多样性、种群结构、种群遗传分化等信息对物种保育具有重要意义[6-8]。为了有效保护和利用带叶兜兰,先前已经开展了一些保护遗传学方面的研究:高丽霞等[9]基于SRAP标记评估了广西木论国家级自然保护区40份野生带叶兜兰资源的遗传多样性;徐言等[10]基于SSR标记对中国西南地区190份带叶兜兰资源(其中13份来自广西木论国家级自然保护区,39份来自广西雅长兰科植物国家级自然保护区)进行了遗传多样性、种群结构和表型多样性分析。广西是兰科植物的天然分布中心之一,拥有相对丰富的兰科植物资源,然而先前对广西带叶兜兰资源的遗传多样性评估都集中在自然保护区中的就地保护种群,可能难以反映存活在自然保护区之外的带叶兜兰野生种群的遗传多样性。迄今为止,还未有关于同色兜兰遗传多样性和群体结构方面的报道。此外,对于这两种不同开花习性的兜兰属物种的遗传分化程度也缺少研究。
因此,本研究通过实地调查,广泛收集了分布于广西的231份同色兜兰和带叶兜兰样品,并利用SSR标记开展保护遗传学研究,重点探究不同开花习性的同色兜兰和带叶兜兰的遗传多样性水平、种群结构及其遗传分化。
1 材料与方法 1.1 材料在广西采集了同色兜兰5个自然种群的80个样品以及带叶兜兰7个自然种群的151个样品(表 1)。为了探究同色兜兰和带叶兜兰的系统发育关系,还采集了同属的其他3个近缘种,分别为长瓣兜兰(P.dianthum)、麻栗坡兜兰(P.malipoense)和硬叶兜兰(P.micranthum),每个近缘种各采集3个样品。由于部分兜兰属物种具有丛生特点,为了避免采集到同一株植株,所有采集植株之间至少间隔10 m。使用变色硅胶分别采集各植株的新鲜叶片样品。
| 物种 Species |
采集地点 Collection site |
经度 Longitude |
纬度 Latitude |
种群号 Population ID |
采样数 Sampling number |
| P.concolor | Pairu Village, White-headed Langur Nature Reserve | 107°42′E | 22°50′N | TS-PR | 3 |
| Huguo Village, Encheng Town, Daxin County, Chongzuo City | 107°60′E | 22°46′N | TS-HG | 30 | |
| Jiuhan Village, Fuxin Town, Tiandeng County, Chongzuo City |
107°10′E | 22°54′N | TS-JH | 10 | |
| Weiping Village, Yalong Town, Dahua County, Hechi City |
107°52′E | 23°59′N | TS-WP | 15 | |
| Minqiang Village, Shanglong Town, Longzhou County, Chongzuo City | 106°53′E | 22°26′N | TS-MQ | 22 | |
| P.hirsutissimum | Laya Gorge, Yarchang Nature Reserve | 106°19′E | 24°48′N | DY-LX | 15 |
| Orchid Germplasm Gene Bank of Yachang Nature Reserve | 106°25′E | 24°51′N | DY-YC | 23 | |
| Xiakairong, Mingmo Station, Mulun Nature Reserve | 107°58′E | 25°60′N | DY-XKR | 30 | |
| Huangjiang Village, Luofu Town, Nandan County, Hechi City | 107°21′E | 25°50′N | DY-HJ | 20 | |
| Jingxi County, Baise City | 106°30′E | 22°55′N | DY-JX | 10 | |
| Poji Town, Tian′e County, Hechi City | 107°80′E | 25°12′N | DY-PJ | 29 | |
| Tianlin County, Baise City | 105°39′E | 24°31′N | DY-TL | 24 | |
| P.micranthum | Tianlin County, Baise City | 105°39′E | 24°31′N | YY-TL | 3 |
| P.dianthum | Tianlin County, Baise City | 105°39′E | 24°31′N | CB-TL | 3 |
| P.malipoense | Tianlin County, Baise City | 105°39′E | 24°31′N | MLP-TL | 3 |
| Note: several populations were not accurate to the towns and villages. | |||||
1.2 DNA提取和SSR分型
使用改良CTAB(Cetyltrimethylammonium Bromide)法[11]提取了240个样品的DNA,并利用1.2%琼脂糖电泳(120 V,20 min)进行质检。使用NanoDROP 8000超微量分光光度计(赛默飞世尔科技公司)检测DNA的浓度和纯度(A260/A280=1.8-2.0,浓度≥30 ng/μL)。利用10对高多态性SSR引物(表 2)对240个样品进行分型。10对高多态性SSR引物筛选自Xu等[12]在亨利兜兰(P.henryanum)中开发的34对EST-SSR引物,该研究已证明34对引物在7种兜兰属(包括同色兜兰和带叶兜兰)植物中均有较好的扩增效果。PCR扩增体系:2×Taq PCR Master Mix 5.0 μL,DNA 1.0 μL(~20 ng),上、下游引物各0.5 μL(浓度均为10 pmol/μL),ddH2O 3.0 μL。PCR扩增程序:95 ℃预变性5 min;95 ℃变性30 s,62-52 ℃退火30 s,72 ℃延伸30 s,反应循环10次;95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸30 s,反应循环25次;72 ℃末端延伸20 min;4 ℃保温。使用GeneMapper v4.1软件在ABI 3730xl DNA分析仪上读取SSR基因型。
| 位点 Locus |
重复基序 Repeat motif |
上游/下游引物 Upstream/Reverse primer |
产物大小/bp Size range/bp |
观测等位基因数(Na) Number of observed alleles(Na) |
有效等位基因数(Ne) Number of effective alleles(Ne) |
香农信息指数(I) Shannon information index (I) |
观测杂合度(Ho) Observation heterozygosity(Ho) |
期望杂合度(He) Expected heterozygosity(He) |
固定系数(F) Fixed index(F) |
| DL14 | (CTC)6 | TTCCTTCCCTACCCTTTCCA/CAGCGGTGTCGTTGATGTT | 259-267 | 2.4 | 1.3 | 0.453 | 0.268 | 0.248 | -0.062 |
| DL20 | (GCC)6 | GGCCAAGTACATGCACCCAT/TTCCCACCTCGGTTATGCAC | 290-314 | 2.0 | 1.3 | 0.317 | 0.208 | 0.185 | -0.104 |
| DL21 | (CAG)6 | GCAAATCCATTCAGCCCTGC/CGACATGGTCTGAGAGGAGC | 95-219 | 4.5 | 2.6 | 1.093 | 0.629 | 0.571 | -0.096 |
| DL23 | (AGA)6 | CTTGGGACTCTTTCCTCGGC/CCAGGAGGCTCTCAGCTTTC | 235-270 | 1.7 | 1.1 | 0.147 | 0.029 | 0.081 | 0.528 |
| DL30 | (CCG)6 | CAGGTTGACAGCAATGTCGC/GCCGCAGCTTTTCGGATAAG | 202-208 | 1.9 | 1.3 | 0.291 | 0.180 | 0.174 | -0.027 |
| DL32 | (AAAC)5 | AGCGTGTTTGGACTAGAGCA/TCGGGGATGCACATGGAAAA | 249-284 | 2.4 | 1.9 | 0.692 | 0.106 | 0.409 | 0.710 |
| DL34 | (CGG)6 | GGGTGGGGAGAGTAGGAGTT/GCCACAACTTGTTTTCCCGG | 239-251 | 2.4 | 1.5 | 0.452 | 0.234 | 0.256 | 0.153 |
| DL36 | (CGT)6 | CCACGTGTGACAGAATCCCA/GGCTCCCGACGAGGAATTAC | 246-255 | 1.3 | 1.2 | 0.154 | 0.132 | 0.102 | -0.209 |
| DL39 | (ATC)6 | CCACCAGCTTTCATATCCTCCA/GCCCATGCTGTGCAAAAAGA | 223-259 | 2.7 | 1.9 | 0.618 | 0.331 | 0.341 | 0.028 |
| DL40 | (TCT)6 | AAGAAGTGGCTTCCATGGCA/GCAAAACCAAGGTGTCGTCC | 227-242 | 2.5 | 1.8 | 0.603 | 0.403 | 0.359 | -0.110 |
| Mean | 2.4 | 1.6 | 0.482 | 0.252 | 0.273 | 0.081 |
1.3 方法 1.3.1 遗传多样性和瓶颈效应分析
使用GenAlEx v6.5.1[13]计算位点和群体的遗传多样性指标,包括观测等位基因数(Na)、有效等位基因数(Ne)、香农信息指数(I)、观测杂合度(Ho)、期望杂合度(He)和固定系数(F)等。利用R语言对同色兜兰和带叶兜兰间各群体的遗传多样性指标差异进行t检验。利用分子方差分析(AMOVA)评估种群间、个体间和个体内的总遗传变异。采用BOTTLENECK v1.2.02[14]软件对各种群进行瓶颈效应分析。根据Piry等[14]的建议使用更适合SSR标记的两相混合模型(TPM)进行检验,并设置方差(Variance)为12,步移式突变(SMM)占模型检测的比例为95%,以及迭代次数为1 000。由于同色兜兰TS-PR种群的采样数量低于Bottleneck软件的最低使用标准,因此在瓶颈效应检验中排除该种群。
1.3.2 系统发育关系和遗传结构分析为了更准确评估同色兜兰和带叶兜兰间的系统发育关系,在分析中加入了长瓣兜兰、麻栗坡兜兰和硬叶兜兰共9个样品的数据,然后利用MEGA v6.0[15]的非加权平均(UPGMA)法和邻接(NJ)法分别构建群体水平和个体水平的系统发育树。
采用不同分析方法检测群体间的遗传结构。首先,通过GeneAlEx v6.5.1计算个体间Nei氏遗传距离矩阵,随后使用主坐标分析(PCoA)来计算样品主坐标成分,并通过主坐标成分来说明个体间遗传聚类。使用STRUCTURE[16]对全部个体进行贝叶斯聚类分析,设置假定分群数(K)为1-10,Burn-in周期为10 000,MCMC(Markov Chain Monte Carlo)设为100 000,每个K值重复运行10次,再利用在线工具Structure Harvester,根据Delta K的最大值确定最佳分群数[17]。
1.3.3 遗传分化、距离隔离(IBD)和环境隔离(IBE)分析利用Arlequin v3.5.2.2[18]计算同色兜兰和带叶兜兰群体间的遗传分化系数(FST)。根据12个种群的地理坐标信息,利用R语言计算种群间的地理距离,并在WorldClim数据库(http://worldclim.org/)中下载了19个气候变量信息,在国家青藏高原数据中心(https://data.tpdc.ac.cn/)的Harmonized World Soil Database v1.1中下载了16个表土理化特性信息作为土壤变量。通过计算35个变量间的Spearman相关性系数(R)来去除冗余变量,当两两变量间的相关性绝对值大于0.8时,仅保留其中1个变量,最终确定了12个代表性环境变量:年平均气温(BIO1)、昼夜温差月均值(BIO2)、等温性(BIO3)、温度季节性变化标准差(BIO4)、最干月降水量(BIO14)、降水量变异系数(BIO15)、表土含砾量(T_GRAVEL)、表土黏土分数(T_CLAY)、表土USDA质地分类(T_USDA_TEX)、表土水pH值(T_PH_H2O)、表土阳离子交换量(T_CEC_CLAY)、表土盐度(T_ECE)。最后,分别对遗传分化系数、地理距离以及12个代表性环境变量进行Mantel检验,计算Spearman相关性系数,并进行9 999次置换检验显著性。
2 结果与分析 2.1 遗传多样性和种群瓶颈在231个同色兜兰和带叶兜兰样品中,10个SSR位点检测的平均Na为2.4,平均Ne为1.6。其中,I最高的是位点DL21(1.093),最低的是DL23(0.147)。Ho和He分别为0.029-0.629和0.081-0.571。各位点的平均F值为0.081 (表 2)。
通过比较物种间的平均遗传多样性水平发现,带叶兜兰的I(0.619)约为同色兜兰(0.290)的2倍,并且其Ho(0.335)和He(0.354)均极显著(P < 0.001)高于同色兜兰(Ho=0.136,He=0.159)。在所有种群中,检测到的平均Na和Ne分别为0.9-3.2和0.8-2.0。从固定系数上看,除DY-XKR和TS-JH种群外,其余种群均表现出杂合缺失(F>0)。此外,瓶颈效应结果显示,同色兜兰的TS-HG和TS-MQ种群存在显著瓶颈效应(P < 0.05),而带叶兜兰所有种群均不存在显著瓶颈效应(P>0.05,表 3)。
| 种群 Population |
观测等位基因数(Na) Number of observed alleles (Na) |
有效等位基因数(Ne) Number of effective alleles (Ne) |
香农信息指数(I) Shannon information index (I) |
观测杂合度(Ho) Observation heterozygosity(Ho) |
期望杂合度(He) Expected heterozygosity(He) |
固定系数(F) Fixed index(F) |
瓶颈效应a Bottleneck effecta |
| TS-HG | 2.3 | 1.3 | 0.269 | 0.105 | 0.130 | 0.116 | * |
| TS-JH | 1.6 | 1.4 | 0.260 | 0.180 | 0.156 | -0.013 | NS |
| TS-MQ | 2.5 | 1.7 | 0.434 | 0.132 | 0.223 | 0.225 | * |
| TS-PR | 0.9 | 0.8 | 0.109 | 0.033 | 0.072 | 0.400 | - |
| TS-WP | 2.0 | 1.5 | 0.376 | 0.232 | 0.214 | 0.050 | NS |
| Meanb | 1.9** | 1.3* | 0.290*** | 0.136*** | 0.159*** | 0.156 NS | - |
| DY-HJ | 2.4 | 1.8 | 0.555 | 0.310 | 0.323 | 0.015 | NS |
| DY-JX | 2.5 | 1.7 | 0.600 | 0.330 | 0.349 | 0.087 | NS |
| DY-LX | 2.9 | 1.7 | 0.634 | 0.353 | 0.359 | 0.029 | NS |
| DY-PJ | 3.2 | 1.8 | 0.650 | 0.327 | 0.360 | 0.028 | NS |
| DY-TL | 2.8 | 2.0 | 0.722 | 0.402 | 0.411 | 0.017 | NS |
| DY-XKR | 2.7 | 1.7 | 0.597 | 0.351 | 0.348 | -0.048 | NS |
| DY-YC | 2.8 | 1.6 | 0.578 | 0.271 | 0.325 | 0.117 | NS |
| Meanb | 2.8** | 1.8* | 0.619*** | 0.335*** | 0.354*** | 0.035 NS | - |
| Note: a represents the significance of the bottleneck effect test; b represents the significance of the t-test; NS, *, **and *** represent the significance of P>0.05, P < 0.05, P < 0.01 and P < 0.001, respectively; “-” indicates missing data. | |||||||
AMOVA分析(表 4)显示,带叶兜兰的遗传变异主要来自个体内(77.5%),同色兜兰个体间(45.9%)和个体内(40.5%)的遗传变异相当,两个物种来自种群间的变异都较少,带叶兜兰仅7.4%,同色兜兰仅13.6%。
| 变异来源 Source of variation |
自由度 Df. |
平方和 Sum of squares |
方差分量 Variance components |
变异百分比/% Percentage of variation/% |
|
| P.concolor | Among populations | 4 | 35.4 | 0.2 | 13.6 |
| Among individuals | 75 | 165.3 | 0.8 | 45.9 | |
| Within individuals | 80 | 54.0 | 0.7 | 40.5 | |
| P.hirsutissimum | Among populations | 6 | 54.1 | 0.2 | 7.4 |
| Among individuals | 144 | 331.0 | 0.3 | 15.1 | |
| Within individuals | 151 | 250.0 | 1.7 | 77.5 | |
2.2 系统发育关系及遗传结构
群体水平的UPGMA树和个体水平的NJ树均显示同色兜兰和带叶兜兰各自聚为1支[图 1(a)、(b)],但相比于长瓣兜兰、麻栗坡兜兰和硬叶兜兰,带叶兜兰与同色兜兰的系统发育关系更为接近。在UPGMA树中发现,同色兜兰的TS-JH和TS-WP遗传距离最近,而带叶兜兰中DY-JX和DY-PJ,以及DY-XKR和DY-TL的遗传距离都较近[图 1(a)]。PCoA的结果与NJ树分析的结果基本一致,可以较好地区分带叶兜兰与同色兜兰个体,其中,第1和第2主坐标成分对总变量的解释率分别为54.8%和12.2% [图 1(c)]。
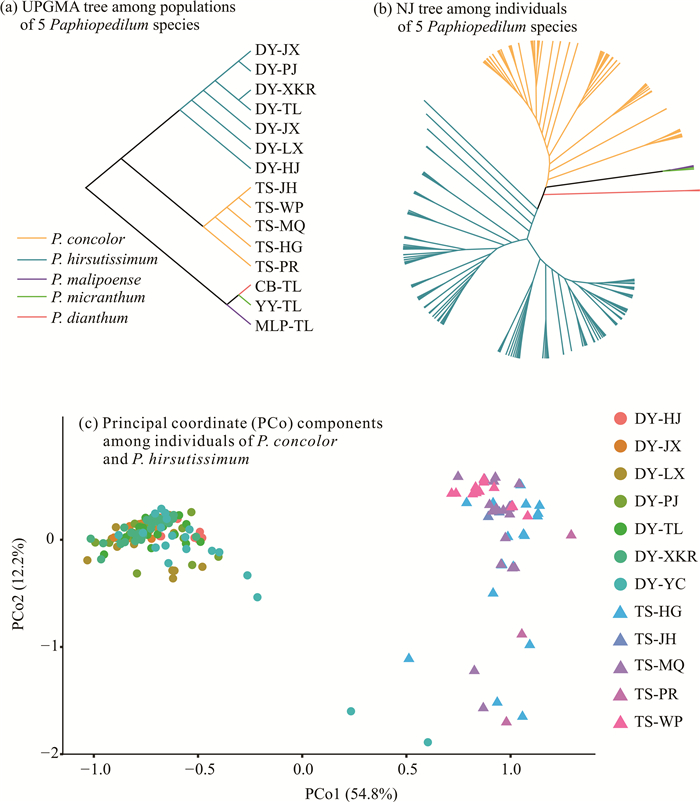
|
| 图 1 系统发育分析和主坐标分析 Fig. 1 Phylogenetic analysis and PCoA |
对两个物种的Structure分析显示K=2的分群结果最佳,此时两个物种的个体被明显分开[图 2(a)、(b)]。同色兜兰和带叶兜兰的独立Structure分析显示,两个物种都进一步检测到两种不同的遗传成分[图 2(c)、(d)]。

|
| 图 2 群体结构分析 Fig. 2 Population structure analysis |
2.3 遗传分化、IBD和IBE的检验
遗传分化分析结果[图 3(a)]显示,带叶兜兰两两种群间的平均FST为0.077,同色兜兰两两种群间的平均FST为0.237,而两个物种的种群间的平均FST为0.572,达到了较高的分化水平。为探究遗传分化的驱动因子,分别进行IBD检验[图 3(b)]和IBE检验[图 3(c)]。其中,IBD检验显示12个种群间的地理距离与遗传距离呈现显著正相关(R=0.300,P < 0.05),表明地理隔离可能促进两个物种间的遗传分化。然而,FST与12个代表性环境因子间的Mantle检验均不显著,表明IBE模式在驱动带叶兜兰和同色兜兰的种群分化过程中的作用可能不太明显。
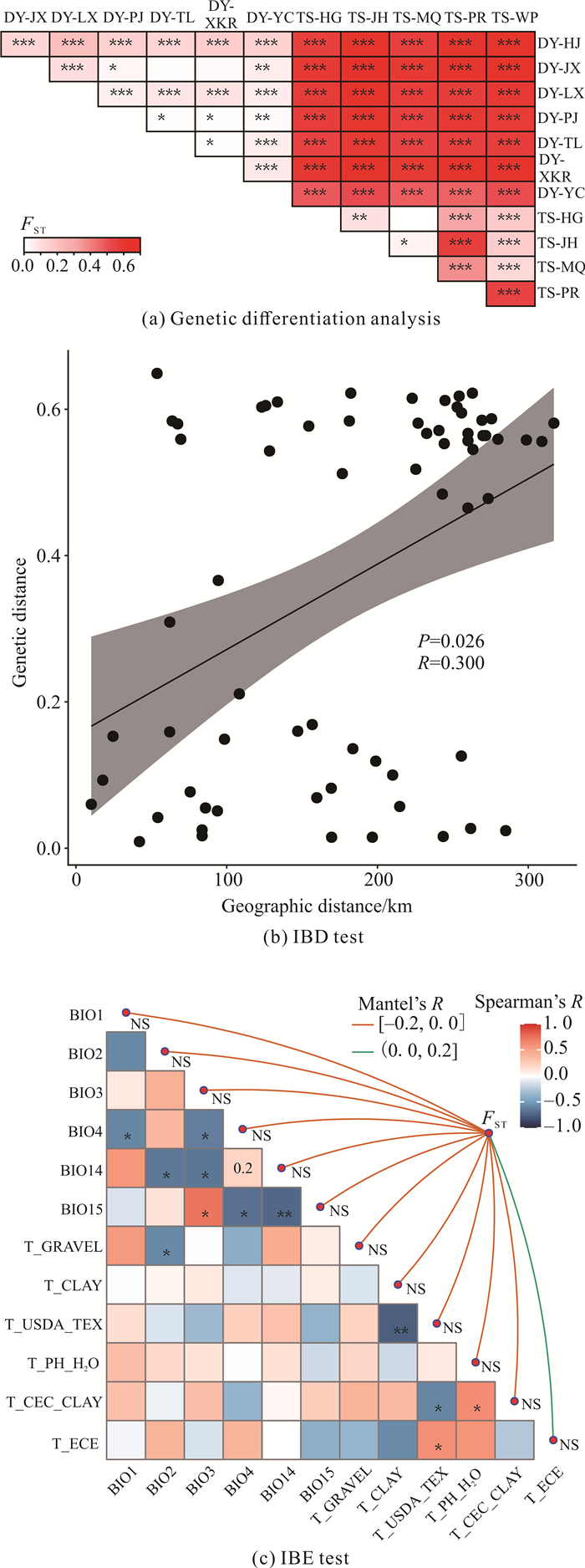
|
| NS, *, **, and *** represent the significance of P>0.05, P < 0.05, P < 0.01, respectively. 图 3 遗传分化分析、IBD检验和IBE检验 Fig. 3 Genetic differentiation analysis, IBD tests and IBE tests |
3 讨论
本研究利用10个多态性EST-SSR位点比较了四季开花型同色兜兰和春季开花型带叶兜兰的遗传多样性水平,并发现前者的遗传多样性水平显著低于后者。物种的遗传多样性可能受到分布范围的影响,许多狭窄分布的濒危物种的遗传多样性水平都较低[19, 20]。张央等[4]对全国兰科兜兰属宽瓣亚属(Paphiopedium Subgen.Brachypelalum)植物的一项专项调查研究结果显示,目前野生同色兜兰的自然分布区域已经缩小到广西西南地区,因此更狭窄的自然分布范围和更小的有效种群数量可能解释了同色兜兰更低的遗传多样性。带叶兜兰的高遗传多样性可能与其个体间丰富的遗传变异有关,AMOVA结果显示,带叶兜兰中来自个体内的遗传变异达77.5%,这与徐言等[10]的研究结果类似;而同色兜兰中来自个体内的遗传变异仅为40.5%。此外,物种的遗传多样性水平还可能受到多样化的种群进化历史的影响[21-23]。例如,孙艺琦等[21]发现单叶蔓荆(Vitex rotundifolia)与蔓荆(V.trifolia)的种群曾经历过瓶颈效应,并导致了两者较低的遗传多样性。种群瓶颈的原因主要包括两方面: 一是人工选择压力,二是奠基者效应[24]。通常,野生植物种群的瓶颈效应主要是由奠基者效应引起的。本研究在同色兜兰的部分种群中检测到了显著的瓶颈效应,但在带叶兜兰中未检测到,表明在种群形成历史中曾遭遇的瓶颈效应可能导致同色兜兰丧失部分的遗传多样性。
本研究中带叶兜兰的DY-YC种群和DY-XKR种群分别与徐言等[10]研究中的GYC和GML种群来源一致,但本研究评估的两个就地保护种群的遗传多样性略低于徐言等[10]的评估结果。本研究使用了兼顾两个物种(带叶兜兰和同色兜兰)多态性的SSR位点,因此这些位点在单一物种中的变异可能相对保守,从而低估了种群杂合度。此外,不同的采样个体和采样数量也可能影响对种群杂合度的评估。对自然保护区内外的带叶兜兰种群的遗传多样性的比较结果表明,3个自然保护区内的就地保护种群的多态性指数(DY-XKR,I=0.597;DY-YC,I=0.578;DY-LX,I=0.634)符合野生种群的标准(I=0.555-0.722),就地保护可以较好地保护野生带叶兜兰的遗传多样性。然而,位于广西崇左白头叶猴国家级自然保护区的同色兜兰种群(TS-PR,I=0.109)遗传多样性则低于其他4个野生同色兜兰种群。因此,为了更好地保护同色兜兰的野生资源,建议进一步扩大自然保护区的范围或者结合迁地保护措施。
AMOVA和遗传分化分析的结果均显示,同色兜兰种群的分化程度要高于带叶兜兰种群。较高的遗传分化通常意味着基因流受限[25, 26]。兜兰属物种大多采用欺诈性授粉的策略[27],这种传粉策略风险很高,通常严重依赖专一的传粉者;并且自然环境中的兜兰属物种还普遍存在种子萌发困难[28]的现象,这些都可能导致由花粉或者种子介导的基因流受限。与同色兜兰不同,带叶兜兰具有非常强的营养繁殖能力,还可以通过走茎进行营养繁殖[5],这种繁殖方式可以有效促进种群规模的扩大,并有可能增加种群间基因流的机会。
基因流受限可能是由地理隔离或者环境隔离导致的,这与许多濒危或特有植物的处境类似,如地中海高山植物Silene ciliate[29]、中国北方濒危的野蔷薇(Rosa rugosa)[30]、中国南方特有的篦子三尖杉(Cephalotaxus oliveri)[31]等。IBD和IBE的结果表明,地理隔离是影响同色兜兰和带叶兜兰种群遗传分化的主要因素,而环境隔离的影响较小。尽管此前一些研究模拟和分析了中国境内兜兰属植物的地理分布格局,发现一些环境变量如昼夜温差、年平均气温变化范围、最暖季度平均气温、太阳辐射、水蒸气压等对兜兰属植物的自然地理分布格局具有重要影响[4, 32],但是本研究并未发现环境变量对同色兜兰和带叶兜兰种群分化具有显著影响。一方面,人为因素的存在可能会削弱IBE的影响,特别是考虑到带叶兜兰和同色兜兰野生植株经常遭受非法采摘;另一方面,由于本研究的采样范围局限于广西,在相对小的尺度下IBE的作用可能并不明显。因此,对IBE模式在兜兰属物种种群分化过程中的作用的进一步探究需要结合更大范围的采样。
同色兜兰是兜兰属中罕见的四季开花型物种,但分布较狭窄。与同色兜兰相比,尽管带叶兜兰花期短,但是其自然分布面积较广,适生区范围较大。因此,通过开展两个物种之间的杂交有望从中选育出一些花期长且适应性强的优良品种,以满足日益增长的市场需求,并且能更好地保护野生兜兰资源。此前对兜兰属物种的杂交研究发现,在带叶兜兰、杏黄兜兰(P.armeniacum)、硬叶兜兰和同色兜兰的16种杂交及自交组合类型中,同色兜兰×带叶兜兰的杂交结实率最高,达93.1%[33]。一般而言,兰科物种间的亲缘关系越近则杂交亲和性越高[34]。而本研究的NJ树和UPGMA树的结果显示,在5个兜兰属物种中,同色兜兰与带叶兜兰间的系统发育关系最近,因此,该结果为同色兜兰和带叶兜兰间的高杂交率提供了有力的系统发育学证据支持。此外,这些系统发育关系信息还可以用于指导今后兜兰属的优良品种选育,例如,在UPGMA树中发现同色兜兰的TS-JH和TS-WP种群与带叶兜兰的DY-HJ种群的遗传距离均较为接近,因此,这些种群可以优先作为开展杂交试验的材料。然而鉴于SSR位点提供的遗传信息有限,对于兜兰属物种间系统发育关系的进一步阐明还需要借助更全面的基因组信息,如简化基因组或全基因组数据。
| [1] |
刘仲健, 陈心启, 陈利君, 等. 中国兜兰属植物[M]. 北京: 科学出版社, 2009.
|
| [2] |
尹玉莹, 房林, 李琳, 等. 兜兰属植物花期调控研究进展[J]. 热带作物学报, 2022, 43(4): 769-778. DOI:10.3969/j.issn.1000-2561.2022.04.013 |
| [3] |
刘仲健, 张建勇, 徐向明, 等. 同色兜兰及其亲缘群的研究[J]. 云南植物研究, 2000, 22(4): 390-394. |
| [4] |
张央, 安明态, 武建勇, 等. 中国兜兰属宽瓣亚属植物地理分布格局及其主导气候因子[J]. 植物生态学报, 2022, 46(1): 40-50. |
| [5] |
王文晓, 程浩, 徐玉凤, 等. 带叶兜兰5个野生居群表型多样性分析[J]. 植物遗传资源学报, 2020, 21(5): 1196-1206. |
| [6] |
杨磊, 曹秋梅, 冯缨, 等. 珍稀濒危植物阜康阿魏的遗传多样性及遗传结构[J]. 植物研究, 2023, 43(1): 51-58. |
| [7] |
HUANG J L, LI S Y, HU H. ISSR and SRAP markers reveal genetic diversity and population structure of an endangered slipper orchid, Paphiopedilum micranthum (Orchidaceae)[J]. Plant Diversity, 2014, 36(2): 209-218. |
| [8] |
TONG Y W, DURKA W, ZHOU W M, et al. Ex situ conservation of Pinus koraiensis can preserve genetic diversity but homogenizes population structure[J]. Forest Ecology and Management, 2020, 465: 117820. DOI:10.1016/j.foreco.2019.117820 |
| [9] |
高丽霞, 覃国乐, 易桂萍. 带叶兜兰遗传多样性的SRAP分析[J]. 南方农业学报, 2014, 45(10): 1734-1738. |
| [10] |
徐言, 陈之光, 徐玉凤, 等. 基于SSR标记的西南地区野生带叶兜兰资源遗传多样性分析[J/OL]. 热带作物学报, 2023: 1-11[2023-08-10]. http://kns.cnki.net/kcms/detail/46.1019.S.20230216.1655.008.html.
|
| [11] |
DOYLE J J, DOYLE J L, BALLENGER J A, et al. The distribution and phylogenetic significance of a 50-kb chloroplast DNA inversion in the flowering plant family Leguminosae[J]. Molecular Phylogenetics and Evolution, 1996, 5(2): 429-438. DOI:10.1006/mpev.1996.0038 |
| [12] |
XU Y F, JIA R D, ZHOU Y H, et al. Development and characterization of polymorphic EST-SSR markers for Paphiopedilum henryanum (Orchidaceae)[J]. Applications in Plant Sciences, 2018, 6(5): e01152. DOI:10.1002/aps3.1152 |
| [13] |
PEAKALL R, SMOUSE P E. GenAlEx 6.5: genetic analysis in Excel.population genetic software for teaching and research: an update[J]. Bioinformatics, 2012, 28(19): 2537-2539. DOI:10.1093/bioinformatics/bts460 |
| [14] |
PIRY S, LUIKART G, CORNUET J M. Computer note. BOTTLENECK: a computer program for detecting recent reductions in the effective size using allele frequency data[J]. Journal of Heredity, 1999, 90(4): 502-503. DOI:10.1093/jhered/90.4.502 |
| [15] |
TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [16] |
PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2): 945-959. DOI:10.1093/genetics/155.2.945 |
| [17] |
EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study[J]. Molecular Ecology, 2005, 14(8): 2611-2620. DOI:10.1111/j.1365-294X.2005.02553.x |
| [18] |
EXCOFFIER L, LAVAL G, SCHNEIDER S. Arlequin (version 3.0): an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 2007, 1: 47-50. |
| [19] |
GITZENDANNER M A, SOLTIS P S. Patterns of genetic variation in rare and widespread plant congeners[J]. American Journal of Botany, 2000, 87(6): 783-792. DOI:10.2307/2656886 |
| [20] |
GONG W, GU L, ZHANG D X. Low genetic diversity and high genetic divergence caused by inbreeding and geographical isolation in the populations of endangered species Loropetalum subcordatum (Hamamelidaceae) endemic to China[J]. Conservation Genetics, 2010, 11(6): 2281-2288. DOI:10.1007/s10592-010-0113-9 |
| [21] |
孙艺琦, 赵露颖, 朱波, 等. 基于SSR分子标记的蔓荆子基原植物的遗传多样性及遗传结构分析[J]. 中国中药杂志, 2021, 46(15): 3824-3831. |
| [22] |
RUAN X X, WANG Z, WANG T, et al. Characterization and application of EST-SSR markers developed from the transcriptome of Amentotaxus argotaenia (Taxaceae), a relict vulnerable conifer[J]. Frontiers in Genetics, 2019, 10: 1014. DOI:10.3389/fgene.2019.01014 |
| [23] |
WU F Q, SHEN S K, ZHANG X, et al. Inferences of genetic structure and demographic history of Rhododendron protistum var.giganteum: the world's largest Rhododendron using microsatellite markers[J]. Flora, 2017, 233: 1-6. DOI:10.1016/j.flora.2017.04.009 |
| [24] |
余意, 王凌, 孙嘉惠, 等. 基于微卫星群体遗传学的栽培枸杞遗传多样性和遗传结构评价[J]. 中国中药杂志, 2020, 45(4): 838-845. |
| [25] |
王天翼, 徐悦, 王罗云, 等. 中国沙棘和云南沙棘的遗传分化及遗传多样性[J]. 林业科学研究, 2021, 34(4): 13-21. |
| [26] |
张宏祥, 郑田勇. 生境片段化对新疆野苹果种群遗传结构的影响[J]. 干旱区研究, 2020, 37(3): 715-721. |
| [27] |
初美静, 宫子惠, 孙纪霞, 等. 肉饼兜兰传粉生物学及开花进程研究[J]. 种子, 2019, 38(3): 88-92, 96. |
| [28] |
张娟娟, 严宁, 胡虹. 三种兜兰属植物种子发育过程及其与无菌萌发的关系[J]. 植物分类与资源学报, 2013, 35(1): 33-40. |
| [29] |
MORENTE-LÓPEZ J, GARCÍA C, LARA-ROMERO C, et al. Geography and environment shape landscape genetics of Mediterranean alpine species Silene ciliata Poiret.(Caryophyllaceae)[J]. Frontiers in Plant Science, 2018, 9: 1698. DOI:10.3389/fpls.2018.01698 |
| [30] |
XU J J, ZANG F Q, WU Q C, et al. Analysis of the genetic diversity and molecular phylogeography of the endangered wild rose (Rosa rugosa) in China based on chloroplast genes[J]. Global Ecology and Conservation, 2021, 28: e01653. DOI:10.1016/j.gecco.2021.e01653 |
| [31] |
LIU H J, WANG Z, ZHANG Y L, et al. Geographic isolation and environmental heterogeneity contribute to genetic differentiation in Cephalotaxus oliveri[J]. Ecology and Evolution, 2023, 13(3): e9869. DOI:10.1002/ece3.9869 |
| [32] |
YE P C, WU J Y, AN M T, et al. Geographical distribution and relationship with environmental factors of Paphiopedilum subgenus Brachypetalum Hallier (Orchidaceae) taxa in southwest China[J]. Diversity, 2021, 13(12): 634. DOI:10.3390/d13120634 |
| [33] |
覃艳芬, 朱根发, 彭永宏. 4种兜兰属植物的杂交结实性研究[C]. 中国观赏园艺研究进展2011. 银川: 中国林业出版社, 2011: 227-232.
|
| [34] |
朱根发. 国际兰属植物杂交育种进展[J]. 广东农业科学, 2005, 32(4): 25-27. |



