珊瑚礁被比喻为“海洋的热带雨林”,是地球上生产力最高、生物多样性最为丰富的海洋生态系统[1],在全球碳循环中扮演着重要角色。然而,珊瑚礁又是非常脆弱的生态系统,全球气候变化、海水酸化、海平面上升等一系列生态问题和人类活动的影响导致全球范围内的珊瑚礁持续退化,且白化频度和严重度不断上升[2, 3]。在过去的几十年里,全球科学家围绕珊瑚礁地质学、生物学、基因组学、共生关系、生态修复、生物多样性、应对气候变化和海洋酸化等方面开展了较多的研究[4-10]。其中,珊瑚礁生物多样性领域的研究多集中于基于形态学的珊瑚多样性[11, 12]、基于分子生物学的珊瑚进化和遗传距离分析[13, 14]、鱼类[15, 16]、潜水可见的底栖动物等[17, 18]方面,而实际上珊瑚礁区存在的生物种类远不止鱼类和大型底栖动物。
珊瑚礁隐居生物(或底内生物,Cryptofauna)是栖息于活珊瑚隐匿空间和死珊瑚碎砾里的后生动物的总称[19, 20]。珊瑚礁内的无脊椎动物已被发现多达16.5万种,而科学家预测有属于31个门、多达900万种的隐居生物还未被描述[21]。隐居生物构成了已知珊瑚礁物种的90%以上[22],单位面积的隐居生物多样性和生物量远高于珊瑚礁环境中的其他生物[23],其群落在珊瑚礁营养动力和能量代谢上行使着重要的生态功能,有不可或缺的作用[24, 25]。
1 珊瑚礁隐居生物的定义和类别隐匿生活于珊瑚分枝、裂隙以及死珊瑚碎砾的缝隙空间的生物即为珊瑚礁隐居生物[19, 20]。珊瑚礁隐居生物或营附着生活或营运动生活,涵盖了几乎所有的后生动物门类,包括多孔动物门、环节动物门、星虫动物门、软体动物门、节肢动物门、棘皮动物门、脊索动物门等[23, 26]。
2 珊瑚礁隐居生物的作用珊瑚礁隐居生物对珊瑚礁生境结构的形成有着重要的影响[27-29]。例如,海绵、贻贝等生物会弱化珊瑚的骨架,促进珊瑚的无性繁殖[30-32];涡虫、海星、海胆、寄居蟹、多毛类、缨鳃虫、海蛞蝓等会摄食珊瑚,这些生物大量聚集时会伤害珊瑚[33];具有生物腐蚀作用的藤壶、贻贝、多毛类、星虫和海绵会钻入死珊瑚骨架中并降解珊瑚碳酸钙物质[30, 34]。此外,海绵和海鞘等隐居生物能将珊瑚碎块胶结并紧固成稳定的底质,利于珊瑚礁体的补充和生长[35, 36];隐居于珊瑚枝条缝隙中的一些虾、蟹会清洁珊瑚[37],促进珊瑚黏液的分泌,并护卫珊瑚免于被食用[38, 39]。
珊瑚礁隐居生物是珊瑚礁生态系统营养网络的支配成员,对珊瑚礁生态系统功能的发挥至关重要。珊瑚礁隐居生物在珊瑚礁生态系统中扮演着不同的角色,包括屑食者、滤食者、捕食者和植食者。碎屑食性的隐居生物(如甲壳动物、棘皮动物、鱼类)能消解有机沉积物和粪便[37, 40],将其转化成生命可以利用的形态。滤食性的珊瑚礁隐居生物(如双壳贝类、海鞘、海绵)在水体中捕食浮游动植物,将其转化为底栖生物的生物量[41, 42]。植食性的珊瑚礁隐居生物(如腹足类和甲壳类)能吃掉珊瑚藻类,也能在珊瑚的遮护下免遭雀鲷、鮋科等礁栖鱼类的捕食而占据优势种的地位[43]。
3 海洋酸化对珊瑚礁隐居生物的影响全球珊瑚礁正面临多方面的威胁,珊瑚礁生物群落结构不同,其对环境压力的响应就不同。科学合理地制订珊瑚礁修复措施需要全方位考虑,其中应包括珊瑚礁的生物多样性因素,如果隐居生物这一重要的因素被忽略,那么珊瑚礁的保护效率就可能被削弱。
海水pH值的降低称为海洋酸化,表现为海水碳饱和浓度的降低,会影响海洋生物的钙增生,降低钙化率和加速生物溶蚀。人们关注到长期的海洋酸化会导致珊瑚礁复杂性结构的简化,但往往忽略了珊瑚礁生态系统中的生物对海洋酸化的响应[44]。一方面,珊瑚礁隐居生物中的贝类、甲壳类等多种钙化生物会因海洋酸化而降低生物钙的增生,其胚胎、幼体的发育以及生活史会因此受到影响,而胚胎和幼体发育是否正常决定种群世代繁衍能否完成。另一方面,海洋酸化引起的珊瑚礁结构的变化会造成栖息在其凹隙内部的隐居生物的生物多样性、丰度、生物量、优势种的变化以及栖息地的迁移[45]。因此,海洋酸化不仅仅会直接影响隐居生物,其引发的造礁珊瑚结构的改变也会间接影响隐居生物,继而进一步影响珊瑚礁生态系统的功能。
因此,只研究造礁珊瑚本身的变化不足以判断海洋酸化对珊瑚礁生态系统造成的影响,还需进一步开展珊瑚礁隐居生物的研究,才能全面认识海洋酸化对珊瑚礁的生态影响[46]。通过对珊瑚礁隐居生物的了解,又可以反过来帮助认识和判断珊瑚礁的状况及变化趋势。目前,人们对珊瑚礁隐居生物群落对海洋酸化的生态响应仍所知甚少,国内相关研究尤为缺乏,概因对珊瑚礁隐居生物这一丰富的动物群落了解不足。
4 珊瑚礁隐居生物的研究方法 4.1 珊瑚礁自动监测装置(ARMS)重视程度不够和研究方法的局限,造成人们对珊瑚礁隐居生物的生物学和生态功能知之甚少[47]。利用箱式采泥器可以无破坏性地采集礁区底栖动物,但无法采集到珊瑚礁内部的隐居生物。采用截线法和样框法拍摄高清珊瑚照片和录像于电脑上进行分析,是研究珊瑚礁生物多样性最常用的方法,可有效用于营固着生活的动物以及活动缓慢的棘皮类、贝类等大型底栖动物的调查研究,但并不适用于运动迅速的鱼类、甲壳类等的调查研究,更无法用于隐居生物的调查研究[48]。鉴于珊瑚礁隐居生物研究的重要性,1978年,联合国教科文组织提出了采用斗式抓泥器、重力采集器对珊瑚礁隐居生物进行研究的方法[49],但这些方法会对礁石区产生不同程度的破坏。
为无破坏性并标准化地对珊瑚礁隐居生物开展研究,美国国家海洋和大气管理局(NOAA)的珊瑚礁生态系统处(Coral Reef Ecosystem Division, CRED)、海洋生物普查计划(Census of Marine Life, CoML)和珊瑚礁生态系统普查计划(Census of Coral Reef Ecosystems, CReefs)共同开发了一种珊瑚礁自动监测装置(Autonomous Reef Monitoring Structures,ARMS),如图 1所示。该装置是由9层22.5 cm×22.5 cm×1.27 cm的聚氯乙烯(PVC) 板组装而成,通过模拟珊瑚礁生境和结构来吸引无脊椎动物以及藻类等生物的栖息[50],用于评估和探明全球尺度下珊瑚礁隐居生物的多样性、分布、丰度和群落结构。
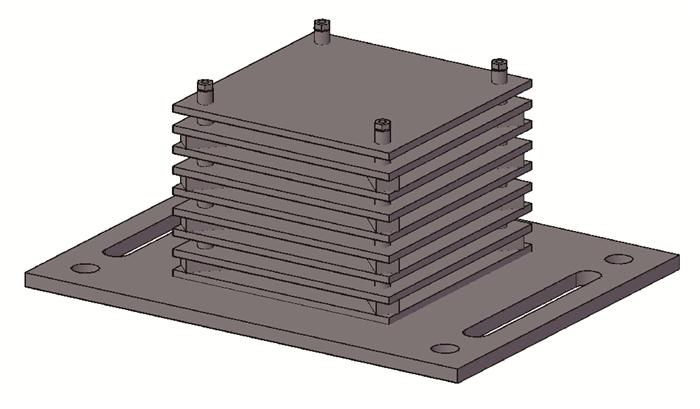
|
| 图 1 珊瑚礁自动监测装置示意 Fig. 1 Coral reef automatic monitoring device |
一个珊瑚礁自动监测装置可收集到的运动和固着隐居生物多达数十种,数量成百上千,可谓理想的采样平台。利用珊瑚礁自动监测装置可实现以标准化、可重复、系统化的方式收集珊瑚礁隐居生物并分析其多样性、栖息密度和丰度等数据,提高研究者监测和研究隐居生物群落的能力,所获得的数据可在全球尺度和时间尺度上进行比对。
珊瑚礁自动监测装置从2006年开始设计,到2008年完成试用[51]。2009年后,全球各国在海底配置了超过2 000个珊瑚礁自动监测装置并开展一系列研究,评估南太平洋、地中海、加勒比海、红海等珊瑚礁区的生物多样性,这些研究不局限于隐居生物,还包括底栖无脊椎动物、藻类、微生物等[52-55]。2020年,中国水产科学研究院南海水产研究所借鉴珊瑚礁自动监测装置收集生物的方式,对大亚湾的底栖生物进行了研究[56]。采用珊瑚礁自动监测装置收集的样本中有多种新种和新纪录种被发现,反映了珊瑚礁隐居生物为人所知甚少,珊瑚礁隐居生物研究仍是海洋生物多样性研究的洼地和宝库[57, 58]。
4.2 珊瑚礁隐居生物的物种鉴定利用珊瑚礁自动监测装置采集到的隐居生物被分离后,先进行形态学鉴定,即将自海底取出的珊瑚礁自动监测装置拆解,拍照并记录每一块PVC板后,用过滤海水洗出或刮下其上所有的生物,随后进行样品的处理、保存、计数以及形态学鉴定等[59]。但由于对珊瑚礁隐居生物认识不足,传统的形态学方法很难分类并鉴定出利用珊瑚礁自动监测装置采集到的所有隐居生物物种,形态分类鉴定存在较大的专业局限性。
个体不完整或研究人员难以通过形态学分类的物种,可通过分子条形码辅助鉴定。分子条形码是指将基因序列作为物种鉴定的标准参照[59, 60]。在大多数物种中,线粒体DNA (mitochondrial DNA, mtDNA)相对于细胞核DNA来说更适合作为条形码靶标:线粒体细胞色素氧化酶Ⅰ(COⅠ)基因有着最丰富的参考序列数据库,对物种的鉴定特异性较高,是最常见的分子条形码基因[61, 62]。此外,12S rRNA、16S rRNA、18S rRNA等也被用作不同门类物种鉴定的分子条形码[63]。
近年来,下一代高通量测序技术高速发展,在宏条形码(Metabarcoding)的助力下,使得从一个样本中测序并分析出数以百万级的分子条形码序列成为可能,因此该技术已被广泛地应用于生物多样性的研究中[64, 65]。将所有获得的物种(特别是小型无脊椎动物以及原生动物、微生物等)一起混合提取DNA后,通过下一代高通量测序和宏分子条形码分析可得到混合样品里所有的物种信息[66]。Pearman等[48]在对红海3个近岸珊瑚礁进行监测时发现,利用珊瑚礁自动监测装置收集到的动物种类远高于水下摄影见到的动物种类,而通过混合样本提取DNA高通量测序后分析分子条形码所得到的物种种类,比形态学鉴定的结果又要高1-2个数量级。
分子条形码和高通量测序技术大大提升了隐居生物研究的效率,但是传统的、基于形态学的分类方法仍是该项研究的基石。特别是隐居生物中存在的诸多新物种,仍需通过形态鉴定初步分类后,再获取分子条形码数据并上传,从而充实隐居生物分子条形码数据库以供全球研究者参考。
5 基于文献检索的国内外隐居生物研究现状使用Web of Science数据库(网址为https://apps.webofknowledge.com)搜索主题为“cryptofauna coral reefs”的文献,共检索到1988-2023年发表的论文91篇(截至2023年10月)。研究的范围覆盖隐居生物多样性的研究[67, 68],分子条形码研究[69],对海洋酸化的响应[70],隐居生物群落结构[71-73],隐居生物和珊瑚礁的营养、生态功能的关联[74, 75]等。按国家分类(图 2),来自澳大利亚、美国和日本的研究机构发文量排名前3,分别发表26、24、11篇论文,研究区域包括大堡礁[72]、印度洋查戈斯群岛[76]、夏威夷群岛[73]、伯利兹堡礁[77]、加勒比海[78]等珊瑚礁区域。来自中国的2篇论文出自南方海洋科学与工程广东省实验室(广州)-香港科技大学的共建香港分部实验室,其中一篇为研究法属波利尼西亚的莫雷阿岛珊瑚礁隐居生物的论文[50],另一篇为关于韩国济州岛珊瑚礁隐居生物的综述[79]。在中国知网中搜索“隐居生物”得到的文献数为0,表明国内的珊瑚礁隐居生物研究仍属于空白领域。

|
| 图 2 Web of Science数据库中检索“cryptofauna coral reefs”的出版物数量(国家,排名前20) Fig. 2 Number of publications searching for "cryptofauna coral reef" in Web of Science database (country, top 20) |
在Web of Science数据库中搜索主题“Autonomous Reef Monitoring Structures ARMS”,得到2013-2023年的34篇论文(截至2023年10月)。论文主要来自美国、沙特阿拉伯、法国和意大利等国家(图 3),研究内容主要集中在珊瑚礁生态系统的生物多样性方面,其中来自中国的论文是前述关于韩国济州岛珊瑚礁隐居生物的综述[79]。在中国知网搜索相关的主题和关键词,得到的文献数量仍为0,说明国内尚未开展此项研究。从以上文献检索的结果来看,珊瑚礁隐居生物和基于珊瑚自动监测装置的研究无论在国内还是在国外都起步较晚,该方法仍有待推广。

|
| 图 3 Web of Science数据库中检索“Autonomous Reef Monitoring Structures ARMS”的出版物数量(国家,排名前10) Fig. 3 Number of publications searching for "Autonomous Reef Monitoring Structures ARMS" in Web of Science database (country, top 10) |
6 展望
珊瑚礁隐居生物被认为是珊瑚礁中多样性最丰富的一群生物,其物种涵盖所有的海洋无脊椎动物门类。由于珊瑚礁隐居生物的隐居特性和采样方法的局限等,珊瑚礁隐居生物的生物多样性认识仍存在诸多空白,需要开展更为广泛深入的研究。采用诸如国际标准的珊瑚礁自动监测装置进行珊瑚礁隐居生物的采样是可取的方法,利于开展无破坏性的珊瑚礁研究工作,所获数据可进行不同时空尺度的比对,从而探索珊瑚礁结构、构造与珊瑚礁隐居生物多样性的关系,揭示珊瑚礁隐居生物的分布格局及其驱动因素。
珊瑚礁隐居生物的生物量远高于其表面栖息的动物类群,表明其是珊瑚礁营养层级结构中极为重要的一环,它们不仅为鱼类提供食物来源,而且其自身也可以是捕食者、植食者、屑食者、滤食者和腐食者,在珊瑚礁食物链和营养流动中起到重要的作用。开展珊瑚礁隐居生物营养动力学的研究,例如水体营养与珊瑚礁隐居生物生物量的关系、珊瑚礁隐居生物与珊瑚礁鱼类的营养关联等,有助于深入认识珊瑚礁高生产力的机理,了解珊瑚礁退化对隐居生物的影响以及对珊瑚礁生态系统功能的影响。
珊瑚礁隐居生物物种多样性丰富、生物量高,在珊瑚礁生态系统的营养结构中作用巨大,因此科学地认知珊瑚礁隐居生物,有助于更加全面地认识珊瑚礁生态系统。因其个体微小和隐匿生活的特性,珊瑚礁隐居生物易被忽略,科学认识仍有缺口,国内外相关研究均很有限,需进一步拓展和深化。
处于人类世的珊瑚礁,面临人类活动的严重干扰及其所引起的环境变化的影响。无疑,更全面科学地认知包括珊瑚礁隐居生物在内的珊瑚礁生态系统,将有助于采取科学有效的应对措施,提高珊瑚礁保护的有效性。
| [1] |
STONE L. Biodiversity and habitat destruction: a comparative study of model forest and coral reef ecosystems[J]. Proceedings of the Royal Society B: Biological Sciences, 1995, 261(1362): 381-388. DOI:10.1098/rspb.1995.0163 |
| [2] |
MORRISON T H, HUGHES T P, ADGER W N, et al. Save reefs to rescue all ecosystems[J]. Nature, 2019, 573(7774): 333-336. DOI:10.1038/d41586-019-02737-8 |
| [3] |
BELLWOOD D R, HUGHES T P, FOLKE C, et al. Confronting the coral reef crisis[J]. Nature, 2004, 429(6994): 827-833. DOI:10.1038/nature02691 |
| [4] |
黄晖, 俞晓磊, 黄林韬, 等. 珊瑚礁生态学研究和展望[J/OL]. 热带海洋学报. (2023-09-08)[2023-10-15]. https://link.cnki.net/urlid/44.1500.P.20230907.0931.002.
|
| [5] |
余克服. 南海珊瑚礁及其对全新世环境变化的记录与响应[J]. 中国科学: 地球科学, 2012, 42(8): 1160-1172. |
| [6] |
HU M, ZHENG X, FAN C M, et al. Lineage dynamics of the endosymbiotic cell type in the soft coral Xenia[J]. Nature, 2020, 582(7813): 534-538. DOI:10.1038/s41586-020-2385-7 |
| [7] |
STEPHENS T G, LEE J, JEONG Y, et al. High-quality genome assembles from key Hawaiian coral species[J]. Gigascience, 2022, 11: giac098. DOI:10.1093/gigascience/giac098 |
| [8] |
TSUCHIYA K, ZAYASU Y, NAKAJIMA Y, et al. Ge- nomic analysis of a reef-building coral, Acropora digitifera, reveals complex population structure and a migration network in the Nansei Islands, Japan[J]. Molecular Ecology, 2022, 31(20): 5270-5284. DOI:10.1111/mec.16665 |
| [9] |
BAIROS-NOVAK K R, HOOGENBOOM M O, VAN OPPEN M J H, et al. Coral adaptation to climate change: meta-analysis reveals high heritability across multiple traits[J]. Global Change Biology, 2021, 27(22): 5694-5710. DOI:10.1111/gcb.15829 |
| [10] |
GISCHLER E, HUDSON J H, EISENHAUER A, et al. 9000 years of change in coral community structure and accretion in Belize reefs, western Atlantic[J]. Scientific Reports, 2023, 13(1): 11349. DOI:10.1038/s41598-023-38118-5 |
| [11] |
黄林韬, 黄晖, 江雷. 中国造礁石珊瑚分类厘定[J]. 生物多样性, 2020, 28(4): 515-523. |
| [12] |
梁文, 周浩郎, 王欣, 等. 涠洲岛西南部海域造礁石珊瑚的群落结构特征分析[J]. 海洋学报, 2021, 43(11): 123-135. |
| [13] |
KERR A M. Molecular and morphological supertree of stony corals (Anthozoa: Scleractinia) using matrix representation parsimony[J]. Biological Reviews of the Cambridge Philosophical Society, 2005, 80(4): 543-558. DOI:10.1017/S1464793105006780 |
| [14] |
QUEK Z B R, JAIN S S, RICHARDS Z T, et al. A hybrid-capture approach to reconstruct the phylogeny of Scleractinia (Cnidaria: Hexacorallia)[J]. Molecular Phylogenetics and Evolution, 2023, 186: 107867. DOI:10.1016/j.ympev.2023.107867 |
| [15] |
BRANDL S J, GOATLEY C H R, BELLWOOD D R, et al. The hidden half: ecology and evolution of cryptobenthic fishes on coral reefs[J]. Biological Reviews of the Cambridge Philosophical Society, 2018, 93(4): 1846-1873. DOI:10.1111/brv.12423 |
| [16] |
代血娇, 张俊, 陈作志. 珊瑚礁鱼类多样性及保护研究进展[J]. 生态学杂志, 2021, 40(9): 2996-3006. |
| [17] |
黄丁勇, 王建佳, 陈甘霖, 等. 亚龙湾珊瑚礁大型礁栖生物的群落结构及生态警示[J]. 生态学杂志, 2021, 40(2): 412-426. |
| [18] |
夏光远, 陈石泉, 谢海群, 等. 西沙群岛珊瑚礁区大型底栖生物群落特征[J]. 热带生物学报, 2023, 14(1): 8-16. |
| [19] |
HUTCHINGS P. Cryptofaunal communities of coral reefs[J]. Acta Oceanologica Sinica, 1986, 5(4): 603-613. |
| [20] |
BIONDI P, MASUCCI G D, REIMER J D. Coral cover and rubble cryptofauna abundance and diversity at outplanted reefs in Okinawa, Japan[J]. PeerJ, 2020, 8(1): e9185. |
| [21] |
STELLA J S, JONES G P, PRATCHETT M S. Variation in the structure of epifaunal invertebrate assemblages among coral hosts[J]. Coral Reefs, 2010, 29: 957-973. DOI:10.1007/s00338-010-0648-8 |
| [22] |
STELLA J S, WOLFE K, ROFF G, et al. Functional and phylogenetic responses of motile cryptofauna to habitat degradation[J]. Journal of Animal Ecology, 2022, 91(11): 2203-2219. DOI:10.1111/1365-2656.13809 |
| [23] |
WOLFE K, DESBIENS A, STELLA J, et al. Length-weight relationships to quantify biomass for motile coral reef cryptofauna[J]. Coral Reefs, 2020, 39: 1649-1660. DOI:10.1007/s00338-020-01993-9 |
| [24] |
KLUMPP D W, MCKINNON A D, MUNDY C. Motile cryptofauna of a coral reef: abundance, distribution and trophic potential[J]. Marine Ecology-Progress Series, 1988, 45(1/2): 95-108. |
| [25] |
WOLFE K, DESBIENS A A, MUMBY P J. Emigration patterns of motile cryptofauna and their implications for trophic functioning in coral reefs[J]. Ecology and Evolution, 2023, 13(3): e9960. DOI:10.1002/ece3.9960 |
| [26] |
PLAISANCE L, CALEY M J, BRAINARD R E, et al. The diversity of coral reefs: what are we missing?[J]. PLoS One, 2011, 6(10): e25026. DOI:10.1371/journal.pone.0025026 |
| [27] |
SIMON-BLECHER N, ACHITUV Y. Relationship between the coral pit crab Cryptochirus coralliodytes Heller and its host coral[J]. Journal of Experimental Marine Biology and Ecology, 1997, 215(1): 93-102. DOI:10.1016/S0022-0981(97)00002-6 |
| [28] |
VAGO R, ACHITUV Y, VAKY L, et al. Colony architecture of Millepora dichotoma Forskal[J]. Journal of Experimental Marine Biology and Ecology, 1998, 224(2): 225-235. DOI:10.1016/S0022-0981(97)00203-7 |
| [29] |
LIU P J, HSIEH H L. Burrow architecture of the spionid polychaete Polydora villosa in the corals Montipora and Porites[J]. Zoological Studies, 2000, 39(1): 47-54. |
| [30] |
GLYNN P W. Bioerosion and coral-reef growth: a dynamic balance[M]//BIRKELAND C. Life and Death of Coral Reefs. [S. l.]: Springer, 1997: 68-95.
|
| [31] |
CARBALLO J L, BAUTISTA-GUERRERO E, LEY-TE-MORALES G E. Boring sponges and the modeling of coral reefs in the east Pacific Ocean[J]. Marine Ecology Progress Series, 2008, 356: 113-122. DOI:10.3354/meps07276 |
| [32] |
BOULAY J N, HELLBERG M E, CORTÉS J, et al. Unrecognized coral species diversity masks differences in functional ecology[J]. Proceedings of the Royal Society B: Biological Science, 2014, 281(1776): 20131580. DOI:10.1098/rspb.2013.1580 |
| [33] |
ROTJAN R D, LEWIS S M. Impact of coral predators on tropical reefs[J]. Marine Ecology Progress Series, 2008, 367: 73-91. DOI:10.3354/meps07531 |
| [34] |
LIU J C W, HØEG J T, CHAN B K K. How do coral barnacles start their life in their hosts?[J]. Biology Letters, 2016, 12(6): 20160124. DOI:10.1098/rsbl.2016.0124 |
| [35] |
WULFF J L. Sponge-mediated coral reef growth and rejuvenation[J]. Coral Reefs, 1984, 3: 157-163. DOI:10.1007/BF00301960 |
| [36] |
BAK R P M, LAMBRECHTS D Y M, JOENJE M, et al. Long-term changes on coral reefs in booming populations of a competitive colonial ascidian[J]. Marine Ecology Progress Series, 1996, 133: 303-306. DOI:10.3354/meps133303 |
| [37] |
GLYNN P W. Increased survivorship in corals harboring crustacean symbionts[J]. Marine Biology Letters, 1983, 4(2): 105-111. |
| [38] |
EL-SAYED A A M, EL KHALEK A A M, AMER M A, et al. The guard coral crabs of Familes Trapeziidae and Domeciidae (Superfamily Trapezoidea) from the Egyptian Red Sea Coasts, and its asscoiated gulfs, Aqaba and Suez, Egypt[J]. Egyptian Academic Journal of Biological Sciences B.Zoology, 2019, 11(3): 43-65. DOI:10.21608/eajbsz.2019.60910 |
| [39] |
GLYNN P W. Defense by symbiotic crustacea of host corals elicited by chemical cues from predator[J]. Oecologia, 1980, 47(3): 287-290. DOI:10.1007/BF00398518 |
| [40] |
BONALDO R A, HOEY A S, BELLWOOD D R. The ecosystem roles of parrotfishes on tropical reefs[J]. Oceanography and Marine Biology: An Annual Review, 2014, 52: 81-132. |
| [41] |
GLYNN P W, ENOCHS I C. Invertebrates and Their Roles in Coral Reef Ecosystems[M]//DUBINSKY Z, STAMBLER N. Coral Reefs: An Ecosystem in Transition. Dordrecht: Springer, 2010.
|
| [42] |
ROTHANS T C, MILLER A C. A link between biologically imported particulate organic nutrients and the detritus food web in reef communities[J]. Marine Biology, 1991, 110: 145-150. DOI:10.1007/BF01313101 |
| [43] |
STIER A C, LERAY M. Predators alter community organization of coral reef cryptofauna and reduce abundance of coral mutualists[J]. Coral Reefs, 2014, 33(1): 181-191. DOI:10.1007/s00338-013-1077-2 |
| [44] |
ALLEMAND D, OSBORN D. Ocean acidification impacts on coral reefs: from sciences to solutions[J]. Regional Studies in Marine Science, 2019, 28: 100558. DOI:10.1016/j.rsma.2019.100558 |
| [45] |
SUNDAY J M, FABRICIUS K E, KROEKER K J, et al. Ocean acidification can mediate biodiversity shifts by changing biogenic habitat[J]. Nature Climate Change, 2017, 7(1): 81-85. DOI:10.1038/nclimate3161 |
| [46] |
WIDDICOMBE S, SPICER J I. Predicting the impact of ocean acidification on benthic biodiversity: what can animal physiology tell us?[J]. Journal of Experimental Marine Biology and Ecology, 2008, 366(1/2): 187-197. |
| [47] |
ENOCHS I C, MANZELLO D P. Species richness of motile cryptofauna across a gradient of reef framework erosion[J]. Coral Reefs, 2012, 31(3): 653-661. DOI:10.1007/s00338-012-0886-z |
| [48] |
PEARMAN J K, ANLAUF H, IRIGOIEN X, et al. Please mind the gap - visual census and cryptic biodiversity assessment at central Red Sea coral reefs[J]. Marine Environmental Research, 2016, 118: 20-30. DOI:10.1016/j.marenvres.2016.04.011 |
| [49] |
HUTCHINGS P A. Non colonial cryptofauna[M]// STODDART D R, JOHANNES R E. Coral Reefs: Research Methods. Paris: UNESCO, 1978: 251-263.
|
| [50] |
RANSOME E, GELLER J B, TIMMERS M, et al. The importance of standardization for biodiversity comparisons: a case study using autonomous reef monitoring structures (ARMS) and metabarcoding to measure cryptic diversity on Mo'orea coral reefs, French Polynesia[J]. PLoS One, 2017, 12(4): e0175066. DOI:10.1371/journal.pone.0175066 |
| [51] |
RANSOME E J, TIMMERS M, HARTMANN A, et al. Cryptic coral reef diversity across the Pacific assessed using autonomous reef monitoring structures and multi-omic methods[C]//American Geophysical Union, Ocean Sciences Meeting. [S. l.: s. n.], 2016.
|
| [52] |
DAVID R, UYARRA M C, CARVALHO S, et al. Lessons from photo analyses of Autonomous Reef Monitoring Structures as tools to detect (bio-)geographical, spatial, and environmental effects[J]. Marine Pollution Bulletin, 2019, 141: 420-429. DOI:10.1016/j.marpolbul.2019.02.066 |
| [53] |
PALOMINO-ALVAREZ L A, VITAL X G, CASTILLO-CUPUL R E, et al. Evaluation of the use of Autonomous Reef Monitoring Structures (ARMS) for describing the species diversity of two coral reefs in the Yucatan Peninsula, Mexico[J]. Diversity, 2021, 13(11): 579. DOI:10.3390/d13110579 |
| [54] |
PENNESI C, DANOVARO R. Assessing marine environmental status through microphytobenthos assemblages colonizing the Autonomous Reef Monitoring Structures (ARMs) and their potential in coastal marine restoration[J]. Marine Pollution Bulletin, 2017, 125(1/2): 56-65. |
| [55] |
PEARMAN J K, AYLAGAS E, VOOLSTRA C R, et al. Disentangling the complex microbial community of coral reefs using standardized Autonomous Reef Monitoring Structures (ARMS)[J]. Molecular Ecology, 2019, 28(15): 3496-3507. DOI:10.1111/mec.15167 |
| [56] |
朱文涛, 秦传新, 段丁毓, 等. 大亚湾典型珊瑚礁区域冬春季节底栖生物分布研究[J]. 海洋渔业, 2020, 42(2): 170-182. |
| [57] |
TOMOMI S, FUJITA Y. A new species of the stenopodidean shrimp genus Odontozona Holthuis, 1946 (Crustacea: Decapoda: Stenopodidea: Stenopodidae) from the Ryukyu Islands, Indo-West Pacific[J]. Zootaxa, 2018, 4550(4): 458-472. |
| [58] |
ANKER A. Pseudalpheopsis guana gen.nov., sp. nov.(Crustacea: Decapoda), a new alpheid shrimp from the British Virgin Islands, Caribbean Sea[J]. Zoological Studies, 2007, 46(4): 428-440. |
| [59] |
HEBERT P D, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society B: Biological Sciences, 2003, 270(1512): 313-321. DOI:10.1098/rspb.2002.2218 |
| [60] |
MORITZ C, CICERO C. DNA barcoding: promise and pitfalls[J]. PLoS Biology, 2004, 2(10): e354. |
| [61] |
COSTA F O, DEWAARD J R, BOUTILLIER J, et al. Biological identifications through DNA barcodes: the case of the Crustacea[J]. Canadian Journal of Fisheries and Aquatic Sciences, 2007, 64(2): 272-295. |
| [62] |
ANDÚJAR C, ARRIBAS P, YU D W, et al. Why the COI barcode should be the community DNA metabarcode for the Metazoa[J]. Molecular Ecology, 2018, 27(20): 3968-3975. |
| [63] |
ZHENG L, HE J, LIN Y, et al. 16S rRNA is a better choice than COI for DNA barcoding hydrozoans in the coastal waters of China[J]. Acta Oceanologica Sinica, 2014, 33(4): 55-76. |
| [64] |
DRUMMOND A J, NEWCOMB R D, BUCKLEY T R, et al. Evaluating a multigene environmental DNA approach for biodiversity assessment[J]. GigaScience, 2015, 4(1): 46. |
| [65] |
VALENTINI A, TABERLET P, MIAUD C, et al. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding[J]. Molecular Ecology, 2016, 25(4): 929-942. |
| [66] |
LERAY M, KNOWLTON N. DNA barcoding and metabarcoding of standardized samples reveal patterns of marine benthic diversity[J]. Proceedings of the National Academy of Sciences, 2015, 112(7): 2076-2081. |
| [67] |
BARONOI M D A, BUCHER D J. Artificial crevice habitats to assess the biodiversity of vagile macro-cryptofauna of subtidal rocky reefs[J]. Marine and Freshwater Research, 2008, 59(8): 661-670. |
| [68] |
KRAMER M J, BELLWOOD D R, BELLWOOD O. Cryptofauna of the epilithic algal matrix on an inshore coral reef, Great Barrier Reef[J]. Coral Reefs, 2012, 31(4): 1007-1015. |
| [69] |
PLAISANCE L, KNOWLTON N, PAULAY G, et al. Reef-associated crustacean fauna: biodiversity estimates using semi-quantitative sampling and DNA barcoding[J]. Coral Reefs, 2009, 28(4): 977-986. |
| [70] |
PLAISANCE L, MATTERSON K, FABRICIUS K, et al. Effects of low pH on the coral reef cryptic invertebrate communities near CO2 vents in Papua New Guinea[J]. PLoS One, 2021, 16(12): e0258725. |
| [71] |
COUNSELL C W W, DONAHUE M J. Protection mutualists affect colonization and establishment of host-associated species in a coral reef cryptofauna community[J]. Oikos, 2021, 130(10): 1823-1833. |
| [72] |
PRESTON N P, DOHERTY P J. Cross-shelf patterns in the community structure of coral-dwelling Crustacea in the central region of the Great Barrier Reef. Ⅱ. Cryptofauna[J]. Marine Ecology Progress Series, 1994, 104(1/2): 47-61. |
| [73] |
BAILEY-BROCK J, BROCK R, KAM A, et al. Anthropogenic disturbance on shallow cryptofaunal communities in a marine life conservation district on Oahu, Hawaii[J]. International Review of Hydrobiology, 2007, 92(3): 291-300. |
| [74] |
CECCARELLI D M, JONES G P, MCCOOK L J. Territorial damselfishes as determinants of the structure of benthic communities on coral reefs[J]. Oceanography and Marine Biology, 2001, 39: 355-389. |
| [75] |
KRAMER M J, BELLWOOD D R, TAYLOR R B, et al. Benthic Crustacea from tropical and temperate reef locations: differences in assemblages and their relationship with habitat structure[J]. Coral Reefs, 2017, 36(3): 971-980. |
| [76] |
HEAD C E I, BONSALL M B, KOLDEWEY H, et al. High prevalence of obligate coral-dwelling decapods on dead corals in the Chagos Archipelago, central Indian Ocean[J]. Coral Reefs, 2015, 34(3): 905-915. |
| [77] |
MACDONALD K S, RIÍOS R, DUFFY J E. Biodiversity, host specificity, and dominance by eusocial species among sponge-dwelling alpheid shrimp on the Belize Barrier Reef[J]. Diversity and Distributions, 2006, 12(2): 165-178. |
| [78] |
RICHARD V P, STANHOPE M J, SHIVJI M S. Island endemism, morphological stasis, and possible cryptic speciation in two coral reef, commensal Leucothoid amphipod species throughout Florida and the Caribbean[J]. Biodiversity and Conservation, 2012, 21: 343-361. |
| [79] |
YANG H S, KIM T, LEE K T, et al. Use of autonomous reef monitoring structures to monitor changes in the marine environment in Jeju, South Korea: a brief review[J]. Ocean Science Journal, 2023, 58(2): 17. |



