2. 广西大学生命科学与技术学院,广西南宁 530004
2. College of Life Science and Technology, Guangxi University, Nanning, Guangxi, 530004, China
结核分枝杆菌(Mycobacterium tuberculosis,MTB)是结核病的致病菌,是最成功的胞内病原菌。近年来,随着卡介苗对结核病预防能力的下降以及艾滋病感染人数的增加,结核病在全球范围内的发病率呈现逐渐上升的趋势。世界卫生组织发布的《2022年全球结核病报告》指出2020-2021年全球结核病发病率增加了3.6%,我国结核病发病率为5.5%,病死率为4.0%[1, 2]。结核病形势如此严峻,主要是因为人们对结核分枝杆菌的致病性与抗逆性的机制了解不够。因此,深入了解结核分枝杆菌的致病性与抗逆性的分子机理可为结核病的防治提供理论基础。
1 结核分枝杆菌在不同理化条件下的抗逆性结核分枝杆菌具有极强的逆境生存能力,包括抗干燥、抗酸碱、抗氧化与抗化学染料等。作为最成功的胞内病原菌,结核分枝杆菌面临着巨噬细胞内的氧化压力、还原压力、酸性条件与铁营养胁迫等逆境。
1.1 结核分枝杆菌对氧化压力的耐受巨噬细胞内NADPH氧化酶和黄嘌呤氧化酶的催化反应会产生超氧化物(O2-),而超氧化物会进一步转换为过氧化物、次氯酸盐、羟自由基和过氧亚硝酸盐。这些超氧化物及其衍生物会破坏细菌的多种细胞组分进而导致细菌死亡[3]。结核分枝杆菌具有非常强的抗氧化生长能力,因而可以在巨噬细胞中长期存活。
结核分枝杆菌内存在两种应对氧化还原压力的缓冲物,分别为分枝硫醇(Mycothiol,MSH)和麦角硫因(Ergothioneine,EGT)。氧化型分枝硫醇(Mycothione, MSSM)可以还原为MSH以应对环境的氧化压力。与MSH不同,EGT多以氧化态的形式存在,以应对还原压力[4]。EGT的氧化还原电位是-60 mV,而MSH的氧化还原电位大于-200 mV,不同电位的缓冲物可以应对不同的氧化压力[5]。
第二信使c-di-GMP通过下游受体转录因子LtmA和HpoR来调控耻垢分枝杆菌(Mycobacterium smegmatis)的抗氧化生长(图 1)。c-di-GMP分别通过以下3条途径共同调控hpoR操纵子进而影响分枝杆菌的抗氧化能力:(1)c-di-GMP刺激LtmA对hpoR操纵子表达的正调控;(2)c-di-GMP解除HpoR对其自身所在基因簇的抑制作用;(3)c-di-GMP促进LtmA与HpoR发生物理上的相互作用,这将进一步增强LtmA的DNA结合活性,并降低HpoR对hpoR操作子的抑制作用[6, 7]。
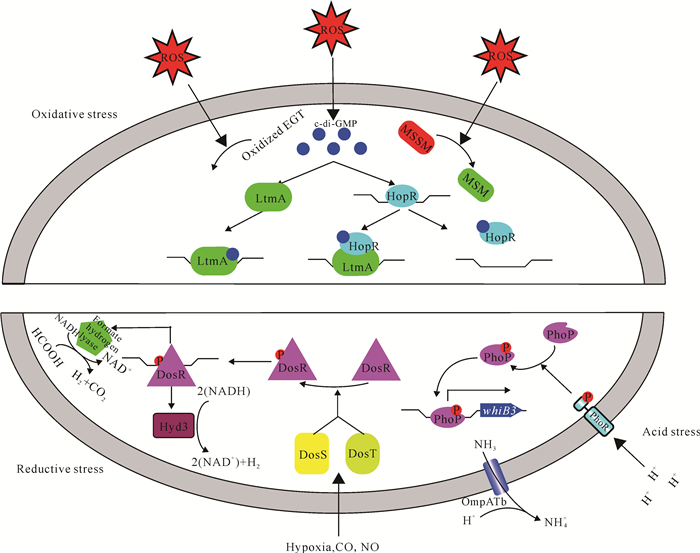
|
| 图 1 结核分枝杆菌在氧化应激、还原应激、酸应激下的应对策略 Fig. 1 MTB′s response strategies under oxidative stress, reductive stress, and acidic stress |
结核分枝杆菌中多种酶类也参与了抗氧化应激的调控,如超氧化物歧化酶(Superoxide Dismutase,SOD)、过氧化氢酶(Catalase-peroxidase,KatG)、硫氧还蛋白还原酶(Thioredoxin Reductase,TrxR)、烷基过氧化氢还原酶(Alkyl hydroperoxide reductase,AhpC),以及烷基氢过氧化物还原酶E(Alkyl hydroperoxide reductase E,AhpE)[3]。
1.2 结核分枝杆菌对缺氧环境的耐受结核分枝杆菌侵染宿主后,宿主肺部会产生肉芽肿。随后,肉芽肿内部会出现干酪样坏死,导致结核分枝杆菌处于缺氧状态[8]。缺氧会导致细胞在氧化磷酸化的过程中丧失电子的最终受体,引发还原物的积累,最终导致细菌发生还原应激[8]。缺氧会影响结核分枝杆菌的细胞生理状态,包括细胞生长受损、蛋白质聚集、蛋白质折叠异常、蛋白质合成受到抑制等[5]。
分枝杆菌的DosRST系统可以响应还原应激(图 1)。DosS与DosT感受到缺氧信号后使DosR发生磷酸化修饰,而DosR可以调控 narX、nark2、fdxA、nrdZ等50多种还原应激相关基因的表达[9]。DosS和DosT的GAF结构域可以与血红素(Heme)共价连接,是感知细胞氧化水平的重要结构。DosS和DosT的激酶活性在氧化状态下受到抑制。然而,当细胞内氧分压降低时,伴侣分子会夺取DosS和DosT的氧气,进而活化其激酶活性。此外,DosS与DosT也可以与NO、CO结合并将其本身锁定在活化状态[5]。另一项研究表明,c-di-GMP可以与DosR结合从而促进DosS对DosR的磷酸化修饰,该研究表明DosRST系统可能参与氧化应激[10]。
还原胁迫必然导致结核分枝杆菌的代谢重塑。在缺氧环境下,最终电子受体缺乏导致NADH和NADPH在细胞中堆积,是引发细菌代谢重塑的重要原因。乙醛酸支路是一种以琥珀酸为最终产物的代谢通路,在还原应激中发挥着重要作用[11]。异柠檬酸裂解酶(Isocitrate Lyases,ICL)可以以丙酰辅酶A为底物合成琥珀酸。在缺氧的环境下,琥珀酸参与ATP的合成与稳定膜电位[11, 12]。结核分枝杆菌的糖代谢由糖酵解途径向磷酸戊糖途径转移,同时甘油三酯合成增加而分枝菌酸(Mycolic Acid,MA)合成降低,琥珀酸脱氢酶、丙氨酸脱氢酶、脯氨酸脱氢酶等多种初级脱氢酶表达量增加[12, 13]。
在代谢重塑的过程中,DosRST对NADH/NADPH的消耗发挥了一定的作用。例如,DosR可以刺激Hyd2产生氢气进而转移电子;DosR可以调控假定的甲酸氢裂解酶(Formate hydrogen lyase),该酶可以氧化甲酸酯并消耗NADH;DosR可以通过调控亚硝酸盐/硝酸盐转运体编码基因 narK2X的表达来消耗NADH[5]。
1.3 结核分枝杆菌对酸性环境的耐受酸性环境不利于细菌的生长,这是由于细胞内外的高浓度质子梯度会影响物质的跨膜运输,另外低pH值也会影响蛋白质的活性,因此结核分枝杆菌进化出一系列策略以应对吞噬体内的酸性环境。
PhoPR调节子由组氨酸激酶传感器PhoR和PhoP组成,PhoR可以感知酸性环境并诱导组氨酸磷酸化,随后该磷酸基团被转移到PhoP的天冬氨酸上,进而活化PhoR (图 1)[14]。PhoPR参与调控150多种基因的表达,包括控制氧化还原的相关基因、ESX-1分泌系统的相关基因、细胞膜脂质合成的相关基因等。值得注意的是,PhoPR双组分系统只在致病菌株中参与调控 whiB3,暗示着该通路可能与结核分枝杆菌的致病性有关[14, 15]。whiB3编码的WhiB3参与调控脂质合成代谢和氧化还原代谢基因的表达,以响应低pH值的环境压力[16]。
对于结核分枝杆菌,酸应激和还原应激引起的代谢变化相似,主要表现为还原性柠檬酸循环的增加导致结核分枝杆菌对外分泌琥珀酸增多。如果添加硝酸盐作为电子的受体则会导致琥珀酸分泌减少[17]。与缺氧型还原应激不同的是,即使给低pH值环境下的结核分枝杆菌提供充足的氧气和甘油,依旧会导致其酸性生长停滞[15]。另外,结核分枝杆菌的OmpATb在应对酸胁迫方面发挥了一定的作用,OmpATb可以通过介导氨的分泌进而中和酸性环境[18]。
1.4 结核分枝杆菌在铁胁迫下的耐受铁与分枝杆菌的致病性高度相关,其作为一种辅助因子参与细菌的电子传递、能量代谢与DNA合成等过程。
细菌依赖于铁载体(Fe3+螯合剂)来摄取铁,结核分枝杆菌的铁载体包括分枝菌素(Mycobactin)与羧基分枝菌素(Carboxymycobactin)。由于疏水性的差异,易溶于水的羧基分枝菌素会被结核分枝杆菌分泌至细胞外去获取铁,而分枝菌素则锚定在外膜上[19]。MmpL4/S4和MmpL5/S5参与了铁载体的分泌,而IrtA/IrtB参与了铁-羧基分枝菌素的输入[19]。宿主的脂运载蛋白2 (Lipocalin 2,LCN2)具有噬铁蛋白的活性,其通过结合铁载体阻止结核分枝杆菌摄取铁从而抑制细菌生长,而结核分枝杆菌可以通过分泌c-di-GMP,竞争性结合LCN2进而抑制噬铁蛋白的抗菌活性(图 2)[20]。

|
| 图 2 结核分枝杆菌在铁胁迫下的应对策略 Fig. 2 MTB′s response strategies under iron stress |
在哺乳动物中,70.0%的铁与血红素辅基结合,这限制了铁载体获取铁的数量。因此,分枝杆菌中还存在依赖血红素的铁摄取系统[21]。有研究提出结核分枝杆菌的血红素获取模型:血红蛋白(Haemoglobin,Hb)释放的血红素被PPE36/PE22捕获,并由PPE62运输进入结核分枝杆菌的周质,DppA将血红素传递至DppBCD并使之跨越内膜,最后进入细胞质的血红素被MhuD降解,并释放铁(图 2)[22]。有研究构建了结核分枝杆菌ΔmbtB、ΔmbtBΔmmpL3、ΔmbtBΔrv0203,在以Fe3+作为铁的唯一来源时,三者的生长情况均正常。而在以血红素作为铁的唯一来源时,后两者相对于ΔmbtB表现出生长缺陷,这提示MmpL3和Rv0203都参与了血红素的摄取[21]。
铁与分枝杆菌的氧化应激密切相关。一方面,缺铁会导致SOD与过氧化氢酶的活性降低,使细胞无法及时处理活性氧(Reactive Oxygen Species,ROS)进而导致氧化损伤[23]。另一方面,Fe3+催化的芬顿反应会产生多种ROS进而引发氧化应激,因此结核分枝杆菌需要动态调控铁的摄取与存储[23]。IdeR是调控结核分枝杆菌铁摄取的关键蛋白,与Fe3+结合的IdeR具有DNA结合活性,可以动态调控结核分枝杆菌体内游离Fe3+的浓度。当Fe3+浓度较高时,IdeR与铁载体合成基因mbtA-J的启动子结合,通过抑制mbtA-J表达来抑制铁载体的合成[23]。IdeR与Lsr2竞争性结合铁储存基因bfrA的启动子,进而促进bfrA的表达从而促进铁的储存(图 2)[24]。此外,IdeR调控多种功能蛋白基因的表达,包括转运蛋白基因 rv0282 - 0284,脂质代谢的相关基因rv1344 - 1345、rv1347,PE/PPE家族成员基因rv0285 - 0286、rv2123、mmpL4与mmpS4 [25]。
2 结核分枝杆菌的耐药性解决结核分枝杆菌的耐药性问题是治疗结核病的关键,根据《2022年全球结核病报告》,2021年新增45万利福平耐药患者,比2020年增加了3.1%,全球耐药结核病治愈率较低,仅为60.0%[1, 2]。异烟肼(Isoniazid,INH)、利福平(Rifampin,RIF)、乙胺丁醇(Ethambutol,EMB)、吡嗪酰胺(Pyrazinamide,PZA)、链霉素(Streptomycin,STR)是治疗结核病的一线药物,文章总结了结核分枝杆菌产生耐药性的关键基因(表 1),深入研究结核分枝杆菌的耐药机制可以为新型抗结核药物研发提供参考和指导。
| 抗生素 Antibiotics |
耐药相关基因 Antibiotic resistance genes |
基因功能/基因注释 Gene function/Gene annotation |
| INH | katG(rv1908c) | Catalase-peroxidase |
| ahpC(rv2428) | Alkyl hydroperoxide reductase subunit | |
| ndh(rv1854c) | NADH dehydrogenase | |
| inhA(rv1484) | NADH-dependent enoyl-[ACP] reductase | |
| nat(rv3566c) | Arylamine N-acetyltransferase | |
| RIF | rpoB(rv0667) | DNA-directed RNA polymerase subunit beta |
| lipX(rv1169c) | Lipase | |
| EMB | embB(rv3795) | Arabinosyltransferase B |
| embR(rv1267c) | Transcriptional regulator | |
| PZA | pncA(rv2043c) | Pyrazinamidase/nicotinamidase |
| rpsA(rv1630) | 30S ribosomal protein S1 | |
| panD(rv3601c) | Aspartate 1-decarboxylase | |
| clpC1(rv3596c) | ATP-dependent protease ATP-binding subunit | |
| STR | rpsL(rv0682) | 30S ribosomal protein S12 |
| rrs(rvnr01) | 16S ribosomal RNA | |
| gidB(rv3919c) | 16S rRNA (guanine(527)-N(7))-methyltransferase |
2.1 异烟肼
异烟肼是一种一线抗结核药物。进入结核分枝杆菌体内的异烟肼由KatG将异烟肼还原成异烟酸,而异烟酸又可以与NAD+反应产生异烟酸-NAD+复合物,该复合物可以与InhA发生作用,抑制InhA的活性[26]。InhA是一种烯酰基载脂蛋白还原酶,是脂肪酸合酶Ⅱ的组成部分,InhA活性受到抑制后会导致细胞壁的重要组分分枝菌酸的合成受阻,最终导致结核分枝杆菌细胞壁组分缺失而引起细菌死亡[26, 27]。
结核分枝杆菌产生异烟肼耐药性的因素有很多,但临床上分离的结核分枝杆菌对异烟肼耐药主要是katG基因突变所致。之前的研究报道了katG基因中存在的多个不同位点的突变介导结核分枝杆菌产生异烟肼耐药,其中315位丝氨酸突变为苏氨酸使KatG的异烟肼结合位点产生空间位阻,从而限制了异烟肼的作用效果[28]。ahpC编码一种烷基氢过氧化物还原酶,ahpC表达水平的上调可能会在一定程度上补偿由katG表达下调或突变而引发的氧化损伤[29]。ndh编码一种NADH脱氢酶,ndh表达上调会导致细菌细胞内NADH水平升高,可能会抑制异烟酸-NAD+复合物的形成,从而抑制异烟肼的作用[30]。此外,inhA启动子的突变也可能导致异烟肼耐药,突变体的inhA表达水平增加引起分枝菌酸的合成量增加,这就补偿了异烟肼对分枝菌酸合成的抑制作用[30]。nat编码的NAT酶可以结合异烟肼并使其发生乙酰化,导致异烟肼无法被活化,从而限制了异烟肼的作用[31]。
除此之外,还有oxyR、kasB、ini、efpA、fadE等多种基因的突变也可能会导致结核分枝杆菌对异烟肼产生耐药性[32]。
2.2 利福平利福平属于利福霉素类抗生素,对革兰氏阳性菌具有较强的抑制作用,而对革兰氏阴性菌的抑制作用较弱。利福平、利福喷汀、利福布汀都属于利福霉素类抗生素,其中利福平是最典型且常见的抗结核药物。
利福平主要作用于结核分枝杆菌RNA聚合酶的β亚基,通过形成空间位阻导致RNA的生物合成受阻。研究发现,利福平不会影响RNA前2 nt的合成,当RNA合成到第3 nt时,利福平会阻碍RNA的进一步延伸[33]。
rpoB是编码RNA聚合酶β亚基的基因。绝大多数的利福平耐药性是rpoB突变所致,这些突变集中在507-533氨基酸残基区间,该区间被称为利福平抗性决定区(Rifampicin-Resistance-Determining Region,RRDR)[34]。进一步研究发现,RRDR区间中最常见的突变是516、526、531位密码子突变[35]。
除了rpoB的突变外,细菌细胞壁通透性改变也是其产生利福平耐药性的重要原因[36]。分枝杆菌的PE11是一种脂酶,其变化可影响质膜的脂质构成[37]。结核分枝杆菌中PE11增加会提高细菌对利福平的抗性[36]。
2.3 乙胺丁醇分枝杆菌的细胞壁外层含有大量的分枝菌酸,这些分枝菌酸与细胞壁内侧的阿拉伯半乳聚糖(Arabinogalactan,AC)共价相连形成分枝菌酸-聚阿拉伯半乳糖-肽聚糖(mycolyl-Arabinogalactan-Peptidoglycan,mAGP),脂阿拉伯甘露聚糖(Lipoarabinomannan,LAM)、脂甘露聚糖(Lipomannan,LM)等特异性脂质存在于分枝菌酸与AC之间,分枝杆菌的这些复杂结构可以与宿主发生相互作用[38, 39]。emb操纵子包含embA、embB、embC,前两者被证明与AC的合成有关,embC则与LAM的合成有关[39]。
乙胺丁醇通过抑制EmbB与EmbC,进而抑制AC与LAM的生物合成,这限制了mAGP的形成从而使得分枝杆菌细胞壁的完整性缺失[40]。因此,分枝杆菌的乙胺丁醇耐药机制主要与emb操纵子有关。
EmbR是调控emb操纵子的转录调控因子,EmbR在被PknH磷酸化后调控emb操纵子中相关基因的表达水平。PknH对EmbR的磷酸化水平增加,以及EmbR与emb操纵子结合能力增强都是分枝杆菌产生EMB耐药性的原因[41]。其中,embB发生突变是分枝杆菌发生乙胺丁醇耐药的主要原因。embB中存在一段被称之为乙胺丁醇抗性决定区(Ethambutol Resistance-Determining Region,ERDR)的区域,包括306、406、497位密码子。这些位点的突变有可能导致EmbB蛋白结构发生改变,因此乙胺丁醇缺失靶标,降低了其杀菌作用[42]。
2.4 吡嗪酰胺吡嗪酰胺是一种在酸性环境下对结核分枝杆菌具有抑菌活性的药物。因此,吡嗪酰胺在对潜伏在巨噬细胞中的结核分枝杆菌具有较好的抑菌效果[26]。吡嗪酰胺的作用机制存在较大的争议,目前公认的观点是吡嗪酰胺可以由吡嗪酰胺酶(Pyrazinamidase,PncA)转化为吡嗪酸(Pyrazinoic Acid,POA),但POA的作用机制仍有争议[43]。主流观点认为,POA在酸性环境下发生质子化并转化为HPOA,随后HPOA进入分枝杆菌体内,使得其pH值降低,破坏细胞质子梯度进而抑制分枝杆菌ATP的生物合成并影响酶的活性[44]。还有研究指出,吡嗪酰胺可能通过抑制30S核糖体的RpsA蛋白活性进而影响细菌的翻译过程[45]。PanD是一种天冬氨酸脱羧酶,在泛酸的生物合成中起重要作用。ClpC1可以识别PanD的降解标签进而使之降解,而POA可以改变PanD的构象并使其降解标签暴露。POA通过降解PanD进而抑制分枝杆菌泛酸的合成,最终导致乙酰辅酶A合成受阻[43]。
pncA编码PncA,该基因的突变是细菌产生吡嗪酰胺耐药性的重要原因。pncA的突变以错义突变为主,主要集中于3-17、61-85和132-142位点[44]。目前也发现RpsA蛋白的438位丙氨酸突变会导致吡嗪酰胺耐药,这可能是由于RpsA的突变影响其与吡嗪酰胺的互作[46]。panD与clpC1的突变也会导致结核分枝杆菌产生吡嗪酰胺耐药性,这可能是由于突变影响了POA与PanD的结合并抑制了其对PanD的降解[43]。
此外,rspA、gpsl、mas/ppsA-E、tap、iprG、fadD2也可能与吡嗪酰胺耐药有关[43]。
2.5 链霉素链霉素属于氨基糖苷类抗生素,其作用位点是30S核糖体。链霉素通过抑制tRNA与核糖体结合进而抑制翻译过程。rrs和rpsL分别编码核糖体蛋白S12、16S rRNA,这些都是链霉素的靶点。rrs的513、517、906、907位点突变和rpsL的128、263位精氨酸突变都会导致细菌产生链霉素耐药性[47]。值得注意的是,16S rRNA的甲基转移酶的编码基因gidB的突变也会引发细菌的链霉素耐药性,这可能是GidB改变了16S rRNA的甲基化位点所导致的耐药性[48]。
2.6 结核分枝杆菌的多药耐药机制结核分枝杆菌对β-内酰胺类、喹诺酮类、大环内酯类等临床上常见的抗生素可能具有多药耐药性,其机制与结核分枝杆菌的氧化还原应激以及药物外排泵密切相关。
whiB7是whiB3的旁系同源物,whiB3对于结核分枝杆菌的酸耐受性和氧化还原耐受性起重要的作用,而whiB7编码的WhiB7则介导了结核分枝杆菌对常见抗生素药物的耐药性。目前已经发现WhiB7可以通过诱导Eis、Tap、Erm等多种蛋白质的表达而影响细菌的耐药性[49]。WhiB7正调控SigA的表达,SigA正调控Eis的表达,表达量增加的Eis可以通过乙酰化氨基糖苷类抗生素进而提高细菌的耐药性[49, 50]。Tap是一种药物外排泵,Tap表达量的变化可以影响细菌对β-内酰胺类、大环内酯类、喹诺酮类抗生素的耐药性。Erm则是一种核糖体RNA甲基转移酶,其介导了细菌对大环内酯类药物的耐药性[51]。
一些参与调控细菌氧化还原应激的物质也与细菌的耐药性相关。ICLs参与调控细菌的还原应激与酸应激。有研究表明,异烟肼、利福平、链霉素的耐药性与ICLs有关,其机制可能是ICLs使底物进入乙醛酸循环,这降低了NADH的量并减少活性氧中间体(Reactive Oxygen Intermediates,ROIs)进而引发耐药[52]。分枝杆菌的MSH系统可以影响利福平、红霉素、阿奇霉素、万古霉素等多种药物的耐药性[53]。
药物外排也是影响结核分枝杆菌多药耐药性的重要因素。结核分枝杆菌的Rv0849、Rv1218c、Rv1258c、Rv3065与β-内酰胺类抗生素的耐药性相关,Rv1747、Rv0341、Rv0343、Rv2942、Rv0933与异烟肼的耐药性相关,Rv2936、Rv2937、Rv2938与四环素、链霉素、乙胺丁醇的耐药性相关[54]。
3 结核分枝杆菌的致病性与免疫性结核分枝杆菌可以侵染巨噬细胞并在巨噬细胞中长期存活,在该过程中致病因子发挥了重要作用。多种细胞因子可以活化巨噬细胞并使其杀死胞内的结核分枝杆菌,然而结核分枝杆菌也存在一些机制以应对这些细胞因子的胁迫。结核病会诱导肉芽肿的产生,对于结核性肉芽肿的作用目前尚有争议,但已经确定的是结核分枝杆菌可以利用肉芽肿帮助其增殖。
3.1 结核分枝杆菌的致病因子结核分枝杆菌中具有多种致病因子,主要包括脂质、蛋白质与多糖。致病因子可以通过不同的途径对宿主造成损伤,包括诱导肉芽肿的形成、抑制免疫细胞的免疫反应、形成生物被膜等。
脂质是结核分枝杆菌的主要致病因子,这些脂质多为细胞的表面成分。分枝菌酸可以诱导巨噬细胞发生脂滴积累进而形成泡沫巨噬细胞,结核分枝杆菌可以在这些脂滴中存活并长时间潜伏于巨噬细胞中[55]。另外MA还与结核分枝杆菌生物被膜的形成密切相关[55]。海藻糖-6, 6′-二分枝菌酸酯(6, 6′-trehalose dimycolate,TDM)又被称为索状因子,是结核分枝杆菌细胞壁中最丰富的糖酯。TDM可以通过抑制中性粒细胞的迁移、抑制磷脂小泡的融合、降低NAD的水平来发挥其致病作用[56]。LAM在抑制宿主免疫反应的过程中起着关键作用,LAM可以通过消除巨噬细胞产生的氧自由基、抑制蛋白质激酶C的活性、阻断IFN-γ的合成来保证结核分枝杆菌的存活[57]。
多种致病蛋白在结核分枝杆菌侵入宿主细胞的过程中发挥作用。纤连蛋白结合蛋白(Fibronectin binding protein,Fbp)通过结合纤连蛋白促使结核分枝杆菌黏附到细胞的黏膜表面,进而侵入宿主细胞[56]。哺乳动物细胞进入蛋白(Mammalian cell entry,Mce)在结核分枝杆菌侵入巨噬细胞以及抑制免疫反应的过程中发挥着重要作用,Mce3C与巨噬细胞表面整合素结合,促进结核分枝杆菌黏附并侵入巨噬细胞[58]。另外Mce2E与Mce3E可以通过抑制MAPK信号转导通路来抑制细胞因子IL-6与TNF-α的表达[58]。
致病蛋白还可以影响巨噬细胞对结核分枝杆菌的吞噬和消化作用。SapM、PknG、EIS、EspB等蛋白参与了对宿主自噬过程的调节[59]。PtpA、TlyA、LpdC、SapM等蛋白抑制了吞噬体的成熟[59]。
3.2 结核分枝杆菌逃逸吞噬溶酶体对其的分解结核分枝杆菌可以逃逸吞噬溶酶体对其的降解作用,主要机制包括抑制吞噬溶酶体的成熟与破坏吞噬体。
巨噬细胞吞噬结核分枝杆菌后在胞内形成新生吞噬体,新生吞噬体随后与早期内体、晚期内体、溶酶体融合形成可以杀灭细菌的吞噬溶酶体。Rab5是一种GTP酶,一方面Rab5可以招募PI3K并催化合成PI3P,随后Rab5进一步与PI3P共同招募EEA1使新生吞噬体与早期内体融合(图 3)[60]。另一方面,Rab5与PI3P可以招募Rab7,而Rab7可以介导吞噬溶酶体的形成[60]。囊泡ATP酶在吞噬体后期酸化的过程中发挥重要的作用,其可以通过水解ATP来降低吞噬体的pH值,进而激活多种酸性水解酶[61]。

|
| 图 3 巨噬细胞吞噬并分解结核分枝杆菌与结核分枝杆菌逃逸吞噬溶酶体的机制 Fig. 3 Mechanism of macrophages phagocytizing and degrading MTB versus MTB′s escape from phagolysosomal degradation |
结核分枝杆菌可以通过多种途径抑制吞噬溶酶体的成熟,甘露糖基脂阿拉伯甘露聚糖(Mannosylated Lipoarabinomannan,ManLAM)可以降低胞内的Ca2+浓度,进而抑制钙调蛋白的活性,阻断钙调蛋白激活PI3K的通路,最终导致吞噬体PI3P数量降低[62]。分枝杆菌也可以分泌磷酸酶SamP,这使得PI3P发生去磷酸化从而抑制其功能[62]。PtpA是一种酪氨酸磷酸酶,可以使Vps33b发生去磷酸化进而抑制Rab7的活性。此外,PtpA还可以结合囊泡ATP酶的H亚基,进而阻止囊泡ATP酶的转运与活化(图 3)[63, 64]。
酸性环境可以激活结核分枝杆菌的ESX-1分泌系统,该分泌系统通过分泌Esx-A/ESAT-6与Esx-B/CFP10引发吞噬体膜损伤,进而导致结核分枝杆菌逃逸(图 3)[63]。
3.3 结核分枝杆菌影响细胞因子对免疫细胞的调控宿主中的细胞因子IL-12、TNF-α和IFN-γ在对抗结核分枝杆菌过程中起关键作用。首先,IL-12诱导Th0细胞分化成为Th1细胞并使之分泌IFN-γ,随后IL-2和IFN-γ促使NK细胞和CTL细胞活化并分泌TNF-α,最后TNF-α诱导巨噬细胞分泌IL-12,这是产生细胞因子分泌的级联放大循环(图 4)[65]。IFN-γ可以通过激活巨噬细胞中的一氧化氮合酶产生活性氮中间体(Reactive Nitrogen Intermediates,RNI)以促进溶酶体成熟来应对结核分枝杆菌。TNF-α则可以通过激活CTL、促进单核细胞成熟为DC或巨噬细胞、辅助建立趋化因子梯度、促进肉芽肿产生来对抗结核分枝杆菌[63, 65]。
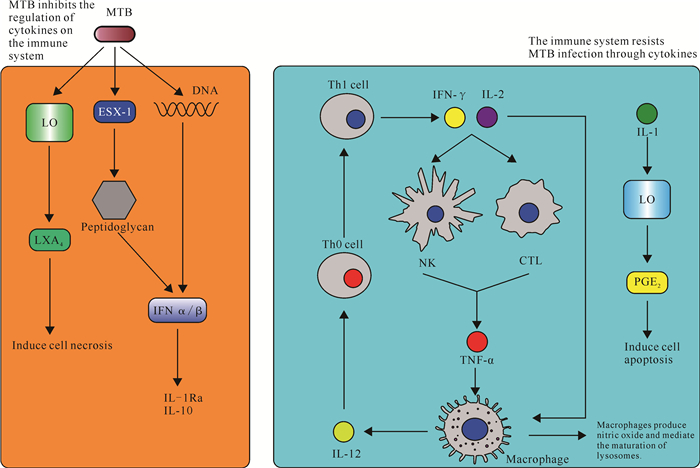
|
| 图 4 结核分枝杆菌与宿主细胞因子的分子交互及其影响 Fig. 4 Molecular interactions and implications of MTB with host immunoregulatory cytokines |
IL-1分为IL-1α与IL-1β。IL-1的缺陷会让小鼠对结核分枝杆菌更敏感,IL-1由炎症单核巨噬细胞、炎症DC和中性粒细胞产生。研究证明,IL-1的主要功能是募集中性粒细胞,但中性粒细胞对结核分枝杆菌的具体作用机制尚不清楚[66]。
结核分枝杆菌的ESX-1分泌系统可以通过分泌肽聚糖来激活Nod2受体并诱导IFN α/β分泌,细菌的DNA也可以诱导IFN α/β分泌[67]。IFNAR1可以被IFN α/β激活并介导细胞分泌IL-1Ra (IL-1的拮抗剂)与IL-10来抑制免疫反应(图 4)。LAM可以诱导IL-10分泌[67, 68]。研究发现IL-10缺陷的小鼠对结核分枝杆菌的抵抗能力更强,IL-10还会诱导动物结核病的再激活[69]。IL-10可以抑制促炎细胞因子IFN-γ、TNF-α、IL-12的产生,并通过抑制MHCⅡ来阻断T细胞的活化(图 4)。在体外实验中,IL-10可以抑制Th1与Th2亚群,因此IL-10对于结核分枝杆菌抵抗宿主细胞因子胁迫至关重要[69]。
花生四烯酸及其代谢产物也在结核分枝杆菌的免疫调控中起重要作用。脂氧合酶(Lipoxygenase,LO)与环氧合酶(Cyclooxygenase,COX)都以花生四烯酸为底物,竞争性合成脂氧素A4(Lipoxin A4,LXA4)与前列腺素E2(Prostaglandin E2,PGE2)。LXA4可以诱导细胞坏死,而PGE2则诱导细胞凋亡。在细胞凋亡的过程中会产生凋亡小体,这些凋亡小体阻碍了结核分枝杆菌的释放并可以被DC吞噬用于抗原呈递[70]。然而,细胞坏死后也会直接释放结核分枝杆菌,这促进了结核分枝杆菌的感染[70]。细胞因子可以调控凋亡和坏死的发生。例如,TNF-α会同时介导凋亡与坏死,IL-1β则可以通过PGE2来促进细胞凋亡,IFN α/β则可以诱导LXA4进一步诱导细胞坏死,并通过诱导分泌IL-1Ra来抑制IL-1β[71]。结核分枝杆菌通过诱导LXA4的产生并抑制COX2 mRNA积累进而使细胞内PGE2水平降低,抑制PGE2介导的膜修复作用,导致线粒体膜损伤发生积累,进而引起其通透性改变并引发线粒体膜电位丧失,这进一步导致ROS积累与ATP合成停滞并最终引发细胞坏死(图 4)[70]。
3.4 肉芽肿对结核分枝杆菌感染的影响结核性肉芽肿又称结核结节,是结核病典型的病理学特征,随着结核分枝杆菌数量的增加肉芽肿会被破坏并产生空洞。结核性肉芽肿的核心是富含脂质的干酪样坏死区域,该区域为结核分枝杆菌提供营养,在其周围具有一层致密的白细胞壁可以阻止结核分枝杆菌的扩散[72]。肉芽肿内部存在着不同的巨噬细胞,包括上皮样巨噬细胞、泡沫巨噬细胞、多核巨噬细胞(Multinucleated Giant Cells,MGCs),而在巨噬细胞周围存在T、B淋巴细胞[63]。
TDM可以保护结核分枝杆菌不被巨噬细胞杀死,同时会介导结核性肉芽肿的发生[73]。经典模型认为肉芽肿的形成产生了物理阻隔,可抑制病原体的传播与增殖。然而,研究发现,缺乏ESX-1的结核分枝杆菌感染宿主后,其肉芽肿表现出发育不良并伴有感染减弱,该结果暗示了肉芽肿的形成可能反而加快了结核分枝杆菌的感染[63]。另外一项研究表明,结核分枝杆菌可以诱导巨噬细胞周围的上皮细胞分泌金属蛋白酶9 (Matrix Metalloproteinase 9,MMP9),而MMP9进一步募集巨噬细胞进入肉芽肿[74]。随后这些未被感染的巨噬细胞识别了被感染巨噬细胞信号后并将之吞噬,这导致被感染的巨噬细胞数量增加,最后被感染的巨噬细胞发生迁移并在其他部位形成新的肉芽肿。
结核分枝杆菌通过细胞表面的邻苯二甲酸二纤维素酯(Phthiocerol Dimycoceroserate,PDIM)来掩盖病原体相关分子模式(Pathogen-Associated Molecular Patterns,PAMPs)。PDIM优先招募不依赖MyD88的巨噬细胞,由于这种巨噬细胞不易产生诱导性一氧化氮合酶(inducible Nitric Oxide Synthase,iNOS),这使得结核分枝杆菌更容易在此类巨噬细胞中存活,因此,在早期肉芽肿中的结核分枝杆菌更易增殖[75]。
肉芽肿的干酪样坏死中心的氧分压较低并且营养较为匮乏。为了应对这种不利环境,部分结核分枝杆菌进入休眠状态,该过程由DosRST系统参与调控。进入休眠期的结核分枝杆菌对异烟肼和吡嗪酰胺具有耐药性,因为这两种抗生素只作用于复制期的结核分枝杆菌[76]。结核分枝杆菌具有5个rpf基因,rpfD缺失被证明可以延缓结核分枝杆菌恢复,此外Rpf可以促进结核病患者痰液中的结核分枝杆菌以及冻干的牛结核分枝杆菌(Mycobacterium bovis)的复苏。一项在兔子体内的研究表明,使用免疫抑制剂会使结核分枝杆菌的rpf转录水平上调,这说明理化因素与免疫因素共同影响了结核分枝杆菌的细胞周期[72, 77]。
4 展望结核分枝杆菌的致病性极强,对不同的胁迫条件具有较高的耐受性。在抗氧化方面,除了存在MSH与EGT两套系统外,c-di-GMP可以通过调控结核分枝杆菌的LtmA与HpoR进而调控其抗氧化水平。在抗还原方面,DosRST系统响应还原应激,同时结核分枝杆菌也可以通过代谢重塑应对还原应激。酸应激也可以导致还原应激,PhoPR可以调控 whiB3以适应酸应激,同时细菌也可以通过代谢重塑适应酸应激。结核分枝杆菌可以通过铁载体来摄取内环境中游离的铁,也可以摄取血红素中的铁。结核分枝杆菌体内铁浓度过高或过低都可能导致ROS的产生,IdeR参与细菌体内游离铁浓度的动态调控。
在耐药性方面,结核分枝杆菌产生异烟肼耐药性主要是由于细菌的katG突变,ahpC、ndh、inhA、nat以及外排泵等多种基因也影响细菌的异烟肼耐药性。分枝杆菌的利福平耐药性主要是rpoB的突变所致。此外,外排泵与细胞膜通透性的改变也影响细菌的利福平耐药性。乙胺丁醇通过抑制EmbB与EmbC来发挥作用,embB突变是结核分枝杆菌对乙胺丁醇产生耐药的主要原因。吡嗪酰胺的作用机制尚有争议,目前主流观点认为其耐药性与 pncA、panD、clpC1、rpsA的突变有关。链霉素属于氨基糖苷类抗生素,细菌产生链霉素耐药性与rrs、rpsl、gidB的突变有关。最后,whiB7、MSH、ICLs、外排泵影响结核分枝杆菌对多抗生素共同的耐药性。
在致病性与免疫性方面,MA、TDM、LAM、Fbp、Mce等致病分子参与了结核分枝杆菌的致病。结核分枝杆菌可以通过ManLAM与PtpA来阻止吞噬溶酶体的成熟,还可以通过分泌Esx-A/ESAT-6与Esx-B/CFP10来破坏吞噬体。宿主的IL-1、IL-12、TNF-α、IFN-γ对于抵抗结核分枝杆菌起关键作用,结核分枝杆菌可以通过诱导宿主产生IL-10、IL-1Ra、IFN α/β来抑制免疫反应。此外,结核分枝杆菌还可以通过诱导LXA4的分泌使巨噬细胞坏死。肉芽肿对结核分枝杆菌的作用尚有争议,一方面肉芽肿可以形成物理屏障阻碍结核分枝杆菌传播,另一方面结核分枝杆菌可以通过诱导上皮细胞分泌MMP9使巨噬细胞聚集并加速结核分枝杆菌的增殖。
虽然之前的研究使人们对结核分枝杆菌的致病性与抗逆性有一定的了解,但目前仍有一些问题有待解决。处于休眠期的结核分枝杆菌对周围环境具有较强的耐受性同时也产生了耐药性,解析结核分枝杆菌休眠与觉醒的机制有助于研发针对休眠菌的药物,防止结核病的复发。另外,仍然没有明确多种免疫细胞在抵抗结核分枝杆菌中的作用机制,这使得人们无法据此研发疫苗和药物。
一些新型技术为攻克结核病提供了帮助,重组BCG疫苗是结核分枝杆菌疫苗开发的一个热点,重组BCG疫苗通过过表达免疫优势抗原进而增强疫苗的保护力。2021年,DeepMind推出的AlphaFold2是生物信息学中的里程碑,基于AlphaFold2对蛋白质结构的精准预测,可以通过虚拟筛选与反向对接技术寻找抗结核新药或解析抗结核药物的作用机制[78]。
通过对结核分枝杆菌的抗逆性与致病性研究,为新型抗结核药物的研发提供理论依据,有助于解决日益严重的结核分枝杆菌耐药性问题。
| [1] |
宋敏, 陆普选, 方伟军, 等. 2022年WHO全球结核病报告: 全球与中国关键数据分析[J]. 新发传染病电子杂志, 2023, 8(1): 87-92. |
| [2] |
World Health Organization. Global tuberculosis report 2022[EB/OL]. (2022-10-19)[2023-04-12]. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2022.
|
| [3] |
NAMBI S, LONG J E, MISHRA B B, et al. The oxidative stress network of Mycobacterium tuberculosis reveals coordination between radical detoxification systems[J]. Cell Host & Microbe, 2015, 17(6): 829-837. |
| [4] |
SAINI V, CUMMING B M, GUIDRY L, et al. Ergothioneine maintains redox and bioenergetic homeostasis essential for drug susceptibility and virulence of Mycobacterium tuberculosis[J]. Cell Reports, 2016, 14(3): 572-585. DOI:10.1016/j.celrep.2015.12.056 |
| [5] |
MAVI P S, SINGH S, KUMAR A, et al. Reductive stress: new insights in physiology and drug tolerance of Mycobacterium[J]. Antioxidants & Redox Signaling, 2020, 32(18): 1348-1366. |
| [6] |
LI W H, LI M, HU L H, et al. HpoR, a novel c-di-GMP effective transcription factor, links the second messenger's regulatory function to the mycobacterial antioxidant defense[J]. Nucleic Acids Research, 2018, 46(7): 3595-3611. DOI:10.1093/nar/gky146 |
| [7] |
LI W H, HU L H, XIE Z W, et al. Cyclic di-GMP integrates functionally divergent transcription factors into a regulation pathway for antioxidant defense[J]. Nucleic Acids Research, 2018, 46(14): 7270-7283. DOI:10.1093/nar/gky611 |
| [8] |
VIA L E, LIN P L, RAY S M, et al. Tuberculous granulomas are hypoxic in guinea pigs, rabbits, and nonhuman primates[J]. Infection and Immunity, 2008, 76(6): 2333-2340. DOI:10.1128/IAI.01515-07 |
| [9] |
RUSTAD T R, SHERRID A M, MINCH K J, et al. Hypoxia: a window into Mycobacterium tuberculosis latency[J]. Cellular Microbiology, 2009, 11(8): 1151-1159. DOI:10.1111/j.1462-5822.2009.01325.x |
| [10] |
HU Q B, ZHANG J X, CHEN Y, et al. Cyclic di-GMP co-activates the two-component transcriptional regulator DevR in Mycobacterium smegmatis in response to oxidative stress[J]. Journal of Biological Chemistry, 2019, 294(34): 12729-12742. DOI:10.1074/jbc.RA119.008252 |
| [11] |
EOH H, RHEE K Y. Multifunctional essentiality of succinate metabolism in adaptation to hypoxia in Mycobacterium tuberculosis[J]. Proceedings of the National Academy of Sciences, 2013, 110(16): 6554-6559. DOI:10.1073/pnas.1219375110 |
| [12] |
CHANG D P S, GUAN X L. Metabolic versatility of Mycobacterium tuberculosis during infection and dormancy[J]. Metabolites, 2021, 11(2): 88. DOI:10.3390/metabo11020088 |
| [13] |
BERNEY M, COOK G M. Unique flexibility in energy metabolism allows mycobacteria to combat starvation and hypoxia[J]. PLoS One, 2010, 5(1): e8614. DOI:10.1371/journal.pone.0008614 |
| [14] |
FENG L P, CHEN S Y, HU Y B, et al. PhoPR positively regulates whiB3 expression in response to low pH in pathogenic mycobacteria[J]. Journal of Bacteriology, 2018, 200(8): e00766-17. |
| [15] |
BAKER J J, DECHOW S J, ABRAMOVITCH R B, et al. Acid fasting: modulation of Mycobacterium tuberculosis metabolism at acidic pH[J]. Trends in Microbiology, 2019, 27(11): 942-953. DOI:10.1016/j.tim.2019.06.005 |
| [16] |
MEHTA M, RAJMANI R S, SINGH A, et al. Mycobacterium tuberculosis WhiB3 responds to vacuolar pH-induced changes in mycothiol redox potential to modulate phagosomal maturation and virulence[J]. Journal of Biological Chemistry, 2016, 291(6): 2888-2903. DOI:10.1074/jbc.M115.684597 |
| [17] |
BAKER J J, ABRAMOVITCH R B. Genetic and metabolic regulation of Mycobacterium tuberculosis acid growth arrest[J]. Scientific Reports, 2018, 8(1): 4168. DOI:10.1038/s41598-018-22343-4 |
| [18] |
SONG H H, HUFF J, JANIK K, et al. Expression of the ompATb operon accelerates ammonia secretion and adaptation of Mycobacterium tuberculosis to acidic environments[J]. Molecular Microbiology, 2011, 80(4): 900-918. DOI:10.1111/j.1365-2958.2011.07619.x |
| [19] |
CHAO A, SIEMINSKI P J, OWENS C P, et al. Iron acquisition in Mycobacterium tuberculosis[J]. Chemical Reviews, 2019, 119(2): 1193-1220. DOI:10.1021/acs.chemrev.8b00285 |
| [20] |
LI W H, CUI T, HU L H, et al. Cyclic diguanylate monophosphate directly binds to human siderocalin and inhibits its antibacterial activity[J]. Nature Communications, 2015, 6(1): 8330. DOI:10.1038/ncomms9330 |
| [21] |
JONES C M, NIEDERWEIS M. Mycobacterium tuberculosis can utilize heme as an iron source[J]. Journal of Bacteriology, 2011, 193(7): 1767-1770. DOI:10.1128/JB.01312-10 |
| [22] |
MITRA A, KO Y H, CINGOLANI G, et al. Heme and hemoglobin utilization by Mycobacterium tuberculosis[J]. Nature Communications, 2019, 10(1): 4260. DOI:10.1038/s41467-019-12109-5 |
| [23] |
RODRIGUEZ G M, SMITH I. Mechanisms of iron regulation in mycobacteria: role in physiology and virulence[J]. Molecular Microbiology, 2003, 47(6): 1485-1494. DOI:10.1046/j.1365-2958.2003.03384.x |
| [24] |
KURTHKOTI K, TARE P, PAITCHOWDHURY R, et al. The mycobacterial iron-dependent regulator IdeR induces ferritin (bfrB) by alleviating Lsr2 repression[J]. Molecular Microbiology, 2015, 98(5): 864-877. DOI:10.1111/mmi.13166 |
| [25] |
RODRIGUEZ G M, VOSKUIL M I, GOLD B, et al. IdeR, an essential gene in Mycobacterium tuberculosis: role of IdeR in iron-dependent gene expression, iron metabolism, and oxidative stress response[J]. Infection and Immunity, 2002, 70(7): 3371-3381. DOI:10.1128/IAI.70.7.3371-3381.2002 |
| [26] |
葛赛, 宋欣怡, 姜惠跃, 等. 结核分枝杆菌药物作用机理及耐药机制研究进展[J/OL]. 海南医学院学报, 2022, 15: 1-13[2023-04-03]. https://doi.org/10.13210/j.cnki.jhmu.20221128.002.
|
| [27] |
VILCHEÈZE C, MORBIDONI H R, WEISBROD T R, et al. Inactivation of the inhA-encoded fatty acid synthase Ⅱ (FAS Ⅱ) enoyl-acyl carrier protein reductase induces accumulation of the FAS Ⅰ end products and cell lysis of Mycobacterium smegmatis[J]. Journal of Bacteriology, 2000, 182(14): 4059-4067. DOI:10.1128/JB.182.14.4059-4067.2000 |
| [28] |
BERTRAND T, EADY N A J, JONES J N, et al. Crystal structure of Mycobacterium tuberculosis catalase-peroxidase[J]. Journal of Biological Chemistry, 2004, 279(37): 38991-38999. DOI:10.1074/jbc.M402382200 |
| [29] |
WILSON T, DE LISLE G W, MARCINKEVICIENE J A, et al. Antisense RNA to ahpC, an oxidative stress defence gene involved in isoniazid resistance, indicates that AhpC of Mycobacterium bovis has virulence properties[J]. Microbiology, 1998, 144(10): 2687-2695. DOI:10.1099/00221287-144-10-2687 |
| [30] |
VILCHÈZE C, WEISBROD T R, CHEN B, et al. Altered NADH/NAD+ ratio mediates coresistance to isoniazid and ethionamide in mycobacteria[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(2): 708-720. DOI:10.1128/AAC.49.2.708-720.2005 |
| [31] |
PAYTON M, GIFFORD C, SCHARTAU P, et al. Evidence towards the role of arylamine N-acetyltransferase in Mycobacterium smegmatis and development of a specific antiserum against the homologous enzyme of Mycobacterium tuberculosis[J]. Microbiology, 2001, 147(12): 3295-3302. DOI:10.1099/00221287-147-12-3295 |
| [32] |
UNISSA A N, SUBBIAN S, HANNA L E, et al. Overview on mechanisms of isoniazid action and resistance in Mycobacterium tuberculosis[J]. Infection, Genetics and Evolution, 2016, 45: 474-492. DOI:10.1016/j.meegid.2016.09.004 |
| [33] |
CAMPBELL E A, KORZHEVA N, MUSTAEV A, et al. Structural mechanism for rifampicin inhibition of bacterial RNA polymerase[J]. Cell, 2001, 104(6): 901-912. DOI:10.1016/S0092-8674(01)00286-0 |
| [34] |
MBOOWA G, NAMAGANDA C, SSENGOOBA W. Rifampicin resistance mutations in the 81 bp RRDR of rpoB gene in Mycobacterium tuberculosis clinical isolates using Xpert® MTB/RIF in Kampala, Uganda: a retrospective study[J]. BMC Infectious Diseases, 2014, 14: 481. DOI:10.1186/1471-2334-14-481 |
| [35] |
ZAW M T, EMRAN N A, LIN Z. Mutations inside rifampicin-resistance determining region of rpoB gene associated with rifampicin-resistance in Mycobacterium tuberculosis[J]. Journal of Infection and Public Health, 2018, 11(5): 605-610. DOI:10.1016/j.jiph.2018.04.005 |
| [36] |
XU G, LIU H C, JIA X D, et al. Mechanisms and detection methods of Mycobacterium tuberculosis rifampicin resistance: the phenomenon of drug resistance is complex[J]. Tuberculosis, 2021, 128: 102083. DOI:10.1016/j.tube.2021.102083 |
| [37] |
RASTOGI S, SINGH A K, PANT G, et al. Down-regulation of PE11, a cell wall associated esterase, enhances the biofilm growth of Mycobacterium tuberculosis and reduces cell wall virulence lipid levels[J]. Microbiology, 2017, 163(1): 52-61. DOI:10.1099/mic.0.000417 |
| [38] |
ESCUYER V E, LETY M A, TORRELLES J B, et al. The role of the embA and embB gene products in the biosynthesis of the terminal hexaarabinofuranosyl motif of Mycobacterium smegmatis arabinogalactan[J]. Journal of Biological Chemistry, 2001, 276(52): 48854-48862. DOI:10.1074/jbc.M102272200 |
| [39] |
BERG S, STARBUCK J, TORRELLES J B, et al. Roles of conserved proline and glycosyltransferase motifs of EmbC in biosynthesis of lipoarabinomannan[J]. Journal of Biological Chemistry, 2005, 280(7): 5651-5663. DOI:10.1074/jbc.M411418200 |
| [40] |
GOUDE R, AMIN A G, CHATTERJEE D, et al. The arabinosyltransferase EmbC is inhibited by ethambutol in Mycobacterium tuberculosis[J]. Antimicrobial Agents and Chemotherapy, 2009, 53(10): 4138-4146. DOI:10.1128/AAC.00162-09 |
| [41] |
XIANG X H, GONG Z, DENG W Y, et al. Mycobacterial ethambutol responsive genes and implications in antibiotics resistance[J]. Journal of Drug Targeting, 2021, 29(3): 284-293. DOI:10.1080/1061186X.2020.1853733 |
| [42] |
BROSSIER F, SOUGAKOFF W, BERNARD C, et al. Molecular analysis of the embCAB locus and embR gene involved in ethambutol resistance in clinical isolates of Mycobacterium tuberculosis in France[J]. Antimicrobial Agents and Chemotherapy, 2015, 59(8): 4800-4808. DOI:10.1128/AAC.00150-15 |
| [43] |
GOPAL P, GRVBER G, DARTOIS V, et al. Pharmacological and molecular mechanisms behind the sterilizing activity of pyrazinamide[J]. Trends in Pharmacological Sciences, 2019, 40(12): 930-940. DOI:10.1016/j.tips.2019.10.005 |
| [44] |
ZHANG Y, MITCHISON D. The curious characteristics of pyrazinamide: a review[J]. The International Journal of Tuberculosis and Lung Disease, 2003, 7(1): 6-21. |
| [45] |
NJIRE M, TAN Y J, MUGWERU J, et al. Pyrazinamide resistance in Mycobacterium tuberculosis: review and update[J]. Advances in Medical Sciences, 2016, 61(1): 63-71. DOI:10.1016/j.advms.2015.09.007 |
| [46] |
SHI W L, CUI P, NIU H X, et al. Introducing RpsA point mutations Δ438A and D123A into the chromosome of Mycobacterium tuberculosis confirms their role in causing resistance to pyrazinamide[J]. Antimicrobial Agents and Chemotherapy, 2019, 63(6): e02681-18. |
| [47] |
JAGIELSKI T, IGNATOWSKA H, BAKUŁA Z, et al. Screening for streptomycin resistance-conferring mutations in Mycobacterium tuberculosis clinical isolates from Poland[J]. PLoS One, 2014, 9(6): e100078. DOI:10.1371/journal.pone.0100078 |
| [48] |
WONG S Y, LEE J S, KWAK H K, et al. Mutations in gidB confer low-level streptomycin resistance in Mycobacterium tuberculosis[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(6): 2515-2522. DOI:10.1128/AAC.01814-10 |
| [49] |
BURIAN J, RAMÓN-GARCÍA S, HOWES C G, et al. WhiB7, a transcriptional activator that coordinates physiology with intrinsic drug resistance in Mycobacterium tuberculosis[J]. Expert Review of Anti-Infective Therapy, 2012, 10(9): 1037-1047. DOI:10.1586/eri.12.90 |
| [50] |
BURIAN J, YIM G, HSING M, et al. The mycobacterial antibiotic resistance determinant WhiB7 acts as a transcriptional activator by binding the primary sigma factor SigA (RpoV)[J]. Nucleic Acids Research, 2013, 41(22): 10062-10076. DOI:10.1093/nar/gkt751 |
| [51] |
SHUR K V, MASLOV D A, MIKHEECHEVA N E, et al. The intrinsic antibiotic resistance to β-lactams, macrolides, and fluoroquinolones of mycobacteria is mediated by the whiB7 and tap genes[J]. Russian Journal of Genetics, 2017, 53: 1006-1015. DOI:10.1134/S1022795417080087 |
| [52] |
NAKUMAR M, NATHAN C, RHEE K Y. Isocitrate lyase mediates broad antibiotic tolerance in Mycobacterium tuberculosis[J]. Nature Communications, 2014, 5(1): 4306. DOI:10.1038/ncomms5306 |
| [53] |
NGUYEN L, THOMPSON C J. Foundations of antibiotic resistance in bacterial physiology: the mycobacterial paradigm[J]. Trends in Microbiology, 2006, 14(7): 304-312. DOI:10.1016/j.tim.2006.05.005 |
| [54] |
BLACK P A, WARREN R M, LOUW G E, et al. Energy metabolism and drug efflux in Mycobacterium tuberculosis[J]. Antimicrobial Agents and Chemotherapy, 2014, 58(5): 2491-2503. DOI:10.1128/AAC.02293-13 |
| [55] |
MARRAKCHI H, LANÉELLE M A, DAFFÉ M, et al. Mycolic acids: structures, biosynthesis, and beyond[J]. Chemistry & Biology, 2014, 21(1): 67-85. |
| [56] |
FORRELLAD M A, KLEPP L I, GIOFFRÉ A, et al. Virulence factors of the Mycobacterium tuberculosis complex[J]. Virulence, 2013, 4(1): 3-66. DOI:10.4161/viru.22329 |
| [57] |
RAJNI, RAO N, MEENA L S. Biosynthesis and virulent behavior of lipids produced by Mycobacterium tuberculosis: LAM and cord factor: an overview[J]. Biotechnology Research International, 2011, 2011: 274693. |
| [58] |
FENN K, WONG C T, DARBARI V C, et al. Mycobacterium tuberculosis uses Mce proteins to interfere with host cell signaling[J]. Frontiers in Molecular Biosciences, 2020, 6: 149. DOI:10.3389/fmolb.2019.00149 |
| [59] |
RAHLWES K C, DIAS B R, CAMPOS P C, et al. Pathogenicity and virulence of Mycobacterium tuberculosis[J]. Virulence, 2023, 14(1): 2150449. DOI:10.1080/21505594.2022.2150449 |
| [60] |
CANTON J. Phagosome maturation in polarized macrophages[J]. Journal of Leukocyte Biology, 2014, 96(5): 729-738. DOI:10.1189/jlb.1MR0114-021R |
| [61] |
KINCHEN J M, RAVICHANDRAN K S. Phagosome maturation: going through the acid test[J]. Nature Reviews Molecular Cell Biology, 2008, 9(10): 781-795. DOI:10.1038/nrm2515 |
| [62] |
BOSEDASGUPTA S, PIETERS J. Macrophage-microbe interaction: lessons learned from the pathogen Mycobacterium tuberculosis[J]. Seminars in Immunopathology, 2018, 40(6): 577-591. DOI:10.1007/s00281-018-0710-0 |
| [63] |
PHILIPS J A, ERNST J D. Tuberculosis pathogenesis and immunity[J]. Annual Review of Pathology, 2012, 7: 353-384. DOI:10.1146/annurev-pathol-011811-132458 |
| [64] |
施旭骏, 王晴岚, 高谦. 结核分枝杆菌耐酸机制的研究进展[J]. 微生物与感染, 2013, 8(3): 192-196. |
| [65] |
YASUI K. Immunity against Mycobacterium tuberculosis and the risk of biologic anti-TNF-α reagents[J]. Pediatric Rheumatology Online Journal, 2014, 12(1): 45. |
| [66] |
RAVESLOOT-CHÁVEZ M M, DIS E V, STANLEY S A. The innate immune response to Mycobacterium tuberculosis infection[J]. Annual Review of Immunology, 2021, 39: 611-637. |
| [67] |
MAYER-BARBER K D, SHER A. Cytokine and lipid mediator networks in tuberculosis[J]. Immunological Reviews, 2015, 264(1): 264-275. |
| [68] |
YUAN C H, QU Z L, TANG X L, et al. Mycobacterium tuberculosis mannose-capped lipoarabinomannan induces IL-10-producing B cells and hinders CD4+ Th1 immunity[J]. iScience, 2019, 11: 13-30. |
| [69] |
CAVALCANTI Y V N, BRELAZ M C A, NEVES J K D A L, et al. Role of TNF-alpha, IFN-gamma, and IL-10 in the development of pulmonary tuberculosis[J]. Pulmonary Tuberculosis, 2012, 2012: 745483. |
| [70] |
BEHAR S M, DIVANGAHI M, REMOLD H G. Evasion of innate immunity by Mycobacterium tuberculosis: is death an exit strategy?[J]. Nature Reviews Microbiology, 2010, 8(9): 668-674. |
| [71] |
AMARAL E P, LASUNSKAIA E B, D'IMPÉRIO-LIMA M R. Innate immunity in tuberculosis: how the sensing of mycobacteria and tissue damage modulates macrophage death[J]. Microbes and Infection, 2016, 18(1): 11-20. |
| [72] |
ULRICHS T, KAUFMANN S H E. New insights into the function of granulomas in human tuberculosis[J]. The Journal of Pathology: a Journal of the Pathological Society of Great Britain and Ireland, 2006, 208(2): 261-269. |
| [73] |
HUNTER R L, OLSEN M R, JAGANNATH C, et al. Multiple roles of cord factor in the pathogenesis of primary, secondary, and cavitary tuberculosis, including a revised description of the pathology of secondary disease[J]. Annals of Clinical & Laboratory Science, 2006, 36(4): 371-386. |
| [74] |
MCCLEAN C M, TOBIN D M. Macrophage form, function, and phenotype in mycobacterial infection: lessons from tuberculosis and other diseases[J]. Pathogens and Disease, 2016, 74(7): ftw068. |
| [75] |
CAMBIER C J, TAKAKI K K, LARSON R P, et al. Mycobacteria manipulate macrophage recruitment through coordinated use of membrane lipids[J]. Nature, 2014, 505(7482): 218-222. |
| [76] |
GENGENBACHER M, KAUFMANN S H E. Mycobacterium tuberculosis: success through dormancy[J]. FEMS Microbiology Reviews, 2012, 36(3): 514-532. |
| [77] |
VEATCH A V, KAUSHAL D. Opening Pandora's box: mechanisms of Mycobacterium tuberculosis resuscitation[J]. Trends in Microbiology, 2018, 26(2): 145-157. |
| [78] |
张强锋, 徐魁. 人工智能预测蛋白质结构[J]. 中国科学基金, 2022, 36(2): 348-349. |



