2. 宁德市富发水产有限公司, 大黄鱼育种国家重点实验室, 福建宁德 352103
2. State Key Laboratory of Large Yellow Croaker Breeding, Ningde Fufa Fisheries Co., Ltd., Ningde, Fujian, 352103, China
大黄鱼Larimichthys crocea是石首鱼科Sciaenidae中主要的经济鱼类之一,俗称黄花鱼、黄鱼、大鲜等,为暖水性集群洄游的中下层鱼类,从山东半岛以南至雷州半岛以东海域均有分布。大黄鱼为我国特有的地方性海水鱼类,素有“国鱼”之称,大黄鱼渔业曾位居中国“四大海洋渔业”之首[1]。早在20世纪50年代,国内学者就开始对大黄鱼的渔业生物学、生态学以及种质资源状况进行了持续调查。以往的研究报道通常将中国沿海的大黄鱼分为3个地理族群:岱衢族(黄东海种群),分布在黄海南部至东海中部;闽粤东族(闽粤东种群),从东海南部至珠江口;硇洲族(粤西种群),限于珠江口以西至琼州海峡以东[2-4]。经过1955-1980年3个时期的过度捕捞,中国近海大黄鱼资源从兴旺繁盛时代逐渐衰退到严重枯竭状态,早已不再形成渔汛[5]。大黄鱼生活史类型从之前的K选择演化为当前的r选择,种群结构趋向简单,资源稳定性变差。
为此,我国政府大力发展东海区的大黄鱼人工养殖和增殖放流,以减轻对野生大黄鱼的捕捞压力[3]。然而,养殖实践和人工放流容易影响鱼类种质资源状况和改变野生种群的遗传背景。基于多种分子标记的大量研究表明,东海区大黄鱼养殖群体遗传多样性处于较低水平[6-8],野生群体的种质资源整体退化严重,可供种质改良所需的遗传基础急剧减少[9]。目前有关大黄鱼粤西种群遗传背景的调查研究较少,仅见Wang等[10]、Han等[11]、Liu等[12]应用线粒体D-loop序列或SNP标记分析了粤西大黄鱼的群体遗传变异,且该种群与闽粤东种群的遗传学关系仍存有疑惑和争议。此外,东海区大黄鱼养殖个体是否对粤西种群的遗传多样性产生影响尚未知晓。因此,有必要使用其他分子标记来进一步开展东海区和粤西海域大黄鱼不同群体间的遗传学研究,以了解养殖和野生群体间的遗传特征差异以及养殖对野生群体的遗传学影响。
线粒体COI作为线粒体DNA中功能稳定而又具有较多变异的基因序列,已成为动物物种DNA条形码与系统进化、谱系地理学和遗传分化等方面研究的重要分子标记。此外,也有研究指出COI基因序列在鱼类群体遗传学研究中具有较好的解析能力,已广泛应用于鲻Mugil cephalus[13]、棘头梅童鱼Collichthys lucidus[14]、弹涂鱼科Periophthalmidae[15]等鱼类的群体遗传多样性研究。本研究通过分析大黄鱼粤西野生群体、闽粤东野生群体、东海区养殖群体的线粒体COI基因序列变异,研究不同群体间的遗传多样性、遗传分化和历史动态,以期为粤西海域和东海区大黄鱼种质资源保护提供基础资料和科学依据,亦可为养殖群体种质资源改良提供参考。
1 材料与方法 1.1 样品采集本研究所用的两个大黄鱼野生群体分别于2017年12月和2019年9月采自粤西的徐闻东岸(XWD,34尾)和硇洲岛近岸(NZD,23尾),两个养殖群体于2011年11月分别采自浙江象山岱衢族大黄鱼养殖实验基地(XSYZ,21尾)和福建宁德三都湾大黄鱼养殖渔排(NDYZ,20尾)。实验鱼经形态学鉴定后,取背部肌肉装入95%酒精溶液中,于-20 ℃保存备用。
1.2 序列扩增和测序采用传统的苯酚-氯仿法提取样品的基因组DNA。利用鱼类COI条码通用引物(FishF1、FishR1)来扩增大黄鱼线粒体COI基因序列。PCR的反应体系和扩增程序参照Wu等[16]报道的实验方法。PCR扩增产物经1.2% 琼脂糖凝胶电泳检测合格后送至苏州金唯智生物科技有限公司测序。
1.3 数据分析除上述4个群体测定的98条COI序列外,本研究还从GenBank上下载了22条闽粤东野生群体(MYD)的大黄鱼COI条码序列,其中9条序列来自福建中南部沿海(MG574434-MG574437、KX777989、KX777990)和台湾西岸(KX777985、KX777986、KX777988),13条序列来自珠江口近海(EU595167-EU595177、FJ237998、HQ564537、HQ564538)。本研究分析了包括上述5个群体共120条大黄鱼的COI条码序列。
群体COI条码序列的碱基组成、序列变异、单倍型确定、单倍型网络关系以及群体间的单倍型分布频率Exact检验和Fst值等分析参照吴仁协等[17]报道的方法。群体基因交流Nm由DnaSP 5.10软件基于Gammast值进行估算。在Arlequin Ver 3.5.1.2软件中,通过3种设定方式来进行群体分子变异分析(AMOVA):一是将5个群体归为一个组群;二是将5个群体划分为野生组群(MYD、XWD、NZD)和养殖组群(XSYZ、NDYZ);三是根据群体遗传分化Fst值将5个群体划分为3个组群(MYD、XWD、NZD,XSYZ,NDYZ)。上述分析所涉及的核苷酸进化模型采用由jModelTest V2.1.10软件运算出的DNA序列最佳替换模型(K-2P+G,G=0.876),其余参数为分析软件设置的默认参数。根据样品来源情况,将3个野生群体合并为一个野生组群(YSQ)、2个养殖群体合并为一个养殖组群(YZQ)进行历史动态分析,其中,中性检验、核苷酸不配对分布、扩张时间分析等参见吴仁协等[17]报道的方法。由于线粒体COI基因分歧速率与Cyt b基因相近。因此,本研究采用其他硬骨鱼类Cyt b基因2%每百万年的突变速率作为大黄鱼COI条码序列的突变速率,以推算群体扩张时间。
2 结果与分析 2.1 遗传多样性所分析的120条大黄鱼COI条码序列经比对后,保留共有序列长度有591 bp,共编码197个氨基酸,序列无插入和缺失或终止密码子。序列T、C、A、G平均碱基含量分别为27.3%、30.0%、22.9%、19.8%,A+T含量(50.2%)稍微高于C+G含量(49.8%)。其C+G含量在3个密码子中的分布差异较大,第1密码子的C+G含量(56.6%)明显高于第2、3密码子(分别为43.4%、49.6%)。在591 bp长的序列中共检测到35个变异位点,其中简约信息位点有13个,单碱基突变位点有22个。这些变异位点共定义了35处核苷酸替换,包括30处替换和5处颠换。
表 1显示,5个群体的单倍型多样性以NZD群体最高(h=0.877 5)、NDYZ群体最低(h=0.194 7),核苷酸多样性以XWD群体最高(π=0.003 1)、XSYZ群体最低(π=0.000 8),表现出明显的群体间差异。粤西2个野生群体(XWD、NZD)的平均单倍型多样性(h=0.841 6)稍微高于闽粤东野生群体(MYD,h=0.757 6),但二者的核苷酸多样性相等(π值均为0.002 8),表明二者的遗传多样性水平大致相当。野生组群(YSQ)的单倍型多样性和核苷酸多样性(h=0.810 8、π=0.002 9)均远高于养殖组群(YZQ,h=0.376 8、π=0.000 9),前者是后者的2-3倍。所有个体的单倍型多样性、核苷酸多样性和平均核苷酸差异数分别为0.690 6、0.002 2、1.327 3。
| 采样群体(群体简称) Sample groups (Group abbreviation) |
样品数量 Number of samples |
单倍型数量 Number of haplotypes |
多态位点数 Polymorphic loci |
单倍型多样性 Haplotype diversity |
核苷酸多样性 Nucleotide diversity |
平均核苷酸差异数 Average number of nucleotide differences |
| Min-Yuedong wild population (MYD) | 22 | 11 | 13 | 0.757 6±0.097 5 | 0.002 8±0.001 9 | 1.686 1±1.026 5 |
| East Xuwen wild population (XWD) |
34 | 17 | 23 | 0.805 7±0.069 4 | 0.003 1±0.002 0 | 1.856 8±1.090 8 |
| Naozhou Island wild population (NZD) |
23 | 13 | 15 | 0.877 5±0.060 7 | 0.002 6±0.001 8 | 1.562 2±0.966 9 |
| Xiangshan farmed population (XSYZ) | 21 | 3 | 2 | 0.495 2±0.092 4 | 0.000 8±0.000 7 | 0.460 2±0.420 9 |
| Ningde farmed population (NDYZ) |
20 | 3 | 4 | 0.194 7±0.114 5 | 0.001 0±0.000 9 | 0.591 1±0.493 8 |
| Wild group (YSQ) | 79 | 32 | 34 | 0.810 8±0.045 5 | 0.002 9±0.001 9 | 1.715 2±1.011 8 |
| Farmed group (YZQ) | 41 | 5 | 6 | 0.376 8±0.088 2 | 0.000 9±0.000 8 | 0.563 5±0.469 1 |
| Total | 120 | 34 | 35 | 0.690 6±0.047 6 | 0.002 2±0.001 6 | 1.327 3±0.831 7 |
2.2 单倍型分布
所分析的120条COI条码序列共定义34个单倍型,单倍型间的核苷酸差异数为1-9。表 2的单倍型分布表明,单倍型H1为5个群体所共享,H8、H15为3个群体所共享,H4、H7、H9、H10、H17为2个群体所共享,其余26个单倍型均为某一个群体所特有。其中单倍型H1出现频率最高,占5个群体总个数的55%,说明该单倍型是大黄鱼种群在长期进化过程中形成的较为稳定的优势基因型。2个养殖群体均只有3个单倍型,且以单倍型H1的个体数为主,分别占XSYZ、NDYZ群体个数的67%、90%;而3个野生群体的单倍型数量较多,有11-17个。单倍型中介邻接网络关系图呈现出以一个主体单倍型H1为中心、其他单倍型呈辐射状分布的星状结构(图 1),未显示出与序列来源地相对应的谱系结构。这种结构与群体发生扩张的谱系特征相一致。
| 单倍型 Haplotypes |
MYD | XWD | NZD | XSYZ | NDYZ | 总计 Total |
| H1 | 11 | 15 | 8 | 14 | 18 | 66 |
| H2 | 1 | 1 | ||||
| H3 | 1 | 1 | ||||
| H4 | 1 | 1 | 2 | |||
| H5 | 1 | 1 | ||||
| H6 | 1 | 1 | ||||
| H7 | 1 | 3 | 4 | |||
| H8 | 1 | 2 | 1 | 4 | ||
| H9 | 2 | 1 | 3 | |||
| H10 | 1 | 1 | 2 | |||
| H11 | 1 | 1 | ||||
| H12 | 1 | 1 | ||||
| H13 | 1 | 1 | ||||
| H14 | 1 | 1 | ||||
| H15 | 2 | 2 | 6 | 10 | ||
| H16 | 2 | 2 | ||||
| H17 | 1 | 1 | 2 | |||
| H18 | 1 | 1 | ||||
| H19 | 1 | 1 | ||||
| H20 | 1 | 1 | ||||
| H21 | 1 | 1 | ||||
| H22 | 1 | 1 | ||||
| H23 | 1 | 1 | ||||
| H24 | 1 | 1 | ||||
| H25 | 1 | 1 | ||||
| H26 | 1 | 1 | ||||
| H27 | 1 | 1 | ||||
| H28 | 1 | 1 | ||||
| H29 | 1 | 1 | ||||
| H30 | 1 | 1 | ||||
| H31 | 1 | 1 | ||||
| H32 | 1 | 1 | ||||
| H33 | 1 | 1 | ||||
| H34 | 1 | 1 |
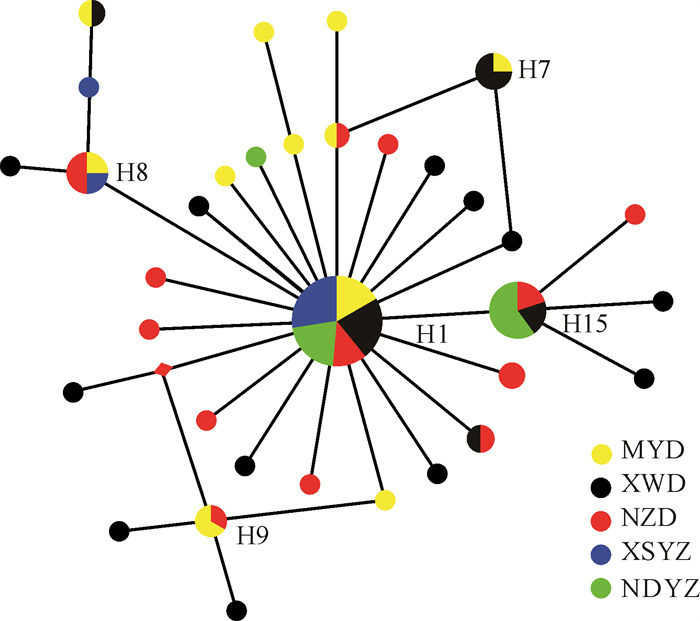
|
| The sizes of circles are proportional to haplotype frequency 图 1 大黄鱼群体线粒体COI单倍型的中介邻接网络关系 Fig. 1 Median-joining network of mitochondrial COI haplotypes in L.crocea populations |
2.3 遗传分化和历史动态
遗传分化分析结果显示(表 3),XSYZ与NDYZ、MYD群体间的Fst值具有显著性差异(Fst=0.128 5、0.086 0,P < 0.05),表明象山养殖群体与宁德养殖群体、闽粤东野生群体均存在显著的遗传分化。3个野生群体间的Fst值均为负值,说明闽粤东野生群体与粤西野生群体内个体间的遗传差异大于群体间的遗传差异水平。3个野生群体与NDYZ间的Fst值不具有显著性差异(Fst=-0.010 6-0.000 2,P>0.05),基因流分析也显示这4个群体间的Nm值较大(19.55-38.04)(表 3),表明3个野生群体与宁德养殖群体间存在遗传同质性,群体间的基因交流明显。
| 群体 Population |
MYD | XWD | NZD | XSYZ | NDYZ |
| MYD | 36.06 | 22.50 | 6.72 | 19.55 | |
| XWD | -0.009 6 | 38.04 | 17.70 | 25.28 | |
| NZD | -0.002 0 | -0.010 3 | 14.91 | 25.97 | |
| XSYZ | 0.086 0* | 0.017 7 | 0.017 4 | 4.86 | |
| NDYZ | 0.000 1 | 0.000 2 | -0.010 6 | 0.128 5* | |
| Note:* indicates statistical test P < 0.05. | |||||
3种AMOVA结果均表明大黄鱼5个群体的遗传变异绝大部分来自群体内部(占总变异的98.03%-99.05%),来自组群间或群体间的遗传变异仅占极少量(0.04%-1.25%),且统计检验差异均不显著(P=0.153-0.723 > 0.05)(表 4)。同时,AMOVA结果不支持5个群体划分为野生组群和养殖组群(Fct=0.000 4, P=0.496),也不支持根据Fst值分为3个组群的假设(Fct=0.012 5, P=0.382)。但是,Exact检验分析表明,单倍型在群体间的分布频率差异具有显著性(P < 0.001),不支持5个群体是一个随机交配种群的零假设。
| 变异来源 Source of variation |
自由度 Degree of freedom |
方差总和 Total variance |
变异组成 Variance components |
变异百分比/% Percentage of variation/% |
分化固定指数 Differentiation fixed index |
P值 P value |
| One group | ||||||
| Among populations | 4 | 3.235 | 0.006 33 | 0.95 | Fst=0.009 5 | 0.153 |
| Within population | 115 | 75.743 | 0.658 64 | 99.05 | ||
| Two groups | ||||||
| Among groups | 1 | 0.814 | 0.000 26 | 0.04 | Fct=0.000 4 | 0.496 |
| Within group | 3 | 2.421 | 0.006 18 | 0.93 | Fsc=0.009 3 | 0.205 |
| Within population | 115 | 75.743 | 0.658 64 | 98.03 | Fst=0.009 7 | 0.166 |
| Three groups | ||||||
| Among groups | 2 | 1.868 | 0.008 33 | 1.25 | Fct=0.012 5 | 0.382 |
| Within group | 2 | 1.368 | 0.000 98 | 0.15 | Fsc=0.001 5 | 0.723 |
| Within population | 115 | 75.743 | 0.658 64 | 98.61 | Fst=0.013 9 | 0.158 |
表 5显示野生组群(YSQ)的Tajima′s D检验(D=-2.274, P=0.002)和Fu′s FS检验(FS=-27.438, P= < 0.001)均为显著的负值,提示野生大黄鱼可能经历了群体扩张事件。野生组群的核苷酸不配对分布呈单峰分布(图 2),其拟合优度检验的SSD值(0.000 3)和Hri指数(0.022)均统计不显著(P=0.94、0.83)(表 5),表明所观测的野生组群核苷酸不配对分布与群体扩张模型相吻合。根据群体数量扩张参数τ值(1.30),推算出野生群体约在10.9万年前即晚更新世时期经历了群体数量扩张事件。群体扩张后、前的θ1/θ0比值为19.4,揭示了野生群体发生扩张后其有效群体数量有实质性的增大。在养殖组群(YZQ)中,Tajima′s D检验(D=-1.445, P=0.062)和Fu′s FS检验(FS=-1.506, P=0.123)均为统计检验不显著的负值(表 5),该结果与群体发生扩张的分子信号不相符。虽然养殖组群的核苷酸不配对分布的拟合优度检验结果(SSD=0.009 3、P=0.50,Hri=0.196、P=0.71)并未显著偏离群体扩张模型(表 5、图 2),但其群体扩张参数θ1/θ0比值仅为1.2,提示养殖群体的有效群体数量在其历史动态过程中变化并不明显。可见,历史动态分析结果支持野生大黄鱼经历了晚更新世的群体数量扩张,但不支持养殖大黄鱼发生群体扩张事件。
| 群体 Population |
Tajima′s D检验 Tajima′s D test |
Fu′s Fs检验 Fu′s Fs test |
数量扩张参数 Demographic expansion parameters |
拟合优度检验 Goodness of fit test |
||||||||||
| D | P | Fs | P | τ | θ0 | θ1 | SSD | PSSD | Hri | PHri | ||||
| Wild group (YSQ) | -2.274 | 0.002 | -27.438 | < 0.001 | 1.30 | 0.768 | 14.908 | 0.000 3 | 0.94 | 0.022 | 0.83 | |||
| Farmed group (YZQ) | -1.445 | 0.062 | -1.506 | 0.123 | 2.75 | 0.486 | 0.581 | 0.009 3 | 0.50 | 0.196 | 0.71 |
|||

|
| The histogram is the observation distribution, and the curve is the expected distribution under the population expansion model. 图 2 大黄鱼线粒体COI单倍型的核苷酸不配对分布 Fig. 2 Nucleotide mismatch distribution of mitochondrial COI haplotypes of L.crocea |
3 讨论 3.1 大黄鱼野生和养殖群体的遗传多样性比较
本研究的大黄鱼野生群体和养殖群体显示出不同的遗传多样性模式,前者为高h(>0.5)、低π(<0.005)类型,后者为低h、低π类型。这种差异在以往的大黄鱼群体线粒体COI序列变异研究中也有相同的报道[18, 19]。历史动态分析结果支持野生大黄鱼经历了晚更新世的群体扩张事件,但不支持养殖大黄鱼发生群体扩张事件。因此,野生大黄鱼的这种遗传多样性模式很可能是因其在种群数量动态演化史中发生了群体扩张, 即经历了一个由较小的有效群体快速扩张成一个较大群体的过程,该过程产生的许多新突变可增加单倍型多样性,但因缺乏足够的进化时间来积累核苷酸序列变异,最终形成高h、低π模式[17]。养殖大黄鱼呈现低h、低π模式可能是因为其群体经历过近期瓶颈效应或由单一、少数群体所产生的奠基者效应引起的[19]。与野生群体相比,本研究的养殖大黄鱼群体遗传多样性下降较为明显(前者是后者的2-3倍)。这种下降与基于其他分子标记研究东海区大黄鱼养殖群体遗传变异的分析结果相一致[6, 9, 20-22]。以往研究普遍认为用于养殖繁育的亲本数量少、遗传背景单一,由此引起的遗传瓶颈效应及其伴随发生的遗传漂变和近交衰退等作用,是导致东海区养殖大黄鱼种质资源退化的主要原因[7, 20-22]。此外,养殖过程中的人为选择也可能是造成养殖大黄鱼群体遗传多样性下降的另一个因素。人工选育通常筛选目标群体的优良性状,随着世代培育及代际遗传,其定向选择势必增强,容易引起群体中某些稀有基因丧失和杂合子缺失,从而造成子代群体遗传多样性的丢失[23]。可见,不同进化力量在大黄鱼野生和养殖群体中的作用机制导致了二者遗传多样性特征的显著差异。
与其他研究报道的大黄鱼线粒体COI序列遗传多样性相比,如吕泗野生群体(h=0.915、π=0.004 12)和浙闽2个养殖群体(h=0.503、π=0.001 53)[18],以及黄东海8个野生群体(h=0.842 5、π=0.003 454)、福建2个养殖群体(h=0.463 5、π=0.001 1)、江浙4个养殖群体(h、π均为0)[19],本研究的野生和养殖群体的遗传多样性均低于上述研究结果(因共同亲本繁育而来的江浙4个养殖群体除外),提示所检测的大黄鱼种质资源状况不容乐观。然而,Han等[11]报道了黄东海的大黄鱼野生群体线粒体D-loop序列具有较高的遗传多样性(h=0.989 5-1.000 0、π=0.021 2-0.024 6)。除了分子标记、样品数量、采样时间的差异引起不同研究的差别之外,一个不争的事实是过度捕捞导致中国近海的大黄鱼野生群体资源早已处于严重枯竭状态。而养殖群体通常来源于少数野生大黄鱼繁育的后代个体,有效亲本数量较小导致种群出现遗传瓶颈效应和近交衰退[20, 21]。因此,本研究未能在野生群体和养殖群体中检测到较为丰富的遗传多样性属于情理之中。
与东海区大黄鱼不同的是,粤西大黄鱼尚无长期的人工养殖或种苗放流增殖历史。因此,影响遗传多样性的瓶颈效应、遗传漂变和近亲杂交等不利因素在大黄鱼粤西种群遗传中的作用应当不明显,粤西种群可能保存相对较多的稀有等位基因。然而,本研究的粤西野生群体遗传多样性水平与闽粤东野生群体相当,明显低于上述报道的黄海野生群体的遗传多样性(h=0.915-0.946、π=0.004 12-0.004 70)[18, 19]。究其原因可能有2个:一是粤西种群是大黄鱼3个地理种群中产卵群体数量、资源量及捕捞产量、栖息地面积等均最少的一个种群[2, 4],其有效群体数量相对较少,所蕴含的遗传多样性丰富度不如黄东海种群;二是浙闽不仅是野生大黄鱼的主要产区,也是主要的养殖区和人工放流区,其养殖逃逸及放流苗种数量较多[19, 22],东海区的这些养殖个体可能通过中国沿岸流进入南海,与南海的野生大黄鱼发生基因交流,从而影响了粤西大黄鱼野生群体的遗传多样性。研究表明养殖鱼类可对野生群体产生遗传稀释或基因渐渗,导致野生群体遗传多样性的降低[23]。
与弹涂鱼科(h= 0.913-0.987、π=0.002-0.005)、鲚属Coilia(h= 0.556-0.933、π=0.002-0.005)、石斑鱼属Epinephelus(h= 0.884-0.955、π=0.002)、银鲳Pampus argenteus(h= 0.619、π=0.002)等鱼类[17]相比,本研究的粤西野生大黄鱼群体遗传多样性(h=0.757 6-0.805 7、π=0.002 8-0.003 1)处于中等水平。这与Wang等[10]和Han等[11]基于线粒体D-loop序列的研究结果相一致。可见,粤西大黄鱼野生群体的遗传多样性水平虽比不上南黄海野生群体,但该群体仍保持了一定程度的遗传变异,具有较强的环境适应能力、生存能力和进化潜力。此外,考虑到粤西大黄鱼的群体资源开发和利用程度,以及受人工养殖影响程度均是大黄鱼3个地理种群中相对较轻的一个,粤西种群可能是中国近海大黄鱼非常宝贵的一个天然种质资源库,可视为大黄鱼增养殖繁育群体的一个潜在的适合补充群体。因此,必须对大黄鱼粤西种群进行有效的资源保护和科学管理,以维护其良好的种质资源状况。
3.2 大黄鱼不同来源群体间的遗传分化Fst值和Exact检验分析表明,大黄鱼5个群体间存在一定程度的遗传差异,主要是象山养殖群体与宁德养殖群体、闽粤东野生群体之间具有显著的遗传分化。这种遗传分化在其他研究报道的大黄鱼群体间也有发现。例如,谌微等[19]的线粒体COI序列分析显示黄东海大黄鱼的养殖和野生两大组群间具有显著的遗传分化,但组群内群体间无遗传差异;Wang等[7]的SSR标记研究也表明大黄鱼养殖和野生群体之间存在明显的遗传结构。研究表明,养殖过程中发生的奠基者效应、随机遗传漂变以及人工选育是造成养殖和野生群体间遗传分化的主要原因,这在其他经济鱼类的群体遗传变异研究中已有广泛的报道,如大西洋鲑Salmo salar[24]、草鱼Ctenopharyngodon idella[25]、斜带石斑鱼Epinephelus coioides[26]等。由于遗传多样性较低的养殖鱼苗大规模放流到野生种群的生活海区中,一旦产生基因交流就会改变野生种群的遗传组成和适合度,从而降低野生种群的生存能力和适应环境能力[26],这对大黄鱼野生群体资源的保护是非常不利的。因此,对于目前东海区大规模开展的大黄鱼苗种增殖放流工程需谨慎实施[27],尤其是在当前养殖群体遗传多样性已出现明显下降之时。此外,本研究发现象山养殖与宁德养殖群体间存在中等偏高程度的遗传分化水平。这两个养殖群体均来源于东海区大黄鱼的主养区,也都经历过长期的人工选育,该结果表明人工选育的强度和作用在东海区大黄鱼的主要养殖群体中已呈现明显的遗传效应。这提示在大黄鱼养殖过程中,除了注重亲本来源外,对养殖实践中的人为选择问题也应当给予足够的重视。
本研究检测到宁德养殖群体与3个野生群体之间具有高度的遗传同质性,即大黄鱼闽粤东种群与粤西种群间存在明显的基因交流。这在以往的大黄鱼群体线粒体DNA序列研究中也有发现。Han等[11]研究表明黄海、东海、南海的4个大黄鱼野生群体间D-loop序列分歧很小,可视为同一个种群。Wang等[8]联合COI和Cyt b序列分析显示黄海、东海5个大黄鱼野生群体的2个谱系遗传分化并不显著。大黄鱼在分布区缺乏显著的地理谱系结构可能反映了冰期后大黄鱼在中国近海陆架区发生重新殖化的历史进程。本研究的历史动态分析表明大黄鱼野生群体经历了晚更新世的群体扩张,且群体间只检测到一个单倍型谱系。考虑到目前野生大黄鱼遗传多样性最丰富的群体位于黄海南部,其分布中心位于东海区。可推测中国近海大黄鱼种群的冰期避难所应该只位于东海的深水区,在晚更新世冰期之后,来自冰期避难所的大黄鱼孑留群体向黄、东、南海陆架区扩张,扩散后新建立的群体尚未在迁移和遗传漂变之间取得平衡[17, 28],这可能是大黄鱼闽粤东种群与粤西种群间遗传差异不显著的主要历史原因。这种历史动态进程在中国近海鱼类种群遗传研究中具有普遍性[17, 28, 29]。
大黄鱼是一种近岸洄游性的海洋鱼类,幼体浮游期较长(3-4周)、游泳能力较强[1],具有长距离扩散能力[11]。对该物种而言,东海和南海北部的海洋环境缺乏明显的隔离因素[15, 17]。因此,东海区的养殖逃逸和人工放流大黄鱼有可能借助中国沿岸流进入南海北部,从而促进两海域间大黄鱼群体的基因交流。通常群体间Nm>1就能发挥遗传均质化作用,可有效抑制由遗传漂变引起的遗传分化[30]。只要群体的每一个世代有一个个体能有效迁移到另一个群体的繁育群体中,就可阻止群体间因为遗传漂变或选择作用而产生的遗传分化[29]。因此,养殖逃逸、人工放流以及大黄鱼较强的扩散能力不但使东海区大黄鱼养殖和野生群体间具有很高的遗传一致性,还可能促进了大黄鱼闽粤东种群和粤西种群间发生遗传均质化。
考虑到本研究所分析的群体数量、样品涵盖范围以及分子标记存在的不足之处,本研究还难以对中国近海大黄鱼的种群划分问题进行深入的讨论。有必要在后续研究中收集更多的大黄鱼野生和养殖群体样品,采用分辨力更高的分子标记(如D-loop、SSR、SNP等)[31, 32],综合探讨中国近海大黄鱼的种群划分和遗传资源保护。
4 结论本研究的线粒体COI序列分析表明,大黄鱼野生群体和养殖群体具有完全不同的遗传多样性特征,这种差异反映了不同进化力量在大黄鱼群体遗传中的作用机制。晚更新世的群体扩张事件是形成现有野生群体遗传多样性的重要因素,而养殖繁育亲本数量少和人工选育则可能是导致目前养殖群体遗传多样性下降的主要原因。大黄鱼粤西野生群体的遗传多样性处于中等水平,具有一定程度的遗传变异,可视为大黄鱼增养殖繁育群体的一个潜在的适合补充群体。当前的养殖实践和人工选育可能导致东海区大黄鱼群体间产生显著的遗传分化,包括养殖和野生群体间、不同养殖群体间。晚更新世冰期后的大黄鱼历史殖化事件以及东海区大黄鱼养殖个体向南海北部的扩散,可能促使大黄鱼闽粤东种群(包括野生和养殖)和粤西种群具有遗传同质性。本研究结果提示,中国近海大黄鱼种质资源总体状况不容乐观,对于当前东海区大规模开展的大黄鱼增殖放流工程需谨慎实施,养殖实践、增殖放流、人工选育等因素对养殖大黄鱼群体遗传特征产生了重要影响,这应当引起高度重视和需要长期、持续的研究监测,以保护好大黄鱼这一我国特有的宝贵鱼类资源。
| [1] |
徐鹏, 柯巧珍, 苏永全, 等. 大黄鱼种质资源保护与利用现状及建议[J]. 水产学报, 2022, 46(4): 674-682. |
| [2] |
田明诚, 徐恭昭, 余日秀. 大黄鱼Pseudosciaena crocea (Richardson)形态特征的地理变异与地理种群问题[J]. 海洋科学集刊, 1962, 2: 79-97. |
| [3] |
LIU M, DE MITCHESON Y S. Profile of a fishery collapse: why mariculture failed to save the large yellow croaker[J]. Fish and Fisheries, 2008, 9: 219-242. DOI:10.1111/j.1467-2979.2008.00278.x |
| [4] |
张其永, 洪万树, 杨圣云, 等. 大黄鱼地理种群划分的探讨[J]. 现代渔业信息, 2011, 26(2): 3-8. |
| [5] |
李立华, 严小军, 王健鑫, 等. 浙江近海大黄鱼和带鱼产量的长期变化及资源评估: 基于历史统计数据分析[J]. 浙江海洋大学学报(自然科学版), 2022, 41(1): 63-69. |
| [6] |
张祖兴, 李明云. 大黄鱼种质资源研究进展[J]. 水产科学, 2006, 25(7): 376-378. DOI:10.3969/j.issn.1003-1111.2006.07.014 |
| [7] |
WANG L, SHI X F, SU Y Q, et al. Loss of genetic diversity in the cultured stocks of the large yellow croaker, Larimichthys crocea, revealed by microsatellites[J]. International Journal of Molecular Sciences, 2012, 13: 5584-5597. DOI:10.3390/ijms13055584 |
| [8] |
WANG L, SHI X F, SU Y Q, et al. Genetic divergence and historical demography in the endangered large yellow croaker revealed by mtDNA[J]. Biochemical Systematics and Ecology, 2013, 46: 137-144. DOI:10.1016/j.bse.2012.09.021 |
| [9] |
贾超峰, 刘海林, 许津, 等. 大黄鱼种质遗传多样性研究进展[J]. 海洋通报, 2017, 36(1): 12-18. |
| [10] |
WANG Z D, CHEN C, GUO Y S, et al. High sequence variation and low population differentiation of mitochondrial control regions of wild large yellow croaker in South China Sea[J]. Biochemical Systematics and Ecology, 2014, 56: 151-157. DOI:10.1016/j.bse.2014.05.019 |
| [11] |
HAN Z Q, XU H X, SHUI B N, et al. Lack of genetic structure in endangered large yellow croaker Larimichthys crocea from China inferred from mitochondrial control region sequence data[J]. Biochemical Systematics and Ecology, 2015, 61: 1-7. DOI:10.1016/j.bse.2015.04.025 |
| [12] |
LIU Q H, LIN H D, CHEN J, et al. Genetic variation and population genetic structure of the large yellow croaker (Larimichthys crocea) based on genome-wide single nucleotide polymorphisms in farmed and wild populations[J]. Fisheries Research, 2020, 232: 105718. DOI:10.1016/j.fishres.2020.105718 |
| [13] |
SUN P, SHI Z H, YIN F, et al. Genetic variation analysis of Mugil cephalus in China Sea based on mitochondrial COI gene sequences[J]. Biochemical Genetics, 2012, 50: 180-191. DOI:10.1007/s10528-011-9460-6 |
| [14] |
赵明, 宋炜, 马春艳, 等. 基于线粒体COI基因序列的棘头梅童鱼7个野生群体遗传结构分析[J]. 中国水产科学, 2015, 22(2): 233-242. |
| [15] |
杨帆, 何利军, 雷光春, 等. 中国东南沿海弹涂鱼科常见鱼类的遗传多样性和DNA条形码[J]. 生态学杂志, 2012, 31(3): 676-683. |
| [16] |
WU R X, ZHANG H R, LIU J, et al. DNA barcoding of the family Sparidae along the coast of China and revelation of potential cryptic diversity in the Indo-West Pacific oceans based on COI and 16S rRNA genes[J]. Journal of Oceanology and Limnology, 2018, 36(5): 1753-1770. |
| [17] |
吴仁协, 梁秀何, 庄志猛, 等. 中国近海银鲳线粒体COI基因序列变异分析[J]. 动物分类学报, 2012, 37(3): 480-488. |
| [18] |
陈淑吟, 徐士霞, 张志勇, 等. 大黄鱼野生群体与养殖群体遗传多样性研究[J]. 海洋科学, 2011, 35(12): 82-87. |
| [19] |
谌微, 张凤英, 王景, 等. 基于COI基因序列的东、黄海区野生与养殖大黄鱼遗传多样性分析[J]. 中国水产科学, 2016, 23(6): 1255-1267. |
| [20] |
王军, 全成干, 苏永全, 等. 宫井洋大黄鱼遗传多样性的RAPD分析[J]. 海洋学报, 2001, 23(3): 87-91. |
| [21] |
王志勇, 王艺磊, 林利民, 等. 福建官井洋大黄鱼AFLP指纹多态性的研究[J]. 中国水产科学, 2002, 9(3): 198-202. |
| [22] |
黎中宝, 方秀, 陈锦, 等. 大黄鱼(Pseudosciaena crocea)养殖群体遗传多样性的降低[J]. 海洋与湖沼, 2009, 40(4): 446-450. |
| [23] |
COSSU P, SCARPA F, SANNA D, et al. Influence of genetic drift on patterns of genetic variation: the footprint of aquaculture practices in Sparus aurata (Teleostei: Sparidae)[J]. Molecular Ecology, 2019, 28: 3012-3024. |
| [24] |
NORRIS A T, BRADLEY D G, CUNNINGHAM E P. Microsatellite genetic variation between and within farmed and wild Atlantic salmon (Salmo salar) populations[J]. Aquaculture, 1999, 180: 247-264. |
| [25] |
LIU F, XIA J H, BAI Z Y, et al. High genetic diversity and substantial population differentiation in grass carp (Ctenopharyngodon idella) revealed by microsatellite analysis[J]. Aquaculture, 2009, 297: 51-56. |
| [26] |
WANG L, MENG Z, LIU X C, et al. Genetic diversity and differentiation of orange-spotted grouper (Epinephelus coioides) between and within cultured stocks and wild populations inferred from microsatellite DNA analysis[J]. International Journal of Molecular Sciences, 2011, 12: 4378-4394. |
| [27] |
张其永, 洪万树, 杨圣云, 等. 大黄鱼增殖放流的回顾与展望[J]. 现代渔业信息, 2010, 25(12): 3-5, 12. |
| [28] |
吴仁协, 柳淑芳, 庄志猛, 等. 基于线粒体Cyt b基因的黄海、东海小黄鱼(Larimichthys polyactis)群体遗传结构[J]. 自然科学进展, 2009, 19(9): 924-930. |
| [29] |
吴仁协, 张浩冉, 牛素芳, 等. 东海近岸带鱼(Trichiurus japonicus)线粒体控制区序列的群体遗传变异研究[J]. 海洋与湖沼, 2019, 50(6): 1318-1327. |
| [30] |
EDWARDS A W F. The fundamental theorem of natural selection[J]. Theoretical Population Biology, 2002, 61(3): 335-337. |
| [31] |
吴利娜, 张凝鋆, 孙松, 等. 微卫星分子标记技术在大黄鱼增殖放流效果评估中的应用[J]. 中国水产科学, 2021, 28(9): 1100-1108. |
| [32] |
彭士明, 王亚冰, 王倩, 等. 大黄鱼分子育种相关技术研究进展[J]. 海洋渔业, 2022, 44(3): 375-384. |



