2. 南京师范大学生命科学学院, 江苏南京 210023;
3. 江苏省淡水水产研究所,江苏南京 210017
2. College of Life Sciences, Nanjing Normal University, Nanjing, Jiangsu, 210023, China;
3. Jiangsu Freshwater Fisheries Research Institute, Nanjing, Jiangsu, 210017, China
生物钟又称“昼夜节律钟”或“近日时间节律钟”,是生物体适应环境周期性变化的内在计时机制,具有重要的功能[1]。Abe等[2]研究证实生物钟基因调节生物体的稳态,与炎症反应、血糖平衡、合成和分解代谢等有着密切的关系。昼夜节律的发生是以时钟基因及其相关蛋白为物质基础[3],例如时钟基因(clock,Clk)与生物体生命健康息息相关,其蛋白产物CLOCK (CLK)属于bHLH/PAS家族的转录因子,驱动目标时钟基因以及与组织特异性转录因子相关的其他基因表达[4]。在生物钟调控系统中,CLK蛋白通常与CYC蛋白形成异源二聚体,激活下游基因转录,在反馈环中起正调控作用。它主要在视交叉上核和视网膜中表达,在外周组织器官(肝脏、心脏、肌肉、肾脏、胰脏、睾丸、卵巢等)中也有表达[5]。在对小鼠的研究中发现,敲除该基因的小鼠表现出生物节律紊乱,多巴胺在中脑顿腹部(Ventral Tegmental Area,VTA)的转运增加,由此推断出CLK蛋白与脑回路中的多巴胺运转有关[6]。敲除斑马鱼Clk基因,会导致其他几个相关基因的表达量下降[7]。
Cryptochrome (Cry)又被称为“隐花色素基因”,其产物蛋白是一种对蓝光极其敏感的黄素蛋白光受体,该蛋白是多细胞动物核心昼夜节律振荡器的组成部分,可调节动物体内昼夜生物钟光导引过程[8, 9]。Cry基因是核心生物钟基因,可分为3种基因类型:Cry1(或称果蝇型Cry)、Cry2(或称哺乳动物型Cry)和Cry3。其中CRY2是昆虫和哺乳动物生物钟的重要组成部分; 而CRY1在果蝇研究中较为深入,在睡眠剥夺所引发的血管炎症影响中发现,CRY1可明显抑制血管炎症的发生和发展[10]。
克氏原螯虾(Procambarus clarkii)肉质结实,味道清新鲜美,是我国重要的水产养殖经济虾类。但在克氏原螯虾规模化人工养殖过程中会遇到很多问题,如苗种繁育不足、个体小型化、疾病防控落后等。谢伟等[11]研究表明克氏原螯虾诸多生理与行为节律均受体内生物钟基因的调控,生物钟基因对克氏原螯虾的生长发育和摄食行为具有很大影响。作为重要的经济虾类,目前对克氏原螯虾生物钟相关基因的种类和功能研究还不够全面,对其调控机制更是缺少了解,因此本研究运用生物信息学方法分析Clk基因(命名为PcClk)与Cry1基因(命名为PcCry1) 的一些理化性质、功能结构,并使用荧光定量PCR技术(qRT-PCR)探究克氏原螯虾这两个生物钟基因在不同温度和光照协同作用模式下的mRNA表达差异情况,并从进化生物学角度对克氏原螯虾PcClk与PcCry1进行选择压力分析,以期了解温度和光照周期对克氏原螯虾生物钟节律的影响,为其健康养殖提供基础资料。
1 材料与方法 1.1 样品采集克氏原螯虾样品均采集于江苏南通野外生境,选取8-12 cm健康的雄性克氏原螯虾个体,以排除性别对实验的干扰。样品带回实验室后对其进行适应驯化培养。
暂养驯化培养:在实验室循环水箱中饲养2周,用全光谱日光灯模拟室外自然光照,光照周期为12 h光照和12 h黑暗(12 LD),用增氧泵24 h不间断充氧并维持室温在25℃左右;同时,在养殖箱底铺满沙石,并放置尼龙管、石块、易拉罐,为克氏原螯虾提供隐蔽处,减少其打斗频率。暂养期间于早(8:00)晚(20:00)投喂虾蟹专用颗粒饲料,并保证每天投喂量一样,及时吸取残饵,2 d换水一次,换水量约为原来的1/3。
驯化2周后,将样品放置在24 LL (24 h光照)、12 LD (12 h光照和12 h黑暗)和24 DD (24 h黑暗)模式下培养。24 h黑暗处理是将实验室所有门窗使用不透光黑薄膜遮蔽,仅在喂食和换水时短暂开灯,其余时间均处于黑暗状态;24 h光照处理是将处于自然光照射下的养殖水箱上方放置全光谱日光灯,并且24 h打开。由于在25℃、12 LD培养模式下克氏原螯虾的生长状况最好,在24 LL、24 DD或者温度过高时,其成活率会降低(数据暂未发表),所以在每种光照周期下设置恒定温度25℃,同时在12 LD的光照周期下设置20℃、25℃和30℃ 3个不同温度进行研究,水温由加热棒控制。在驯化期间正常喂食,驯化结束后分别挑取52只健康雄性克氏原螯虾转入各模式下继续培养,3 d后于时间点8:00、12:00、16:00、20:00、00:00,次日4:00和8:00分别对每组取样。每个时间点取两只一组,共取3组平行实验组。每组取脑、眼柄、肝胰腺各约30 mg的组织,加入RNA保护试剂,匀浆后于-80℃冰箱保存备用。
1.2 总RNA提取和cDNA合成将状态良好的克氏原螯虾肝胰腺、脑、眼柄组织分别取出,按Trizol试剂盒(赛默飞世尔科技有限公司)说明书的方法提取总RNA,用焦碳酸二乙酯(Diethyl Pyrocarbonate,DEPC)处理水稀释,使RNA沉淀溶解,并用1.0%琼脂糖凝胶电泳检测RNA质量;使用超微量紫外分光光度计对RNA进行浓度定量测定。据PrimeScriptTM RT Master Mix (上海力敏实业有限公司)说明,将获得的总RNA反转录为cDNA,-80℃冰箱保存备用。
1.3 cDNA序列和氨基酸序列分析用cDNA末端快速扩增方法获得的PcClk基因和PcCry1基因全长,于美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)进行PcClk基因(MN908585)和PcCry1基因(MG659714.1) cDNA全长完整性验证。使用ExPASy服务器(https://web.expasy.org/)[12]的ProtParam和ProtScale工具分析PcCLK蛋白、PcCRY1蛋白的理化性质和疏水性。使用SignalP程序[13]预测信号肽,MotifScan程序查找motif,SMART软件[14]分析结构功能域,PSORT Ⅱ软件进行亚细胞定位,TMHMM v.2.0软件[15]预测跨膜区,SWISS-MODEL网站预测蛋白质三级结构[16]。使用MEGA Ⅹ软件的邻接法构建系统发育树,验证PcCLK蛋白与PcCRY1蛋白在进化过程中与其他同源蛋白的关系[17]。
1.4 荧光定量PCR检测使用Prime Primer 5软件设计引物[表 1,以β-actin作为内参基因,引物均由生工生物工程(上海)股份有限公司合成],并对PcClk基因、PcCry1基因在不同光照和温度协同作用模式下7个时间点的mRNA表达情况进行检测。使用StepOnePlusTM实时荧光定量PCR仪(Applied Biosystems,美国应用生物系统公司)进行定量PCR实验,按照实时定量PCR试剂盒SYBRⓇ Premix-TaqTM Ⅱ (TliRNaseH Plus)说明书操作,荧光定量PCR反应体系如下:SYBRⓇ Premix ExTaqTM Ⅱ (2×) 10 μL,PCR Forward Primer (10 μmol/L) 0.8 μL,PCR Reverse Primer (10 μmol/L) 0.8 μL,cDNA (100 ng/μL) 1 μL,再加上去离子水7.4 μL。反应程序为95℃ 5 min,95℃ 5 s,62℃ 30 s,72℃ 15 s,72℃ 1 min,16℃ 15 s。反应结束后分析相关数据,采用比较CT值的方法(基因相对表达量=2-ΔΔCT)[18]来分析PcClk基因、PcCry1基因的相对表达水平。采用SPSS17.0软件对所有数据进行统计分析,利用单因素方差分析法(ANOVA)对不同处理组样品进行差异显著性分析,P < 0.05表示差异显著。
| 基因 Gene |
引物用途 Primer purpose |
引物名称 Primer name |
引物序列(5′→3′) Primer sequence (5′→3′) |
登录号 Accession |
| PcClk | RT-qPCR | qClock-F | AAGATGGAGTGCTGGACTTTAG | MN908585 |
| RT-qPCR | qClock-R | GGATAAGGGAGCTCTGAAACAA | ||
| PcCry1 | RT-qPCR | qCryptochrome-F | AGTCAGTGCAACGAGCTGCGAAAT | MN908586 |
| RT-qPCR | qCryptochrome-R | AGGTGGAGGTGATGGGTAGTGGTC | ||
| β-actin | RT-qPCR | qβ-actin-F | GCACCATCCACCATGAAGATTA | KR135165 |
| RT-qPCR | qβ-actin-R | GGTAGAAAGGGAAGCCAAGATG |
1.5 适应性进化分析 1.5.1 序列获取
为研究克氏原螯虾PcClk基因和PcCry1基因的适应性进化,选择代表性节肢动物物种进行分析,涉及甲壳亚门、螯肢亚门和六足亚门。所有选取物种的Clk基因和Cry基因序列均从NCBI数据库直接下载,相关信息如表 2、表 3所示。
| 亚门 Subphylum |
纲 Class |
物种 Species |
登录号 Accession |
| 脊椎动物亚门 Vertebrata | 鸟纲 Aves | 藏香鸡 Gallus gallus | AF246959.1 |
| 哺乳纲 Mammalia | 褐家鼠 Rattus norvegicus | AB019258.1 | |
| 哺乳纲 Mammalia | 绵羊 Ovis aries | EU016223.1 | |
| 爬行纲 Reptile | 中华鳖 Pelodiscus sinensis | KY624634.1 | |
| 辐鳍鱼纲 Actinopterygii | 斑马鱼 Danio rerio | AAI63244.1 | |
| 甲壳动物亚门 Crustacea | 软甲纲 Malacostraca | 罗氏沼虾 Macrobrachium rosenbergii | AY842303.1 |
| 软甲纲 Malacostraca | 等足目虫 Eurydice pulchra | AGV28723.1 | |
| 甲壳纲 Crustacea | 南美白对虾 Penaeus vannamei | ROT60838.1 | |
| 甲壳纲 Crustacea | 克氏原螯虾 Procambarus clarkii | MN908585.1 | |
| 六足亚门 Hexapoda | 昆虫纲 Insecta | 柞蚕 Antheraea pernyi | AY330486.1 |
| 昆虫纲 Insecta | 米虫 Sitophilus oryzae | MH070263.1 | |
| 昆虫纲 Isecta | 赤拟谷盗 Tribolium castaneum | EF174303.1 | |
| 昆虫纲 Isecta | 二化螟 Chilo suppressalis | MH933743.1 | |
| 螯肢亚门 Chelicerata | 肢口纲 Merostomata | 美洲鲎 Limulus polyphemus | KX014718.1 |
| 亚门 Subphylum |
纲 Class |
物种名 Name of species |
登录号 Accession |
| 甲壳动物亚门 Crustacea | 甲壳纲 Crustacea | 南美白对虾 Penaeus vannamei | ROT79736.1 |
| 甲壳纲 Crustacea | 克氏原螯虾 Procambarus clarkii | MN908586.1 | |
| 鳃足纲 Branchiopoda | 大型溞 Daphnia magna | LC027222.1 | |
| 软甲纲 Malacostraca | 等足目虫 Eurydice pulchra | KC885970.1 | |
| 软甲纲 Malacostraca | 欧洲沙蚤 Talitrus saltator | JQ413343.1 | |
| 软甲纲 Malacostraca | 中华绒螯蟹 Eriocheir sinensis | MG659714.1 | |
| 六足亚门 Hexapoda | 昆虫纲 Insecta | 黑腹果蝇 Drosophila melanogaster | AF099734.1 |
| 昆虫纲 Insecta | 叶榕传粉榕小蜂 Ceratosolen solmsi | JX409893.1 | |
| 昆虫纲 Insecta | 棉铃虫 Helicoverpa armigera | GQ896502.1 | |
| 昆虫纲 Insecta | 甜菜夜蛾 Spodoptera exigua | HQ234484.1 | |
| 昆虫纲 Insecta | 黑脉金斑蝶 Danaus plexippus | AY860425.1 | |
| 昆虫纲 Insecta | 麦田灰飞虱 Laodelphax striatella | AVP27638.1 | |
| 昆虫纲 Insecta | 球菜夜蛾 Agrotis ipsilon | JQ616846.1 | |
| 昆虫纲 Insecta | 欧洲熊蜂 Bombus terrestris | MK250529.1 | |
| 昆虫纲 Insecta | 若虫 Modicogryllus siamensis | LC320665.1 | |
| 脊椎动物亚门 Vertebrata | 两栖纲 Amphibian | 日本树蟾 Hyla japonica | AB206651.1 |
| 昆虫纲 Insecta | 双斑蟋蟀 Gryllus bimaculatus | LC202047.1 | |
| 辐鳍鱼纲 Actinopterygii | 斑马鱼 Danio rerio | AB042248.1 | |
| 鸟纲 Aves | 原鸽 Columba livia | MG839692.1 |
1.5.2 构建系统发育树
去除所有序列的终止密码子,使用将获取的节肢动物PcClk基因、PcCry1基因序列通过PRANK软件进行密码子比对[19]。使用Trimal软件处理密码子的比对结果后,删除一些低质量或高变异度的序列区域,仅保留进化保守的区域用于后续分析[20]。使用IQ-TREE自动测试功能并选择最佳替代模型,构建系统发育树[21]。该系统发育树同时也被用于后续构建选择压力分析的树文件。
1.5.3 选择压力分析使用PAML 4.9软件包的CODEML程序计算ω值,使用位点模型、分支模型和分支位点模型进行进化分析[22]。
首先使用位点模型检测PcClk基因和PcCry1基因在节肢动物数据集中受正选择的位点,并比较Ma和Ma0,M0和M3,M1a和M2a,M7和M8,M8和M8a模型。接着,利用似然率检测方法计算2ΔlnL值与自由度之间的卡方分布。然后,利用贝叶斯方法BEB (Bays Empirical Bays)计算位点的后验概率(Posterior Probability,PP),若后验概率>0.8,则被认为是潜在的正选择位点[23]。此外,使用Datamonkey中的FEL、SLAC和FUBRA检测正选择位点,设置FEL和SLAC显著水平小于0.2,FUBAR后验概率值大于0.8[24]。最后,整合PAML和Datamonkey筛选的正选择位点,将同时被PAML和Datamonkey检测到的位点视为显著的正选择位点,并标注在模拟的克氏原螯虾PcClk基因和PcCry1基因的蛋白质三维结构中。
2 结果与分析 2.1 PcClk、PcCry1基因序列与蛋白结构特征将PcClk核苷酸序列在NCBI公共数据库中比对,发现其与其他物种的Clk核苷酸同源性相似度区间为68%-92%,其中与十足目通讯螯虾(Pacifastacus leniusculus)的Clk基因最为相似,相似度为92%,氨基酸序列比对结果类似。同样地,将PcCry1核苷酸序列在NCBI公共数据库中进行比对后发现,其与其他物种的PcCry1核苷酸同源性相似度区间为73%-84%,其核苷酸序列与美洲海螯虾(Homarus americanus)的Cry1基因最为相似,而氨基酸序列比对结果显示PcCRY1蛋白与美洲海螯虾(Homarus americanus)CRY2蛋白相似性最高,相似度为96%。
2.2 PcClk基因和PcCry1基因正选择位点的定位通过SWISS-MODEL网站预测得到克氏原螯虾PcCLK和PcCRY1三级结构(图 1),强烈正选择位点在三级结构中的定位如图 1标记所示。图中显示PcCLK蛋白与模板4f3l.1.B蛋白的匹配度最高,序列相似度为54.60%;PcCRY1蛋白与模板7d1c.1.A匹配度最高,序列相似度为71.72%。
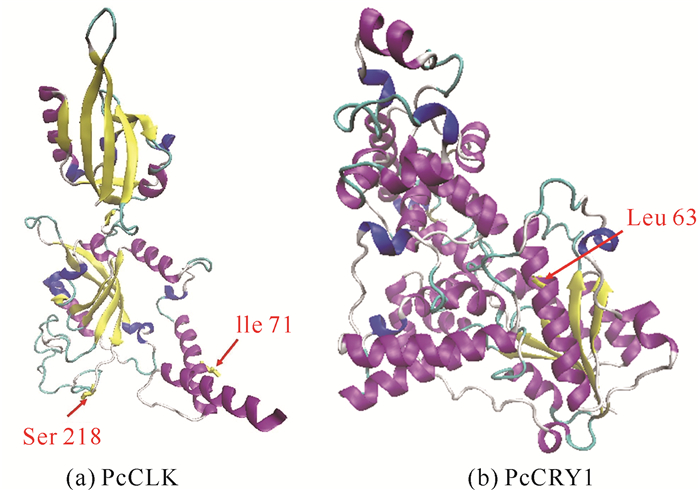
|
| 图 1 克氏原螯虾PcCLK和PcCRY1蛋白质三维结构预测结果(标注为强烈正选择位点) Fig. 1 Three-dimensional structure prediction results of PcCLK and PcCRY1 proteins of Procambarus clarkii (The strongly positive selection sites were labeled) |
2.3 多序列比对和系统发育分析
基于MEGA Ⅹ软件邻接法的系统发育分析显示,PcCLK与甲壳类动物的CLK聚在一起,而PcCRY1与甲壳类动CRY1聚在一起(图 2)。克氏原螯虾PcCLK与南美白对虾(Penaeus vannamei)、罗氏沼虾(Macrobrachium rosenbergii)、等足目虫(Eurydice pulchra)的CLK蛋白形成甲壳动物亚门分支;克氏原螯虾PcCRY1与南美白对虾、大型溞(Daphnia magna)、等足目虫、欧洲沙蚤(Talitrus saltator)、中华绒螯蟹(Eriocheir sinensis)的CRY1蛋白形成甲壳动物亚门分支(图 2)。
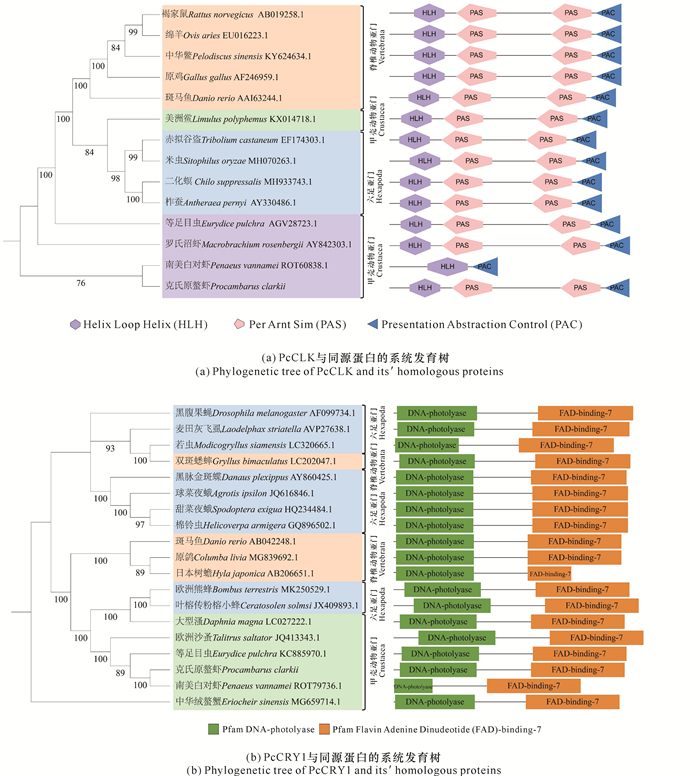
|
| 图 2 基于邻接法构建的PcCLK和PcCRY1及其同源蛋白的系统发育树 Fig. 2 Phylogenetic trees of PcCLK and PcCRY1 and their homologous proteins based on neighbor-joining method |
2.4 PcClk基因和PcCry1基因表达水平分析
在12 LD、不同温度条件下,PcClk基因、PcCry1基因在脑、眼柄、肝胰腺中的mRNA表达情况如图 3所示。PcClk基因在脑、眼柄、肝胰腺中的mRNA表达基本保持24 h的振荡规律。随着温度的升高,PcClk基因在脑、眼柄中的mRNA表达特征均发生变化,表现为振荡波动增强、节律性更明显,尤其在30℃条件下,PcClk的mRNA表达量整体上明显上调。而在肝胰腺中,随着温度的升高,PcClk基因的mRNA表达波动减弱,节律性相对20℃时变得不明显。同样地,随着温度的升高,PcCry1基因在脑和眼柄中的mRNA表达振荡特征均发生变化,波动增强,节律性更明显。与PcClk基因类似的是,相对于20℃,25℃、30℃情况下PcCry1基因在肝胰腺中的mRNA表达趋于平缓,振荡波动减弱,节律不明显。
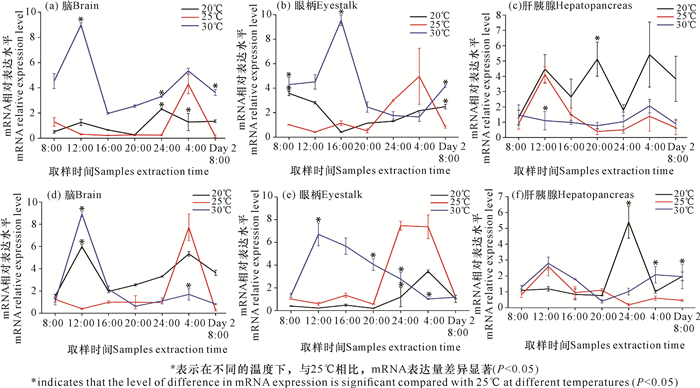
|
| 图 3 克氏原螯虾PcClk基因[(a)-(c)]和PcCry1基因[(d)-(f)]在不同温度、不同组织中的mRNA表达水平(12 LD) Fig. 3 mRNA expression levels of PcClk [(a)-(c)] and PcCry1 [(d)-(f)] genes of Procambarus clarkii under different temperatures and in different tissues (12 LD) |
在25℃、不同光照周期培养下,PcClk基因、PcCry1基因在脑、眼柄、肝胰腺中的mRNA表达情况如图 4所示。在12 LD的光照周期下,脑、眼柄、肝胰腺中PcClk基因的mRNA表达有一定的振荡节律;而在24 LL和24 DD光照周期下,振荡节律发生改变,尤其是眼柄中PcClk基因的mRNA表达表现出无振荡节律。PcCry1基因的mRNA在脑和眼柄中的表达都基本遵循着一定的振荡节律,在12 LD和24 LL光照周期下,PcCry1的表达量在白天缓慢堆积,在客观夜晚期间达到峰值,随后缓慢下降。而PcCry1基因的mRNA在肝胰腺的表达与脑、眼柄有所区别:在12 LD的光照周期下mRNA表达有一定的振荡节律,但在24 LL和24 DD光照周期下振荡波动减弱,节律不明显。
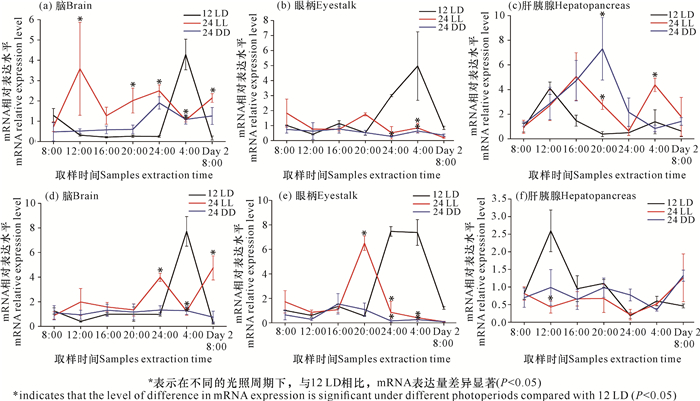
|
| 图 4 克氏原螯虾PcClk基因[(a)-(c)]和PcCry1基因[(d)-(f)]在不同光照周期、不同组织中的mRNA表达水平(25℃) Fig. 4 mRNA expression levels of PcClk [(a)-(c)] and PcCry1 [(d)-(f)] genes in different tissues of Procambarus clarkii under different photoperiods (25℃) |
2.5 克氏原螯虾PcClk基因、PcCry1基因的适应性进化分析结果 2.5.1 系统发育关系
利用包括克氏原螯虾在内的节肢动物PcClk基因和PcCry1基因序列,基于最大似然法构建系统发育树。结果如图 5所示,PcClk基因和PcCry1基因的系统发育树分为3个主要的进化枝:甲壳动物亚门进化枝(Crustacea clade)、六足动物亚门进化枝(Hexapoda clade)和螯肢动物亚门进化枝(Chelicerata clade)。
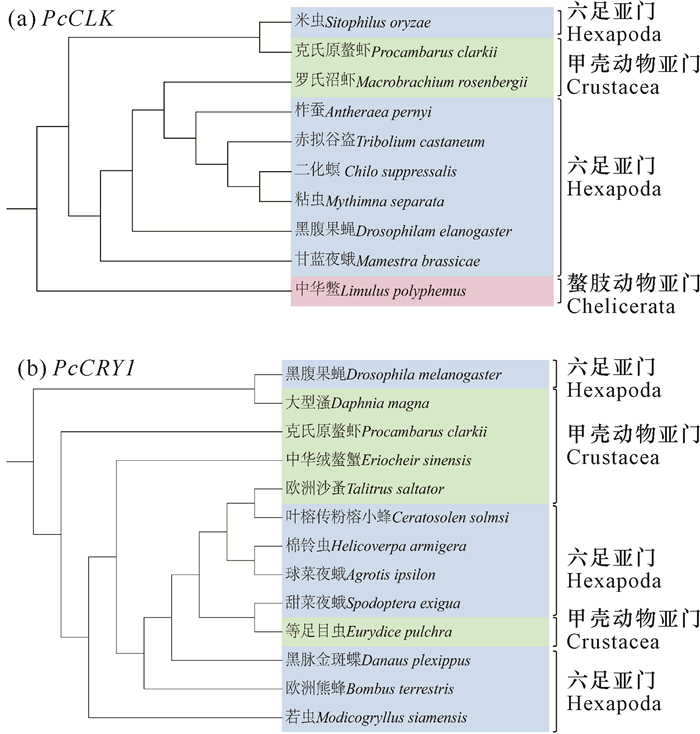
|
| 图 5 节肢动物PcClk基因(a)和PcCry1基因(b)的系统发育树 Fig. 5 Phylogenetic trees of PcClk (a) and PcCry1 (b) genes in arthropods |
2.5.2 选择压力分析
为研究克氏原螯虾PcClk基因和PcCry1基因是否受到正选择作用,首先假设同一个基因的系统发育树所有分支都有同一个ω值。PcClk基因和PcCry1基因one-ratio模型结果显示,ω值分别为0.023和0.029,显著小于1,表明该基因整体上受纯化选择作用(表 4、表 5)。Free-ratio模型假定系统发育树上每一分支和节点都具有独立的ω值,结果显示free-ratio模型显著优于one-ratio模型(P < 0.05),提示不同支系具有不同的进化速率。以克氏原螯虾为前景枝,这两个基因two-ratio模型拟合效果并没有明显优于one-ratio模型(P>0.05),表明克氏原螯虾PcClk基因、PcCry1基因与其他近缘甲壳动物有相似的进化速率(表 4、表 5)。其次,使用位点模型,发现这两个基因的M1a模型和M2a模型、M7模型和M8a模型、M8模型和M8a模型之间均没有显著差异(P>0.05),而PcClk基因的M0模型和M3模型有显著差异(P < 0.05)。最后,使用分支位点模型检测PcClk基因、PcCry1基因,PcCry1基因的Ma模型优于Ma0模型(P < 0.01)。
| 模型 Models |
模型比较 Model comparison |
-lnL | 2ΔlnL | df | P值 P values |
ω值 ω values |
正选择位点 Positive selection sites (PP>0.95) |
| 位点模型 Site model |
M3/M0 | 11 341.852/11 437.834 | 191.963 | 4 | Nearly 0 | — | Not allowed |
| M2a/M1a | 11 728.389/11 728.392 | 0.006 | 2 | 0.997 | — | Not allowed | |
| M7/M8 | 11 347.926/11 347.928 | 0.003 | 2 | 0.997 | — | Not allowed | |
| M8a/M8 | 11 360.329/11 347.928 | 24.802 | 1 | Nearly 0 | — | Not allowed | |
| 分支模型 Branch model |
Free-ratio/one-ratio | 11 419.506/11 437.834 | 36.655 | 16 | 0.002 | — | — |
| Two-ratio/one-ratio | 11 437.829/11 437.834 | 0.010 | 1 | 0.921 | ωBackground=0.023,ωForeground=0.011 | — | |
| 分支位点模型 Branch-site model |
Ma/Ma0 | 11 728.389/11 728.389 | 2.000 | 1 | 0.999 | Background(a/b): 0.000/1.000 | Not allowed |
| 注:“-”表示无此项内容 Note: "-" indicates that the content does not exist |
|||||||
| 模型 Models |
模型比较 Model comparison |
-lnL | 2ΔlnL | df | P值 P values |
ω值 ω values |
正选择位点 Positive selection sites (PP>0.95) |
| 位点模型 Site model |
M3/M0 | 21 566.245/21 566.245 | 0.000 | 4 | 1.000 | — | Not allowed |
| M2a/M1a | 22 029.630/22 029.635 | 0.009 | 2 | 0.995 | — | Not allowed | |
| M7/M8 | 21 558.841/22 029.635 | 941.588 | 2 | Nearly 0 | — | Not allowed | |
| M8a/M8 | 22 029.635/22 029.635 | 0.000 | 1 | 1.000 | — | Not allowed | |
| 分支模型 Branch model |
Free-ratio/one-ratio | 21 548.131/21 566.245 | 36.227 | 2 | 0.029 | — | — |
| Two-ratio/one-ratio | 21 566.166/21 566.245 | 0.159 | 1 | 0.690 | ωBackground=0.029, ωForeground=0.022 | — | |
| 分支位点模型 Branch-site model |
Ma/Ma0 | 22 021.236/22 029.630 | 16.788 | 1 | Nearly 0 | Background(a/b): 0.000/1.000,Foreground(a/b): 105.267/105.267 | 2 A 0.953*, 3 D 0.988*, 7 G 0.996**, 9 A 0.985*, 14 F 0.985*, 16 F 0.977*,et al, a total of 121 |
| 注:“-”表示无此项内容;2A、3D、7G、9A……表示氨酸;*表示后验概率大于0.95;**表示后验概率大于0.99 Note: "-" indicates that the content does not exist;2A, 3D, 7G, 9A…indicate amino acid; *indicates a posterior probability is greater than 0.95;** indicates a posterior probability is greater than 0.99 |
|||||||
通过PAML筛选,结果显示PcClk基因没有正选择位点,而PcCry1基因中鉴定有121个正选择位点(表 6)。为得到更加精确的结果,进一步使用Datamonkey网站中的FEL、FUBAR和SLAC 3种方法检测,共检测到PcClk基因有2个正选择位点,PcCry1基因有1个正选择位点。最终,PcClk基因中的71,218位点可被FEL、SLAC同时检测出来,而PcCry1基因中的63位点可被PAML、FEL、SLAC同时检测出来,故这些正选择位点被认为是强烈的正选择位点(表 6,几种方法同时检测出的正选择位点以粗体加波浪线标注)。
| Gene | PAML | Datamokey | ||
| FEL (P < 0.2) |
FUBAR (PP>0.8) |
SLAC (P < 0.2) |
||
| PcClk |  ,124,145,157,163,201,217, ,124,145,157,163,201,217,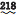 ,222,364,376,406,432,433,445,455,458,460,471,507,513,521,522,525 ,222,364,376,406,432,433,445,455,458,460,471,507,513,521,522,525 |
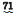 ,136, ,136,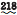 ,346 ,346 |
||
| PcCry1 | 6,18,21,26,27,29,31,33,34,37,40,41,43,47,51,52,53,56,57,62, ,64,66,68,69,76,80,81,85,65,99,105,108,110,112,122,123,130,133,138,139,140,141,147,152,155,157,158,163,167,168,171,178,183,185,195,200,205,207,208,209,211,216,218,226,230,232,237,238,241,242,243,246,252,254,256,257,261,271,276,281,285,293,300,302,303,304,305,306,310,311,312,318,319,323,371,385,388,399,404,409,413,424,425,427,432,434,439,441,443,447,448,452,453,454,460,463,475,477,484,489 ,64,66,68,69,76,80,81,85,65,99,105,108,110,112,122,123,130,133,138,139,140,141,147,152,155,157,158,163,167,168,171,178,183,185,195,200,205,207,208,209,211,216,218,226,230,232,237,238,241,242,243,246,252,254,256,257,261,271,276,281,285,293,300,302,303,304,305,306,310,311,312,318,319,323,371,385,388,399,404,409,413,424,425,427,432,434,439,441,443,447,448,452,453,454,460,463,475,477,484,489 |
3,9,15,17,18,21,39,45,52, ,64,65,95,99,133,134,148,159,177,181,186,191,197,209,215,222,237,240,247,250,261,266,271,272,273,285,309,319,334,352,360,361,374,380,389,390,405,406,407,408,409,411,414,430,437,449,453,470,477 ,64,65,95,99,133,134,148,159,177,181,186,191,197,209,215,222,237,240,247,250,261,266,271,272,273,285,309,319,334,352,360,361,374,380,389,390,405,406,407,408,409,411,414,430,437,449,453,470,477 |
169 | 1,50,53,55,57, ,70,77,103,125,131,178,213,239,280,302,303,340,400,410,417,462,478,479 ,70,77,103,125,131,178,213,239,280,302,303,340,400,410,417,462,478,479 |
| 注:粗体加波浪线数字为强烈的正选择位点 Note: Bold plus wavy line numbers are strong positive selection sites |
||||
3 讨论
生物钟是生物体对地球上环境因子周期变化长期适应而演变的内在调节机制[25]。本研究发现PcCry1基因的71与213位点受到强烈正选择作用,该位点位于CLOCK蛋白的PAS结构域中。该功能结构域可介导CLOCK蛋白与另一个生物钟基因的二聚体作用,对昼夜节律的产生和维持有一定作用,这与先前对其他甲壳类动物如罗氏沼虾[26]的研究结果相似。PcCry1基因的63位点受到强烈正选择作用,提示克氏原螯虾可能通过增强自身的调节来适应外界环境的变化,这与李敬[27]发现鲸CRY蛋白对昼夜节律的调控有一定作用的研究结果一致。
迄今关于甲壳动物生物钟基因表达的数据仍然很少。本研究中,在12 LD光照周期下,温度升高,脑、眼柄中的PcClk基因和PcCry1基因mRNA表达增强,节律性更明显,但两个基因在肝胰腺中的mRNA表达量减弱,节律性不明显。因此,推测克氏原螯虾的PcClk基因和PcCry1基因都是由内源性节律驱动。脑作为核心生物钟调控元件组织,在生物钟基因调控中发挥中央指挥者的作用,其时钟基因mRNA的表达最先受到调控; 眼柄临近脑组织,因此时钟基因mRNA的表达相似; 而外周组织器官离脑较远,所以对节律性的保持没有脑组织明显。在对果蝇的研究中发现,Clk表达的节律性在脑中比在心脏中更明显[28],而在豌豆蚜(Acyrthosiphon pisum)和冈比亚疟蚊(Anopheles gambiae)的研究中,Cry在各自的头部存在更明显的节律性表达[29]。这与本研究发现的克氏原螯虾的生物钟基因在脑中节律性更明显的结果一致。
在温度为25℃时,相对于24 LL和24 DD的光照周期,12 LD的光照周期下,克氏原螯虾脑、眼柄、肝胰腺中PcClk基因的mRNA表达振荡节律更明显;同样地,在25℃、12 LD培养模式下,脑和眼柄中PcCry1基因的mRNA表达节律性也较明显。这可能是因为25℃、12 LD对克氏原螯虾来说是适宜的温度和光照,而适宜的温度和光暗循环更有利于克氏原螯虾PcClk和PcCry1基因节律性的保持。李龙[30]研究显示光照的改变对斑马鱼生物钟基因的调控有很大影响;蔡明岐[31]在研究光照和温度对蚤状溞(Daphnia pulex)的影响时发现,在25℃且光照和黑暗比为8∶16时最有利于蚤状溞生物钟基因昼夜节律的保持,也最适合蚤状溞生长;徐加元等[32]发现在适宜的温度和光照周期下,适当延长光照周期可促进克氏原螯虾雌虾卵巢的发育,但过长的光照不仅不会促进雌虾的性腺发育,反而会抑制其性腺发育。适宜的光照和温度有利于克氏原螯虾生物钟基因的调节,而生物钟调节的稳定对生物体健康状况意义重大。本研究中,25℃、12 LD培养模式最有利于克氏原螯虾PcClk和PcCry1基因节律性的保持,而生物钟节律的稳定可以让克氏原螯虾保持更稳定的健康状态。由此可见,在克氏原螯虾的养殖过程中选择合适的温度和光照尤为重要。
4 结论在温度25℃、12 LD光照周期下,克氏原螯虾脑中的PcClk和PcCry1的mRNA表达都呈现出一定的振荡节律,说明PcClk基因和PcCry1基因是内源性的节律基因。选择压力分析显示,通过PAML和Datamonkey共同筛选出的PcCLK正选择位点为71与213,PcCRY1正选择位点为63。这些位点在节肢动物CLK和CRY1蛋白进化中起重要作用,调控mRNA翻译过程和蛋白质相互作用,可能参与节肢动物内在节律的调控。
| [1] |
WOELFLE M A, OUYANG Y, PHANVIJHITSIRI K, et al. The adaptive value of circadian clocks: An experimental assessment in cyanobacteria[J]. Current Biology, 2004, 14(16): 1481-1486. DOI:10.1016/j.cub.2004.08.023 |
| [2] |
ABE M, HERZOG E D, YAMAZAKI S, et al. Circadian rhythms in isolated brain regions[J]. The Journal of Neuroscience, 2002, 22(1): 350-356. DOI:10.1523/JNEUROSCI.22-01-00350.2002 |
| [3] |
黎小梅, 金风. 时钟基因CLOCK在恶性肿瘤中的研究进展[J]. 现代肿瘤医学, 2020, 28(8): 1370-1374. DOI:10.3969/j.issn.1672-4992.2020.08.032 |
| [4] |
JOSKA T M, ZAMAN R, BELDEN W J. Regulated DNA methylation and the circadian clock: Implications in cancer[J]. Biology, 2014, 3(3): 560-577. DOI:10.3390/biology3030560 |
| [5] |
DODD A N, SALATHIA N, HALL A, et al. Plant circadian clocks increase photosynthesis, growth, survival, and competitive advantage[J]. Science, 2005, 309(5734): 630-633. DOI:10.1126/science.1115581 |
| [6] |
INADA Y, UCHIDA H, UMEMURA Y, et al. Cell and tissue-autonomous development of the circadian clock in mouse embryos[J]. FEBS Letters, 2014, 588(3): 459-465. DOI:10.1016/j.febslet.2013.12.007 |
| [7] |
WHITMORE D, FOULKES N S, STRÄHLE U, et al. Zebrafish Clock rhythmic expression reveals independent peripheral circadian oscillators[J]. Nature Neuroscience, 1998, 1(8): 701-707. DOI:10.1038/3703 |
| [8] |
SANCAR A. Structure and function of DNA photolyase and cryptochrome blue-light photoreceptors[J]. Chemical Reviews, 2003, 103(6): 2203-2237. DOI:10.1021/cr0204348 |
| [9] |
张达燕, 梁辉, 司马杨虎, 等. 温度与光照节律对家蚕成虫生物钟基因Cry1和Cry2表达的影响[J]. 蚕业科学, 2013, 39(3): 453-459. |
| [10] |
LIN C T, ROBERTSON D E, RAIBEKAS A, et al. Association of flavin adenine dinucleotide with the Arabidopsis blue light receptor CRY1[J]. Science, 1995, 269(5226): 968-970. DOI:10.1126/science.7638620 |
| [11] |
谢伟, 蒋琦辰, 孙宾, 等. 克氏原螯虾PcCyc基因克隆及其对光周期的表达响应[J]. 水产科学, 2021, 40(1): 72-80. |
| [12] |
GASTEIGER E, HOOGLAND C, GATTIKER A, et al. Protein identification and analysis tools on the ExPASy server[M]//WALKER J M. The proteomics protocols handbook. Springer protocols handbooks. Totowa NJ: Humana Press Inc., 2005: 571-607.
|
| [13] |
ARMENTEROS J J A, TSIRIGOS K D, SØNDERBY C K, et al. SignalP 5.0 improves signal peptide predictions using deep neural networks[J]. Nature Biotechnology, 2019, 37(4): 420-423. DOI:10.1038/s41587-019-0036-z |
| [14] |
IVICA L, PEER B. 20 years of the SMART protein domain annotation resource[J]. Nucleic Acids Research, 2018, 46(D1): D493-D496. DOI:10.1093/nar/gkx922 |
| [15] |
KROGH A, LARSSON B, VON HEIJNE G, et al. Predicting transmembrane protein topology with a hidden markov model: Application to complete genomes[J]. Journal of Molecular Biology, 2001, 305(3): 567-580. DOI:10.1006/jmbi.2000.4315 |
| [16] |
BORDOLI L, SCHWEDE T. Automated protein structure modeling with SWISS-MODEL workspace and the protein model portal[M]//ORRY A, ABAGYAN R. Homology modeling. Methods in molecular biology (Methods and Protocols), Totowa NJ: Humana Press, 2012, 857: 107-136.
|
| [17] |
KUMAR S, STECHER G, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. DOI:10.1093/molbev/msy096 |
| [18] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [19] |
LÖYTYNOJA A. Phylogeny-aware alignment with PRANK[M]//RUSSELL D J. Multiple sequence alignment methods. Methods in molecular biology (Methods and protocols). Totowa NJ: Humana Press, 2014, 1079: 155-170.
|
| [20] |
TALAVERA G, CASTRESANA J. Improvement of phylogenies after removing divergent and ambiguously aligned blocks from protein sequence alignments[J]. Systematic Biology, 2007, 56(4): 564-577. DOI:10.1080/10635150701472164 |
| [21] |
NGUYEN L T, SCHMIDT H A, VON HAESELER A, et al. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Molecular Biology and Evolution, 2015, 32(1): 268-274. DOI:10.1093/molbev/msu300 |
| [22] |
YANG Z H. PAML 4:Phylogenetic analysis by maximum likelihood[J]. Molecular Biology and Evolution, 2007, 24(8): 1586-1591. DOI:10.1093/molbev/msm088 |
| [23] |
YANG Z H, WONG W S W, NIELSEN R. Bayes empirical bayes inference of amino acid sites under positive selection[J]. Molecular Biology and Evolution, 2005, 22(4): 1107-1118. DOI:10.1093/molbev/msi097 |
| [24] |
WEAVER S, SHANK S D, SPIELMAN S J, et al. Datamonkey 2.0:A modern web application for characterizing selective and other evolutionary processes[J]. Molecular Biology and Evolution, 2018, 35(3): 773-777. DOI:10.1093/molbev/msx335 |
| [25] |
DUNLAP J C. Molecular bases for circadian clocks[J]. Cell, 1999, 96(2): 271-290. DOI:10.1016/S0092-8674(00)80566-8 |
| [26] |
CHRISTIE A E, YU A, PASCUAL M G. Circadian signaling in the northern krill Meganyctiphanes norvegica: In silico prediction of the protein components of a putative clock system using a publicly accessible transcriptome[J]. Marine Genomics, 2018, 37(1): 97-113. |
| [27] |
李敬. 海洋哺乳动物睡眠进化的遗传机制[D]. 南京: 南京师范大学, 2018.
|
| [28] |
李秋芳. 增龄与Clk基因表达下调对果蝇睡眠-活动节律和心跳节律的影响及运动干预效果研究[D]. 长沙: 湖南师范大学, 2018.
|
| [29] |
CORTÉS T, ORTIZ-RIVAS B, MARTÍNEZ-TORRES D. Identification and characterization of circadian clock genes in the pea aphid Acyrthosiphon pisum[J]. Insect Molecular Biology, 2010, 19(Suppl 2): 123-139. |
| [30] |
李龙. 斑马鱼血管活性肠肽Ⅱ型受体Vipr2/Vpac2参与生物钟调控进程[D]. 苏州: 苏州大学, 2019.
|
| [31] |
蔡明岐. 光暗循环对蚤状溞衰老的影响[D]. 上海: 华东师范大学, 2020.
|
| [32] |
徐加元, 岳彩锋, 戴颖, 等. 水温、光周期和饲料对克氏原螯虾雌虾成活和性腺发育的影响[J]. 华中师范大学学报(自然科学版), 2008, 42(1): 97-101. |



