神经胶质瘤起源于神经胶质细胞,是最常见的中枢神经系统原发性恶性肿瘤,占所有中枢神经系统恶性肿瘤的80%左右[1]。近年来,尽管神经胶质瘤的外科手术治疗、靶向治疗与免疫治疗等治疗方法取得了重大突破,但神经胶质瘤患者的预后仍然较差[2, 3]。随着科学技术的不断发展,神经胶质瘤的诊断与治疗趋于基因和分子水平,寻找特异性的临床标志物有助于神经胶质瘤的早期诊断和准确预后,为个体化治疗提供了可能[4]。
铁死亡是不同于自噬与调亡的新型细胞死亡方式,其依赖于细胞内铁离子与活性氧含量的变化,引起细胞脂质过氧化,导致调节性细胞坏死[5]。Long non-coding RNA(lncRNA)是一类长度大于200个核苷酸、以缺少或者无开放阅读框架为特征的RNA分子,是体内重要的信号分子,调控基因的表达,参与各种生物调控过程如转录沉默、染色体修饰等,与肿瘤发生、发展和转移等密切相关[6]。目前,国内外对铁死亡相关lncRNA的研究较少,有研究表明,LINC00618可以增加铁离子与活性氧的水平并调控相关基因导致肿瘤细胞铁死亡,同时改善抗肿瘤药物的耐药性,提示铁死亡相关lncRNA在肿瘤治疗中的重要性[7]。lncRNA可以在外泌体中选择性地包装、分泌和在细胞之间转移,且外泌体能够穿过血脑屏障(Blood-Brain Barrier,BBB)并调节神经胶质瘤增殖、侵袭、血管生成、免疫逃逸和治疗抵抗等,展现lncRNA作为神经胶质瘤新型的生物标志物与潜在治疗靶点的巨大潜力[8]。然而,通过铁死亡相关lncRNA评估神经胶质瘤患者预后与生存率的研究仍较少。
本研究将通过生物信息学方法,分析从癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库(https://portal.gdc.cancer.gov/)中获得的神经胶质瘤患者的转录组数据与临床数据,借助基因共表达等方法寻找铁死亡相关lncRNA,通过生存分析、独立预后分析、受试者工作特征曲线(Receiver Operating Characteristic Curve,ROC Curve)、列线图、基因富集分析(Gene Set Enrichment Analysis,GSEA)等方法,评估铁死亡相关lncRNA在神经胶质瘤预后中的价值及临床意义,通过TIMER、CIBERSORT、CIBERSORT-ABS等方法分析免疫浸润情况,进一步对免疫功能、免疫检查点以及N6-甲基腺苷(m6A)甲基化差异进行分析,为神经胶质瘤患者的基因治疗与靶向治疗提供新策略。
1 材料与方法 1.1 获得神经胶质瘤转录组数据与筛选铁死亡相关lncRNA从TCGA数据库抽取698名神经胶质瘤患者的转录组数据与临床数据,通过FerrDB数据库(http://www.zhounan.org/ferrdb/)获得目前已知的259个铁死亡基因,通过基因共表达的方法计算铁死亡基因与lncRNA的关联系数,根据|cor|>0.4,P-value < 0.05进行初步筛选,再通过单因素Cox回归分析结果再次筛选,得到预后相关的铁死亡相关lncRNA,根据LASSO Cox回归分析与铁死亡相关lncRNA的表达情况计算每个样本的风险评分,通过cytoscape 3.8.2软件对上述结果绘制lncRNA-基因网络图。
1.2 风险评分独立预后分析将风险评分与临床特征(年龄、性别、疾病分型)进行单因素独立预后分析与多因素独立预后分析,通过R软件绘制森林图,并通过绘制ROC曲线验证观察该模型预测患者生存情况的准确性,验证独立预后分析结果,绘制列线图预测患者生存率。
1.3 高、低风险组生存分析根据风险得分的中位值将698个样本分为高风险组与低风险组,对高、低风险组结合临床生存数据进行Kaplan-Meier生存分析,通过R软件绘制生存曲线与风险曲线,绘制热图,对lncRNA在高、低风险组总的表达情况进行可视化。
1.4 高、低风险组基因富集分析对高、低风险组神经胶质瘤患者的基因表达矩阵进行基因ID转换,通过GSEA 4.1.0软件对高、低风险组进行基因富集分析,设置GSEA分析参数,得到GSEA结果后根据归一化富集分数(Normalized Enrichment Score, NES)>1、名义上(Nominal, NOM)P < 0.05,错误发现率(False Discovery Rate, FDR)q < 0.25进行筛选。
1.5 高、低风险组免疫与m6A相关基因差异分析从TIMER数据库获得TCGA数据库免疫浸润样本,通过TIMER、CIBERSORT、CIBERSORT-ABS、QUANTISEQ、MCPCOUNTER、XCELL、EPIC等方法,分析包括B细胞、CD4+T细胞、CD8+T细胞等免疫细胞在高、低风险组神经胶质瘤患者中的免疫浸润情况,通过R软件制作热图,对高、低风险组的免疫浸润情况进行可视化分析,计算高、低风险组免疫功能、免疫检查点、m6A相关基因的差异,通过R软件绘制箱线图进行可视化分析。
2 结果与分析 2.1 铁死亡相关lncRNA的筛选经过初步筛选,通过基因共表达的方法获得铁死亡相关lncRNA共2 536个,再经过单因素Cox回归分析,共确定14个预后相关的铁死亡相关lncRNA,绘制lncRNA-基因网络图(图 1),该14个lncRNA(包括AC002116.2、LINC02100、LINC00900、AL441992.1、LINC01426、CYTOR、AP001486.2、LINC01503、MIR4435-2HG、OSMR-AS1、CRNDE、AC022613.1、AL133415.1、WAC-AS1)与94个铁死亡基因有关联,将这14个lncRNA作为后续的研究对象,对其在698个胶质瘤样本中的表达情况与LASSO Cox回归分析结果进行风险评分。
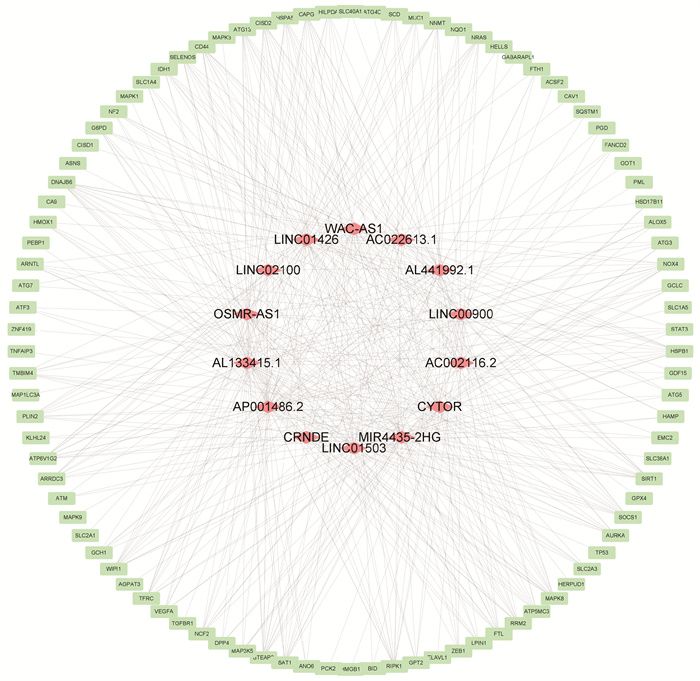
|
| 图 1 14个铁死亡相关lncRNA-基因网络图 Fig. 1 14 ferroptosis-related lncRNA-gene network map |
2.2 风险评分独立预后分析
风险评分与各种临床特征进行单因素、多因素独立预后分析,如图 2(a)、图 2(b)所示,P值均小于0.001,说明风险评分结果可以作为一个独立的特征来预测神经胶质瘤患者的预后。图 2(c)、图 2(d)的ROC曲线表明,风险评分的曲线下面积(Area Under the Curve,AUC)为0.882,高于年龄、性别、疾病分型这些临床特征,表明风险评分相较于年龄、性别、疾病分型这些临床特征,更可以预测患者疾病生存率,且风险评分1,2,3 a的ROC曲线中的AUC值均大于0.7,均可以有效预测患者生存率。根据以上结果,如图 2(e)所示,绘制列线图来预测临床中患者的生存率,若为53岁左右的G3期女性高风险患者,根据列线图可以评分为196分,其对应的生存时间小于5 a的概率为0.873,小于3年的概率为0.614,小于1年的概率为0.157,可为临床中预测患者的生存率提供参考。
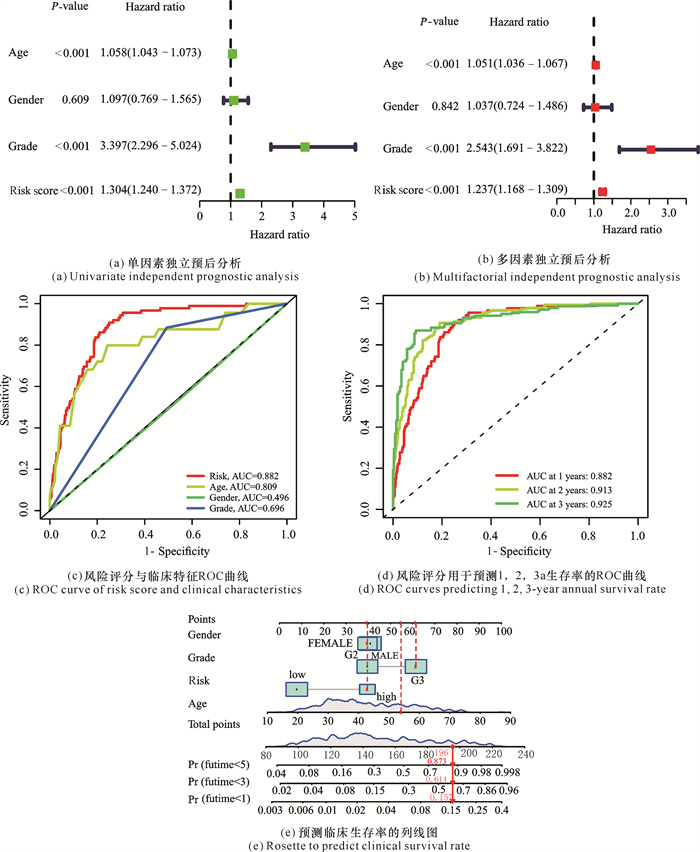
|
| 图 2 风险评分预后分析结果 Fig. 2 Prognosic analysis results of risk scores |
2.3 高、低风险组生存分析
根据风险评分的中位值将698个样本分为高、低风险两组,根据Kaplan-Meier生存分析,如图 3(a)所示,高风险组的生存率显著低于低风险组,差异具有统计学意义。同时在风险曲线图 3(b)中可以看到,698个神经胶质瘤患者中高风险组的患者死亡数更高,且AC002116.2、LINC02100、LINC00900、AL441992.1、LINC01426、CYTOR、LINC01503、MIR4435-2HG、OSMR-AS1、CRNDE、AC022613.1、AL133415.1在高风险组中的表达相对低风险组显著升高,而AP001486.2、WAC-AS1在低风险组的表达相对高风险组显著升高,且在高、低风险组中,年龄和性别均有显著差异。

|
| 图 3 高、低风险组生存分析结果 Fig. 3 Survival analysis results of high and low risk groups |
2.4 高、低风险组基因富集分析
高、低风险组的GSEA结果如图 4所示。高风险组中同种异体移植排斥、DNA复制、错配修复等较低风险组显著活跃[图 4: (a)-(c)],而低风险组中长时程增强效应、磷脂酰肌醇信号系统、长时程抑制效应等较高风险组显著活跃[图 4: (d)-(f)]。
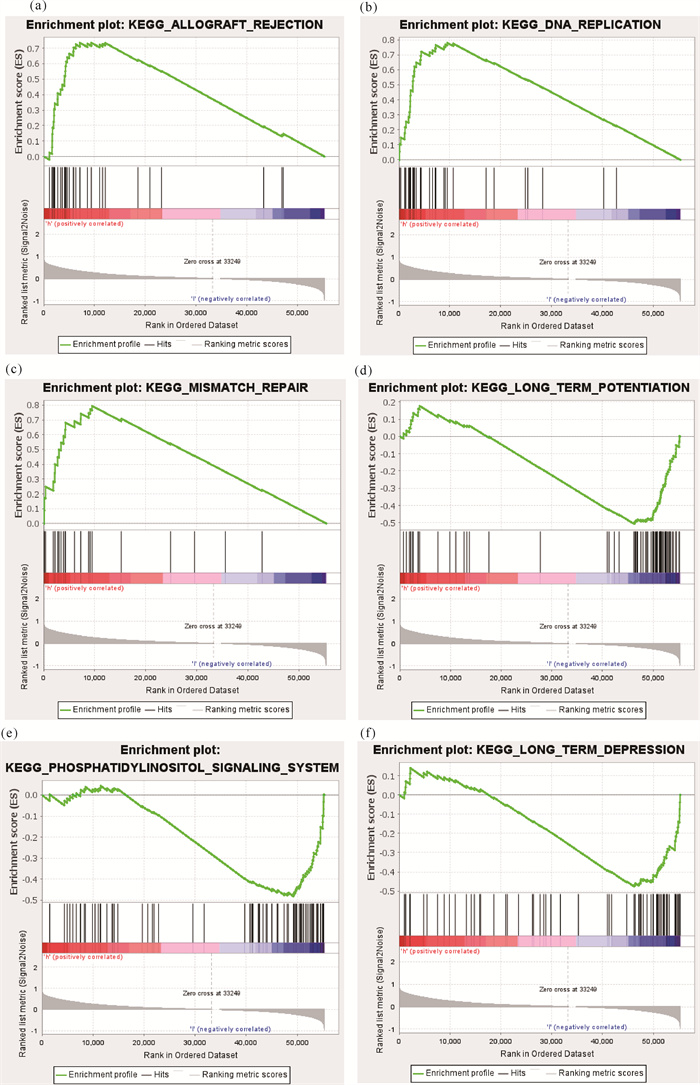
|
| (a)-(c)上调前三位;(d)-(f)下调前三位 (a)-(c) The top three up-regulated; (d)-(f) The top three down-regulated 图 4 基因富集分析结果 Fig. 4 Results of GSEA |
2.5 高、低风险组免疫及m6A相关基因的差异
基于TIMER、CIBERSORT、CIBERSORT-ABS、QUANTISEQ、MCPCOUNTER、XCELL、EPIC等方法,高、低风险组的免疫浸润如图 5所示,在免疫功能上高、低风险组的免疫检查点(Check-point)、趋化因子受体(C-C Chemokine Receptor,CCR)、人类白细胞抗原(Human Leukocyte Antigen,HLA)等均具有显著差异,考虑高、低风险组在免疫检查点上有显著差异,对免疫检查点作进一步分析,可以观察到免疫检查点PDCD-1(即PD-1)、CD44、CTLA4等在高、低风险组均具有显著差异,且大部分的免疫检查点在高风险组中显著高于低风险组。m6A相关基因FTO、ALKBH5、METTL14、METTL3、HNRNPC、WTAP、YTHDF2、RBM15、YTHDC1、YTHDF1、ZC3H13、YTHDC2的表达在高、低风险组中也具有显著差异。
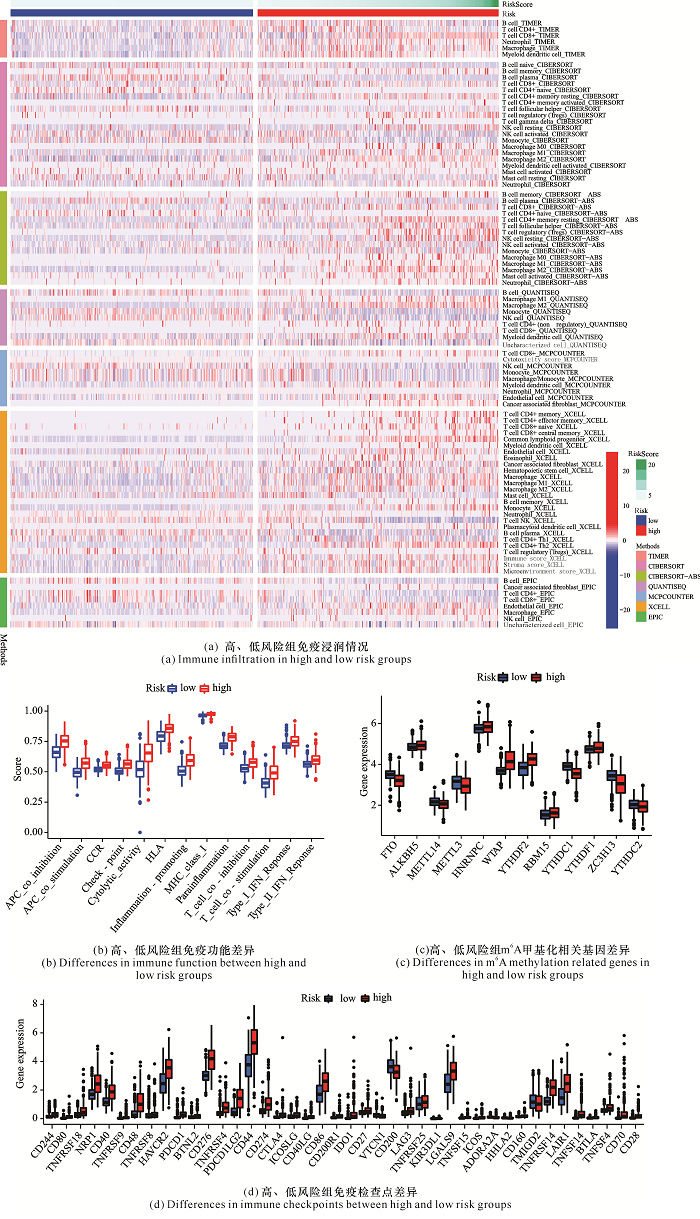
|
| 图 5 高、低风险组免疫及m6A相关基因的差异 Fig. 5 Differences of immunity and m6A related genes in the high and low risk groups |
3 讨论
神经胶质瘤是一种恶性程度极高、侵袭性极强的恶性肿瘤,目前的治疗手段仍无法根治,且不同的患者其预后也不相同。随着肿瘤的基因治疗与免疫治疗的不断发展,目前迫切需要寻找新的生物标志物协助临床医生完善神经胶质瘤的治疗方案。生物信息学是信息技术与生物医药交叉融合发展的学科,应用生物信息学方法,对生物学数据进行分析,研究其生物过程[9],同时可预测有效的治疗靶点,指导疾病的诊断、治疗、预后等,对药物的研发、个体化治疗有重大意义[10]。
肿瘤研究的主要挑战之一是如何有效杀死肿瘤细胞,同时不影响正常细胞。肿瘤细胞为了促进生长,与正常细胞相比,肿瘤细胞内铁的需求增加,这种铁依赖性的特性会使肿瘤细胞更容易受到铁催化的坏死,引起肿瘤细胞铁死亡,同时增敏化学治疗药物,减少耐药,提示铁死亡作用机制在治疗肿瘤领域的巨大潜力[11]。lncRNA虽然不编码蛋白质,但是在生物体内可以与某些miRNA结合,作为竞争内源性RNA调控相关基因的表达,参与生物体内各项生物过程[12]。近年来,有研究报道lncRNA可以通过影响铁死亡相关的p53信号通路,抑制铁死亡相关基因SLC7A11的表达来促进肿瘤细胞铁死亡[13],进一步表明lncRNA对调控肿瘤细胞铁死亡有重要作用。lncRNA在多种癌症诊断、预后与治疗中有非常重要的意义,有研究表明lncRNA与神经胶质瘤患者的肿瘤大小、WHO分级和预后相关,并且提出lncRNA可以作为神经胶质瘤临床诊断、治疗和预后的潜在分子生物标志物[14]。因此,本研究旨在研究铁死亡相关lncRNA在神经胶质瘤预后中的价值及临床意义。
本研究通过生物信息学的分析方法共筛选出14个铁死亡相关lncRNA,包括AC002116.2、LINC02100、LINC00900、AL441992.1、LINC01426、CYTOR、AP001486.2、LINC01503、MIR4435-2HG、OSMR-AS1、CRNDE、AC022613.1、AL133415.1、WAC-AS1,它们在高、低风险组中的表达均有差异。除AP001486.2、WAC-AS1外,其余12个lncRNA在高风险组中的表达显著高于低风险组。已有研究表明CRNDE、CYTOR可作为肿瘤患者的预后生物标志物[15, 16],本研究结果与现有研究结果相符,证明通过生物信息学方法筛选出的14个lncRNA作为预后生物标志物的可靠性。
为了进一步探讨筛选出来的14个lncRNA在神经胶质瘤中预后的临床意义,本研究通过单因素与多因素Cox分析,得到的风险评分可作为独立的预后因子,与年龄、疾病分型相同,可以评估患者预后情况,ROC曲线验证独立预后分析的结果,同时绘制列线图可预测临床中神经胶质瘤患者的生存率。Kaplan-Meier生存曲线与风险曲线进一步验证筛选出来的14个lncRNA在神经胶质瘤患者中的预后价值,高风险组的患者表现出较低的生存率,低风险组的患者表现出较高的生存率,两者之间的差异具有统计学意义。最后通过基因富集分析,研究高、低风险组的患者其生物学功能与相关信号通路的差异,发现高风险组中同种异体移植排斥、DNA复制、错配修复等显著活跃,而低风险组磷脂酰肌醇信号系统显著活跃。考虑到磷脂酰肌醇信号系统涉及广泛的细胞过程,包括生长、代谢、分化、增殖等,且与免疫细胞的正常功能和分化有着重要关系[17],本研究进一步对高、低风险组患者中免疫相关的差异进行分析。
肿瘤浸润免疫细胞是促进和调节肿瘤发展和生长的复杂微环境的重要部分,影响着肿瘤的诊断、生存和临床治疗敏感性等,近年来在中枢神经系统肿瘤中浸润免疫细胞取得了重要进展,有研究表明巨噬细胞、淋巴细胞(特别是T细胞和NK细胞,以及较少见的B细胞)对中枢神经系统肿瘤的浸润程度显著[18]。本研究通过不同的免疫浸润分析方法分析高、低风险组的免疫浸润情况,高风险组的免疫浸润程度较低风险组显著。进一步对高、低风险组的免疫功能进行比较,高风险组的免疫检查点(Check-point)、趋化因子受体(CCR)、人类白细胞抗原(HLA)等免疫功能均显著高于低风险组,考虑到高、低风险组存在显著差异,且近年来免疫检查点抑制剂对肿瘤治疗的重要性,本研究进一步分析高、低风险组免疫检查点的差异。
免疫检查点是在免疫应答过程中,调节T细胞受体抗原识别的共刺激或者抑制信号,在正常生理条件下,免疫检查点可以调节免疫功能,防止机体免疫功能过度活化,避免自身免疫性疾病的发生。但免疫检查点过度表达时,免疫功能就会受到抑制;反之,当免疫检查点表达下降时,机体的免疫功能也会异常。肿瘤细胞可以影响免疫检查点,使得抗原不能有效被提呈,机体免疫功能不能被有效激活,从而抑制免疫细胞的功能,起到免疫逃逸的作用[19]。目前, 临床中已出现取得较好治疗效果的免疫检查点抑制剂,如PD-1单克隆抗体、CTLA-4抗体等,可以解除肿瘤细胞对免疫检查点的作用,激活机体的免疫系统,有效杀伤肿瘤细胞[20]。通过对高、低风险组免疫检查点的差异进行分析,发现PDCD-1(即PD-1)、CD44、CTLA-4等均有显著差异,且高风险组中大部分的免疫检查点显著高于低风险组。
RNA甲基化是一种可逆的RNA转录后修饰,对许多生物过程有重大的影响。m6A被认为是最常见的真核细胞mRNA甲基化修饰类型之一,存在于多种生物体中。m6A甲基化在大脑的不同发育阶段控制神经干细胞的发育及其向神经元、胶质细胞(如少突胶质细胞和星形胶质细胞)的分化[21]。有研究表明,m6A修饰可以通过多种机制在胶质瘤中发挥关键作用,为胶质瘤的早期诊断、靶向治疗、改善患者预后提供更多可能,具有重要的临床意义[22]。因此, 本研究对高、低风险组中m6A甲基化相关基因的差异作进一步分析,发现高、低风险组中FTO、ALKBH5、METTL14、METTL3、HNRNPC、WTAP、YTHDF2、RBM15、YTHDC1、YTHDF1、ZC3H13、YTHDC2均有显著差异,提示靶向上述基因m6A甲基化的基因治疗可为神经胶质瘤的治疗提供新策略。
4 结论根据筛选出的14个铁死亡相关lncRNA在样本中的表达情况与LASSO Cox回归分析结果进行风险评分,将样本分为高风险组与低风险组,高、低风险组的生存曲线、风险曲线等均具有显著差异,风险评分结果可作为独立预后因素,提示上述铁死亡相关lncRNA可以作为神经胶质瘤新的生物标志分子。此外,本研究对高、低风险组免疫浸润、免疫功能、免疫检查点、m6A甲基化的差异进行了探讨,为神经胶质瘤患者的基因治疗与免疫治疗提供了有价值的参考。
| [1] |
XU S C, TANG L, LI X Z, et al. Immunotherapy for glioma: Current management and future application[J]. Cancer Letters, 2020, 476: 1-12. DOI:10.1016/j.canlet.2020.02.002 |
| [2] |
MIYAUCHI J T, TSIRKA S E. Advances in immunotherapeutic research for glioma therapy[J]. Journal of Neurology, 2018, 265(4): 741-756. DOI:10.1007/s00415-017-8695-5 |
| [3] |
中国医师协会脑胶质瘤专业委员会, 上海市抗癌协会神经肿瘤分会. 中国中枢神经系统胶质瘤免疫和靶向治疗专家共识(第二版)[J]. 中华医学杂志, 2020, 100(43): 3388-3396. DOI:10.3760/cma.j.cn112137-20200714-02117 |
| [4] |
LUO Y, HOU W T, ZENG L, et al. Progress in the study of markers related to glioma prognosis[J]. European Review for Medical and Pharmacological Sciences, 2020, 24(14): 7690-7697. |
| [5] |
MOU Y H, WANG J, WU J C, et al. Ferroptosis, a new form of cell death: Opportunities and challenges in cancer[J]. Journal of Hematology & Oncology, 2019, 12: 34. DOI:10.1186/s13045-019-0720-y |
| [6] |
ANASTASIADOU E, FAGGIONI A, TRIVEDI P, et al. The nefarious nexus of noncoding RNAs in cancer[J]. International Journal of Molecular Sciences, 2018, 19(7): 2072. DOI:10.3390/ijms19072072 |
| [7] |
WANG Z L, CHEN X W, LIU N, et al. A nuclear long non-coding RNA LINC00618 accelerates ferroptosis in a manner dependent upon apoptosis[J]. Molecular Therapy, 2021, 29(1): 263-274. DOI:10.1016/j.ymthe.2020.09.024 |
| [8] |
CHENG J, MENG J L, ZHU L, et al. Exosomal noncoding RNAs in glioma: Biological functions and potential clinical applications[J]. Molecular Cancer, 2020, 19: 66. DOI:10.1186/s12943-020-01189-3 |
| [9] |
石桃雄, 赵佳利, 田桃, 等. 苦荞CCT基因家族生物信息学及表达分析[J]. 广西科学, 2021, 28(3): 301-309. |
| [10] |
佟春晓, 陈海英, 孙曼妮, 等. 子痫前期患者胎盘组织中环状RNA差异表达的生物信息学分析[J]. 中国医科大学学报, 2021, 50(8): 673-677. |
| [11] |
HASSANNIA B, VANDENABEELE P, VANDEN BERGHE T. Targeting ferroptosis to iron out cancer[J]. Cancer Cell, 2019, 35(6): 830-849. DOI:10.1016/j.ccell.2019.04.002 |
| [12] |
WANG L Y, CHO K B, LI Y, et al. Long noncoding RNA (lncRNA)-mediated competing endogenous RNA networks provide novel potential biomarkers and therapeutic targets for colorectal cancer[J]. International Journal of Molecular Sciences, 2019, 20(22): 5758. DOI:10.3390/ijms20225758 |
| [13] |
MAO C, WANG X, LIU Y T, et al. A G3BP1-interac- ting lncRNA promotes ferroptosis and apoptosis in cancer via nuclear sequestration of p53[J]. Cancer Research, 2018, 78(13): 3484-3496. |
| [14] |
ZHOU Q, LIU J, QUAN J, et al. lncRNAs as potential molecular biomarkers for the clinicopathology and prognosis of glioma: A systematic review and meta-analysis[J]. Gene, 2018, 668: 77-86. DOI:10.1016/j.gene.2018.05.054 |
| [15] |
LIANG J Y, WEI X, LIU Z H, et al. Long noncoding RNA CYTOR in cancer: A TCGA data review[J]. Clinica Chimica Acta, 2018, 483: 227-233. DOI:10.1016/j.cca.2018.05.010 |
| [16] |
MOMTAZMANESH S, REZAEI N. Long non-coding RNAs in diagnosis, treatment, prognosis, and progression of glioma: A state-of-the-art review[J]. Frontiers in Oncology, 2021, 11: 712786. DOI:10.3389/fonc.2021.712786 |
| [17] |
PREITE S, GOMEZ-RODRIGUEZ J, CANNONS J L, et al. T and B-cell signaling in activated PI3K delta syndrome: From immunodeficiency to autoimmunity[J]. Immunological Reviews, 2019, 291(1): 154-173. DOI:10.1111/imr.12790 |
| [18] |
DOMINGUES P, GONZÁLEZ-TABLAS M, OTERO Á, et al. Tumor infiltrating immune cells in gliomas and meningiomas[J]. Brain, Behavior, and Immunity, 2016, 53: 1-15. DOI:10.1016/j.bbi.2015.07.019 |
| [19] |
DARVIN P, TOOR S M, NAIR V S, et al. Immune checkpoint inhibitors: Recent progress and potential biomarkers[J]. Experimental & Molecular Medicine, 2018, 50(12): 1-11. |
| [20] |
李向敏, 于莉, 张晓晔. 免疫检查点在肿瘤治疗中的作用机制及研究进展[J]. 现代肿瘤医学, 2018, 26(12): 1962-1965. DOI:10.3969/j.issn.1672-4992.2018.12.041 |
| [21] |
PAN T, WU F, LI L L, et al. The role m6A RNA me- thylation is CNS development and glioma pathogenesis[J]. Molecular Brain, 2021, 14(1): 119. DOI:10.1186/s13041-021-00831-5 |
| [22] |
GALARDI S, MICHIENZI A, CIAFRÈ S A. Insights into the regulatory role of m6A epitranscriptome in glioblastoma[J]. International Journal of Molecular Sciences, 2020, 21(8): 2816. DOI:10.3390/ijms21082816 |



