2. 广西高校并行分布式计算技术重点实验室,广西南宁 530004
2. Key Laboratory of Parallel and Distributed Computing in Guangxi Colleges and Universities, Nanning, Guangxi, 530004, China
蛋白质是组成生物有机体细胞、组织的重要成分,是生命的物质基础,也是生命活动的执行者。虽然有些蛋白质是以单体的形式发挥作用,但是大部分生物有机体蛋白质是和伴侣分子或与其他蛋白质一起发挥作用。在生命活动中,蛋白质及其相互作用是必不可少的,它们是细胞进行一切代谢活动的基础。蛋白质组学从整体角度分析细胞内动态变化的蛋白质组分、表达水平与修饰状态,了解蛋白质相互作用与联系,揭示蛋白质功能与细胞生命活动规律。在后基因组时代,揭示蛋白质相互作用关系、建立相互作用关系网络图,并从中挖掘功能子结构和预测蛋白质功能,已成为蛋白质组学研究的热点。
随着酵母双杂交(Y2H)[1]技术、串联亲和纯化-质谱(TAP-MS)[2]技术和蛋白质芯片(Protein Chip)[3]技术等高通量实验技术的飞速发展,研究人员掌握了大量的蛋白质相互作用(Protein-Protein Interaction,PPI)数据。同时,基于上述湿式实验室技术产生的PPI数据,研究人员利用计算机手段进一步推断出更多的PPI数据,这些推断出来的PPI数据和经实验核实的PPI数据共同被收录在开放数据库中。目前,收录PPI数据的开放数据库有酵母蛋白质组数据库(YPD)[4]、慕尼黑蛋白质序列信息数据库(MIPS)[5]、分子交互数据库(MINT)[6]、相互作用数据库(IntAct)[7]、相互作用蛋白质数据库(DIP)[8]、生物分子交互网络数据库(BIND)[9]、生物网格数据库(BioGRID)[10]、人类蛋白质参考数据库(HPRD)[11]、人类蛋白质交互数据库(HPID)[12]和果蝇蛋白质交互数据库(DroID)[13]等。此外,数据库Stitch[14]和STRING[15]还提供文本挖掘分析服务。这些开放数据库收录的PPI数据为分析挖掘蛋白质复合物及功能模块提供了基础。
蛋白质及其相互作用可用蛋白质相互作用网络PPIN表示。而PPIN可用无向简单图(Graph)来建模。一个无向简单图可表示为G=(V, E),其中V表示结点集,E表示结点间连接的边集,即E={(i, j)|i, j∈V}。蛋白质相互作用网络图的结点表示蛋白质,边表示蛋白质相互作用。将PPI数据建模为蛋白质相互作用网络后,则可利用图理论对蛋白质相互作用网络进行深入分析,以揭示生物过程中蛋白质复合物、功能模块的拓扑结构特征和功能组织机理。
蛋白质复合物是在细胞内生物过程中同时同地物理绑定彼此的蛋白质组,它对应蛋白质相互作用网络中具有生物学意义的功能子图。蛋白质功能模块则是参与某一特定生物过程的全体蛋白质,其中的蛋白质可以在不同时间不同场所相互作用[16]。在过去二十多年里,基于蛋白质相互作用网络预测蛋白质复合物和功能模块的算法层出不穷。随着AI技术的发展和注入,蛋白质复合物和功能模块预测必将迎来新一轮的研究热潮。
1 蛋白质复合物和功能模块预测算法按照历史发展脉络,蛋白质复合物和功能模块预测算法的研究先后形成两个并存发展方向:静态蛋白质相互作用网络(SPIN)方向和动态蛋白质相互作用网络(DPIN)方向。随着研究的深入,蛋白质复合物和功能模块的生物特性及其在蛋白质相互作用网络中的拓扑特征不断被用于预测算法中。稠密连接和核心-附件结构是蛋白质复合物和功能模块在蛋白质相互作用网络中呈现出的基本拓扑特征,而蛋白质复合物和功能模块预测算法所利用的生物特性有基因共表达、蛋白质共定位、基因本体(GO)相似性、互斥相互作用、结构域相互作用等。下面将围绕拓扑特征和生物特性回顾基于静态蛋白质相互作用网络的复合物预测算法。
1.1 基于SPIN的蛋白质复合物预测算法在静态蛋白质相互作用网络中,蛋白质复合物呈现稠密连接的特征,这是其在静态蛋白质相互作用网络中的基本特征。因此,早期预测蛋白质复合物的算法大多数依靠蛋白质复合物的拓扑特性挖掘稠密连接子图,并以此作为蛋白质复合物。为进一步提高预测的准确性,不同的生物学特征陆续被引入预测算法设计策略中。
1.1.1 基于复合物拓扑特征的SPIN蛋白质复合物预测算法有研究基于“团”的概念设计算法,在蛋白质相互作用网络中预测蛋白质复合物[16-21]。为发现蛋白质网络中稠密连接子图,Spirin等[16]利用极大团枚举、超顺磁性聚类(Super Paramagnetic Clustering,SPC)和蒙特卡洛(Monte Carlo,MC)等方法来预测蛋白质复合物/功能模块。由于缺少时空信息,Spirin等[16]预测的结果无法区分复合物和功能模块。Liu等[17]基于极大团的概念提出聚类算法CMC,该方法首先使用深度优先搜索DFS策略枚举所有的极大团,然后对搜索得到的团打分并按降序排列,最后将两个重叠团中的低分团合并到高分团中,以获得稠密连接的大子图来生成复合物。CMC的打分机制使得算法对随机噪声交互具鲁棒性,从而提高其预测蛋白质复合物的能力。众所周知,搜索极大团是NP-难(Non-deterministic Polynomial time-Hard, NP-hard)问题,所以枚举极大团的算法仅适用于小规模且稀疏的蛋白质相互作用网络。为获得可靠的蛋白质相互作用网络,Chua等[18]提出蛋白质复合物预测算法PCP,该算法利用功能相似度(Functional Similarity,FS)过滤低权值直接相互作用并引入高权值间接相互作用,以改善蛋白质相互作用网络,并在以这种方式修改的蛋白质相互作用网络中获得较好的复合物预测精度。与上述算法不同的是,局部团合并算法LCMA基于稠密连接图搜索局部团,然后合并局部团以预测蛋白质复合物,该方法对不完整交互数据不敏感,并能平衡查全率(Recall)和查准率(Precision),可以获得较高的F值(F-Measure)和效率[19]。考虑交互的不完整,DECAFF算法将搜索极大团松弛为搜索局部稠密邻域[20]。相比而言,DECAFF算法的整体性能优于LCMA算法。PE-WCC算法以最大团作为复合物的核心,添加与核心蛋白质连接数达自身连接度一半以上的蛋白质,最后形成复合物[21]。虽然该算法能以较高准确度预测更多新复合物,但是对交互数据的可靠性评估会带来额外的时间开销。
基于功能关联的思想,可以使用种子扩展策略设计算法来预测蛋白质复合物。MCODE算法始于高权重结点,以顶点权百分率VWP扩展结点来形成初始聚类,并删掉密度低的子图以生成重叠聚类[22],但MCODE算法产生的重叠聚类数量较少且规模较大。DPClus算法同样选择高权重结点作为种子,扩展能维持一定稠密度水平的外部高连接度结点以形成聚类,从而在蛋白质相互作用网络中预测蛋白质复合物[23]。与MCODE算法类似,ClusterONE算法[24]始于选定的种子蛋白质并采用贪心策略扩张分组,以获得内连接比例高的聚类,在合并高度重叠的分组后产生蛋白质复合物。由于考虑重叠复合物,ClusterONE算法获得的结果质量比MCODE算法更好。同样地,为维持一定的稠密度水平,SPICi算法以边为种子,按贪心策略扩展高支持度顶点以形成聚类。SPICi算法的快速性使其能很好地适应规模渐增的稠密功能性生物网络,但缺点是不能检测重叠聚类[25]。PROCODE算法采用贪心策略扩展最大共邻边以产生初始聚类,然后合并初始聚类以产生维持一定稠密度水平的蛋白质复合物[26]。Wang等[27]在提出的ClusterM算法中考虑拓扑特性和算法可扩展性,整合网络拓扑结构和蛋白质序列相似性信息,以识别多物种蛋白质相互作用网络中的保守蛋白质复合物。
马尔可夫聚类(MCL)算法以模拟网络流的随机游走方式,对网络转换概率矩阵交替地执行扩张和膨胀操作,以强化稠密连接区域的网络流,弱化稀疏连接区域的网络流,从而实现网络流随机游走概率的分配与分化,最终根据不同的概率完成图的划分并达到聚类的目的[28]。Brohee等[29]指出,MCL算法因对图变化具有显著鲁棒性且使用参数相对较少而广为流行。Vlasblom等[30]、R-MCL[31, 32]、SR-MCL[33]和F-MCL[34]对PPI网络的(加权)邻接矩阵交替地执行扩张和膨胀操作,以实现PPI网络的划分,从而预测蛋白质复合物和功能模块。
酵母复合物在蛋白质相互作用网络中呈现核心-附件结构(Core-attachment structure)特征,其核心是指构成复合物中心单元的稠密连接功能性蛋白质,而附件则是指围绕在核心蛋白质周围并协助参与相应生物过程的蛋白质[35]。Ahmed等[36]提出一个与“核心-附件结构”同义的术语“核心-外围结构”,并指出蛋白质复合物由核心和外围两部分组成:核心部分是一个稠密连接区域,该区域的蛋白质彼此高度连接,而外围部分则是与核心连接较弱的蛋白质。文献[37-41]根据核心-附件结构特性预测蛋白质复合物。Leung等[37]提出的CORE算法按两蛋白质共邻数确定共核心概率并形成双蛋白核心,随后迭代地合并双蛋白核心、三蛋白核心等,依次类推以生成相互不重叠的蛋白质核心集,最后将与半数核心蛋白质交互的附件蛋白质添加到核心中以形成复合物。COACH算法首先确定高连接度结点,并从其稠密邻域中选定结点作为蛋白质复合物核心,然后用类似于CORE算法的方式将附件添加到核心中,从而获得蛋白质复合物[38]。不同于CORE算法,COACH算法产生的不同复合物核心存在重叠。MCL-CAw算法利用MCL能划分网络的特点,将MCL检测到的稠密区域作为蛋白质核心,然后选择与核心连接度高的结点作为附件进行添加,以生成蛋白质复合物[39, 40]。由于不同蛋白质核心的外围存在相同的蛋白质,所以MCL-CAw算法有可能将相同的附件蛋白质添加到不同的蛋白质核心中,从而允许形成重叠复合物。Peng等[41]提出的WPNCA算法根据核心-附件结构并采用加权页序-蚕食策略,首先选择排序靠前的m个顶点来形成稠密连接子图,然后以形成的稠密连接子图作为核心,继而添加与核心有足够强相互作用的附件蛋白质,最终获得可能重叠的蛋白质复合物。通过利用核心-附件结构,上述几种蛋白质复合物预测算法在F-Measure指标上获得不同程度的提高。
1.1.2 基于生物学特征加权的SPIN蛋白质复合物预测算法复合物在蛋白质相互作用网络中对应于具有生物功能的拓扑子结构,因此在算法中可以融合基因本体(GO)[42]功能标注、基因表达和蛋白质亚细胞定位等生物学数据以预测蛋白质复合物。
RNSC算法用基于GO功能标注的功能同质度、聚类规模和密度3个指标,对被划分的子网进行筛选,并预测蛋白质复合物[43]。但由于策略过于简化且不完善,RNSC算法无法预测功能同质程度低的已知复合物。相互作用蛋白质间基于GO功能标注的相似性和共邻数在OIIP算法中被用于加权蛋白质相互作用网络,从而使得蛋白质复合物预测算法具有较高的精确度,并获得较高的F-Measure指标[44]。Price等[45]分析比较6种预测算法在基于GO功能标注相似性加权的蛋白质相互作用网络中预测蛋白质复合物的优劣,结果表明绝大多数算法在经GO相似性加权后的蛋白质相互作用网络中能较准确地预测蛋白质复合物。
编码相互作用的蛋白质的基因有着相似的基因表达谱。同样地,编码复合物中蛋白质的基因更可能有相似的基因表达谱[46]。因此,根据基因表达数据的相似性可以推断蛋白质功能,也可用于预测蛋白质-蛋白质交互[47-49]。Feng等[50]和Tang等[51]利用基因表达数据研究复合物预测算法。GFA算法使用微阵列基因表达数据加权蛋白质,并保持一定的密度水平预测蛋白质复合物[50]。但GFA算法为提高预测性能而采用的多微阵列样本措施,使得算法在规模大而稠密的蛋白质相互作用网络中运行比较耗时。CMBI算法使用基因表达数据计算蛋白质间的皮尔森相关系数,再组合边聚类系数加权蛋白质相互作用网络,然后采用种子扩展策略检测蛋白质复合物,所预测的蛋白质复合物具有均衡的查准率和查全率,并有较高的F-Measure[51]。
在细胞中,蛋白质是在特定的亚细胞定位中发挥其生物学功能[52, 53],而UniProt数据库存储有蛋白质亚细胞定位数据[54, 55]。SMILE算法[56]利用蛋白质亚细胞定位数据构造亚细胞蛋白质相互作用子网,在检测出蛋白质功能模块后与蛋白质复合物对比,在敏感度Sn、阳性预测值PPV及精度Acc指标上胜过ClusterONE算法[24]和MCL算法[28]。Cheng等[57]则把蛋白质亚细胞定位数据集成至SPIN中以构造共定位蛋白质网络CLPIN,并进一步结合拓扑重叠特征构造局部拓扑重叠蛋白质网络LTOPIN,随后在LTOPIN上取得优越的蛋白质复合物预测性能。蛋白质亚细胞定位数据提供蛋白质及其相互作用的空间信息,在设计蛋白质复合物和功能模块预测算法时使用该数据是必要且值得深入研究的[58]。
此外,Rehman等[59]分析计算氨基酸的出现频度来提取复合物中蛋白质的生物学特征,并结合13个拓扑结构特征来预测蛋白质复合物。Liu等[60]运用GO功能标注、结构域相互作用、基因共表达和STRING数据库的蛋白质相互作用可靠性得分来分析6个蛋白质相互作用网络的生物学特征,并比较这些生物学特征对6个复合物检测算法的影响。Abdulateef等[61]基于基因表达数据和GO功能标注构造局部微调策略,提出优化的辅助启发模型来搜索边界内外局部空间,以提高进化算法检测复合物的可靠性,并收敛获得更多的可靠解,以提高复合物预测准确性。蛋白质复合物由多个蛋白质组成,其中蛋白质间的关系是一种群体关系,因此Zhang等[62]利用GO功能标注、基因表达和蛋白质亚细胞定位等生物特征数据,从群体关系的角度量化判定复合物中蛋白质的功能相似、联合共定位和联合共表达,并在精确匹配数、综合得分及生物显著性上优于对比算法。Younis等[63]提出一个新的序列前向特征选择算法SFFS,该算法提取13个在蛋白质相互作用网络中呈现出的拓扑特征和150个氨基酸序列特征以预测蛋白质复合物,并在查准率、查全率及F-Measure上胜过对比算法。
在蛋白质相互作用网络中仅利用拓扑特征不足以准确预测蛋白质复合物。前述融合的方法利用GO功能标注、基因表达和蛋白质亚细胞定位等生物特征数据加权蛋白质间二元关系,在一定程度上提高了预测精度。但是,针对蛋白质复合物的群体关系特性,更应从群体关系的角度量化判定复合物中蛋白质的功能相似、联合共定位和联合共表达等特征。
1.1.3 融合蛋白质结构域相互作用的SPIN蛋白质复合物预测算法蛋白质物理地相互作用是通过蛋白质结构域相互作用DDI (Domain-Domain Interaction)来实现的[64]。Jung等[64, 65]使用蛋白质结构域交互界面残基数据,根据蛋白质结构域相互作用的互斥性或竞争性提出蛋白质互斥相互作用MEIs (Mutually Exclusive Interactions)的概念,在排除互斥或竞争的蛋白质相互作用后构造同时相互作用蛋白质网络,从而在预测蛋白质复合物时排除互斥相互作用。Jung等[64, 65]利用蛋白质相互作用的相容性确保复合物中蛋白质相互作用是同时发生而不是分时出现的。如果预测复合物中蛋白质的每一个结构域仅为一个蛋白质相互作用所使用,那么所预测的复合物很可能形成一个真的蛋白质复合物[66, 67]。因此,Ozawa等[68]在排除结构域竞争的基础上,基于一个DDI支持一个PPI的假设,运用二元整数规划搜索DDIs的最佳组合来核实预测的蛋白质复合物是否为真复合物,并将来源于公共数据库的高置信DDI数据用于蛋白质复合物预测算法的后处理阶段,使复合物预测算法获得两倍精度的提高和超过25%的性能改善。基于同样的假设和复合物预测流程,Ma等[69]增加DDI预测阶段,然后按最大匹配问题求解DDI的最佳组合,从而获得比Ozawa等[68]更多的DDI和更高的查全率、查准率。由此可见,从结构域竞争引起的蛋白质互斥相互作用MEIs到最佳组合或最大匹配实现DDI支持的PPI,无论是预处理还是后处理,结构域相互作用DDI数据都对蛋白质复合物的准确预测起促进作用。
综上所述,从静态蛋白质相互作用网络的拓扑结构来看,蛋白质复合物具有稠密连接、核心-附件结构等特征;从生物学角度来看,复合物的形成需要相互作用的蛋白质满足共定位、共表达、DDI支持和GO功能标注等基本条件。
1.2 基于SPIN的蛋白质功能模块预测算法蛋白质功能模块预测算法的研究也经历着丰富的发展过程,采用了与复合物预测算法类似的策略。与蛋白质复合物不同的是,构成功能模块的蛋白质及其相互作用没有同一时空约束。预测蛋白质功能模块的算法主要有基于图聚类的算法、基于层次聚类的算法、基于流模拟的算法和基于群智能聚类算法等。
1.2.1 基于图聚类预测SPIN蛋白质功能模块算法为发现蛋白质相互作用网络中稠密连接子图,Spirin等[16]提出3种经典算法。使用团枚举的算法受到蛋白质相互作用网络数据不完整的限制,超顺磁性聚类SPC算法和蒙特卡洛MC算法则可用于预测功能模块。Adamcsek等[70]在所提的Cfinder算法中首先定义k-团和双k-团的概念,并进一步定义k-团链,然后利用团渗透预测k-团,最后组合邻接k-团形成双k-团继而形成k-团链,最终实现功能模块检测。该算法能准确检测出重叠功能模块,但过高的紧密连接条件导致某些符合条件的功能模块无法被检测。Jia等[71]利用团松弛技术和2-club结构[72]对功能模块进行建模,然后按功能模块拓扑结构的属性与作用之间的关系预测功能模块。SCAN算法将大于指定阈值的两个蛋白质共邻相似性定义为结构可达,然后将多个彼此结构可达的蛋白质结点称为核心结点,最后反复添加可达结点到核心结点来扩展聚类以形成功能模块[73]。Abdullah等[74]将功能模块检测分数据预处理、团预测和最近邻搜索3个阶段进行:数据预处理阶段删除蛋白质相互作用网络中的自环和冗余交互;团预测阶段运用扩展方法获得功能富集蛋白质团;最近邻搜索阶段基于聚类系数计算模块密度,搜索与团相连且最近邻的蛋白质并加以添加,从而获得功能模块。该算法能查找到数量相当的重叠模块。Chen等[75]运用社区模块度递增策略扩展蛋白质结点来形成初始社区,然后以功能性内聚测量为指标,合并初始社区形成聚类,从而获得结构模块化和功能性内聚兼具的蛋白质功能模块。NCMine算法按照核心-外围结构,对经加权的结点使用结点度中心性指标提取近似完全子图作为功能模块[76]。Manners等[77]提出一个基于种子扩展策略的聚类算法,该算法使用相对关联得分量化基因功能同形度,构造加权共表达网络,并检测阿兹海默症共表达网络中本质重叠的功能富集调控模块。TICONE算法使用基因表达数据分析计算皮尔森相关系数,然后聚类蛋白质相互作用网络中基因表达模式相似的蛋白质结点,以预测功能富集的功能模块[78]。Shen等[79]用密度模块度取代全局模块度以评估一个功能模块内的紧密程度,并提出ADM算法。该算法克服模块屏障在模块间移动结点,并分析计算移动结点与模块的内外关联度来决定被移动结点的模块归属,然后以最大化密度模块度为目标划分网络,最终检测蛋白质功能模块。He等[80]基于核心-附件结构提出一个贪心搜索算法GSM-CA,该算法基于边权值和核心结点-附件结点判断准则,以最高权值边为种子并采用贪心策略添加核心结点,然后添加附件结点以形成功能模块。GSM-CA算法虽然具有高检测精度但是耗时,为此He等[80]进一步提出改进算法GSM-FC,该算法仅需对边遍历一次以划分功能模块,使得其在保持与GSM-CA算法同样高预测精度的同时显著减少计算时间。Jeong等[81]运用的图熵GE算法按照种子扩张过程,采用贪心策略最小化熵以优化子图模块来搜索局部最优聚类,最终形成功能模块。GE算法独立搜索聚类的过程能获得重叠功能模块,且在功能模块的预测精度和同质性的比较中优于对比算法。Zhao等[82]提出进化算法ECTG,通过组合拓扑系数和基因表达模式相似性,将蛋白质相互作用网络分解为紧密连接的子图以识别功能模块。Ying等[83]基于解旅行商问题算法LKH组合GO功能标注提出一个新预测模型LKHM,该模型首先用基于邻域的CD-distance加权PPI网络,然后用分治法求最短周游路径形成模块,最后合并GO相似模块并删除低密度模块以检测功能模块。模型LKHM继承了LKH低时间复杂度、高精度和高鲁棒性的优点,以最大化内聚度和分离度为目标检测功能内聚模块。
从团、团链、团松弛到聚集系数、功能内聚、结点度中心性、密度模块度、图熵、种子扩展等概念、指标及策略,上述算法将网络局部拓扑特征用于聚类以实现功能模块预测。
1.2.2 基于层次聚类预测SPIN蛋白质功能模块算法基于层次聚类的预测算法可对给定SPIN中的蛋白质结点集按拓扑模块性和生物功能性进行层次分解,直至实现功能内聚的模块化聚类为止,其具体实施过程可分为凝聚[84]和分裂[85]两种方案。MINE算法是一个凝聚式层次聚类的预测算法,它使用修正顶点加权策略并考虑网络模块度,通过在聚类扩张过程中避免伪邻结点的干扰,以确定模块边界[86]。UVCluster是基于距离的凝聚式层次聚类的预测算法,它基于最短路径计算两个蛋白质之间的距离,然后通过逐渐凝聚过程迭代地合并蛋白质以形成聚类并预测蛋白质功能模块[87]。Jerarca套件是UVCluster的扩展版,它融合RCluster算法和SCluster算法计算加权距离,并采用系统进化树算法UPGMA[88]和Neighbor-Joining[89]构建树状层次图,在蛋白质相互作用网络转换成树状层次图后,根据连接分布给出树状层次图的最优划分[90]。Wang等[91]提出的快速层次聚类算法HC-PIN按凝聚方案聚类以发现蛋白质相互作用网络的功能模块,该算法针对无/加权的SPIN计算边聚类值,按贪心策略检查聚类值高的边,根据内聚度将边关联的结点以凝聚方式聚类。HC-PIN算法对假阳性交互不敏感,所发现的功能模块层次与GO层次大致对应,且能发现低密度的功能模块,因此能适应较大规模的蛋白质相互作用网络。
1.2.3 基于流模拟聚类预测SPIN蛋白质功能模块算法TRIBE-MCL算法是一个以MCL原型为基础的功能模块检测算法,它使用序列相似度计算随机游走概率,利用交替执行的扩张和膨胀操作,增强密集连接区域内网络流的分布,并削弱跨密集连接区域网络流,以划分蛋白质相互作用网络,从而实现蛋白质功能模块预测[92]。Gu等[93]提出的MLS算法采用连接相似度矩阵量化蛋白质相互作用的关联强度,并利用马尔可夫聚类机制分化关联强度,从而划分连接相似度矩阵以预测功能模块。Hwang等[94]首先对蛋白质相互作用网络中每个蛋白质扰动后的信号传导行为建模为动态信号传导模型,该模型合理集成了反应率、蛋白质浓度和交互化学当量,随后组合动态信号传导模型和图拓扑设计STM算法。该算法基于簇的相似性迭代地合并高度互连的蛋白质簇以形成聚类,从而以较低的放弃率兼顾检测小而稠密或大而稀疏的生物学相关功能Gu模块。CASCADE算法用蛋白质之间的准全路径取代最短路径从而发展了STM的思想,继而在整个蛋白质相互作用网络中传播分配结点的出现概率[95]。CASCADE算法继承STM算法的优点:以较少的放弃率检测小而稠密或大而稀疏的生物学相关功能Gu模块。Inoue等[96]提出的ADMSC算法将蛋白质相互作用网络聚类作为扩散过程中的随机游走问题来分析求解,该算法使用几何映射后的结点间角度距离来度量结点间相似度,为适应网络异构性引入幂因子构造可调整扩散矩阵,并利用矩阵分解划分蛋白质相互作用网络,以预测蛋白质功能模块。
1.2.4 基于群智能聚类预测SPIN蛋白质功能模块算法基于以下事实——具有短距离的两个蛋白质靠近的可能性很大,Sallim等[97]提出一个蚁群聚类预测算法ACOPIN,该算法首次将蚁群算法运用于蛋白质相互作用网络的功能模块检测。Ji等[98]运用蚁群算法结合功能信息和拓扑特征,以检测蛋白质相互作用网络中的功能模块。然而,蚁群算法易陷于早熟的缺点会影响功能模块检测的结果。因此,在Ji等[98]的研究基础上,Ji等[99]组合蚁群优化策略和多智能体进化策略提出ACO-MAE算法,该算法在搜索可行解空间时自适应扩展子空间以删除局部最优解,从而在检测功能模块过程中克服早熟的不足。Ji等[100]提出的ACC-FMD算法以高聚类系数蛋白质为蚁群种子结点,基于蚁群概率模型将蛋白质添加到相应聚类中,通过更新相似度函数对每次迭代的最佳聚类结果进行信息遗传。Yang等[101]提出的BFO-FMD算法利用细菌觅食的5个优化机制:趋化、结合、繁殖、消除和分散,以检测蛋白质相互作用网络中的功能模块,且在确保收敛速度的同时获得较高的准确性。基于蛋白质相互作用网络结点间的最短路径,Zheng等[102]在一个简化的群体优化算法SSO中分割和过滤搜索最短路径,以生成功能模块。Lei等[103]基于传播机制提出一个人工蜂群聚类算法ABC以检测蛋白质相互作用网络中的蛋白质模块。HFADE-FMD是一个差分进化策略与烟花算法相结合的混合算法,它基于标签传播机制并按拓扑和功能信息初始化烟花个体为候选功能模块,然后运用烟花算法的爆炸操作和差分进化算法的变异、交叉、选择策略迭代地搜索较佳的功能模块划分[104]。
综上所述,基于图聚类功能模块预测算法侧重于利用拓扑结构的稠密特征发现功能模块;基于层次聚类算法以蛋白质间相似性度量为基础,迭代合并相似蛋白质形成功能模块;基于流模拟聚类算法以流的分布差异来发现拓扑结构的稠密区域,通过划分SPIN来生成功能模块;基于群智能聚类算法模拟群智能体行为搜索可行解空间,以检测功能模块。以上几种聚类算法都在各自理论模型下预测结构性模块,但蛋白质功能模块并不完全遵循拓扑结构模块化的特点。这些聚类算法提出不同的蛋白质相似性度量方法,并以不同的方式融入蛋白质功能信息,以提高功能模块的预测精度,但如何预测生物相关性显著的蛋白质功能模块尚待深入研究。
如图 1所示,按编年史方式,可将基于静态蛋白质相互作用网络SPIN预测算法的研究划分为3条并行时间线:预测SPIN中的蛋白质复合物(PPC-SPIN)、预测SPIN中的蛋白质功能模块(PFM-SPIN)、预测SPIN中的蛋白质复合物/功能模块(PPC/FM-SPIN)。

|
| 图 1 静态蛋白质相互作用网络复合物和功能模块预测算法研究的3条并行时间线 Fig. 1 Three parallel time lines of algorithms study on predicting protein complexes and functional modules in static protein interaction network |
1.3 基于DPIN的复合物和功能模块预测算法
细胞周期或细胞响应环境刺激都会引发不同的生物过程,在此过程中蛋白质会根据功能的需要参与蛋白质复合物的装配和解配[105]。当前开放数据库中的蛋白质相互作用数据是在不同的时间、地点、条件下产生的,这些蛋白质相互作用数据仅说明蛋白质之间存在相互作用,却没有说明这些相互作用在何时何地发生。事实上,蛋白质之间的相互作用是随时空环境变化而呈动态性[106]。
大量的PPI数据集由于缺乏时空信息而无法反映蛋白质相互作用的动态性。如何描述蛋白质相互作用网络的动态行为以及同时出现的蛋白质交互,成为蛋白质复合物和功能模块预测算法首要解决的问题。众多研究者将时序基因表达数据与蛋白质相互作用网络组合,从而引入时间因素;而蛋白质亚细胞定位数据与蛋白质相互作用网络组合则使空间因素得以引入[58]。De Lichtenberg等[107]使用这两类数据研究酿酒酵母细胞周期内蛋白质复合物的变化,结果发现蛋白质复合物具有即时装配、即时合成、动态调控等瞬时行为,且几乎所有的蛋白质复合物均包含动态和静态亚基。Han等[108]在酵母蛋白质相互作用网络中发现两种中心蛋白质:party hub蛋白质和date hub蛋白质,其中party hub蛋白质在模块内同时与大多数蛋白质交互而起作用,而date hub蛋白质为实现特定生物过程在不同时间或地点与蛋白质绑定并形成蛋白质组。Mucha等[109]介绍一个可用于时间相关、多尺度且含任意多幅网络的动态网络社区预测流程,其中每幅网络代表一个特定时间点的网络。Party hub蛋白质可从每幅蛋白质相互作用网络中预测出来,而通过考虑时序多幅蛋白质相互作用网络可预测出date hub蛋白质。因此,通过检查被检测出来的社区是否在某幅蛋白质相互作用网络中,则有可能从蛋白质相互作用网络中区分出蛋白质复合物和功能模块[108]。
综上所述,构建动态蛋白质相互作用网络DPIN能在一定程度上反映细胞系统中蛋白质及其相互作用的动态性,所以基于DPIN预测蛋白质复合物和功能模块比基于SPIN更具优势。构建DPIN为设计蛋白质复合物和功能模块预测算法开辟了新的思路与方向。
基于DPIN预测蛋白质复合物和功能模块的算法研究分为两个步骤:第一步是构建动态蛋白质相互作用网络DPIN,第二步是设计从构建的DPIN中预测蛋白质复合物和功能模块的算法。
1.3.1 动态蛋白质相互作用网络DPIN构建算法在一个细胞生命周期内,随着基因表达的时序关停,基因编码的蛋白质也时序地表现活性[110]。因此确定蛋白质表现活性的时间,即所谓的活跃时间点,是构造动态蛋白质相互作用网络的关键。Tang等[111]在构造时间过程蛋白质相互作用网络(Time Course Protein Interaction Network,TC-PIN)时,采用全局阈值过滤3个连续代谢周期中的非活跃酵母蛋白质。相比于静态蛋白质网络SPIN和伪随机网络,在TC-PIN上运用MCL算法[28]识别出的蛋白质复合物数量更多、生物意义更显著。针对采用全局阈值难以适应不同物种表达水平差异的问题,Wang等[112]提出3-sigma阈值原则以确定每个蛋白质的活跃时间点,构造动态蛋白质相互作用网络DPIN,然后运用算法MCL[28]、CPM[113]和Core[37]从DPIN中识别蛋白质复合物。Shen等[114]指出3-sigma阈值原则的过高阈值将有可能过滤基因表达水平不低的蛋白质,于是通过使用偏差度方法,构造(加权)时间演进蛋白质相互作用网络TEPIN和WTEPIN,然后运用算法ClusterONE[24]、MCL[28]和CAMSE[115]检测时序蛋白质复合物。Xiao等[116]提出使用k-sigma阈值原则过滤基因表达谱噪声数据,以确定蛋白质活跃时间点,继而构造噪声过滤活跃蛋白质相互作用网络NF-APIN,最后运用MCL算法[28]从NF-APIN中检测蛋白质复合物。
上述研究均提出构造动态蛋白质相互作用网络的方法,但是这些方法有可能会忽略一些蛋白质相互作用。王希等[117]在不丢失蛋白质相互作用的前提下,删除那些表达水平低的活跃时间点,从而构造蛋白质相互作用全覆盖的动态蛋白质网络。这种方法不需要设置阈值,使蛋白质相互作用数据得以最大限度地保留,但有可能丢失多次出现的蛋白质相互作用。无论如何,构造动态蛋白质相互作用网络是建模细胞系统中蛋白质动态的有效手段。关于动态蛋白质相互作用网络DPIN的构建方法及应用可参阅文献[58, 118, 119]。
1.3.2 基于DPIN的蛋白质复合物预测算法针对蛋白质相互作用网络的动态性,一些学者首先研究动态蛋白质网络的构造,然后设计基于动态蛋白质相互作用网络的蛋白质复合物预测算法。Li等[120]构建时间序列子网TSNs并运用所提出的TSN-PCD算法从中识别蛋白质复合物,然后基于识别的复合物构建复合物-复合物交互网络,最后设计DFM-CIN算法检测功能模块。该算法不仅能区分蛋白质复合物和功能模块,而且能揭示蛋白质复合物和功能模块之间的关系。通过融合时序基因表达数据和蛋白质交互数据构建动态蛋白质相互作用网络,Ou-yang等[121]提出一个时间平滑重叠复合物检测模型TS-OCD来预测时序蛋白质复合物,并利用基于非负矩阵分解的算法来合并那些在不同时间点预测出的相似蛋白质复合物。通过以基因表达谱的平均值为活性阈值来构造时序蛋白质相互作用网络,Lakizadeh等[122]提出一种基于核心-附件模式、加权聚类系数和最大加权密度等方法并能从时序蛋白质相互作用网络中检测蛋白质复合物的PCD-GED算法。基于k-sigma阈值原则,Zhang等[123, 124]通过计算不同时间点每个蛋白质的活性概率以确定蛋白质活跃时间点,构造动态概率蛋白质相互作用网络,并进一步叠加PPI皮尔森相关系数构造新的动态蛋白质相互作用网络,然后基于核心-附件结构分别在这两种动态蛋白质相互作用网络中检测蛋白质复合物。Lei等[125-127]和Zhao等[128]利用3-sigma阈值原则构造动态蛋白质相互作用网络,设计基于群智能体行为的算法以识别蛋白质复合物。此外,Lei等[129]运用3-sigma阈值原则构造动态蛋白质相互作用网络,基于核心-附件结构,按种子扩张策略先后生成蛋白质核心和附件,以检测蛋白质复合物。Shen等[130]构建邻近亲和度动态蛋白质相互作用网络,选择高聚类系数蛋白质及其邻居构成初始簇,通过迭代扩展邻居蛋白质到簇中来检测蛋白质复合物。为处理不确定数据,Zhang等[131]利用k-sigma阈值原则计算结点活性概率,针对PPI拓扑结构计算边的存在概率,依据结点和边的存在性概率构造动态不确定蛋白质相互作用网络,进而依照核心-附件结构开发蛋白质复合物预测算法。Lei等[132]依据3-sigma阈值原则构造动态蛋白质相互作用网络,组合皮尔森相关系数、边聚类系数、GO功能标注和区室共定位,对所构造的网络进行加权,在此基础上提出一个基于拓扑势能的种子扩展算法以识别蛋白质复合物。为获得更多的动态信息,Zhang等[133]按基因表达波动幅度来确定蛋白质活跃时间点,并构造时间区间动态蛋白质网络TI-PINs,然后设计算法ICJointLE-DPN并从TI-PINs中精确预测出相对多的蛋白质复合物。Xie等[134]按3-sigma阈值原则构造动态蛋白质相互作用网络,以模块紧密度和启发式蚁群优化算法获得聚类,通过过滤合并聚类以形成蛋白质复合物。Lei等[135]通过组合共必要、共定位、共标注和共聚类4种关系,重构多关系动态蛋白质相互作用网络,按稠密度发现候选蛋白质核心,并给出改进的鲜花授粉算法以发现外围蛋白质,进而实现蛋白质复合物的预测。Wang等[136]同样根据3-sigma阈值原则确定蛋白质活跃时间点和概率,通过组合基因表达、GO功能标注和高阶共邻测量构造动态网络,然后运用贪心启发搜索检测蛋白质复合物。上述报道的研究特点是,组合基因表达数据和蛋白质相互作用网络构造动态蛋白质相互作用网络,然后设计从动态蛋白质相互作用网络中识别复合物的算法。值得注意的是,有些静态蛋白质相互作用网络预测复合物的算法可以向动态蛋白质相互作用网络移植。
1.3.3 基于DPIN的蛋白质功能模块预测算法由于动态蛋白质相互作用网络有望区分复合物和功能模块,因此一些学者以预测功能模块为目标而构建动态蛋白质相互作用网络。Lin等[137]在静态蛋白质相互作用网络中集成生物学标注和基因表达谱,以构造扩张型心肌病共表达动态蛋白质相互作用网络,并揭示心肌收缩阶段和器官形态建成阶段的蛋白质功能模块的动态变化。Jin等[138]指出动态蛋白质相互作用网络功能模块中蛋白质具有两个特点:一是蛋白质在静态蛋白质相互作用网络中是连通的,二是结点的表达谱在时域形成特定结构。通过使用时序基因表达数据构建网络,Tang等[111]提出一个时序PPI模型以预测功能模块。Zhang等[139]组合蛋白质活性、基因共表达和PPI数据,构造动态共调控蛋白质相互作用网络,并基于非负矩阵分解的贝叶斯图模型检测功能模块。Lei等[140]按3-sigma阈值原则构造动态蛋白质相互作用网络DPIN,并将萤火虫算法FA分别与算法MCL、R-MCL和SR-MCL融合,提出算法F-MCL、FR-MCL和FSR-MCL以检测动态蛋白质相互作用网络DPIN中的蛋白质功能模块。
基于动态蛋白质相互作用网络预测复合物和功能模块的算法研究起步相对较晚,报道的成果相对较少,但基于动态蛋白质相互作用网络的研究方向已掀起新热潮,并将与基于静态蛋白质相互作用网络的研究一起促进预测算法的发展。如图 2所示,动态蛋白质相互作用网络复合物和功能模块预测算法研究有3条并行时间线:构造动态蛋白质相互作用网络(C-DPIN),预测动态蛋白质相互作用网络复合物(PPC-DPIN)和预测动态蛋白质相互作用网络功能模块(PFM-DPIN)。
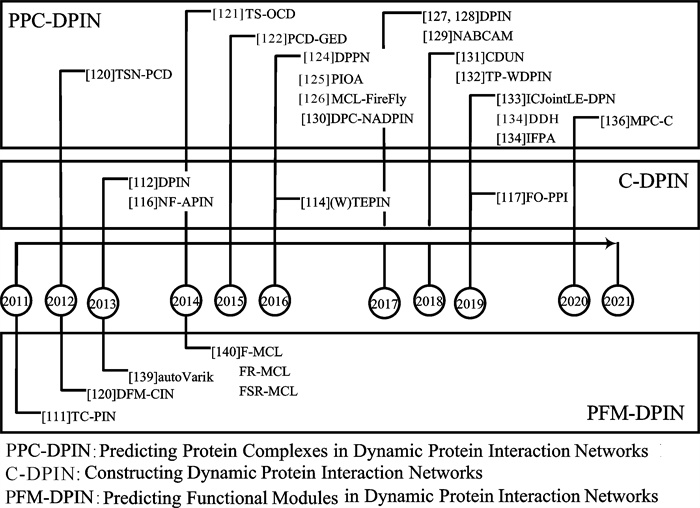
|
| 图 2 动态蛋白质相互作用网络复合物和功能模块预测算法研究的3条并行时间线 Fig. 2 Three parallel time lines of algorithms study on predicting protein complexes and functional modules in dynamic protein interaction networks |
2 数据集
本节介绍基于蛋白质相互作用网络的蛋白质复合物和功能模块预测算法研究所涉及的PPI数据集、复合物数据集、功能模块数据集、基因表达数据集和蛋白质共定位数据集。
PPI数据是蛋白质复合物和功能模块预测算法研究的基础数据,表 1列出一些常用于预测复合物和功能模块的PPI数据集。
| 数据集/数据库Dataset/database | 网址URL | 参考文献Reference |
| MIPS | ftp://ftpmips.gsf.de/fungi/Saccharomycetes/CYGD/]]> | [5] |
| MINT | http://cbm.bio.uniroma2.it/mint/index.html]]> | [6] |
| IntAct | https://www.ebi.ac.uk/intact]]> | [7] |
| DIP | https://dip.doe-mbi.ucla.edu/dip/Main.cgi]]> | [8] |
| BIND | http://binddb.org]]> | [9] |
| BioGrid | https://downloads.thebiogrid.org/BioGRID/Release-Archive/]]> | [10] |
| HPRD | http://www.hprd.org]]> | [11] |
| HPID | http://www.hpid.org]]> | [12] |
| STRING | https://string-db.org/cgi/download]]> | [15] |
| Krogan | http://tap.med.utoronto.ca/exttap/downloads/TAP_core.txt]]> | [141] |
| Gavin | http://yeast-complexes.russelllab.org/complexes.shtml]]> | [142] |
| HuRI | http://www.interactome-atlas.org]]> | [143] |
| PathwayCommons | http://www.pathwaycommons.org]]> | [144] |
高通量蛋白质组和生物信息学算法方面的进展,使得不少高质量的蛋白质复合物数据集得以建立,这些复合物数据集可作为金标准数据集。表 2列出一部分包含蛋白质复合物组成的常用数据库。
| 数据库Database | 模式有机体Model organism | 网址URL | 参考文献Reference |
| Corum | 智人、鼠、蝙蝠Homo sapiens, mouse, bat | http://mips.helmholtz-muenchen.de/ genre/proj/corum]]> | [145] |
| PCDq | 智人Homo sapiens | http://h-invitational.jp/hinv/pcdq/]]> | [146] |
| CYC2008 | 酿酒酵母Saccharomyces cerevisiae | http://wodaklab.org/cyc2008/]]> | [147] |
| YHTP2008 | 酿酒酵母S.cerevisiae | http://wodaklab.org/cyc2008/]]> | [147] |
| MIPS | 智人、鼠、蝙蝠Homo sapiens, mouse, bat | http://mips.helmholtz-muenchen.de/proj/ppi/]]> | [148] |
| SGD | 酿酒酵母S.cerevisiae | http://www.yeastgenome.org/]]> | [149] |
| Yeast complexes | 酿酒酵母S.cerevisiae | http://yeast-complexes.russelllab.org/]]> | [150] |
| Complex Portal | 智人Homo sapiens | http://ftp.ebi.ac.uk/pub/databases/intact/complex/current/complextab/]]> |
蛋白质功能模块是按照生物功能进行划分的蛋白质集合,因此功能模块是根据功能分类进行界定的。功能目录FunCat[151]提供层次化的功能分类,一些全世界开放的数据库存储有典型模式生物蛋白质的FunCat功能类别标注。例如,模式生物酿酒酵母的蛋白质功能类别标注可从MIPs数据库(http://mips.gsf.de/proj/funcatDB)中获取[151],而人类的蛋白质功能类别标注可从Corum数据库(http://mips.helmholtz-muenchen.de/genre/proj/corum)中获得[152]。研究者们将具有相同FunCat功能类别标注的蛋白质分为一类,从而形成基于FunCat功能类别的蛋白质类,这些蛋白质分类可为各种蛋白质功能模块预测算法提供金标准数据集。
基因表达数据是一组基因在若干时间点上的mRNA丰度采样值,它可以反映一组基因在整个采样过程的动态表达模式。由于包含时间信息,基因表达数据成为构造动态蛋白质相互作用网络必不可少的重要数据,同时可用于分析相互作用蛋白质之间表达相关性和疾病基因的差异表达等。表 3给出来源于Omnibus的常用基因表达数据集。
| 数据集Data set | 模式有机体Model organism | 描述Description | 参考文献Reference |
| GSE3431 | 酿酒酵母S.cerevisiae | 3个连续代谢周期上的基因表达数据,每个周期有12个时间间隔,每个时间间隔约25 min Gene expression data over three successive metabolic cycles, 12 time intervals per cycle, ~25 min per time interval | [152] |
| GSE4987 | 酿酒酵母S.cerevisiae | 2个细胞周期上按5 min间隔持续采样2 h的转录谱数据Transcription profiles sampled every 5 min for 2 h across 2 cell cycles | [153] |
| GSE2361 | 智人Homo sapiens | 36种正常人体组织的表达谱Expression profiles of 36 types of normal human tissues | [154] |
| GSE20039 | 拟南芥Arabidopsis thaliana | 弯曲卷叶阶段拟南芥6个种子区室的14个样本14 samples from 6 A. thaliana seed compartments at the bending coyledon stage | [155] |
| GSE40371 | 秀丽隐杆线虫Caenorhabditis elegans | 秀丽隐杆线虫L1幼虫的72个样本72 samples from C.elegans L1 animals | [156] |
| GSE174813 | 黑腹果蝇Drosophila melanogaster | 5-7日龄雄性黑腹果蝇整个头部的48个样本48 samples from whole heads of 5-7 d old male D.melanogaster | [157] |
蛋白质定位数据记录细胞周期中蛋白质组在不同亚细胞区室的出现情况,反映一个细胞周期中蛋白质为发挥生物功能而曾经出现的亚细胞场所。显然,蛋白质定位数据为构造动态蛋白质相互作用网络提供了空间信息。表 4给出常用的蛋白质亚细胞定位标注数据集。
| 数据库Database | 模式有机体Model organism | 网址URL | 参考文献Reference |
| Yeastgfp | 酿酒酵母S.cerevisiae | http://yeastgfp.ucsf.edu]]> | [158] |
| Proteinatlas | 智人Homo sapiens | https://www.proteinatlas.org/download/subcellular_location.tsv.zip]]> | [159] |
| Proteinpedia | 智人Homo sapiens | http://www.humanproteinpedia.org/]]> | [160] |
| NSort/DB | 鼠Mouse | http://www.nsort.org/db/]]> | [161] |
| ComPPI | 酿酒酵母,秀丽隐杆线虫,黑腹果蝇,智人S.cerevisiae, C.elegans, D.melanogaster, homo sapiens | http://ComPPI.LinkGroup.hu]]> | [162] |
| NPD | 脊椎动物Vertebrate | http://npd.hgu.mrc.ac.uk]]> | [163] |
| COMPARTMENTS | 智人,酿酒酵母Homo sapiens, S.cerevisiae | https://compartments.jensenlab.org/Downloads]]> | [164] |
3 展望
对蛋白质组学数据的正确预测可以揭示蛋白质在不同生物学背景下的新功能。相互作用组学进一步揭示了真正参与生物过程的蛋白质复合物和功能模块,以及它们的改变如何导致功能障碍,因此相互作用组学的研究对于解密蛋白质复合物的分子功能尤为重要[165]。然而,在细胞周期或响应外界刺激时,一个蛋白质可与数个宏分子复合物装配,这使得根据PPI数据库中相互作用结果的解释变得复杂[166]。为提取更多的功能信息,进一步开发和实现系统生物学工具预测蛋白质复合物和功能模块,将有助于理解生物过程的结构组织和作用机理,从而在临床上促进和疾病过程相关的研究和靶向药物设计的发展。因此,基于蛋白质相互作用网络研究预测蛋白质复合物和功能模块算法具有深远的意义。
当前,基于蛋白质相互作用网络研究蛋白质复合物和功能模块预测算法需要解决以下问题。
第一,实验技术的局限性使得PPI数据集存在一定程度的假阳性和假阴性数据。假阳性数据的存在给准确预测蛋白质复合物和功能模块带来干扰,基因共表达、蛋白质共定位和结构域相互作用的竞争性可以在一定程度上排除假阳性交互的干扰。假阴性则需要借助其他类型的生物学数据间接推断来排除。因此,在湿式实验方法之外,利用生物学数据建立计算模型并设计算法,以排除假阳性交互、减少假阴性交互并预测蛋白质真交互是一个有待深入研究的课题。
第二,复合物中蛋白质的共定位、共表达特性以及蛋白质相互作用的相容性是复合物形成的必要条件。蛋白质翻译后修饰和空间构象的形成决定蛋白质所发挥的生物功能,生物过程中蛋白质物理地绑定彼此形成复合物以实施相应功能是受内在生化机理所驱动。因此,基于蛋白质相互作用网络设计算法预测复合物有待融入更有力的生物学数据,在缺乏直接有力的生物学数据的情况下,设计准确有效的蛋白质复合物预测算法仍然是一个开放的问题。
第三,涉及同一生物过程的蛋白质及其相互作用表现出级联信号转导的时序性,即执行生物过程的功能模块中的蛋白质及其相互作用并不局限于同一时间、同一空间。因此,在缺少时间信息的蛋白质相互作用网络中检测功能模块将难以获得较高的准确性。在已知蛋白质功能的前提下,采用基于主题的社区发现算法可以检测已知功能模块,但却失去了预测蛋白质功能的作用。虽然相互作用的两个蛋白质是共表达的,但是时序相互作用的多个蛋白质却不是集体共表达的。因此,预测蛋白质功能模块要解决蛋白质表达及其相互作用的时序相关性分析问题。
第四,基因表达数据和蛋白质定位数据的引入使蛋白质交互满足时空约束,因此,基于动态蛋白质相互作用网络预测复合物或功能模块,相比于基于静态蛋白质相互作用网络具有一定的优势。但动态蛋白质相互作用网络构造首先要解决蛋白质活跃时间点的问题,也就是蛋白质活跃的判定问题。另外,当前动态蛋白质相互作用网络构造方法仅考虑共表达的蛋白质及其正相关互调控的相互作用,对于反相关负调控的抑制作用无法反映。存在相互作用的蛋白质在某两个时刻同时活跃并不意味着两个时刻都相互作用,但目前已有的动态蛋白质相互作用网络构造方法却无法区别该情况而导致假阳性交互的增加。因此,针对这些问题设计新的动态蛋白质相互作用网络构造方法也是一个有待解决的课题。
第五,已有的大多数预测算法忽略了只由2个蛋白质构成的复合物和功能模块,对于准确识别规模较大复合物和功能模块也存在较大的难度。在实际中,由2个蛋白质构成的蛋白质复合物和功能模块大量存在,因此准确预测规模为2的复合物和功能模块具有重要意义[167]。对于规模较大复合物和功能模块的准确预测目前尚无公认的有效算法,这表明设计开发兼顾规模为2和规模较大的蛋白质复合物/功能模块预测算法仍然是一个挑战。
| [1] |
UETZ P, GIOT L, CAGNEY G, et al. A comprehensive analysis of protein-protein interactions in Saccharomyces cerevisiae[J]. Nature, 2000, 403(6770): 623-627. DOI:10.1038/35001009 |
| [2] |
HO Y, GRUHLER A, HEIBUT A, et al. Systematic identification of protein complexes in Saccharomyces cerevisiae by mass spectrometry[J]. Nature, 2002, 415(6868): 180-183. DOI:10.1038/415180a |
| [3] |
ZHU H, BILGIN M, BANGHA M R, et al. Global analysis of protein activities using proteome chips[J]. Science, 2001, 293(5537): 2101-2105. DOI:10.1126/science.1062191 |
| [4] |
HODGES P E, MCKEE A H, DAVIS B P, et al. The Yeast Proteome Database (YPD): A model for the organization and presentation of genome-wide functional data[J]. Nucleic Acids Research, 1999, 27(1): 69-73. DOI:10.1093/nar/27.1.69 |
| [5] |
MEWES H W, AMID C, ARNOLD R, et al. MIPS: Analysis and annotation of proteins from whole genomes[J]. Nucleic Acids Research, 2004, 32(suppl 1): D41-D44. |
| [6] |
ZANZONI A, MONTECCHI-PALAZZI L, QUONDAM M, et al. MINT: A molecular INTeraction database[J]. FEBS Letters, 2002, 513(1): 135-140. DOI:10.1016/S0014-5793(01)03293-8 |
| [7] |
KERRIEN S, ALAM-FARUQUE Y, ARANDA B, et al. IntAct-open source resource for molecular interaction data[J]. Nucleic Acids Research, 2007, 35(suppl 1): D561-D565. |
| [8] |
SALWINSKI L, MILLER C S, SMITH A J, et al. The database of interacting proteins: 2004 update[J]. Nucleic Acids Research, 2004, 32(suppl 1): D449-D451. |
| [9] |
BADER G D, DONALDSON I, WOLTING C, et al. BIND-The biomolecular interaction network database[J]. Nucleic Acids Research, 2001, 29(1): 242-245. DOI:10.1093/nar/29.1.242 |
| [10] |
STARK C, BREITKREUTZ B J, REGULY T, et al. BioGRID: A general repository for interaction datasets[J]. Nucleic Acids Research, 2006, 34(suppl 1): D535-D539. |
| [11] |
MISHRA G R, SURESH M, KUMARAN K, et al. Human protein reference database-2006 update[J]. Nucleic Acids Research, 2006, 34(suppl 1): D411-D414. |
| [12] |
HAN K, PARK B, KIM H, et al. HPID: The human protein interaction database[J]. Bioinformatics, 2004, 20(15): 2466-2470. DOI:10.1093/bioinformatics/bth253 |
| [13] |
LIU G Z, PACIFICO S, YU J K, et al. DroID: The Drosophila interactions database, a comprehensive resource for annotated gene and protein interactions[J]. BMC Genomics, 2008, 9: 461. DOI:10.1186/1471-2164-9-461 |
| [14] |
KUHN M, SZKLARCZYK D, FRANCESCHINI A, et al. STITCH 2:An interaction network database for small molecules and proteins[J]. Nucleic Acids Research, 2010, 38(suppl 1): D552-D556. |
| [15] |
JENSEN L J, KUHN M, STARK M, et al. STRING 8-a global view on proteins and their functional interactions in 630 organisms[J]. Nucleic Acids Research, 2009, 37(suppl 1): D412-D416. |
| [16] |
SPIRIN V, MIRNY L A. Protein complexes and functional modules in molecular networks[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(21): 12123-12128. DOI:10.1073/pnas.2032324100 |
| [17] |
LIU G M, WONG L, CHUA H N. Complex discovery from weighted PPI networks[J]. Bioinformatics, 2009, 25(15): 1891-1897. DOI:10.1093/bioinformatics/btp311 |
| [18] |
CHUA H N, NING K, SUNG W K, et al. Using indirect protein-protein interactions for protein complex prediction[J]. Journal of Bioinformatics and Computational Biology, 2008, 6(3): 435-466. DOI:10.1142/S0219720008003497 |
| [19] |
LI X L, TAN S H, FOO C S, et al. Interaction graph mining for protein complexes using local clique merging[J]. Genome Informatics, 2005, 16(2): 260-269. |
| [20] |
LI X L, FOO C S, NG S K. Discovering protein complexes in dense reliable neighborhoods of protein interaction networks[J]. Computational Systems Bioinformatics, 2007, 6: 157-168. |
| [21] |
EFIMOV D, ZAKI N, BERENGUERES J. Detecting protein complexes from noisy protein interaction data [C]//ACM Press the 11th International Workshop. BIOKDD′12: Proceedings of the 11th International Workshop on Data Mining in Bioinformatics. New York: Association for Computing Machinery, 2012: 1-7.
|
| [22] |
BADER G D, HOGUE C W V. An automated method for finding molecular complexes in large protein interaction networks[J]. BMC Bioinformatics, 2003, 4: 2. DOI:10.1186/1471-2105-4-2 |
| [23] |
ALTAF-UL-AMIN M, SHINBO Y, MIHARA K, et al. Development and implementation of an algorithm for detection of protein complexes in large interaction networks[J]. BMC Bioinformatics, 2006, 7: 207. DOI:10.1186/1471-2105-7-207 |
| [24] |
NEPUSZ T, YU H Y, PACCANARO A. Detecting overlapping protein complexes in protein-protein interaction networks[J]. Nature Methods, 2012, 9(5): 471-472. DOI:10.1038/nmeth.1938 |
| [25] |
JIANG P, SINGH M. SPICi: A fast clustering algo-rithm for large biological networks[J]. Bioinformatics, 2010, 26(8): 1105-1111. DOI:10.1093/bioinformatics/btq078 |
| [26] |
HAQUE M, SARMAH R, BHATTACHARYYA D K. A common neighbor based technique to detect protein complexes in PPI networks[J]. Journal of Genetic Engineering and Biotechnology, 2018, 16(1): 227-238. DOI:10.1016/j.jgeb.2017.10.010 |
| [27] |
WANG Y J, JEONG H, YOON B J, et al. ClusterM: A scalable algorithm for computational prediction of conserved protein complexes across multiple protein interaction networks[J]. BMC Genomics, 2020, 21(suppl 10): 615. |
| [28] |
VAN DONGEN S M. Graph clustering by flow simulation [D]. Utrecht: Utrecht University, 2000.
|
| [29] |
BROHEE S, VAN HELDEN J. Evaluation of clustering algorithms for protein-protein interaction networks[J]. BMC Bioinformatics, 2006, 7: 488. DOI:10.1186/1471-2105-7-488 |
| [30] |
VLASBLOM J, WODAK S J. Markov clustering versus affinity propagation for the partitioning of protein interaction graphs[J]. BMC Bioinformatics, 2009, 10: 99. DOI:10.1186/1471-2105-10-99 |
| [31] |
SATULURI V, PARTHASARATHY S. Scalable gra-ph clustering using stochastic flows: Applications to community discovery [C]//ACM Press the 15th ACM SIGKDD international conference. KDD′09: Proceedings of The 15th ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. New York: Association for Computing Machinery, 2009: 737-746.
|
| [32] |
SATULURI V, PARTHASARATHY S, UCAR D. Markov clustering of protein interaction networks with improved balance and scalability [C]//ACM International Conference. BCB′10: Proceedings of the First ACM International Conference on Bioinformatics and Computational Biology. New York: Association for Computing Machinery, 2010: 247-256.
|
| [33] |
SHIH Y K, PARTHASARATHY S. Identifying functional modules in interaction networks through overlapping Markov clustering[J]. Bioinformatics, 2012, 28(18): i473-i479. DOI:10.1093/bioinformatics/bts370 |
| [34] |
LEI X J, WANG F, WU F X, et al. Protein complex identification through Markov clustering with firefly algorithm on dynamic protein-protein interaction networks[J]. Information Sciences, 2016, 329: 303-316. DOI:10.1016/j.ins.2015.09.028 |
| [35] |
GAVIN A C, ALOY P, GRANDI P, et al. Proteome survey reveals modularity of the yeast cell machinery[J]. Nature, 2006, 440(7084): 631-636. DOI:10.1038/nature04532 |
| [36] |
AHMED H A, BHATTACHARYYA D K, KALITA J K. Core and peripheral connectivity based cluster analysis over PPI network[J]. Computational Biology and Chemistry, 2015, 59(part B): 32-41. |
| [37] |
LEUNG H C M, YIU S M, XIANG Q, et al. Predicting protein complexes from PPI Data: A core-attachment approach[J]. Journal of Computational Biology, 2009, 16(2): 133-144. DOI:10.1089/cmb.2008.01TT |
| [38] |
WU M, LI X L, KWOH C K, et al. A core-attachment based method to detect protein complexes in PPI networks[J]. BMC Bioinformatics, 2009, 10: 169. DOI:10.1186/1471-2105-10-169 |
| [39] |
SRIHARI S, NING K, LEONG H W. Refining markov clustering for complex detection by incorporating core-attachment structure[J]. Genome Information, 2009, 23(1): 159-168. |
| [40] |
SRIHARI S, NING K, LEONG H W. MCL-CAw: A refinement of MCL for detecting yeast complexes from weighted PPI networks by incorporating core-attachment structure[J]. BMC Bioinformatics, 2010, 11: 504. DOI:10.1186/1471-2105-11-504 |
| [41] |
PENG W, WANG J X, ZHAO B H, et al. Identification of protein complexes using weighted PageRank-Nibble algorithm and core-attachment structure[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2015, 12(1): 179-192. DOI:10.1109/TCBB.2014.2343954 |
| [42] |
Gene Ontology Consortium. The Gene Ontology (GO) project in 2006[J]. Nucleic Acids Research, 2006, 34(suppl 1): D322-D326. |
| [43] |
KING A D, PRZULJ N, JURISICA I. Protein complex prediction via cost-based clustering[J]. Bioinformatics, 2004, 20(17): 3013-3020. DOI:10.1093/bioinformatics/bth351 |
| [44] |
XU B, LIN H F, YANG Z H. Ontology integration to identify protein complex in protein interaction networks[J]. Proteome Science, 2011, 9(suppl 1): S7. DOI:10.1186/1477-5956-9-S1-S7 |
| [45] |
PRICE T, PEÑA Ⅲ F I, CHO Y R. Survey: Enhancing protein complex prediction in PPI networks with GO similarity weighting[J]. Interdisciplinary Sciences, Computational Life Sciences, 2013, 5(3): 196-210. DOI:10.1007/s12539-013-0174-9 |
| [46] |
GE H, LIU Z, CHURCH G M, et al. Correlation between transcriptome and interactome mapping data from Saccharomyces cerevisiae[J]. Nature Genetics, 2001, 29(4): 482-486. DOI:10.1038/ng776 |
| [47] |
JANSEN R, YU H, GREENBAUM D, et al. A Bayesian networks approach for predicting protein-protein interactions from genomic data[J]. Science, 2003, 302(5644): 449-453. DOI:10.1126/science.1087361 |
| [48] |
BHARDWAJ N, LU H. Correlation between gene expression profiles and protein-protein interactions within and across genomes[J]. Bioinformatics, 2005, 21(11): 2730-2738. DOI:10.1093/bioinformatics/bti398 |
| [49] |
LI X L, TAN Y C, NG S K. Systematic gene function prediction from gene expression data by using a fuzzy nearest-cluster method[J]. BMC Bioinformatics, 2006, 7(suppl 4): S23. DOI:10.1186/1471-2105-7-S4-S23 |
| [50] |
FENG J X, JIANG R, JIANG T. A max-flow based approach to the identification of protein complexes using protein interaction and microarray data[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2011, 8(3): 621-634. DOI:10.1109/TCBB.2010.78 |
| [51] |
TANG X W, WANG J X, PAN Y. Predicting protein complexes via the integration of multiple biological information [C]//IEEE. Proceedings of 2012 IEEE 6th International Conference on Systems Biology (ISB). Xi′an: IEEE, 2012, 174-179.
|
| [52] |
CHENG L, LEUNG K S. Quantification of non-coding RNA target localization diversity and its application in cancers[J]. Journal of Molecular Cell Biology, 2018, 10(2): 130-138. DOI:10.1093/jmcb/mjy006 |
| [53] |
CHENG L, FAN K, HUANG Y, et al. Full characterization of localization diversity in the human protein interactome[J]. Journal of Proteome Research, 2017, 16(8): 3019-3029. DOI:10.1021/acs.jproteome.7b00306 |
| [54] |
THE UNIPROT CONSORTIUM. Activities at the universal protein resource (UniProt)[J]. Nucleic Acids Research, 2014, 42(D1): D191-D198. DOI:10.1093/nar/gkt1140 |
| [55] |
THE UNIPROT CONSORTIUM. The universal protein resource (UniProt) in 2010[J]. Nucleic Acids Research, 2010, 38(suppl 1): D142-D148. |
| [56] |
CHENG L X, LIU P F, LEUNG K S. SMILE: A novel procedure for subcellular module identification with localization expansion[J]. IET Systems Biology, 2018, 12(2): 55-61. DOI:10.1049/iet-syb.2017.0085 |
| [57] |
CHENG L X, LIU P F, WANG D, et al. Exploiting locational and topological overlap model to identify modules in protein interaction networks[J]. BMC Bioinformatics, 2019, 20: 23. DOI:10.1186/s12859-019-2598-7 |
| [58] |
李敏, 孟祥茂. 动态蛋白质网络的构建、分析及应用研究进展[J]. 计算机研究与发展, 2017, 54(6): 1281-1299. |
| [59] |
REHMAN Z U, IDRIS A, KHAN A. Multi-Dimensional Scaling based grouping of known complexes and intelligent protein complex detection[J]. Computational Biology and Chemistry, 2018, 74: 149-156. DOI:10.1016/j.compbiolchem.2018.03.023 |
| [60] |
LIU X X, YANG Z H, ZHOU Z W, et al. The impact of protein interaction networks′ characteristics on computational complex detection methods[J]. Journal of Theoretical Biology, 2018, 439: 141-151. DOI:10.1016/j.jtbi.2017.12.002 |
| [61] |
ABDULATEEF A H, ATTEA B A, RASHID A N, et al. A new evolutionary algorithm with locally assisted heuristic for complex detection in protein interaction networks[J]. Applied Soft Computing, 2018, 73: 1004-1025. DOI:10.1016/j.asoc.2018.09.031 |
| [62] |
ZHANG J X, ZHONG C, HUANG Y R, et al. A method for identifying protein complexes with the features of joint co-localization and joint co-expression in static PPI networks[J]. Computers in Biology and Medicine, 2019, 111: 103333. DOI:10.1016/j.compbiomed.2019.103333 |
| [63] |
YOUNIS H, ANWAR M W, KHAN M U G, et al. A new sequential forward feature selection (SFFS) algorithm for mining best topological and biological features to predict protein complexes from Protein-Protein Interaction Networks (PPINs)[J]. Interdisciplinary Sciences: Computational Life Sciences, 2021, 13(3): 371-388. DOI:10.1007/s12539-021-00433-8 |
| [64] |
JUNG S H, HYUN B, JANG W H, et al. Protein complex prediction based on simultaneous protein interaction network[J]. Bioinformatics, 2010, 26(3): 385-391. DOI:10.1093/bioinformatics/btp668 |
| [65] |
JUNG S H, JANG W H, HUR H Y, et al. Protein complex prediction based on mutually exclusive interactions in protein interaction network [C]//. Proceedings of the 19th International Conference Genome informatics. International Conference on Genome Informatics. London: Imperial College Press, 2008, 21: 77-88.
|
| [66] |
KIM P M, LU L J, XIA Y, et al. Relating three-dimensional structures to protein networks provides evolutionary insights[J]. Science, 2006, 314(5807): 1938-1941. DOI:10.1126/science.1136174 |
| [67] |
SPRINZAK E, ALTUVIA Y, MARGALIT H. Characterization and prediction of protein-protein interactions within and between complexes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(40): 14718-14723. DOI:10.1073/pnas.0603352103 |
| [68] |
OZAWA Y, SAITO R, FUJIMORI S, et al. Protein complex prediction via verifying and reconstructing the topology of domain-domain interactions[J]. BMC Bioinformatics, 2010, 11: 350. DOI:10.1186/1471-2105-11-350 |
| [69] |
MA W J, MCANULLA C, WANG L S. Protein complex prediction based on maximum matching with domain-domain interaction[J]. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, 2012, 1824(12): 1418-1424. DOI:10.1016/j.bbapap.2012.06.009 |
| [70] |
ADAMCSEK B, PALLA G, FARKAS I J, et al. CFinder: Locating cliques and overlapping modules in biological networks[J]. Bioinformatics, 2006, 22(8): 1021-1023. DOI:10.1093/bioinformatics/btl039 |
| [71] |
JIA S W, GAO L, GAO Y, et al. Viewing the meso-scale structures in protein-protein interaction networks using 2-clubs[J]. IEEE Access, 2018, 6: 36780-36797. DOI:10.1109/ACCESS.2018.2852275 |
| [72] |
JIA S W, GAO L, GAO Y, et al. Exploring triad-rich substructures by graph-theoretic characterizations in complex networks[J]. Physica A: Statistical Mechanics and Its Applications, 2017, 4: 53-69. |
| [73] |
METE M, TANG F, XU X W, et al. A structural approach for finding functional modules from large biological networks[J]. BMC Bioinformatics, 2008, 9(suppl 9): S19. DOI:10.1186/1471-2105-9-S9-S19 |
| [74] |
ABDULLAH A, DERIS S, HASHIM S Z M, et al. Graph partitioning method for functional module detections of protein interaction network [C]//IEEE 2009 International Conference. Proceedings of the 2009 International Conference on Computer Technology and Development. Washington, DC: IEEE Computer Society, 2009: 230-234.
|
| [75] |
CHEN J G, LI K L, BILAL K, et al. Parallel protein community detection in large-scale PPI networks based on multi-source learning[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2018. DOI:10.1109/TCBB.2018.2868088 |
| [76] |
TADAKA S, KINOSHITA K. NCMine: Core-peripheral based functional module detection using near-clique mining[J]. Bioinformatics, 2016, 32(22): 3454-3460. |
| [77] |
MANNERS H N, ROY S, KALITA J K. Intrinsic-overlapping co-expression module detection with application to Alzheimer′s Disease[J]. Computational Biology and Chemistry, 2018, 77: 373-389. DOI:10.1016/j.compbiolchem.2018.10.014 |
| [78] |
WIWIE C, KUZNETSOVA I, MOSTAFA A, et al. Time-resolved systems medicine reveals viral infection-modulating host targets[J]. Systems Medicine, 2019, 2(1): 1-9. DOI:10.1089/sysm.2018.0013 |
| [79] |
SHEN X J, YI L, YI Y, et al. Dynamic identifying protein functional modules based on adaptive density modularity in protein-protein interaction networks[J]. BMC Bioinformatics, 2015, 16(suppl 12): S5. DOI:10.1186/1471-2105-16-S12-S5 |
| [80] |
HE J Y, LI C J, YE B L, et al. Efficient and accurate greedy search methods for mining functional modules in protein interaction networks[J]. BMC Bioinformatics, 2012, 13(suppl 10): S19. DOI:10.1186/1471-2105-13-S10-S19 |
| [81] |
JEONG H, KIM Y, JUNG Y S, et al. Entropy-based graph clustering of PPI networks for predicting overlapping functional modules of proteins[J]. Entropy, 2021, 23(10): 1271. DOI:10.3390/e23101271 |
| [82] |
ZHAO Z H, XU W J, CHEN A W, et al. Protein functional module identification method combining topological features and gene expression data[J]. BMC Genomics, 2021, 22: 423. DOI:10.1186/s12864-021-07620-3 |
| [83] |
YING K C, LIN S W. Maximizing cohesion and separation for detecting protein functional modules in protein-protein interaction networks[J]. PLoS ONE, 2020, 15(10): e0240628. DOI:10.1371/journal.pone.0240628 |
| [84] |
NEWMAN M E J. Fast algorithm for detecting community structure in networks[J]. Physical Review E, 2004, 69(6): 066133. DOI:10.1103/PhysRevE.69.066133 |
| [85] |
GIRVAN M, NEWMAN M E J. Community structure in social and biological networks[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(12): 7821-7826. DOI:10.1073/pnas.122653799 |
| [86] |
RHRISSORRAKRAI K, GUNSALUS K C. MINE: Module identification in networks[J]. BMC Bioinformatics, 2011, 12: 192. DOI:10.1186/1471-2105-12-192 |
| [87] |
ARNAU V, MARS S, MARIN I. Iterative cluster analysis of protein interaction data[J]. Bioinformatics, 2005, 21(3): 364-378. DOI:10.1093/bioinformatics/bti021 |
| [88] |
MICHENER C D, SOKAL R R. A quantitative approach to a problem in classification[J]. Evolution, 1957, 11(2): 130-162. DOI:10.1111/j.1558-5646.1957.tb02884.x |
| [89] |
GASCUEL O, STEEL M. Neighbor-joining revealed[J]. Molecular Biology and Evolution, 2006, 23(11): 1997-2000. DOI:10.1093/molbev/msl072 |
| [90] |
ALDECOA R, MARIN I. Jerarca: Efficient analysis of complex networks using hierarchical clustering[J]. PLos ONE, 2010, 5(7): e11585. DOI:10.1371/journal.pone.0011585 |
| [91] |
WANG J X, LI M, CHEN J E, et al. A fast hierarchical clustering algorithm for functional modules discovery in protein interaction networks[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2011, 8(3): 607-620. DOI:10.1109/TCBB.2010.75 |
| [92] |
ENRIGHT A J, VAN DONGEN S, OUZOUNIS C A. An efficient algorithm for large-scale detection of protein families[J]. Nucleic Acids Research, 2002, 30(7): 1575-1584. DOI:10.1093/nar/30.7.1575 |
| [93] |
GU L, HAN Y, WANG C, et al. Module overlapping structure detection in PPI using an improved link similarity-based Markov clustering algorithm[J]. Neural Computing and Applications, 2019, 31(5): 1481-1490. DOI:10.1007/s00521-018-3508-z |
| [94] |
HWANG W, CHO Y R, ZHANG A, et al. A novel functional module detection algorithm for protein-protein interaction networks[J]. Algorithms for Molecular Biology, 2006, 1: 24. DOI:10.1186/1748-7188-1-24 |
| [95] |
HWANG W, CHO Y R, ZHANG A, et al. CASCADE: A novel quasi all paths-based network analysis algorithm for clustering biological interactions[J]. BMC Bioinformatics, 2008, 9: 64. DOI:10.1186/1471-2105-9-64 |
| [96] |
INOUE K, LI W J, KURATA H. Diffusion model based spectral clustering for protein-protein interaction networks[J]. PLoS ONE, 2010, 5(9): e12623. DOI:10.1371/journal.pone.0012623 |
| [97] |
SALLIM J, ABDULLAH R, KHADER A T. ACOPIN: An ACO algorithm with TSP approach for clustering proteins from protein interaction network [C]//Proceedings of 2008 Second UKSIM European Symposium on Computer Modeling and Simulation. Liverpool, UK: IEEE, 2008: 203-208.
|
| [98] |
JI J Z, LIU Z J, ZHANG A D, et al. Improved ant colony optimization for detecting functional modules in protein-protein interaction networks [C]//International Conference on Information Computing and Applications 2012, Part of the Communications in Computer and Information Science. Berlin, Heidelberg: Springer, 2012, 308: 404-413.
|
| [99] |
JI J Z, LIU Z J, ZHANG A D, et al. Ant colony optimization with multi-agent evolutionary for detecting functional modules in protein-protein interaction networks [C]//International Conference on Information Computing and Applications, Part of the Lecture Notes in Computer Science. Berlin, Heidelberg: Springer, 2012, 7473: 445-453.
|
| [100] |
JI J Z, LIU H X, ZHANG A D, et al. ACC-FMD: Ant colony clustering for functional module detection in protein-protein interaction networks[J]. International Journal of Data Mining and Bioinformatics, 2015, 11(3): 331-363. DOI:10.1504/IJDMB.2015.067323 |
| [101] |
YANG C C, JI J Z, ZHANG A. BFO-FMD: Bacterial foraging optimization for functional module detection in protein-protein interaction networks[J]. Soft Computing-A Fusion of Foundations, Methodologies and Applications, 2018, 22(10): 3395-3416. |
| [102] |
ZHENG X H, WU L T, YE S Z, et al. Simplified swarm optimization-based function module detection in protein-protein interaction networks[J]. Applied Sciences, 2017, 7(4): 412. DOI:10.3390/app7040412 |
| [103] |
LEI X J, TIAN J F, GE L, et al. The clustering model and algorithm of PPI network based on propagating mechanism of artificial bee colony[J]. Information Sciences, 2013, 247: 21-39. DOI:10.1016/j.ins.2013.05.027 |
| [104] |
JI J Z, XIAO H H, YANG C C. HFADE-FMD: A hybrid approach of fireworks algorithm and differential evolution strategies for functional module detection in protein-protein interaction networks[J]. Applied Intelligence, 2021, 51(2): 1118-1132. DOI:10.1007/s10489-020-01791-4 |
| [105] |
LEVY E D, PEREIRA-LEAL J B. Evolution and dynamics of protein interactions and networks[J]. Current Opinion in Structure Biology, 2008, 18(3): 349-357. DOI:10.1016/j.sbi.2008.03.003 |
| [106] |
PRZYTYCKA T M, SINGH M, SLONIM D K. Toward the dynamic interactome: It′s about time[J]. Briefings in Bioinformatics, 2010, 11(1): 15-29. DOI:10.1093/bib/bbp057 |
| [107] |
DE LICHTENBERG U, JENSEN L J, BRUNAK S, et al. Dynamic complex formation during the yeast cell cycle[J]. Science, 2005, 307(5710): 724-727. DOI:10.1126/science.1105103 |
| [108] |
HAN J D, BERTIN N, HAO T, et al. Evidence for dynamically organized modularity in the yeast protein-protein interaction network[J]. Nature, 2004, 430(6995): 88-93. DOI:10.1038/nature02555 |
| [109] |
MUCHA P J, RICHARDSON T, MACON K, et al. Community structure in time-dependent, multiscale, and multiplex networks[J]. Science, 2010, 328(5980): 876-878. DOI:10.1126/science.1184819 |
| [110] |
CHEN B L, FAN W W, LIU J, et al. Identifying protein complexes and functional modules-from static PPI networks to dynamic PPI networks[J]. Briefings in Bioinformatics, 2014, 15(2): 177-194. DOI:10.1093/bib/bbt039 |
| [111] |
TANG X W, WANG J X, LIU B B, et al. A comparison of the functional modules identified from time course and static PPI network data[J]. BMC Bioinformatics, 2011, 12: 339. DOI:10.1186/1471-2105-12-339 |
| [112] |
WANG J X, PENG X Q, LI M, et al. Construction and application of dynamic protein interaction network based on time course gene expression data[J]. Proteomic, 2013, 13(2): 301-312. DOI:10.1002/pmic.201200277 |
| [113] |
PALLA G, DERÉNYI I, FARKAS I, et al. Uncovering the overlapping community structure of complex networks in nature and society[J]. Nature, 2005, 435(7043): 814-818. DOI:10.1038/nature03607 |
| [114] |
SHEN X J, YI L, JIANG X P, et al. Mining temporal protein complex based on the dynamic PIN weighted with connected affinity and gene co-expression[J]. PLoS ONE, 2016, 11(4): e0153967. DOI:10.1371/journal.pone.0153967 |
| [115] |
HE T T, LI P, HU X H, et al. A novel proteins complex identification based on connected affinity and multi-level seed extension[J]. International Journal of Data Mining and Bioinformatics, 2016, 14(1): 51-70. DOI:10.1504/IJDMB.2016.073346 |
| [116] |
XIAO Q H, WANG J X, PENG X Q, et al. Detecting protein complexes from active protein interaction networks constructed with dynamic gene expression profiles[J]. Proteome Science, 2013, 11(suppl 1): S20. DOI:10.1186/1477-5956-11-S1-S20 |
| [117] |
王希, 潘理, 胥晓莎, 等. 一种基于全覆盖的动态蛋白质相互作用网络构建方法[J]. 湖南理工学院学报(自然科学版), 2019, 32(4): 21-27. |
| [118] |
李彬, 孙静, 王希, 等. 一种构建动态蛋白质相互作用网络的阈值方法[J]. 湖南理工学院学报(自然科学版), 2021, 34(1): 40-44. DOI:10.3969/j.issn.1672-5298.2021.01.009 |
| [119] |
胡赛, 熊慧军, 赵碧海, 等. 动态加权蛋白质相互作用网络构建及其应用研究[J]. 自动化学报, 2015, 41(11): 1894-1900. |
| [120] |
LI M, WU X H, WANG J X, et al. Towards the identification of protein complexes and functional modules by integrating PPI network and gene expression data[J]. BMC Bioinformatics, 2012, 13: 109. DOI:10.1186/1471-2105-13-109 |
| [121] |
OU-YANG L, DAI D Q, LI X L, et al. Detecting temporal protein complexes from dynamic protein-protein interaction networks[J]. BMC Bioinformatics, 2014, 15: 335. DOI:10.1186/1471-2105-15-335 |
| [122] |
LAKIZADEH A, JALILI S, MARASHI S A. PCD-GED: Protein complex detection considering PPI dynamics based on time series gene expression data[J]. Journal of Theoretical Biology, 2015, 378: 31-38. DOI:10.1016/j.jtbi.2015.04.020 |
| [123] |
ZHANG Y J, LIN H F, YANG Z H, et al. Construction of dynamic probabilistic protein interaction networks for protein complex identification[J]. BMC Bioinformatics, 2016, 17: 186. DOI:10.1186/s12859-016-1054-1 |
| [124] |
ZHANG Y J, LIN H F, YANG Z H, et al. A method for predicting protein complex in dynamic PPI networks[J]. BMC Bioinformatics, 2016, 17(suppl 7): 229. DOI:10.1186/s12859-016-1101-y |
| [125] |
LEI X J, DING Y L, WU F X. Detecting protein complexes from DPINs by density based clustering with Pigeon-Inspired Optimization Algorithm[J]. Science China Information Sciences, 2016, 59(7): 070103. DOI:10.1007/s11432-016-5578-9 |
| [126] |
LEI X J, WANG F, WU F X, et al. Protein complex identification through Markov clustering with firefly algorithm on dynamic protein-protein interaction networks[J]. Information Sciences, 2016, 329: 303-316. DOI:10.1016/j.ins.2015.09.028 |
| [127] |
LEI X J, LI H, ZHANG A D, et al. IOPTICS-GSO for identifying protein complexes from dynamic PPI networks[J]. BMC Medical Genomics, 2017, 10(suppl 5): 80. DOI:10.1186/s12920-017-0314-x |
| [128] |
ZHAO J, LEI X J, WU F X. Predicting protein complexes in weighted dynamic PPI networks based on ICSC[J]. Complexity, 2017, 4120506. DOI:10.1155/2017/4120506 |
| [129] |
LEI X J, LIANG J. Neighbor affinity-based core-attachment method to detect protein complexes in dynamic PPI networks[J]. Molecules, 2017, 22(7): 1223. DOI:10.3390/molecules22071223 |
| [130] |
SHEN X J, YI L, JIANG X P, et al. Neighbor affinity based algorithm for discovering temporal protein complex from dynamic PPI network[J]. Methods, 2016, 110: 90-96. DOI:10.1016/j.ymeth.2016.06.010 |
| [131] |
ZHANG Y J, LIN H F, YANG Z H, et al. An uncertain model-based approach for identifying dynamic protein complexes in uncertain protein-protein interaction networks[J]. BMC Genomics, 2017, 18(suppl 7): 743. DOI:10.1186/s12864-017-4131-6 |
| [132] |
LEI X J, ZHANG Y C, CHENG S, et al. Topology potential based seed-growth method to identify protein complexes on dynamic PPI data[J]. Information Sciences, 2018, 425: 140-153. DOI:10.1016/j.ins.2017.10.013 |
| [133] |
ZHANG J X, ZHONG C, LIN H X, et al. Identifying protein complexes from dynamic temporal interval protein-protein interaction networks[J]. Biomed Research International, 2019, 2019: 3726721. DOI:10.1155/2019/3726721 |
| [134] |
XIE D, YI Y, ZHOU J, et al. A novel temporal protein complexes identification framework based on density-distance and heuristic algorithm[J]. Neural Computing and Applications, 2019, 31(9): 4693-4701. DOI:10.1007/s00521-018-3660-5 |
| [135] |
LEI X J, FANG M, GUO L, et al. Protein complex detection based on flower pollination mechanism in multi-relation reconstructed dynamic protein networks[J]. BMC Bioinformatics, 2019, 20(suppl 3): 131. DOI:10.1186/s12859-019-2649-0 |
| [136] |
WANG R Q, WANG C X, LIU G X. A novel graph clustering method with a greedy heuristic search algorithm for mining protein complexes from dynamic and static PPI networks[J]. Information Sciences, 2020, 522: 275-298. DOI:10.1016/j.ins.2020.02.063 |
| [137] |
LIN C C, HSIANG J T, WU C Y, et al. Dynamic functional modules in co-expressed protein interaction networks of dilated cardiomyopathy[J]. BMC Systems Biology, 2010, 4: 138. DOI:10.1186/1752-0509-4-138 |
| [138] |
JIN R, MCCALLEN S, LIU C C, et al. Identifying dynamic network modules with temporal and spatial constraints[J]. Pacific Symposium on Biocomputing, 2009, 14: 203-214. |
| [139] |
ZHANG Y, DU N, LI K, et al. Co-regulated protein functional modules with varying activities in dynamic PPI networks[J]. Tsinghua Science and Technology, 2013, 18(5): 530-540. DOI:10.1109/TST.2013.6616526 |
| [140] |
LEI X J, WANG F, WU F X, et al. Detecting functional modules in dynamic protein-protein interaction networks using markov clustering and firefly algorithm [C]//2014 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). Belfast, UK: IEEE, 2014: 75-81.
|
| [141] |
KROGAN N J, CAGNEY G, YU H Y, et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae[J]. Nature, 2006, 440(7084): 637-643. DOI:10.1038/nature04670 |
| [142] |
ALOY P, RUSSELL R B. Structural systems biology: Modelling protein interactions[J]. Nature Reviews Molecular Cell Biology, 2006, 7(3): 188-197. DOI:10.1038/nrm1859 |
| [143] |
LUCK K, KIM D K, LAMBOURNE L, et al. A reference map of the human binary protein interactome[J]. Nature, 2020, 580(7803): 402-408. DOI:10.1038/s41586-020-2188-x |
| [144] |
RODCHENKOV I, BABUR O, LUNA A, et al. Pathway Commons 2019 Update: Integration, analysis and exploration of pathway data[J]. Nucleic Acids Research, 2020, 48(D1): D489-D497. |
| [145] |
GIURGIU M, REINHARD J, BRAUNER B, et al. CORUM: The comprehensive resource of mammalian protein complexes-2019[J]. Nucleic Acids Research, 2019, 47(D1): D559-D563. DOI:10.1093/nar/gky973 |
| [146] |
KIKUGAWA S, NISHIKATA K, MURAKAMI K, et al. PCDq: Human protein complex database with quality index which summarizes different levels of evidences of protein complexes predicted from H-invitational protein-protein interactions integrative dataset[J]. BMC Systems Biology, 2012, 6(suppl 2): S7. DOI:10.1186/1752-0509-6-S2-S7 |
| [147] |
PU S, WONG J, TURNER B, et al. Up-todate catalogues of yeast protein complexes[J]. Nucleic Acids Research, 2009, 37(3): 825-831. DOI:10.1093/nar/gkn1005 |
| [148] |
MEWES H W, DIETMANN S, FRISHMAN D, et al. MIPS: Analysis and annotation of genome information in 2007[J]. Nucleic Acids Research, 2008, 36(suppl 1): D196-D201. |
| [149] |
CHERRY J M, HONG E L, AMUNDSEN C, et al. Saccharomyces Genome Database: The genomics resource of budding yeast[J]. Nucleic Acids Research, 2012, 40(D1): D700-D705. DOI:10.1093/nar/gkr1029 |
| [150] |
GAVIN A C, ALOY P, GRANDI P, et al. Proteome survey reveals modularity of the yeast cell machinery[J]. Nature, 2006, 440(7031): 631-636. |
| [151] |
RUEPP A, ZOLLNER A, MAIER D. The FunCat, a functional annotation scheme for systematic classification of proteins from whole genomes[J]. Nucleic Acids Research, 2004, 32(18): 5539-5545. DOI:10.1093/nar/gkh894 |
| [152] |
TU B P, KUDLICKI A, ROWICKA M, et al. Logic of the yeast metabolic cycle: Temporal compartmentalization of cellular processes[J]. Science, 2005, 310(5751): 1152-1158. DOI:10.1126/science.1120499 |
| [153] |
PRAMILA T, WU W, MILES S, et al. The Forkhead transcription factor Hcm1 regulates chromosome segregation genes and fills the S-phase gap in the transcriptional circuitry of the cell cycle[J]. Genes & Development, 2006, 20(16): 2266-2278. |
| [154] |
GE X J, YAMAMOTO S, TSUTSUMI S, et al. Interpreting expression profiles of cancers by genome-wide survey of breadth of expression in normal tissues[J]. Genomics, 2005, 86(2): 127-141. DOI:10.1016/j.ygeno.2005.04.008 |
| [155] |
BELMONTE M F, KIRKBRIDE R C, STONE S L, et al. Comprehensive developmental profiles of gene activity in regions and subregions of the Arabidopsis seed[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(5): E435-E444. |
| [156] |
LOVE D C, GHOSH S, MONDOUX M A, et al. Dynamic O-GlcNAc cycling at promoters of Caenorhabditis elegans genes regulating longevity, stress, and immunity[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(16): 7413-7418. DOI:10.1073/pnas.0911857107 |
| [157] |
RAMNARINE T J S, GRATH S, PARSCH J. Natural variation in the transcriptional response of Drosophila melanogaster to oxidative stress[J]. G3:Genes|Genomes|Genetics, 2022, 12(1): jkab366. DOI:10.1093/g3journal/jkab366 |
| [158] |
HUH W K, FALVO J V, GERKE L C, et al. Global analysis of protein localization in budding yeast[J]. Nature, 2003, 425(6959): 686-691. DOI:10.1038/nature02026 |
| [159] |
THUL P J, ÅKESSON L, WIKING M, et al. A subcellular map of the human proteome[J]. Science, 2017, 356(6340): eaal3321. DOI:10.1126/science.aal3321 |
| [160] |
KANDASAMY K, KEERTHIKUMAR S, GOEL R, et al. Human proteinpedia: A unified discovery resource for proteomics research[J]. Nucleic Acids Research, 2009, 37(suppl 1): D773-D781. |
| [161] |
WILLADSEN K, MOHAMAD N, BODÉN M. NS-ort/DB: An Intranuclear Compartment Protein Database[J]. Genomics, Proteomics & Bioinformatics, 2012, 10(4): 226-229. |
| [162] |
VERES D V, GYURKÓ D M, THALER B, et al. ComPPI: A cellular compartment-specific database for protein-protein interaction network analysis[J]. Nucleic Acids Research, 2015, 43(D1): D485-D493. DOI:10.1093/nar/gku1007 |
| [163] |
DELLAIRE G, FARRALL R, BICKMORE W A. The Nuclear Protein Database(NPD): Sub-nuclear localisation and functional annotation of the nuclear proteome[J]. Nucleic Acids Research, 2003, 31(1): 328-330. DOI:10.1093/nar/gkg018 |
| [164] |
BINDER J X, PLETSCHER-FRANKILD S, TSAFO-U K, et al. COMPARTMENTS: Unification and visualization of protein subcellular localization evidence[J]. The Journal of Biological Databases & Curation 2014, 2014, bau012.. DOI:10.1093/database/bau012 |
| [165] |
MONTI C, ZILOCCHI M, COLUGNAT I, et al. Proteomics turns functional[J]. Journal of Proteomics, 2019, 198: 36-44. DOI:10.1016/j.jprot.2018.12.012 |
| [166] |
MONTI M, COZZOLINO M, COZZOLINO F, et al. Puzzle of protein complexes in vivo: A present and future challenge for functional proteomics[J]. Expert Review Proteomics, 2009, 6(2): 159-169. DOI:10.1586/epr.09.7 |
| [167] |
张锦雄. 基于多源生物学数据的蛋白质复合物与功能模块识别算法研究[D]. 广州: 华南理工大学, 2020.
|



