2. 广西民族大学海洋与生物技术学院, 广西南宁 530008
2. College of Marine and Biotechnology, Guangxi University for Nationalities, Nanning, Guangxi, 530008, China
新发传染病(Emerging Infectious Diseases, EIDs)和多重耐药性病原菌(Multidrug-resistant pathogens)对人类健康造成严重威胁[1, 2]。然而,近年来由于陆生微生物的新型抗生素逐渐减少,且分离重复性逐渐升高[3],从新的、未开发的、极端的环境中分离新菌株成为开发新型天然产物的有效来源[4-6]。在海平面以下,压力随深度的增加而增加,温度、氧气、光照强度随深度的增加而减少。因高压、低温、缺乏光照、盐度和氧气浓度可变,海洋成为一个独特的极端环境[7]。为适应极端环境,海洋生物已进化出独特的生理、生化功能[8, 9]。独特的海洋环境,使海洋微生物具备与陆生微生物不同的特征,可能产生新的生物活性成分和新型抗生素[10]。目前有关海洋微生物的研究表明:海洋微生物,特别是海洋放线菌能产生新型代谢产物,且部分代谢产物具有新的生物活性,可用于新药的开发[11, 12]。
地处热带和亚热带的广西北部湾海域,蕴含着丰富的海洋细菌资源[13]。但目前对广西北部湾海域海洋微生物的研究主要集中于北部湾滩涂的红树林保护区,关于该海域海洋沉积物细菌多样性及生物活性潜力的研究鲜见报道。因此,本研究利用高通量测序技术结合纯培养分离技术,对广西北部湾局部海域海洋沉积物的细菌多样性进行研究,同时对纯培养菌株的生物活性潜力进行评估,为该海域海洋细菌资源的开发和利用提供参考依据。
1 材料与方法 1.1 主要试剂与仪器溶菌酶、蛋白酶K、Taq DNA聚合酶、dNTPs、PCR Buffer、Marker购自昆明云科生物技术有限公司,其余试剂为国产分析纯试剂。由昆明擎科生物科技有限公司合成PCR所用引物。PCR扩增仪,Biometra公司;电泳仪,Bio-Rad公司;凝胶成像分析系统,Gene Company Limited公司。
1.2 样品采集2020年11月,用抓斗取样器从广西北部湾海域采集海洋沉积物。采样的间隔距离为50 m,停船后在船头用抓斗取样5-10次,5-10个点混为一个样品(共4份),并将样品全部装入无菌密封袋中,立即用冰盒运输至实验室,4.0℃冰箱保存。样品信息如表 1所示。
| 样品 Sample |
经度 Longitude (E) |
纬度 Latitude (N) |
深度 Depth (m) |
样品特征 Characteristics of samples |
| A | 108°09′ | 21°00′ | 22 | 灰色砂(含贝类遗骸) Grey sand (including shellfish remains) |
| B | 108°56′ | 20°99′ | 22 | 灰色砂质粉砂 Grey sandy silt |
| C | 109°28′ | 21°19′ | 22 | 灰色粉砂 Grey silt |
| D | 109°56′ | 21°30′ | 22 | 灰色粉砂 Grey silt |
1.3 高通量测序与分析 1.3.1 高通量测序样品处理
所采集的4份样品为混合样品,为确保结果的准确性,每份样品做3个重复。
1.3.2 PCR扩增与高通量测序利用试剂盒提取海洋沉积物样品总DNA。根据编码细菌核糖体RNA的核酸序列保守区设计引物338F (5′-ACTCCTACGGGAGGCAGCA-3′)和806R (5′-GGACTACHVGGGTWTCTAAT-3′),对样品总DNA进行PCR扩增。30.0 μL PCR扩增体系:Buffer 15.0 μL、dNTPs 6.0 μL、338F 0.9 μL、806F 0.9 μL、KOD FX Neo酶0.6 μL、基因组DNA 50.0 ng,加ddH2O补至30.0 μL。PCR扩增程序:98℃ 2 min;98℃ 0.5 min,50℃ 0.5 min,72℃ 1.0 min,30个循环;72℃ 5.0 min。扩增产物经过纯化、定量和均一化后形成测序文库,质检合格后用Illumina HiSeq 2500进行测序。
1.3.3 数据处理与分析高通量测序获得的数据经过预处理后,用Usearch软件[14]对在97.0%相似度水平上的序列进行聚类,获得操作分类单元(Operational Taxonomic Unit, OTUs)。对OTUs进行物种注释,在界、门、纲、目、科、属、种水平上分析各样品的细菌群落组成及组间差异。采用α多样性分析单个样品的物种丰度及物种多样性,其中用Chao1和Ace指数衡量物种丰度,其值与物种丰度成正比;用Shannon和Simpson指数衡量物种多样性,其值与物种多样性成正比。采用β多样性分析不同样品间细菌群落组成的相似性及差异。
1.4 纯培养细菌的分离与鉴定 1.4.1 培养基采用6种分离培养基:腐殖酸(HV)[15]、YIM 91[16]、YIM 171[17]、M3[18]、海藻糖-天冬酰胺[19]、几丁质。几丁质培养基配方如下:几丁质2.0 g/L,K2HPO4 0.7 g/L,KH2PO4 0.3 g/L,微量盐溶液(FeSO4·7H2O 0.1 g,MnCl2·4H2O 0.1 g,ZnSO4·7H2O 0.1 g,溶于100.0 mL水) 1.0 mL,海盐25.0 g/L,琼脂10.0 g/L,pH值为7.2。
上述6个培养基中添加组合抑制剂(25.0 mg/L萘啶酸+50.0 mg/L重铬酸钾+100.0 mg/L制霉菌素)抑制生长较快的细菌和真菌。
纯化培养基YIM 38 (g/L):酵母提取物4.0,无水葡萄糖4.0,麦芽提取物2.5,复合维生素10.0 mg,琼脂12.0,pH值为7.2。
发酵培养基YIM 61(g/L):大豆粉20.0,蛋白胨2.0,葡萄糖20.0,可溶性淀粉5.0,酵母膏2.0,NaCl 4.0,K2HPO4 0.5,MgSO4·7 H2O 0.5,CaCO3 2.0,pH值为7.2。
LB培养基(g/L):胰蛋白胨10.0,酵母提取物5.0,NaCl 10.0,pH值为7.2。
PDA培养基(g/L): 马铃薯200.0,葡萄糖20.0,pH值为7.2。
1.4.2 样品预处理与菌株分离4份灰色砂质沉积物样品分别混合均匀后置于无菌培养皿中,28℃自然干燥7 d。研磨后称取2.0 g样品,置于80℃烘箱中干热处理1.0 h;然后加到18.0 mL无菌0.1% Na4P2O7溶液中,用摇床180.0 r/min震荡1.0 h;最后用超声波清洗器处理40.0 s得到样品悬浮液,并用0.1% Na4P2O7溶液按10-2,10-3比例稀释。
纯培养采用稀释涂布法,取200.0 μL充分混匀后的10-2,10-3样品稀释液涂布于分离培养基上,28℃倒置培养30 d。培养期间挑取菌落形态不一样的单菌落接种至YIM 38斜面,28℃培养7 d。培养结束后根据菌株形态特征去除重复菌株及污染菌株,对剩余菌株进行编号,然后用YIM 38培养基对编号菌株进行扩大培养及纯化,用20%甘油管于-20℃冰箱内保存。
1.4.3 菌株鉴定与系统发育分析纯化后的菌株用酶法提取DNA[20]。采用细菌通用引物PA (5′-CAGAGTTTGATCCTGGCT-3′)和PB (5′-AGGAGGTGATCCAGCCGCA-3′)进行16S rRNA基因扩增。50 μL PCR扩增体系:Buffer 5.0 μL、dNTPs 4.0 μL、PA 1.0 μL、PB 1.0 μL、Taq DNA聚合酶0.3 μL、ddH2O 37.7 μL、模板DNA 1 μL。PCR扩增程序:94℃ 4 min;94℃ 1.0 min,56℃ 1 min,72℃ 2.0 min,30个循环;72℃ 10.0 min。PCR扩增产物用1.0%琼脂糖凝胶进行电泳检测,将验证结果为阳性的PCR产物送至昆明擎科生物科技有限公司进行测序。使用EZ Biocloud数据库(https://www.ezbiocloud.net/)将测序得到的16S rRNA基因序列进行比对,确定菌株与已发表菌株的相似性。采用MAGE 7.0[21]软件以邻接法(Neighbour-Joining)[22]构建菌株的系统发育树,确定菌株系统发育地位。
1.5 抗菌活性筛选 1.5.1 指示菌本研究选用9种临床和农业生产上常见的病原菌作为指示菌进行抗菌活性实验。革兰氏阳性菌:金黄色葡萄球菌(Staphylococcus aureus CGMCC 1.2386)、枯草芽孢杆菌(Bacillus subtilis CGMCC 1.1849)。革兰氏阴性菌:大肠杆菌(Escherichia coli CGMCC 1.2385)、铜绿假单胞菌(Pseudomonas aeruginosa)。真菌:黑曲霉(Aspergillus niger)、白色念珠菌(Candida albicans CGMCC 2.2086)、小麦赤霉病菌(Gibberella saubinetii)、番茄灰霉病菌(Botrytis cinerea)、马铃薯干腐病菌(Fusarium coeruleum)。其中有保藏号的指示菌由中国普通微生物菌种保藏管理中心提供,无保藏号的指示菌由云南省微生物研究所提供。
1.5.2 抗菌活性检测待测菌株和指示菌用YIM 38固体培养基进行活化。活化后的待测菌株接种到YIM 61液体培养基,28℃少量(6.0 mL)发酵7 d,取发酵液进行活性测试,以空白YIM 61培养基作为阴性对照。活化后的革兰氏阳性、革兰氏阴性指示菌接种到LB培养基,活化后的真菌指示菌接种到PDA培养基,28℃培养3 d。
抗菌活性检测采用双琼脂扩散法。培养基下层为水琼脂,琼脂凝固后加入含有指示菌的YIM 38培养基,并将自制的打孔器置于培养基中。待培养基凝固后取出打孔器,每个孔中加入100.0 μL待测菌株发酵液,28℃培养24 h,观察是否产生抑菌圈并记录抑菌圈的大小。
1.6 功能基因筛选 1.6.1 功能基因引物结合相关文献和本实验室前期研究,设计并合成7种功能基因的简并引物,引物信息见表 2。
| 功能基因 Functional gene |
引物 Primer |
引物序列 Primer sequence |
片段长度 Fragment length (bp) |
退火温度 Annealing temperature (℃) |
参考文献 References |
| Diterpenes | GGDPS-F | 5′-CTSMTSCACGACGACGTSATGGAC-3′ | |||
| GGDPS-R | 5′-SAGSAGGTCGTCSCGSAKCTGGAA-3′ | 500 | 65.0 | [23] | |
| Lugdunin | lug F1 | 5′-AAACTGTCTGGAGGTATT-3′ | |||
| lug R1 | 5′-TTTGGCTATCTATTCTGC-3′ | 758 | 43.0 | [24] | |
| Teixobactin | Tox F2 | 5′-CACTGGCAGCAGGTGGAAGC-3′ | |||
| Tox R2 | 5′-GCAGGTGCAAGGACAGGTCG-3′ | 885 | 64.0 | [25] | |
| Salinomycin | 5 F | 5′-AACGCACGCCGTCGTTCTC-3′ | |||
| 5 R | 5′-ACCAGCCACTTGCCGTCCTC-3′ | 750 | 55.0 | [26] | |
| PKS-Ⅱ | KSα | 5′-TSGCSTGCTTGGAYGCSATC-3′ | |||
| KSβ | 5′-TGGAANCCGCCGAABCCTCT-3′ | 600 | 54.0 | [27] | |
| Leinamycin | lnmJ-F | 5′-GTACCGACCAGGAGATCACCAC-3′ | |||
| lnmJ-R | 5′-GCACAGATACAGCGGCAGATT-3′ | 460 | 57.0 | [28] | |
| Glutarimide | chxBCD-F | 5′-GACACCCAGGTCGTCATCGC-3′ | |||
| chxBCD-R | 5′-CCCGTCATCCAGGTAGTAGCC-3′ | 820 | 60.0 | [29, 30] | |
1.6.2 功能基因检测
根据设计的引物,用PCR扩增技术筛选功能基因。50 μL PCR扩增体系:Buffer 5.0 μL,dNTPs 4.0 μL,正向、反向引物各1.0 μL,Taq DNA聚合酶0.3 μL,ddH2O 37.7 μL,模板DNA 1 μL。PCR扩增程序如下:94℃ 4.0 min;94℃ 1.0 min,退火(温度见表 2)1 min,72℃ 2.0 min,30个循环;72℃ 10.0 min。
用1.0%琼脂糖凝胶对扩增产物进行电泳检测,并将验证后结果为阳性的PCR产物送至昆明擎科生物科技有限公司进行测序。
2 结果与分析 2.1 高通量测序细菌多样性分析 2.1.1 细菌群落组成海洋沉积物测序样品在97.0%相似度水平上进行聚类,共获得1 407个OTUs。就单份样品而言,OTUs在组内存在较大差异(图 1),表明组内样品间细菌群落组成不同,可能是由于测序样品是混合样,未混合均匀造成的。
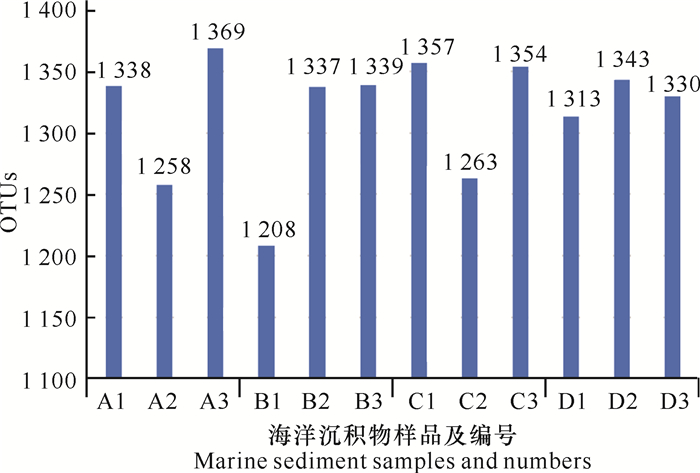
|
| 图 1 高通量测序样品OTUs数目 Fig. 1 OTUs number of high-throughput sequencing samples |
对获得的OTUs进行物种注释,门分类水平上部分物种的分布及丰度见图 2。结果显示, 在门水平上,所有样品中微生物的优势类别相同,均包括变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)和绿屈扰菌门(Chloroflexi),其中变形菌门的丰度最高。另外,梭杆菌门(Fusobacteria)只在B样品(B1、B2、B3)中检测出且有较高的丰度,在其他3个样品中均没有检测出,表明样品的特异性对梭杆菌门的菌株具有选择性。在种分类水平上,注释的OTUs大部分为免培养菌株,表明样品还具有巨大的可培养细菌的潜力。

|
| 图 2 门分类水平上高通量测序样品的物种注释相对丰度 Fig. 2 Relative abundance of species annotation of high-throughput sequencing samples at the phylum classification level |
2.1.2 α多样性分析
海洋沉积物样品α多样性指数如表 3所示。样品Ace指数为1 263.782 7-1 375.893 8,Chao1指数为1 278.069 0-1 381.254 2,Simpson指数为0.880 7-0.988 0,Shannon指数为5.828 7-8.221 0。A3样品4个指数均较高,表明该样品α多样性最高;B1样品4个指数均最低,表明该样品α多样性最低。
| 样品 Sample |
样品编号 Sample number |
Ace指数 Ace index |
Chao1指数 Chao1 index |
Simpson指数 Simpson index |
Shannon指数 Shannon index |
| A | A1 | 1 361.533 8 | 1 371.133 3 | 0.977 1 | 7.685 2 |
| A2 | 1 298.461 5 | 1 308.091 7 | 0.961 9 | 7.059 2 | |
| A3 | 1 375.893 8 | 1 381.097 6 | 0.988 0 | 8.221 0 | |
| B | B1 | 1 263.782 7 | 1 278.069 0 | 0.880 7 | 5.828 7 |
| B2 | 1 354.218 1 | 1 358.863 0 | 0.954 7 | 7.200 3 | |
| B3 | 1 357.809 6 | 1 367.651 5 | 0.962 7 | 7.335 6 | |
| C | C1 | 1 370.922 3 | 1 381.254 2 | 0.978 7 | 7.847 7 |
| C2 | 1 314.123 1 | 1 342.780 2 | 0.970 9 | 7.418 2 | |
| C3 | 1 366.915 2 | 1 370.333 3 | 0.977 9 | 7.724 6 | |
| D | D1 | 1 331.570 7 | 1 334.279 1 | 0.957 7 | 7.026 0 |
| D2 | 1 363.299 2 | 1 375.137 0 | 0.961 3 | 6.997 2 | |
| D3 | 1 353.308 3 | 1 361.662 7 | 0.952 5 | 6.909 1 |
2.1.3 β多样性分析
β多样性分析样品间物种组成相似性的结果如图 3所示。B样品与其余3种样品聚类关系最远,样品间细菌的组成差异最大;B样品和D样品的3个重复均聚类在一起,体现出较近的亲缘关系,说明同一样品内微生物群落的组成及分布较均匀。但是A样品和C样品的3个重复没有完全聚类到一起,存在一定的交叉性,即A2和C2样品表现出较强的聚类关系,表明这两个样品中微生物的组成更为相似。A样品和C样品3个重复样品没有完全聚类的原因可能是:(1)A样品和C样品的3个重复组没有充分混合均匀;(2)两个采样地之间物质和能量的流动性较强,所形成的环境相似,所以样品间的微生物组成也比较相似。
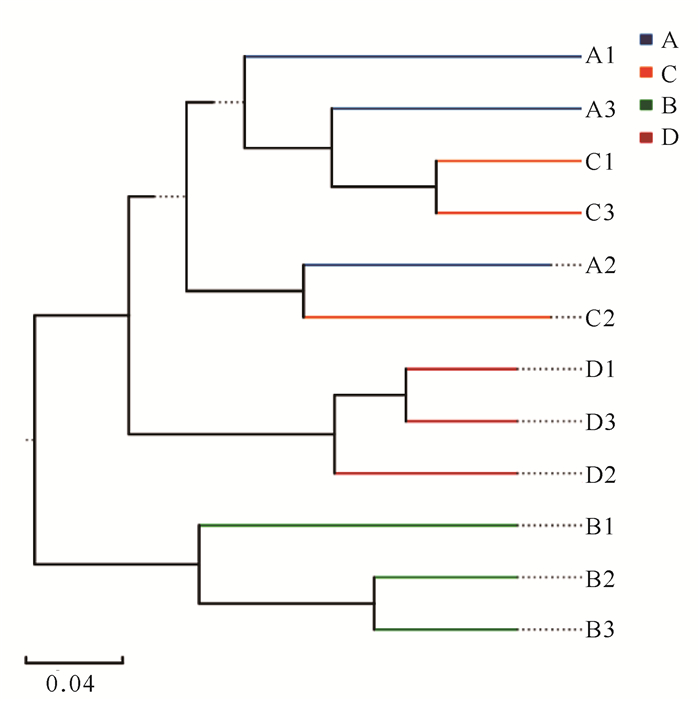
|
| 图 3 基于bray curtis算法构建的UPGMA聚类树 Fig. 3 UPGMA clustering tree based on bray curtis algorithm |
2.2 纯培养多样性分析 2.2.1 细菌多样性
采用6种分离培养基对广西北部湾局部海域采集的4份海洋沉积物样品进行分离,共分离出680株菌。根据菌株形态特征去重复后,对278株菌进行16S rRNA基因测序。经EZ Biocloud数据库比对,278株菌分布于3个门:放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)、变性菌门(Proteobacteria),5个纲:放线菌纲(Actinomycetia)、杆菌纲(Bacilli)、α-变性杆菌纲(Alpha-proteobacteria)、β-变性杆菌纲(Beta-proteobacteria)、γ-变性杆菌纲(Gamma-proteobacteria), 16个目:链霉菌目(Streptomycetales)、微球菌目(Micrococcales)、分枝杆菌目(Mycobacteriales)、假诺卡氏菌目(Pseudonocardiales)、丙酸杆菌目(Propionibacteriales)、动孢囊菌目(Kineosporiales)、链孢菌目(Streptosporangiales)、微杆菌目(Microbacteriales)、短杆菌目(Brevibacteriales)、嗜皮菌目(Dermatophilales)、纤维单孢菌目(Cellulomonadales)、芽孢杆菌目(Bacillales)、根瘤菌目(Rhizobiales)、丙杆菌目(Caulobacterales)、伯克霍尔德氏菌目(Burkholderiales)、假单胞菌目(Pseudomonadales), 24个科:链霉菌科(Streptomycetaceae)、微球菌科(Micrococcaceae)、戈登氏菌科(Gordoniaceae)、类诺卡氏菌科(Nocardiaceae)、迪茨氏菌科(Dietziaceae)、假诺卡氏菌科(Pseudonocardiaceae)、丙酸杆菌科(Propionibacteriaceae)、类诺卡氏菌科(Nocardioidaceae)、动孢囊菌科(Kineosporiaceae)、拟诺卡氏菌科(Nocardiopsaceae)、微杆菌科(Microbacteriaceae)、短杆菌科(Brevibacteriaceae)、皮生球菌科(Dermacoccaceae)、原小单孢菌科(Promicromonosporaceae)、微小杆菌科(Exiguobacteriumf)、游动球菌科(Planococcaceae)、芽孢杆菌科(Bacillaceae)、葡萄球菌科(Staphylococcaceae)、类芽孢杆菌科(Paenibacillaceae)、Aurantimonadaceae、丙杆菌科(Caulobacteraceae)、草酸杆菌科(Oxalobacteraceae)、莫拉氏菌科(Moraxellaceae)、假单胞菌科(Pseudomonadaceae), 31个属:链霉菌属(Streptomyces)、微球菌属(Micrococcus)、考克氏菌属(Kocuria)、节杆菌属(Arthrobacter)、戈登氏菌属(Gordonia)、红球菌属(Rhodococcus)、迪茨氏菌属(Dietzia)、假诺卡氏菌属(Pseudonocardia)、糖多孢菌属(Saccharopolyspora)、Brevilactibacter、类诺卡氏菌属(Nocardioides)、Thalassiella、拟诺卡氏菌属(Nocardiopsis)、微杆菌属(Microbacterium)、农球菌属(Agrococcus)、短杆菌属(Brevibacterium)、巴里恩托斯单孢菌属(Barrientosiimonas)、原小单孢菌属(Promicromonospora)、微小杆菌属(Exiguobacterium)、游动球菌属(Planococcus)、金黄色微菌属(Chryseomicrobium)、芽孢杆菌属(Bacillus)、葡萄球菌属(Staphylococcus)、短芽孢杆菌属(Brevibacillus)、橙色单孢菌属(Aurantimonas)、Aureimonas、短波单孢菌属(Brevundimonas)、马赛菌属(Massilia)、不动杆菌属(Acinetobacter)、嗜冷杆菌属(Psychrobacter)、假单胞菌属(Pseudomonas)。不同属水平上的菌选取一株菌为代表菌与该属的典型菌种用邻接法(Neighbour-Joining)构建系统发育树,如图 4所示。依据16S rRNA序列相似性小于98.65%用于界定物种界限[31],序列比对结果显示,YIM 150853与Nocardioides glacieisoli HLT3-15T (JQ673418)的相似性最高,但也只有98.23%,表明YIM 150853为1个潜在新分类单元,但具体的分类地位需通过多相分类法[32]确定。
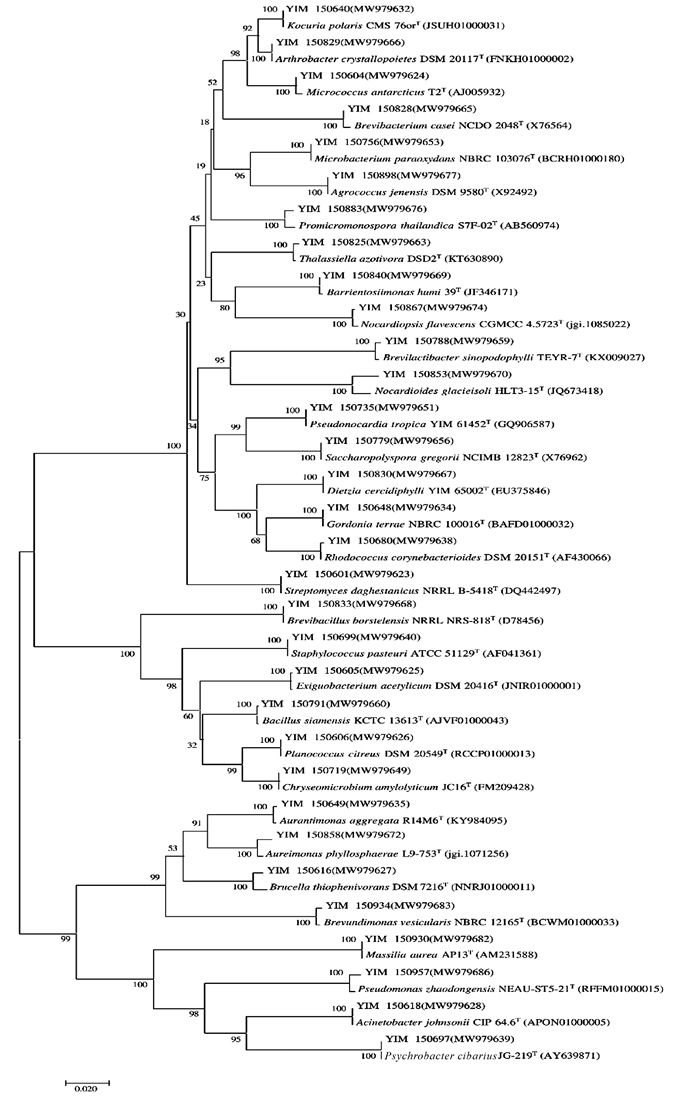
|
| 图 4 基于16S rRNA基因序列构建广西北部湾海域海洋沉积物部分细菌的系统发育树 Fig. 4 Neighbour-Joining phylogenetic tree based on 16S rRNA gene sequences of partial bacteria isolated from marine sediments collected from Guangxi Beibu Gulf |
2.2.2 培养基分离效果
通过6种培养基从4份海洋沉积物样品中分离鉴定出278株菌(31个属65个种),每种培养基均分离出一定数量的细菌。从种水平上来说,HV培养基分离出19种菌,YIM 91培养基和海藻糖-天冬酰胺培养基各分离出15种菌,YIM 171培养基和几丁质培养基各分离出7种菌,而M3培养基仅分离出3种菌。从属水平上来说,HV培养基分离出14个属,YIM 91培养基和海藻糖-天冬酰胺培养基各分离出11个属,YIM 171培养基分离出6个属,几丁质培养基和M3培养基各分离出3个属(图 5)。上述结果表明,HV作为分离培养基获得的菌株数量最多,种属多样性最丰富;其次是YIM 91培养基和海藻糖-天冬酰胺培养基,但YIM 91培养基容易生长真菌,不利于细菌的分离。另外,几丁质培养基虽然分离出4个属的7种菌,但这些菌株仅从B号样品中分离得到,表明几丁质培养基有较强的分离特异性,大部分菌株不能利用几丁质作为碳源,作为分离培养基时普适性较差。

|
| 图 5 不同培养基分离得到的细菌属 Fig. 5 Bacteria genera isolated from different medium |
2.3 抗菌活性及功能基因筛选 2.3.1 抗菌活性
采用双层琼脂扩散法筛选278株细菌的抗菌活性,结果表明, 有212株(76.25%)细菌具有抗1种及1种以上指示菌的活性,另外有168株(60.43%)、159株(57.19%)、158株(56.83%)、151株(54.31%)、139株(50.00%)、131株(47.12%)、131株(47.12%)、22株(7.91%)、5株(1.80%)分别对大肠杆菌、枯草芽孢杆菌、金黄色葡萄球菌、马铃薯干腐病菌、白色念珠菌、黑曲霉、小麦赤霉病菌、铜绿假单胞菌、番茄灰霉病菌有抑制作用。以上说明,广西北部湾局部海域海洋沉积物纯培养细菌具有开发抗生素药物的巨大潜力。
本实验中,链霉菌属的菌株表现出广谱的抗菌活性,且抑菌活性更强。链霉菌YIM 150601对除铜绿假单胞菌、番茄灰霉病菌以外的7种指示菌均有抑菌活性,其中对白色念珠菌的抑菌活性最强,抑菌圈直径达16 mm;链霉菌YIM 150634对大肠杆菌、枯草芽孢杆菌、白色念珠菌、黑曲霉、马铃薯干腐病菌、小麦赤霉病菌、番茄灰霉病菌7种病原菌均有抗菌活性,其中对黑曲霉、马铃薯干腐病菌、小麦赤霉病菌、番茄灰霉病菌的抑菌活性最强,抑菌圈直径均达15 mm以上(表 4)。
| 菌株 Strain |
相似菌株(相似性,%) Closest strain (Similarity,%) |
指示菌抑菌圈直径 Diameter of indicator bacteriostatic zone (mm) |
||||||||
| 金黄色葡萄球菌 S.aureus |
枯草芽孢杆菌 B.subtilis |
大肠杆菌 E.coli |
铜绿假单胞菌 P.aeruginosa |
黑曲霉 A.niger |
白色念珠菌 C.albicans |
马铃薯干腐病菌 F.coeruleum |
番茄灰霉病菌 B.cinerea |
小麦赤霉病菌 G.saubinetii |
||
| YIM 150601 | Streptomyces hydroge- nans (100) | 11 | 10 | 12 | - | 8 | 16 | 10 | - | 12 |
| YIM 150605 | Exiguobacterium acetylicum (99.63) | - | - | - | 9 | - | - | - | - | - |
| YIM 150618 | Acinetobacter johnsonii (99.20) | 4 | - | - | - | - | - | 10 | 6 | - |
| YIM 150619 | Bacillus zhangzhouensis (100) | 6 | - | - | - | - | 6 | - | - | - |
| YIM 150624 | Streptomyces glomer- oaurantiacus (99.53) | 7 | 6 | 6 | - | - | - | 9 | - | - |
| YIM 150634 | Streptomyces youssou- fiensis (99.88) | - | 9 | 8 | - | 15 | 10 | 15 | 16 | 17 |
| YIM 150644 | Streptomyces globis- porus (99.88) | - | - | - | - | - | - | 8 | - | - |
| YIM 150654 | Planococcus maritimus (99.63) | - | - | - | - | - | 7 | - | - | - |
| YIM 150655 | Bacillus altitudinis (100) | - | 8 | - | - | - | - | - | - | - |
| YIM 150680 | Rhodococcus corynebacterioides (99.88) | - | - | - | - | - | - | 11 | - | - |
| YIM 150699 | Staphylococcus pasteuri (100) | - | 9 | - | - | - | - | - | - | - |
| YIM150701 | Psychrobacter fozii (99.64) | - | - | - | 7 | - | - | - | - | - |
| YIM 150709 | Streptomyces albogriseolus (100) | 6 | 11 | 12 | - | - | - | - | - | - |
| YIM 150719 | Chryseomicrobium amylolyticum (99.88) | - | - | - | 7 | - | - | - | - | - |
| YIM 150781 | Kocuria rosea (99.76) | - | 8 | - | - | - | - | - | - | - |
| YIM 150791 | Bacillus siamensis (99.88) | - | - | 13 | 6 | - | - | 7 | - | - |
| YIM 150806 | Streptomyces coelicoflavus (100) | - | 9 | 8 | - | - | - | - | - | - |
| YIM 150827 | Psychrobacter celer (99.76) | - | - | 6 | - | - | - | - | - | - |
| YIM 150833 | Brevibacillus borstelensis (100) | - | - | 10 | - | - | - | - | 8 | - |
| YIM 150840 | Barrientosiimonas humi (99.88) | 8 | - | - | - | - | - | - | - | - |
| YIM 150866 | Bacillus licheniformis (99.63) | - | 4 | 6 | - | - | - | - | - | - |
| YIM 150867 | Nocardiopsis flavescens (99.50) | 7 | - | 7 | - | - | - | - | - | - |
| YIM 150875 | Streptomyces coelicofla- vus (100) | 9 | - | 7 | - | - | - | - | - | - |
| YIM 150957 | Pseudomonas zhaodon- gensis (99.25) | - | 10 | 4 | 6 | - | - | - | - | - |
| YIM 150963 | Pseudomonas oryzihabi- tans (100) | 4 | - | - | - | - | - | 7 | 6 | - |
| YIM 150993 | Brevundimonas terrae (99.40) | 4 | 8 | 8 | - | - | - | - | - | - |
| 注:“-”表示无抑菌圈 Note: “-” means no indicator bacteriostatic zone |
||||||||||
2.3.2 功能基因
用PCR扩增的方法对可培养菌株的基因组进行功能基因筛选,去重复后的实验结果如表 5所示。功能基因筛选结果表明:36株菌(12.90%)至少含有1类功能基因,11株菌(3.94%)含有2类功能基因。二萜类功能基因的阳性率最高,达7.89%,其次依次为雷纳霉素(4.30%)、盐霉素(3.94%)、环缩肽类(1.79%)、PKS-Ⅱ(1.08%)、路邓素(0.36%),但未能检测到含戊二酰亚胺类功能基因的菌株。所筛选到的20株阳性菌株中,15株为放线菌,表明与非放线菌相比,放线菌含有更为丰富的生物合成基因簇,其合成新型天然产物的潜力更大,开发应用价值更大。
| 菌株 Strain |
相似菌株(相似性, %) Closest strain (Similarity, %) |
二萜类 Diterpenes |
路邓素 Lugdunin |
环缩肽类 Teixobactin |
盐霉素 Salinomycin |
聚酮合酶-Ⅱ PKS-Ⅱ |
雷纳霉素 Leinamycin |
戊二酰亚胺类 Glutarimide |
| YIM 150606 | Planococcus citreus (99.88) | - | - | + | + | - | - | - |
| YIM 150640 | Kocuria polaris (100.00) | - | - | - | + | - | - | - |
| YIM 150680 | Rhodococcus corynebacterioides (99.88) | - | - | - | - | - | + | - |
| YIM 150711 | Streptomyces althioticus (99.64) | - | - | - | - | - | + | - |
| YIM 150712 | Streptomyces flavovirens (100.00) | - | - | - | - | + | - | - |
| YIM 150735 | Pseudonocardia tropica (100.00) | + | - | - | - | - | - | - |
| YIM 150756 | Microbacterium paraoxydans(100.00) | + | - | - | - | - | - | - |
| YIM 150757 | Streptomyces badius (99.76) | + | - | - | + | - | - | - |
| YIM 150778 | Rhodococcus cerastii (100.00) | - | - | - | - | - | + | - |
| YIM 150779 | Saccharopolyspora gregorii (99.75) | + | - | - | - | - | - | - |
| YIM 150791 | Bacillus siamensis (99.88) | + | - | - | - | - | + | - |
| YIM 150829 | Arthrobacter crystallopoietes(99.77) | + | - | - | - | - | - | - |
| YIM150830 | Dietzia cercidiphylli (100.00) | + | - | - | - | - | - | - |
| YIM 150853 | Nocardioides glacieisoli (98.23) | + | - | - | - | - | - | - |
| YIM 150856 | Psychrobacter faecalis (99.75) | - | + | - | - | - | - | - |
| YIM 150867 | Nocardiopsis flavescens (99.50) | + | - | - | - | - | - | - |
| YIM 150906 | Rhodococcus cercidiphylli (100.00) | - | - | - | - | + | + | - |
| YIM 150909 | Streptomyces puniceus (100.00) | + | - | - | + | - | - | - |
| YIM 150927 | Psychrobacter maritimus (99.76) | + | - | - | + | - | - | - |
| YIM 150963 | Pseudomonas oryzihabitans (100.00) | + | - | - | + | - | - | - |
| 注:“+”阳性;“-”阴性 Note: "+" Positive; "-" Negative |
||||||||
3 讨论
本研究通过高通量测序技术对广西北部湾局部海域海洋沉积物4份混合样进行测序,结果显示3个平行样品的测序结果均存在一定差异,可能是样品混合不均匀所导致。高通量测序旨在客观、全面地揭示环境样品中细菌多样性和群落组成,由于高通量测序所需样品量少,若样品混合不均匀,则实验结果不能真实反映样品中微生物群落组成。因此,为确保测序结果的准确性,较为全面地揭示环境样品中微生物群落的组成及多样性,采集的样品应充分混合后再进行测序,尽可能减少取样测序时的人为误差。
纯培养结果揭示,广西北部湾局部海域海洋沉积物蕴藏着丰富的细菌资源。本研究从4份海洋沉积物样品中分离出278株细菌,就放线菌而言,链霉菌是优势菌,但稀有放线菌(非链霉菌)有17个属,表明海洋生境蕴含着丰富的稀有放线菌资源,与Subramani等[12]报道结果一致。在属分类水平上,除放线菌有18个属外,芽孢杆菌有6个属,该结果可能与样品预处理有关:放线菌抗逆性强,80.0℃热处理1.0 h有利于放线菌孢子萌发[33];而芽孢有很强的热抵抗力,所以分离到的芽孢杆菌也较多[34]。此外,除几丁质培养基外,本实验所用的5种培养基均是由石松标等[16]、王聪等[18]、彭云霞等[19]、姜怡等[35]、曹艳茹等[36]研究证实的分离细菌效果较好的培养基,尤其是放线菌。但就本实验而言,HV培养基分离效果最好,其次是海藻糖-天冬酰胺培养基,而M3培养基的分离效果最差。结果表明,培养基的分离效果可能受样品类型、采样位置等因素的影响。
北部湾海洋微生物次级代谢产物多样性丰富。截至2020年,多位研究者已从北部湾海洋微生物中分离出110个化合物,其中53个为新型天然产物[37]。本研究通过抗菌活性和功能基因筛选实验评估广西北部湾局部海域海洋沉积物纯培养菌株的生物活性潜力。抗菌活性结果表明,76.34%的菌株至少对1种指示菌有抑菌活性,其中链霉菌YIM 150601、YIM 150634的抑菌谱最广、抑菌活性最强,YIM 150853为类诺卡氏菌属(Nocardioides)的1个潜在新分类单元。基于“新产地、新菌种、新基因、新产物”的观点[38],YIM 150853应该有较好的生物活性,但抗菌活性和功能基因筛选结果却表明,YIM 150853对9种指示菌均无抑菌活性,仅筛选到基因组中含有二萜类化合物的基因簇,该菌株具体的生物活性还有待进一步研究。此外,功能基因筛选和抗菌活性结果显示,与非放线菌相比,放线菌产生生物活性代谢物的潜力更大,更具有开发利用的价值。
近年来,来源于海洋放线菌的新型天然产物逐年增加。1997-2017年,研究者从28个属的海洋放线菌中分离出108种新型天然产物,其中从Salinispora分离的2种天然产物和从链霉菌属分离的1种天然产物处于临床试验阶段[12, 39, 40]。2017-2020年,研究者从5个属的海洋放线菌中分离出50种环肽类化合物,这些化合物对枯草芽孢杆菌、大肠杆菌、白色念珠菌和金黄色葡萄球菌均有抑菌活性,且对鳗弧菌有强烈抑菌活性的环二肽化合物及其衍生物占68%[41-46]。因此,加强广西北部湾海域海洋沉积物细菌资源的研究,特别是放线菌的生物活性研究,有望提高发现新先导化合物的概率,可为新型药物的开发提供候选菌株。
4 结论本研究使用高通量测序和纯培养技术对广西北部湾局部海域海洋沉积物细菌多样性进行研究,结果表明,该海域海洋沉积物中蕴藏着丰富的细菌资源,但由于现阶段纯培养技术的缺陷,仅分离纯化出极少部分菌株。对分离出的可培养菌株进行生物活性评估,结果表明,大量纯培养菌株具有抗菌活性,且部分菌株具有产生活性代谢物的基因簇。综上可知,广西北部湾海域海洋沉积物除了蕴含丰富的细菌资源外,还含有丰富多样的生物合成基因簇及生物活性代谢物。挖掘更多的海洋细菌资源对于今后开发具有新的生物活性化合物具有重大意义。
| [1] |
MORENS D M, FOLKERS G K, FAUCI A S. The challenge of emerging and re-emerging infectious diseases[J]. Nature, 2004, 430(6996): 242-249. DOI:10.1038/nature02759 |
| [2] |
KHAN S N, KHAN A U. Breaking the spell: Combating multidrug resistant superbugs[J]. Frontiers in Microbiology, 2016, 7: 174-184. |
| [3] |
ZOTCHEV S B. Marine actinomycetes as an emerging resource for the drug development pipelines[J]. Journal of Biotechnology, 2012, 158(4): 168-175. DOI:10.1016/j.jbiotec.2011.06.002 |
| [4] |
JENSEN P R, DWIGHT R, FENICAL W. Distribution of actinomycetes in near-shore tropical marine sediments[J]. Applied and Environmental Microbiology, 1991, 57(4): 1102-1108. DOI:10.1128/aem.57.4.1102-1108.1991 |
| [5] |
GOODFELLOW M, FIEDLER H P. A guide to successful bioprospecting: Informed by actinobacterial systematics[J]. Antonie Van Leeuwenhoek, 2010, 98(2): 119-142. DOI:10.1007/s10482-010-9460-2 |
| [6] |
JENSEN P R. Linking species concepts to natural product discovery in the post-genomic era[J]. Journal of Industrial Microbiology and Biotechnology, 2010, 37(3): 219-224. DOI:10.1007/s10295-009-0683-z |
| [7] |
BULL A T, WARD A C, GOODFELLOW M, et al. Search and discovery strategies for biotechnology: The paradigm shift[J]. Microbiology and Molecular Biology Reviews, 2000, 64(3): 573-606. DOI:10.1128/MMBR.64.3.573-606.2000 |
| [8] |
FENICAL W. Chemical studies of marine bacteria: Developing a new resource[J]. Chemical Reviews, 1993, 93(5): 1673-1683. DOI:10.1021/cr00021a001 |
| [9] |
SKROPETA D, WEI L. Recent advances in deep-sea natural products[J]. Natural Product Reports, 2014, 31(8): 999-1025. DOI:10.1039/C3NP70118B |
| [10] |
ELLAIAH P, REDDY A P C. Isolation of actinomycetes from marine sediments off Visakhapatnam, east coast of India[J]. Indian J Mar Sci, 1987, 16(2): 134-135. |
| [11] |
LAM K S. Discovery of novel metabolites from marine actinomycetes[J]. Current Opinion in Microbiology, 2006, 9(3): 245-251. DOI:10.1016/j.mib.2006.03.004 |
| [12] |
SUBRAMANI R, SIPKEMA D. Marine rare actinomycetes: A promising source of structurally diverse and unique novel natural products[J]. Marine Drugs, 2019, 17(5): 249-288. DOI:10.3390/md17050249 |
| [13] |
于清武. 北部湾(广西海域)海洋微生物多样性研究现状与对策[J]. 南方农业学报, 2014, 45(12): 2293-2296. DOI:10.3969/j:issn.2095-1191.2014.12.2293 |
| [14] |
EDGAR R C. UPARSE: Highly accurate OTU sequen-ces from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [15] |
HAYAKAWA M, NONOMURA H. Humic acid-vitamin agar, a new medium for the selective isolation of soil actinomycetes[J]. Journal of Fermentation Technology, 1987, 65(5): 501-509. DOI:10.1016/0385-6380(87)90108-7 |
| [16] |
石松标, 杨立芳, 姜明国, 等. 广西北部湾茅尾海红树林生境放线菌分离培养基的比较[J]. 微生物学通报, 2018, 45(11): 2331-2340. |
| [17] |
SHIRLING E B, GOTTLIEB D. Methods for characterization of Streptomyces species[J]. International Journal of Systematic Bacteriology, 1966, 16(3): 313-340. DOI:10.1099/00207713-16-3-313 |
| [18] |
王聪, 王坤, 姜明国, 等. 广西北部湾放线菌的分离筛选及活性产物的鉴定[J]. 天然产物研究与开发, 2019, 31(7): 1170-1176. |
| [19] |
彭云霞, 姜怡, 段淑蓉, 等. 稀有放线菌的选择性分离方法[J]. 云南大学学报: 自然科学版, 2007, 29(1): 86-89. |
| [20] |
ORSINI M, ROMAN-SPICA V. A microwave-based method for nucleic acid isolation from environmental samples[J]. Letters in Applied Microbiology, 2001, 33(1): 17-20. DOI:10.1046/j.1472-765X.2001.00938.x |
| [21] |
KIMURA M. A simple method for estimating evolu-tionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16(2): 111-120. DOI:10.1007/BF01731581 |
| [22] |
SAITOU N, NEI M. The neighbor-joining method: A new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4): 406-425. |
| [23] |
LUDLOW E J, VASSILIADIS S, EKANAYAKE P N, et al. Analysis of the indole diterpene gene cluster for biosynthesis of the epoxy-janthitrems in Epichloë endophytes[J]. Microorganisms, 2019, 7(11): 560-581. DOI:10.3390/microorganisms7110560 |
| [24] |
ZIPPERER A, KONNERTH M C, LAUX C, et al. Human commensals producing a novel antibiotic impair pathogen colonization[J]. Nature, 2016, 535(7613): 511-516. DOI:10.1038/nature18634 |
| [25] |
LING L L, SCHNEIDER T, PEOPLES A J, et al. A new antibiotic kills pathogens without detectable resistance[J]. Nature, 2015, 517(7535): 455-459. DOI:10.1038/nature14098 |
| [26] |
KNIRSCHOVÁ R, NOVÁKOVÁ R, FECKOVÁ L, et al. Multiple regulatory genes in the salinomycin biosynthetic gene cluster of Streptomyces albus CCM 4719[J]. Folia Micro Biologica, 2007, 52(4): 359-365. DOI:10.1007/BF02932090 |
| [27] |
METSÄ-KETELÄ M, SALO V, HALO L, et al. An efficient approach for screening minimal PKS genes from Streptomyces[J]. FEMS Microbiology Letters, 1999, 180(1): 1-6. DOI:10.1111/j.1574-6968.1999.tb08770.x |
| [28] |
PAN G, XU Z, GUO Z, et al. Discovery of the leinamycin family of natural products by mining actinobacterial genomes[J]. PNAS, 2017, 114(52): E11131-E11140. DOI:10.1073/pnas.1716245115 |
| [29] |
SANDAL I, BHATTACHARYA A, SAINI U, et al. Chemical modification of L-glutamine to alpha-amino glutarimide on autoclaving facilitates Agrobacterium infection of host and non-host plants: A new use of a known compound[J]. BMC Chemical Biology, 2011, 11(1): 1-13. DOI:10.1186/1472-6769-11-1 |
| [30] |
邓洪斌, 李电东, 蔡年生, 等. 戊二酰亚胺类抗生素S632最新研究进展[J]. 中国新药杂志, 2011, 20(23): 2321-2325. |
| [31] |
KIM M, OH H S, PARK S C, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(2): 346-351. |
| [32] |
VANDAMME P, POT B, GILLIS M, et al. Polyphasic taxonomy, a consensus approach to bacterial systematics[J]. Microbiological Reviews, 1996, 60(2): 407-438. DOI:10.1128/mr.60.2.407-438.1996 |
| [33] |
原野, 何山. 海洋稀有放线菌的选择性分离方法[J]. 宁波大学学报: 理工版, 2014, 27(3): 12-17. |
| [34] |
黄忠民, 郭鼐, 潘治利, 等. 生孢梭菌芽孢萌发条件的优化[J]. 现代食品科技, 2021, 37(2): 49-55. |
| [35] |
姜怡, 曹艳茹, 王茜, 等. 波罗的海放线菌的多样性[J]. 微生物学报, 2011, 51(11): 1461-1467. |
| [36] |
曹艳茹, 姜怡, 王茜, 等. 川滇四区森林土壤纯培养放线菌多样性及生物活性[J]. 微生物学报, 2010, 50(8): 995-1000. |
| [37] |
徐新亚, 杨宏, 宁小清, 等. 北部湾海洋微生物物种多样性与化学多样性研究进展[J]. 广西科学, 2020, 27(5): 433-450, 461. |
| [38] |
JIANG Y, CAO Y R, ZHAO L X, et al. Large numbers of new bacterial taxa found by Yunnan Institute of Microbiology[J]. Chinese Science Bulletin, 2011, 56(8): 709-712. DOI:10.1007/s11434-010-4341-7 |
| [39] |
ZOTCHEV S B. Marine actinomycetes as an emerging resource for the drug development pipelines[J]. Journal of Biotechnology, 2012, 158(4): 168-175. DOI:10.1016/j.jbiotec.2011.06.002 |
| [40] |
SUBRAMANI R, AALBERSBERG W. Marine actinomycetes: An ongoing source of novel bioactive metabolites[J]. Microbiological Research, 2012, 167(10): 571-580. DOI:10.1016/j.micres.2012.06.005 |
| [41] |
姚蓉, 马明, 付晖, 等. 海洋放线菌Kocuria sp.次级代谢产物的研究[J]. 中国海洋药物, 2017, 36(3): 11-17. |
| [42] |
林真亭, 叶子坚, 庄玲萍, 等. 一株海洋放线菌抗菌活性物质的分离与结构解析[J]. 江西农业大学学报, 2017, 39(3): 559-566. |
| [43] |
张岗, 庄玲萍, 许莉, 等. 海洋来源放线菌HN-E31次生代谢产物的分离与鉴定[J]. 江西农业大学学报, 2018, 40(5): 1055-1061. |
| [44] |
吉才娟, 王佩, 梅文莉, 等. 海洋放线菌Streptomyces sp.HNWSW-49的次生代谢产物研究[J]. 热带作物学报, 2018, 39(4): 758-762. DOI:10.3969/j.issn.1000-2561.2018.04.023 |
| [45] |
谢春兰, 苏瑞强, 杨全. 海洋放线菌Nocardiopsis sp.SCSIO 11492的抗肿瘤活性成分研究[J]. 广东药科大学学报, 2017, 33(4): 448-452. |
| [46] |
于鑫, 韦霞, 冯婵, 等. 海洋放线菌Streptomyces novaecaesareae次生代谢产物[J]. 中山大学学报: 自然科学版, 2019, 58(3): 63-70. |



