荞麦是一种廖科(Polygonaceae)荞麦属(Fagopyrum Mill.)的双子叶小杂粮作物,甜荞(Fagopyrum esculentum Moench)和苦荞(Fagopyrum tataricum Gaertn.)是其常见的两个栽培种[1]。苦荞含有丰富的多酚化合物、类黄酮和不饱和脂肪酸等生物活性物质,能够降低胆固醇、抑制肿瘤、调节高血压、抗炎和治疗糖尿病等[2, 3]。此外,苦荞还含有丰富的蛋白质、氨基酸、多种维生素和纤维素等成分,是谷类作物中唯一具有七类营养物质的作物[4]。因此,苦荞是一种很好的药食两用作物,具有非常高的药用价值和营养价值,且具有广泛的环境适应能力,能带来良好的经济效应[3, 5, 6]。
花期是植物生命周期中一个十分重要的发育阶段,对经济作物的生态适应性、产量和品质等有着非常重要的影响。苦荞花期的早晚与产量呈极显著负相关关系[7],因此研究荞麦开花的调控机理对提高苦荞产量至关重要。据报道,植物CCT(即CO、COL和TOC1)转录因子家族具有调控花期、生物钟和响应光周期等功能[8, 9]。此外,CTT基因还通过激素信号转导途径参与抵抗干旱、盐、高温、低温等非生物性胁迫,减少这些非生物性胁迫对植物造成的危害[10, 11]。因此CCT基因在植物的生长发育及抗逆境胁迫方面发挥着重要的作用。
CCT基因编码的蛋白都含有CCT结构域,是由CO-LIKE蛋白、CONSTANS (CO)蛋白和TIMING OF CABl (TOCl)蛋白的C末端的43个保守的氨基酸残基组成[12]。根据保守结构域的不同,CCT基因家族分为3个亚家族,分别为CONSTANS-like(COL)亚家族、CCT motif (CMF)亚家族和PRR亚家族。COL亚家族所编码蛋白均包含1-2个B-box结构域和1个CCT结构域,CMF亚家族所编码蛋白仅含有1个CCT结构域,而PRR亚家族所编码蛋白均含有1个CCT结构域和1个Receiver-like (REC)结构域[13]。目前,CCT基因家族已在拟南芥[14]、水稻[8]、小麦[11]、玉米[15]和毛竹[16]等多个物种中被报道。拟南芥的CCT基因家族有40个成员,其中CO基因是最先被发现的,它在长日照条件下能促进开花,突变后拟南芥表现出晚花性状[17];TOC1基因通过昼夜节律调控开花光周期[18];COL1基因在抵抗盐、干旱和温度等非生物胁迫中起到重要作用[14]。水稻中有41个CCT基因,最新研究发现至少有18个成员参与了水稻的开花过程,其中有4个新发现的成员参与抽穗期的调控;3个基因OsCCT22、OsCCT38和OsCCT41表现出在长日照条件下抑制开花,在短日照条件下促进开花的作用,而OsCCT03在长、短日照条件下均能促进水稻开花[8]。玉米中的CCT基因家族成员比其他物种稍多,共有57个,其中有15个成员与花期显著相关[15],而且ZmCOL3基因对开花、茎节数、总叶片株高和穗位高均有影响,呈现出多功能的特点[19]。
在模式植物和大宗作物中CCT基因家族调控花期的研究报道已有不少,但在荞麦中还未见报道。本研究利用生物信息学工具对苦荞CCT基因家族进行全基因组范围的鉴定,并对其理化特性、染色体定位、基因结构、保守基序、保守结构域、系统进化和表达模式等进行分析,拟为进一步探究苦荞CCT基因家族功能,并利用该基因家族改良荞麦品种奠定基础和提供理论依据。
1 材料与方法 1.1 苦荞基因组中CCT基因的筛选及理化性质分析苦荞品种‘cv.Pinku1’的基因组测序数据及其注释信息均从NCBI基因组数据库下载(http://www.mbkbase.org/Pinku1/)。从拟南芥信息资源数据库TAIR (https://www.arabidopsis.org/)下载CCT基因家族序列作为本地BLAST的参考序列(query序列)。运用TBtools(v1.082)软件进行BLAST比对,得到苦荞CCT基因家族候选序列。再运用Conserved Domain Database (CDD)数据库(https://www.ncbi.nlm.nih.gov/Structu-re/bwrpsb.cgi)对这些CCT基因的结构域进行分析,从候选序列中筛选出含有CCT结构域的序列,确认为苦荞的CCT基因家族成员。
运用ExPaSy提供的在线Protparam软件(https://web.expasy.org/protparam/),分析苦荞CCT基因编码的蛋白质的理化性质,包括氨基酸残基的个数、相对分子量、等电点和亲水性等。
1.2 CCT基因染色体分布运用TBtools (v1.082)软件绘制苦荞CCT基因在染色体上的分布图并对其进行共线性分析。
1.3 CCT基因结构、保守基序及结构域分析首先运用MEME在线工具(http://meme-suite.org/)分析苦荞CCT蛋白的氨基酸序列的保守基序,基序个数设置为10个;然后用TBtools(v1.082)软件将预测结果xml文件进行可视化;最后利用TBtools(v1.082)软件分析苦荞CCT基因结构,并利用CDD数据库分析CCT基因保守结构域。
1.4 CCT基因家族系统进化树构建利用MEGA 7.0软件的Cluster W程序对苦荞和拟南芥的CCT基因家族进行多序列比对,采用Neighbor Joining (NJ)法构建系统进化树。
1.5 苦荞CCT基因的表达分析采用Zhang等[20]发表的苦荞‘cv.Pinku1’的转录组数据,根据4种组织(根、茎、叶和花)中的FPKM值,利用TBtools(v1.082)软件绘制CCT基因表达水平的热图。
2 结果与分析 2.1 苦荞CCT基因家族成员的鉴定及在染色体的定位以拟南芥40个CCT基因家族成员为参考序列,并结合CCT基因家族特有的CCT结构域,在苦荞基因组数据库中共鉴定出35个CCT基因家族成员。染色体定位分析表明,苦荞35个CCT基因在8条染色体上均有分布,其中7号染色体上分布的CCT基因数目最多,有9个;其次是8号染色体上,分布了8个成员(图 1)。分别有5个、4个和3个CCT基因定位在2号、1号和5号染色体上。3号、4号和6号染色体上各分布了2个CCT基因。共有8对CCT基因发生片段复制现象(图 1),分别为FtPinG0006138300.01.T01和FtPinG0004072200.01.T01、FtPinG0004072200.01.T01和FtPinG0009564600.01.T01、FtPinG0009564600.01.T01和FtPinG0003531700.01.T01、FtPinG0001582600.01.T01和FtPinG0004578000.01.T01、FtPinG0008789300.01.T01和FtPinG006802300.01.T01、FtPinG0008422500.01.T01和FtPinG0007624400.01.T01、FtPinG0007624400.01.T01和FtPinG0004084100.01.T01以及FtPinG0004084100.01.T01和FtPinG0008422500.01.T01。

|
| 图 1 苦荞FtCCT基因在染色体上的分布及片段复制 Fig. 1 Distribution and fragment duplication of FtCCT genes of F.tataricum on chromosome |
2.2 苦荞FtCCT系统发育、基因结构、保守基序及结构域
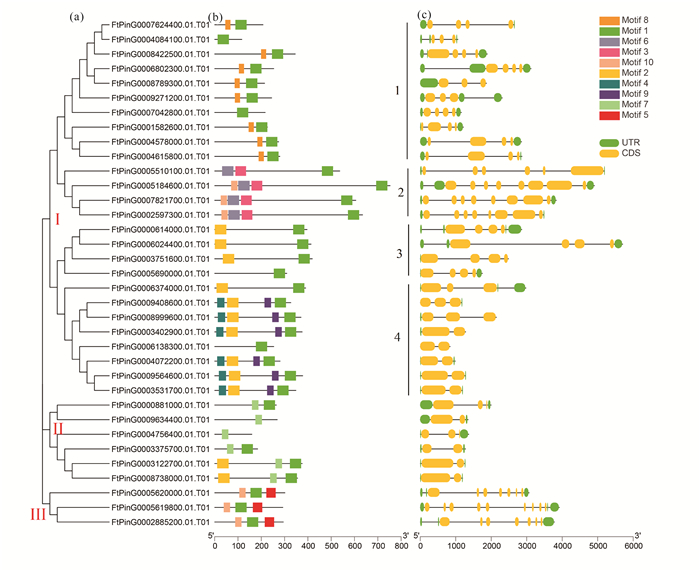
系统发育分析可将苦荞35个CCT基因分成三大类(图 2a),共含有10个保守基序(图 2b)。第Ⅰ类包含了大部分成员,共26个,又可细分为4小组: 第1小组有10个CCT基因,除FtPinG0004084100.01.T01和FtPinG0007042800.01.T01只含有Motif 1,其他都含有Motif 1和Motif 8;第2小组含4个成员,除FtPinG0005510100.01.T01缺少Motif 10,其他3个都具有4个Motif (Motif 1、Motif 5、Motif 6、Motif 10);第3小组由4个CCT基因组成,其中FtPinG0005690000.01.T01只含有Motif 1,其他3个含有Motif 1和Motif 2;第4小组由8个成员组成,其中FtPinG0006138300.01.T01仅含有Motif 1,FtPinG0006374000.01.T01含有Motif 1、Motif 2,其他6个CCT基因由Motif 1、Motif 2、Motif 4和Motif 9组成。第Ⅱ类由6个CCT基因组成,其中2个CCT基因仅含有Motif 7,2个含有Motif 1和Motif 7,还有2个含有Motif 1、Motif 2和Motif 7。第Ⅲ类只有3个CCT基因成员,都含有Motif 1、Motif 5和Motif 10 (图 2a, b)。
|
| 图 2 苦荞FtCCT基因家族的系统发育树(a)、保守基序(b)及基因结构(c) Fig. 2 Phylogenetic tree (a), conserved motif (b) and gene structure (c) of FtCCT gene family in F.tataricum |
35个苦荞CCT基因中,6个CCT基因(FtPinG0004084100.01.T01、FtPinG0007042800.01.T01、FtPinG0005690000.01.T01、FtPinG0006138300.01.T01、FtPinG0009634400.01.T01和FtPinG0004756400.01.T01) 只含有1个基序,其他29个CCT基因含有2-4个基序(图 2a, b)。除了两个成员(FtPinG0009634400.01.T01和FtPinG0004756400.01.T01) 不具有Motif 1, 其他33个CCT基因成员都具有Motif 1,占比达到94%;12个CCT基因成员具有Motif 2的基因,比例为34%。这说明Motif 1和Motif 2可能为苦荞CCT基因家族最保守的基序(图 2b)。在Pfam数据库中(http://pfam.xfam.org/),Motif 1被注释为CCT结构域,Motif 2和Motif 4被注释为B-box结构域,Motif 3和Motif 6被注释为和调节信号响应相关的结构域,Motif 5被注释为GATA zinc finger,Motif 10被注释为tify结构域,Motif 7、Motif 8和Motif 9的注释未知。
苦荞的35个CCT基因内含子数目为1-8,其中FtPinG0005619800.01.T01含有的内含子数目最多,有8个;其次是FtPinG0005184600.01.T01、FtPinG0007821700.01.T01和FtPinG0002597300.01.T01 (均位于第Ⅰ类第2小组中),均含有7个内含子(图 2c),9个CCT基因成员只有1个内含子。
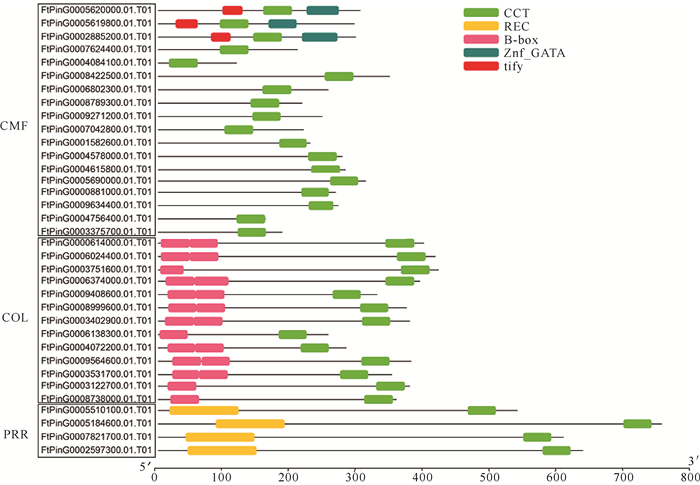
苦荞CCT基因家族所编码蛋白中含有5种主要的保守结构域,分别是CCT、B-box、Znf-GATA、tify和REC (图 3),其中CTT结构域在所有苦荞CCT基因编码蛋白中都有出现,说明该结构域在植物进化过程中高度保守。苦荞CCT基因家族中的CMF亚家族的成员最多(18个),此亚族所编码的蛋白都含有1个CCT结构域,其中还有3个含有tify结构和Znf-GATA结构域,并且这3个蛋白的大小相似;COL亚家族由13个CCT基因组成,所编码蛋白中除了4个成员只含有1个B-box和1个CCT结构域外,其他成员都含有2个B-box和1个CCT结构域;PRR亚家族中的CCT基因数目最少,只有4个,所编码蛋白由1个CCT和1个REC结构域组成。
|
| 图 3 苦荞FtCCT基因家族保守结构域 Fig. 3 Conserved domain of FtCCT gene family in F.tataricum |
2.3 苦荞FtCCT基因家族的理化性质
对35个FtCCT基因编码的蛋白质的理化性质分析表明,CCT基因家族编码的蛋白质含有117-753个氨基酸残基,相对分子质量为4 082.90-82 741.52 Da,预测的理论等电点为4.96-9.51,且以偏酸性和中性居多。预测的苦荞CCT基因家族蛋白质的亲水指数均为负值,表明蛋白质均属于亲水性蛋白,其中以FtPinG0007624400.01.T01的亲水性最强,平均亲水指数为-9.180 (表 1)。
| 基因ID Gene ID |
氨基酸个数 Number of amino acids (aa) |
分子量 Molecular weight (Da) |
理论等电点 Theoretical pI |
平均亲水指数 Grand average of hydropathicity |
| FtPinG0006802300.01.T01 | 254 | 28 506.62 | 5.40 | -0.070 |
| FtPinG0008789300.01.T01 | 251 | 24 460.34 | 5.86 | -0.675 |
| FtPinG0007624400.01.T01 | 280 | 23 431.93 | 7.56 | -9.180 |
| FtPinG0008422500.01.T01 | 346 | 39 349.17 | 5.28 | -0.776 |
| FtPinG0004084100.01.T01 | 117 | 13 882.15 | 5.81 | -1.475 |
| FtPinG0007042800.01.T01 | 217 | 24 969.69 | 7.17 | -0.925 |
| FtPinG0009271200.01.T01 | 245 | 28 135.74 | 6.17 | -1.049 |
| FtPinG0004578000.01.T01 | 275 | 30 855.76 | 5.31 | -0.468 |
| FtPinG0004615800.01.T01 | 280 | 31 326.54 | 5.53 | -0.380 |
| FtPinG0001582600.01.T01 | 227 | 26 143.52 | 5.44 | -0.530 |
| FtPinG0009564600.01.T01 | 378 | 41 234.24 | 5.66 | -0.334 |
| FtPinG0004072200.01.T01 | 281 | 31 151.97 | 7.36 | -0.371 |
| FtPinG0003402900.01.T01 | 376 | 4 082.90 | 5.52 | -0.432 |
| FtPinG0003531700.01.T01 | 394 | 38 323.00 | 6.01 | -0.255 |
| FtPinG0006138300.01.T01 | 254 | 27 914.32 | 5.51 | -0.345 |
| FtPinG0009408600.01.T01 | 327 | 36 302.66 | 6.31 | -0.522 |
| FtPinG0008999600.01.T01 | 371 | 41 072.56 | 5.91 | -0.631 |
| FtPinG0003122700.01.T01 | 376 | 42 553.85 | 6.05 | -0.668 |
| FtPinG0000881000.01.T01 | 265 | 29 461.10 | 6.26 | -0.752 |
| FtPinG0008738000.01.T01 | 356 | 40 364.42 | 7.71 | -0.790 |
| FtPinG0007821700.01.T01 | 606 | 68 023.00 | 6.04 | -0.691 |
| FtPinG0006374000.01.T01 | 391 | 43 083.77 | 4.96 | -0.556 |
| FtPinG0005510100.01.T01 | 537 | 60 598.89 | 6.00 | -0.745 |
| FtPinG0004756400.01.T01 | 160 | 18 352.63 | 7.74 | -0.827 |
| FtPinG0003375700.01.T01 | 185 | 21 025.74 | 9.51 | -0.908 |
| FtPinG0005620000.01.T01 | 302 | 32 932.18 | 6.11 | -0.768 |
| FtPinG0002597300.01.T01 | 635 | 70 527.61 | 5.87 | -0.783 |
| FtPinG0003751600.01.T01 | 419 | 45 833.02 | 4.96 | -0.455 |
| FtPinG0005184600.01.T01 | 753 | 82 741.52 | 6.01 | -0.821 |
| FtPinG0005690000.01.T01 | 310 | 35 031.36 | 5.92 | -1.083 |
| FtPinG0000614000.01.T01 | 397 | 43 516.39 | 5.42 | -0.620 |
| FtPinG0006024400.01.T01 | 414 | 45 045.81 | 4.98 | -0.519 |
| FtPinG0009634400.01.T01 | 269 | 20 528.68 | 7.66 | -0.710 |
| FtPinG0005619800.01.T01 | 293 | 32 063.96 | 6.05 | -0.545 |
| FtPinG0002885200.01.T01 | 295 | 32 443.75 | 5.87 | -0.889 |
2.4 苦荞FtCCT基因家族进化分析结果
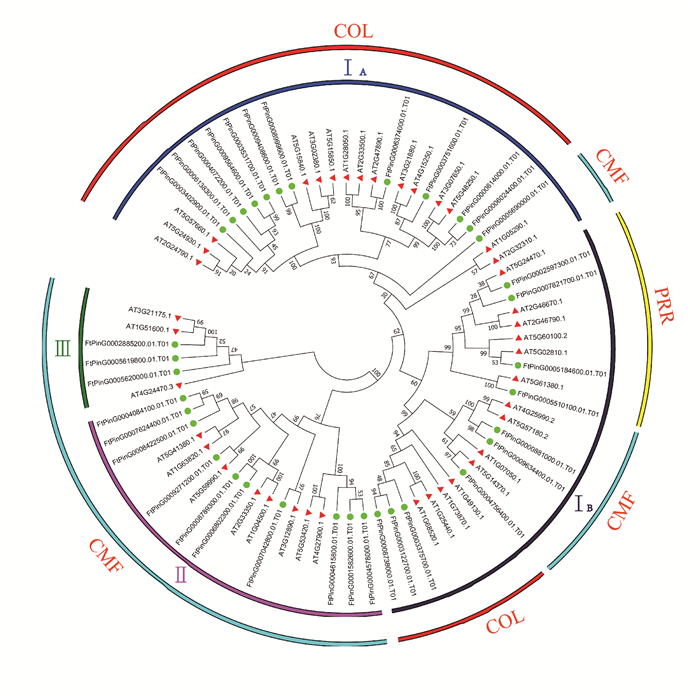
系统进化分析发现苦荞和拟南芥CCT基因家族成员可分为三大类: Ⅰ、Ⅱ和Ⅲ(图 4)。其中,Ⅰ类中的基因数目最丰富,由29个拟南芥CCT基因和22个苦荞CCT基因组成,可以进一步分为ⅠA和ⅠB两个亚类;Ⅱ类包含8个拟南芥和10个苦荞的CCT基因成员;Ⅲ类中拟南芥和苦荞各3个CCT基因成员。
|
| 图 4 苦荞和拟南芥CCT基因家族系统进化 Fig. 4 Phylogenetic tree of CCT gene family from F.tataricum and Arabidopsis |
根据姚新转等[21]对拟南芥CCT基因家族的分类方法,可以将苦荞的35个CCT基因家族分为COL、CMF和PRR 3个亚家族(图 4),与图 3的结果基本一致。其中CMF亚家族包含了分布在进化树上的Ⅱ类和Ⅲ类中的全部CCT成员,以及IA和IB两个亚类中的小分支;COL亚家族主要分布在进化树中的IA亚类以及IB亚类中的一个分支上;PRR亚家族由IB的一个分支组成。
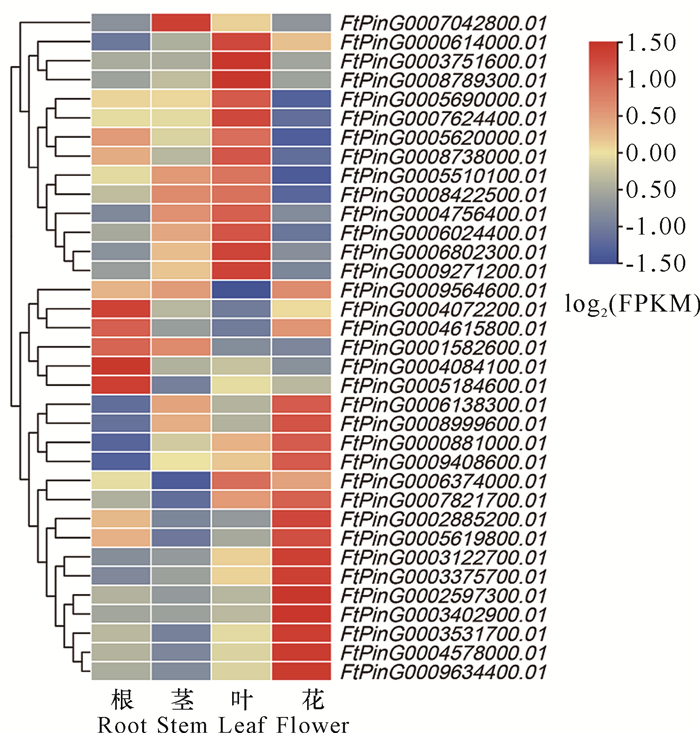
2.5 苦荞CCT基因家族在组织中的表达模式苦荞35个CCT基因在根、茎、叶和花4个组织中的表达模式如图 5所示,有5个CCT基因在根中表达水平高,推测可能与根的生长发育调控有关;只有1个CCT基因在茎中显著高表达,推测其可能参与茎的生长发育过程。在叶中表达水平较高的CCT基因的数目较多,而且大多数是CMF亚家族成员,他们可能参与叶的生长发育与光形态建成。在花中表达水平较高的CCT基因的数目也较多,主要是COL和CMF亚家族成员,这些基因可能参与调控开花过程,比如开花时间、花期长短等。
|
| 图 5 苦荞CCT基因家族在不同组织中的表达模式 Fig. 5 Expression pattern of CCT genes in different tissues of F.tataricum |
3 讨论
植物CCT基因家族在调节植物的生长发育和抗逆境胁迫中起着至关重要的作用,并且大多数成员参与开花调控,与作物的产量也息息相关[8, 16, 22, 23]。本研究在全基因组水平上共鉴定到35个苦荞FtCCT基因,数目与其他物种相差不大,含有1-8个内含子(图 2)。这些苦荞CCT蛋白等电点为4.96-9.51,绝大多数以酸性为主,均为亲水性蛋白(表 1)。35个苦荞FtCCT基因在8条染色体上均有分布,且8对FtCCT基因发生了片段复制事件(图 1)。片段重复在CCT基因家族成员的扩增中可能起着重要的作用,推测这些同源性较高的复制基因具有相似的生物学功能。苦荞CCT蛋白与其他物种一样含CCT、B-box、Znf-GATA、tify和REC等5种主要的保守结构域(图 3)。所有的苦荞CCT蛋白都含有CCT结构域,说明该结构域在植物进化过程中高度保守。
为更好地了解苦荞FtCCT基因家族成员的分类和可能具有的功能,本研究将40个拟南芥AtCCT家族成员与35个苦荞FtCCT家族成员进行蛋白序列比对并构建系统进化树(图 4)。结果表明,两者的CCT基因具有较高的同源性,说明CCT基因在物种间比较保守,可能具有相似的功能。根据姚新转等[21]对拟南芥AtCCT基因家族的分类方法,苦荞的35个FtCCT基因家族也可以分为COL、CMF和PRR 3个亚家族,且CCT基因家族的功能可能与Motif以及结构域相关。Putterill等[17]研究表明,拟南芥的AtCO基因(At15840.1) 属于COL亚家族,具有促进开花的功能,苦荞中两个AtCO同源基因FtPinG0008999600.01.T01和FtPinG0009408600.01.T01在花组织中具有较高的表达水平(图 5),因此推测FtPinG0008999600.01.T01和FtPinG0009408600.01.T01可能具有相似的功能,并且苦荞的COL亚家族的12个成员中有7个在花组织中高表达(图 5),这与拟南芥的COL亚家族中的大部分成员参与拟南芥开花时间的调控一样[18]。属于拟南芥PRR亚家族的TOC1被报道影响昼夜节律[24],苦荞中的同源基因FtPinG0005510100.01.T01可能也参与生物钟的调控。PRR亚家族的4个成员中有2个在花中高表达,在根和叶中各有1个表达水平较高(图 5),因此,不同的成员可能对不同组织的生长发育起着调控作用。CMF亚家族对谷类作物的驯化和适应起着重要作用,且对开花起着抑制作用[25, 26],而苦荞中的CMF亚家族的成员最丰富,其中有8个成员在叶中高表达,有6个在花中高表达,还有少数成员在根和茎中具有较高的表达水平,这些基因可能调控不同组织的生长发育。因此,苦荞CCT基因家族中的有些成员可能与拟南芥的同源基因具有相似的功能,而特异的Motif和结构域可能是不同亚家族成员之间功能差异的主要原因。
4 结论植物CCT基因家族在调节植物的生长发育、花期和抗逆境胁迫中起着至关重要的作用。本研究在全基因组水平上共鉴定出35个苦荞CCT基因,在8条染色体上均有分布。苦荞CCT基因编码的蛋白的等电点为4.96-9.51,绝大多数以酸性为主,均为亲水性蛋白。基于CCT、B-box、Znf-GATA、tify和REC等5种主要的保守结构域,将苦荞的CCT基因家族划分为CMF、COL和PRR 3个亚家族。苦荞和拟南芥CCT基因具有较高的同源性。在叶和花中高表达的CCT基因数目较多,推测这些基因可能参与了生长发育、光形态建成或开花调控过程。以上研究结果为进一步探究苦荞CCT基因家族功能,以及利用该基因家族改良荞麦花期奠定了基础。
| [1] |
盛晋华, 张雄杰, 陕方, 等. 内蒙古自治区荞麦生产开发现状与对策[J]. 作物杂志, 2009(6): 1-5. DOI:10.3969/j.issn.1001-7283.2009.06.001 |
| [2] |
吴韬, 肖丽, 李伟丽. 苦荞的营养与功能成分研究进展[J]. 西华大学学报: 自然科学版, 2021, 40(2): 91-96. |
| [3] |
JOSHI D C, ZHANG K, WANG C, et al. Strategic enhancement of genetic gain for nutraceutical development in buckwheat: A genomics-driven perspective[J]. Biotechnology Advances, 2020, 39: 107479. DOI:10.1016/j.biotechadv.2019.107479 |
| [4] |
何伟俊, 曾荣, 白永亮, 等. 苦荞麦的营养价值及开发利用研究进展[J]. 农产品加工, 2019(23): 69-75. |
| [5] |
TOLAINI V, DEL FIORE A, NOBILI C, et al. Exploitation of tartary buckwheat as sustainable ingredient for healthy foods production[J]. Agriculture and Agricultural Science Procedia, 2016, 8: 455-460. DOI:10.1016/j.aaspro.2016.02.043 |
| [6] |
刘邻渭, 章华伟, 姜丽. 中国荞麦深加工的探索[J]. 西北农林科技大学学报: 自然科学版, 2002, 30(增刊): 83-85. |
| [7] |
吕丹, 黎瑞源, 郑冉, 等. 213份苦荞种质资源主要农艺性状分析及高产种质筛选[J]. 南方农业学报, 2020, 51(10): 2429-2439. DOI:10.3969/j.issn.2095-1191.2020.10.014 |
| [8] |
ZHANG J, FAN X, HU Y, et al. Global analysis of CCT family knockout mutants identifies four genes involved in regulating heading date in rice[J]. Journal of Integrative Plant Biology, 2021, 63(5): 913-923. DOI:10.1111/jipb.13013 |
| [9] |
袁玺垒, 王振山, 贾小平, 等. 光周期调控植物开花分子机制以及CCT基因家族研究进展[J]. 浙江农业学报, 2020, 32(6): 1133-1140. DOI:10.3969/j.issn.1004-1524.2020.06.21 |
| [10] |
MA L, YI D, YANG J, et al. Genome-wide identification, expression analysis and functional study of CCT gene family in Medicago truncatula[J]. Plants, 2020, 9(4): 513. DOI:10.3390/plants9040513 |
| [11] |
田书军. 小麦TaCCT基因家族的全基因组鉴定及非生物胁迫下的表达模式分析[D]. 杨凌: 西北农林科技大学, 2020.
|
| [12] |
ROBSON F, COSTA M M, HEPWORTH S R, et al. Functional importance of conserved domains in the flowering-time gene CONSTANS demonstrated by analysis of mutant alleles and transgenic plants[J]. The Plant Journal, 2001, 28(6): 619-631. |
| [13] |
ZHANG L, LI Q, DONG H, et al. Three CCT domain-containing genes were identified to regulate heading date by candidate gene-based association mapping and transformation in rice[J]. Scientific Reports, 2015, 5(1): 7663. DOI:10.1038/srep07663 |
| [14] |
刘芳, 肖衡, 金玉环, 等. 短命植物小拟南芥光周期调控基因CONSTANS的克隆与表达特征分析[J]. 石河子大学学报: 自然科学版, 2019, 37(2): 190-199. |
| [15] |
JIN M, LIU X, JIA W, et al. ZmCOL3, [STB1] a CCT gene represses flowering in maize by interfering with the circadian clock and activating expression of ZmCCT[J]. Journal of Integrative Plant Biology, 2018, 60(6): 465-480. DOI:10.1111/jipb.12632 |
| [16] |
张文祥, 李敏, 王汇源, 等. 毛竹CCT基因家族成员的鉴定及表达分析[J/OL]. 分子植物育种: 1-12. [2021-05-09]. http://kns.cnki.net/kcms/detail/46.1068.S.20210308.1708.028.html.
|
| [17] |
PUTTERILL J, ROBSON F, LEE K, et al. The CONSTANS gene of Arabidopsis promotes flowering and encodes a protein showing similarities to zinc finger transcription factors[J]. Cell, 1995, 80(6): 847-57. DOI:10.1016/0092-8674(95)90288-0 |
| [18] |
STRAYER C, OYAMA T, SCHULTZ T F, et al. Cloning of the Arabidopsis clock geneTOC1, [STB1] an autoregulatory response regulator homolog[J]. Science, 2000, 289(5480): 768-771. DOI:10.1126/science.289.5480.768 |
| [19] |
贾伟. 玉米光周期敏感基因ZmCOL3的功能解析[D]. 沈阳: 吉林农业大学, 2018.
|
| [20] |
ZHANG L, LI X, MA B, et al. The Tartary buckwheat genome provides insights into rutin biosynthesis and abiotic stress tolerance[J]. Molecular Plant, 2017, 10(9): 1224-1237. DOI:10.1016/j.molp.2017.08.013 |
| [21] |
姚新转, 张宝会, 赵德刚, 等. 茶树CsCCT基因家族的鉴定和表达模式分析[J/OL]. 分子植物育种: 1-27. [2021-05-13]. http://kns.cnki.net/kcms/detail/46.1068.S.20210225.1749.011.html.
|
| [22] |
李晓华, 丁明亮, 乔玲, 等. 粗山羊草CCT基因家族进化及节律表达分析[J]. 植物遗传资源学报, 2017, 18(6): 1151-1158. |
| [23] |
MENGARELLI D A, ZANOR M I. Genome-wide characterization and analysis of the CCT motif family genes in soybean (Glycine max)[J]. Planta, 2021, 253(1): 15. DOI:10.1007/s00425-020-03537-5 |
| [24] |
TAKATA N, SAITO S, SAITO C T, et al. Phylogenetic footprint of the plant clock system in angiosperms: Evolutionary processes of pseudo-response regulators[J]. BMC Evolutionary Biology, 2010, 10(1): 126. DOI:10.1186/1471-2148-10-126 |
| [25] |
LI Y, XU M. CCT family genes in cereal crops: A current overview[J]. The Crop Journal, 2017, 5(6): 449-458. DOI:10.1016/j.cj.2017.07.001 |
| [26] |
章佳. 水稻CCT家族基因的功能研究和Hd1[STB1] 的重新克隆[D]. 武汉: 华中农业大学, 2017.
|



