2. 广西中医药大学海洋药物研究院, 广西南宁 530200;
3. 广西水产科学研究院, 广西南宁 530021
2. Institute of Marine Drugs, Guangxi University of Chinese Medicine, Nanning, Guangxi, 530200, China;
3. Guangxi Academy of Fishery Sciences, Nanning, Guangxi, 530021, China
星虫动物(Sipuncula)是一类圆筒状不分节、具体腔、营穴居生活的海洋蠕虫,是海洋动物独有的门类。目前,已有研究报道的星虫动物超过238种,它们广泛分布于世界各大洋中,从热带海域到极地海域,从深海区到潮间带[1]。中国沿海已发现的星虫动物有2纲4目6科41种,从南至北均有分布,南方热带海域的种数最多[2, 3]。其中,广西北部湾海域常见星虫动物为裸体方格星虫(Sipunculus nudus)、可口革囊星虫(Phascolosoma esculenta)和厥目革囊星虫(P.scolops)等,它们生物资源总量大, 肉质鲜美, 营养和药用保健价值高,是当地广受欢迎的海产珍品[4, 5]。然而,随着市场价格的逐年升高,广西北部湾星虫动物遭到过度采捕,再加上栖息地环境被破坏,其野生资源急剧减少[6, 7]。星虫动物的进化地位至今仍存在争议,其物种形态鉴别特征容易受到环境因素的影响,再加上相似物种的趋同进化,往往导致星虫物种分类的错误和混乱[8]。因此,急需开展星虫动物遗传多样性、生物条形码和系统进化分析等种质资源保护和可持续开发利用方面的基础研究。
线粒体基因组作为核外遗传物质,具有拷贝数量多、母系遗传和进化速率快等特点[9],随着高通量测序技术的快速发展,越来越多的研究开始应用线粒体基因组开展后生动物分子系统进化和种群遗传多样性研究[10, 11]。相比单个线粒体基因,线粒体基因组具有更丰富的遗传进化信息,其基因排列重组也蕴含丰富的进化信息,在解决后生动物系统进化关系和种群遗传多样性的研究中具有较高的应用价值[12]。然而,针对星虫动物门线粒体基因组的研究长期滞后,2009年才有首个线粒体基因组见报道[13],至今Genbank上仅有3种星虫动物线粒体基因组序列可供公开下载[14],远不能满足星虫动物系统进化和遗传多样性的研究需求。为探明广西北部湾常见星虫动物线粒体基因组信息,本研究应用高通量测序技术测定5种星虫动物线粒体基因组序列,并统计分析其遗传变异信息和系统进化关系,为广西北部湾星虫动物渔业资源多样性调查和保护提供分子遗传数据,并为星虫动物系统进化研究提供科学参考。
1 材料与方法 1.1 样品采集和鉴定研究样品为裸体方格星虫、邱米管体星虫(Siphonosoma cumanense)、可口革囊星虫、厥目革囊星虫、类革囊星虫(Phascolosoma similis)等5种广西北部湾常见星虫动物,采自广西北海市银海区和铁山港区近海潮间带滩涂。每个物种采集6个以上个体带回实验室,解剖后参考文献[15]开展形态鉴定,确定种类后采集体壁肌肉组织放置于-80℃超低温冰箱保存。
1.2 基因组DNA提取和高通量测序参考陈建琴等[16]的方法略做修改,每个个体取肌肉组织约30 mg,采用TaKaRa MiniBEST Universal Genomic DNA Extraction Kit (TAKARA BIO INC.)技术从肌肉组织提取基因组DNA,并采用琼脂糖凝胶电泳法检测基因组DNA降解程度与蛋白质污染程度,同时使用NanoDrop 2000超微量分光光度计检测基因组DNA浓度和质量。选取符合高通量测序要求的DNA样品,同一物种的6个个体基因组DNA混合后,用干冰保存送往深圳华大基因股份有限公司开展简化基因组建库测序。测序文库片段长度为350 bp,使用Illumina PE150双末端测序平台完成简化基因组测序。
1.3 线粒体基因组序列组装和基因注释采用Trimmomatic软件对原始测序数据进行质量筛选[17],以GenBank上已经公布的法国群体的裸体方格星虫线粒体基因组序列(登录号:NC_011826)作为参考序列,使用MITObim软件完成广西北部湾5种常见星虫动物线粒体基因组序列组装[18]。利用在线的线粒体基因注释工具tRNAscan-SE、MITOS等分析预测线粒体蛋白质编码基因(PCGs)序列[19]、核糖体RNA基因序列(rRNAs)、转运RNA基因序列(tRNAs)的编码区域和结构特征,完成线粒体基因组序列的识别和注释。
1.4 线粒体基因组遗传变异信息和系统进化分析采用Clustal X软件对5种广西北部湾常见星虫动物线粒体基因组蛋白质编码基因进行多重序列比对,使用DnaSP v6和PAML等软件统计分析线粒体基因组蛋白质编码基因变异位点数、非同义替换率(Ka)和同义替换率(Ks)等遗传变异信息。从GenBnak下载30个物种的线粒体基因组序列,包括5个星虫动物、7个环节动物、2个腕足动物、2个线虫动物、6个软体动物、1个轮形动物、3个刺胞动物、2个螠虫动物、2个扁盘动物,再加上本次获得的5个星虫动物的线粒体基因组,使用PhyML 3.0软件的最大似然法(ML)构建系统发育树的最佳进化模型[20],分析星虫动物的系统进化。
2 结果与分析 2.1 线粒体基因组基因序列特征星虫动物肌肉组织基因组DNA提取结果显示:样品轻微降解,DNA浓度高于100 ng/μL,符合建库测序要求。高通量测序共产生原始数据42 G,其中碱基测序质量高于Q20的数据超过90%,经过质量筛选去除低质量测序碱基,使用MITObim软件组装拼接5个星虫动物的线粒体全基因序列。
基因组装和基因注释结果显示,广西北部湾的裸体方格星虫、邱米管体星虫、可口革囊星虫、厥目革囊星虫和类革囊星虫的线粒体基因组序列全长分别为15 375,15 917,15 494,15 515,16 237 bp,线粒体基因组碱基组成均呈AT偏好,其A+T含量分别为57.5%、60.8%、65.6%、65.4%和63.5%。可口革囊星虫、厥目革囊星虫、类革囊星虫的A+T含量高于裸体方格星虫和邱米管体星虫。注释后的线粒体基因组序列上传至GenBnak,其接收号分别为MG873457、MN813483、MG873458、MT239480和MN813482。方格星虫属裸体方格星虫线粒体基因组由37个基因组成(图 1),包括13个蛋白质编码基因、22个tRNAs和2个rRNAs,与无脊椎动物线粒体基因组原始基因组成一致。然而,管体星虫属的邱米管体星虫(图 2)与可口革囊星虫属的可口革囊星虫(图 3)、厥目革囊星虫(图 4)、类革囊星虫(图 5)这4种星虫的线粒体基因组由38个基因组成,比裸体方格星虫线粒体基因组多一个重复的tRNAMet。此外,本研究中广西北部湾5种常见星虫的线粒体基因组的基因均编码在H链上,星虫动物的基因排列具有高度的保守性,存在3个高度保守的基因排列区块:(1) nad6-cob- tRNAPro;(2) nad4l-nad4- tRNALeu;(3) nad1- tRNATrp- atp6- tRNAArg-tRNAHis- nad5。蛋白质编码基因和2个rRNAs基因排列顺序保持一致,仅有部分tRNAs发生易位。
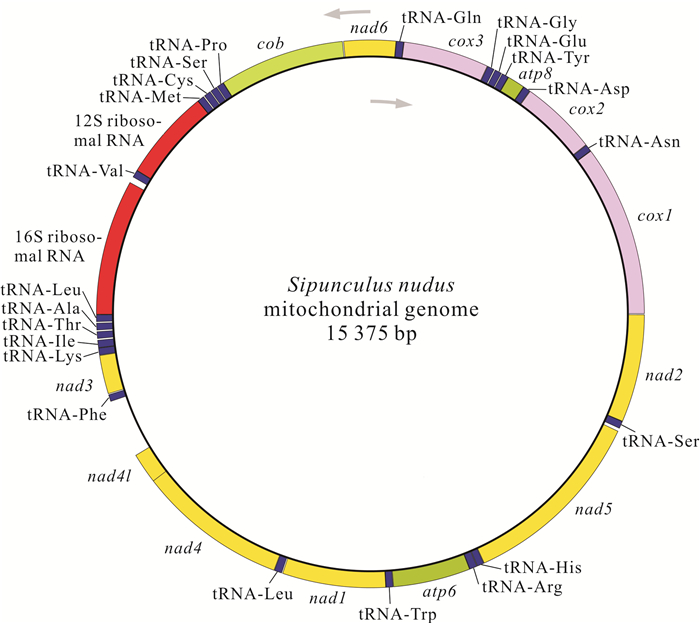
|
| 图 1 裸体方格星虫线粒体基因组的基因图谱 Fig. 1 Gene map of mitochondrial genome in S.nudus |
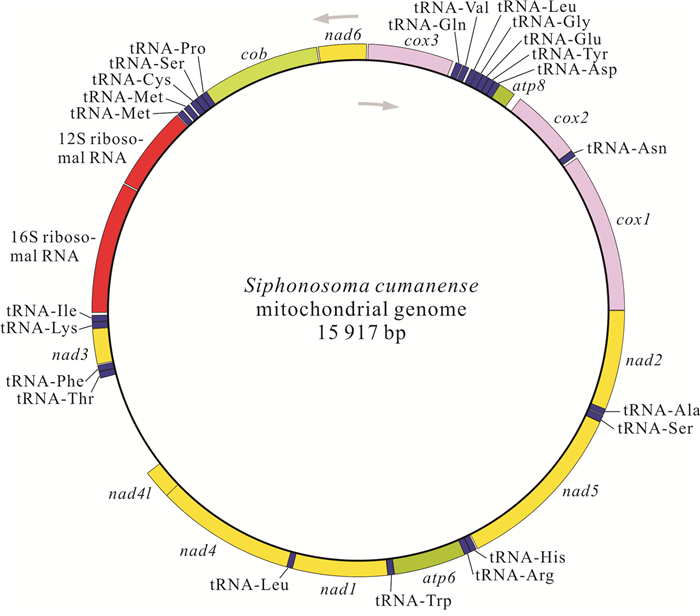
|
| 图 2 邱米管体星虫线粒体基因组的基因图谱 Fig. 2 Gene map of mitochondrial genome in S.cumanense |

|
| 图 3 可口革囊星虫线粒体基因组的基因图谱 Fig. 3 Gene map of mitochondrial genome in P.esculenta |

|
| 图 4 厥目革囊星虫线粒体基因组的基因图谱 Fig. 4 Gene map of mitochondrial genome in P.scolops |

|
| 图 5 类革囊星虫线粒体基因组的基因图谱 Fig. 5 Gene map of mitochondrial genome in P.similis |
2.2 线粒体基因组遗传变异信息
采用DnaSP v6软件,统计分析5种星虫动物线粒体15个主编码基因(13个蛋白质编码基因和2个rRNAs基因)的变异位点,结果显示,cox1基因最保守,遗传变异水平最低,为40.53%;其次是cox2和cob基因,遗传变异水平分别为46.57%和46.83%;nad6、nad4、nad5和nad2等4个线粒体NADH脱氢酶亚基基因遗传变异水平较高,为60%—70%;atp8基因遗传变异水平最高,为75.16%(表 1)。
| 基因 Gene |
总位点数 Total number of sites |
保守位点数 Number of invariable sites |
变异位点数 Number of variable sites |
变异位点比例 Proportion of variable sites (%) |
| cox1 | 1 537 | 914 | 623 | 40.53 |
| cox2 | 687 | 367 | 320 | 46.57 |
| atp8 | 153 | 38 | 115 | 75.16 |
| cox3 | 777 | 388 | 389 | 50.06 |
| nad6 | 461 | 148 | 313 | 67.90 |
| cob | 1 134 | 603 | 531 | 46.83 |
| nad3 | 342 | 140 | 202 | 59.06 |
| nad4l | 279 | 86 | 193 | 69.18 |
| nad4 | 1 339 | 496 | 843 | 62.96 |
| nad1 | 898 | 420 | 478 | 53.23 |
| atp6 | 690 | 287 | 403 | 58.41 |
| nad5 | 1 676 | 630 | 1046 | 62.41 |
| nad2 | 947 | 291 | 656 | 69.27 |
| 12S rRNA | 774 | 354 | 420 | 54.26 |
| 16S rRNA | 1 143 | 547 | 596 | 52.14 |
采用PAML软件,分析蛋白质编码基因非同义替换率(Ka)和同义替换率(Ks),结果显示,5种星虫动物线粒体13个蛋白质编码基因的Ka/Ks比值均低于1 (0.058 2—0.726 6),表明星虫动物中蛋白质编码基因遗传进化中存在一定的纯化(负)选择(表 2)。其中,Ka/Ks比值最高的是nad2基因(0.726 6),nad2基因的Ka和Ks比较接近,表明nad2基因在遗传进化中大多数为无害的中性选择,承受的自然选择压力较弱。其次是atp8基因,Ka/Ks比值为0.577 3,表明atp8基因承受一定的选择压力。cox1、cox3和cob等3个基因的Ka/Ks比值最低(均小于0.1),表明这3个蛋白质编码基因承受强烈的自然选择压力,受基因编码的蛋白质功能束缚,从而保证cox1、cox3和cob基因编码的蛋白质生物功能正常,意味着这3个基因对星虫动物生存进化有重要作用。
| 基因 Gene |
总位点数 Total number of sites |
非同义替换率 Non-synonymous substitution rate (Ka) |
同义替换率 Synonymous substitution rate (Ks) |
Ka/Ks比值 Ka/Ks ratio |
| cox1 | 1 533 | 0.095 8 | 1.645 8 | 0.058 2 |
| cox2 | 684 | 0.159 4 | 1.441 3 | 0.110 6 |
| atp8 | 150 | 0.650 4 | 1.264 4 | 0.577 3 |
| cox3 | 777 | 0.185 0 | 2.077 8 | 0.089 0 |
| nad6 | 456 | 0.410 0 | 1.690 9 | 0.242 5 |
| cob | 1 134 | 0.156 1 | 2.060 0 | 0.075 8 |
| nad3 | 339 | 0.299 6 | 1.751 8 | 0.171 0 |
| nad4l | 279 | 0.409 9 | 1.661 5 | 0.246 7 |
| nad4 | 1 323 | 0.353 4 | 1.626 4 | 0.217 3 |
| nad1 | 873 | 0.284 4 | 1.107 0 | 0.256 9 |
| atp6 | 678 | 0.298 7 | 1.477 7 | 0.202 1 |
| nad5 | 1 659 | 0.412 0 | 1.202 8 | 0.342 6 |
| nad2 | 927 | 0.575 9 | 0.792 5 | 0.726 6 |
2.3 星虫动物门系统进化分析
采用PhyML 3.0软件估算,构建星虫动物系统进化树的最佳进化模型为GTR+G+I。以刺胞动物为外群,星虫动物及其他8个门共35个线粒体基因组的13个蛋白质编码基因的核苷酸序列为原始数据,利用该进化模型构建系统进化树(图 6)。研究结果显示,不同地理种群的裸体方格星虫与不同地理种群的可口革囊星虫首先聚成一支,然后革囊星虫纲的4种和方格星虫纲的2种聚合成平行进化的两个小分支,接着再形成门独立的进化分支。该结果符合星虫动物分类上的亲缘关系,并与传统的形态学鉴定分类结果一致。由图 6可见,星虫动物与环节动物、螠虫动物聚合成一支大进化分支,软体动物则是单独聚合成另一支独立的平行进化分支,表明星虫动物与环节动物存在近缘关系,而不是与软体动物或腕足动物近缘。
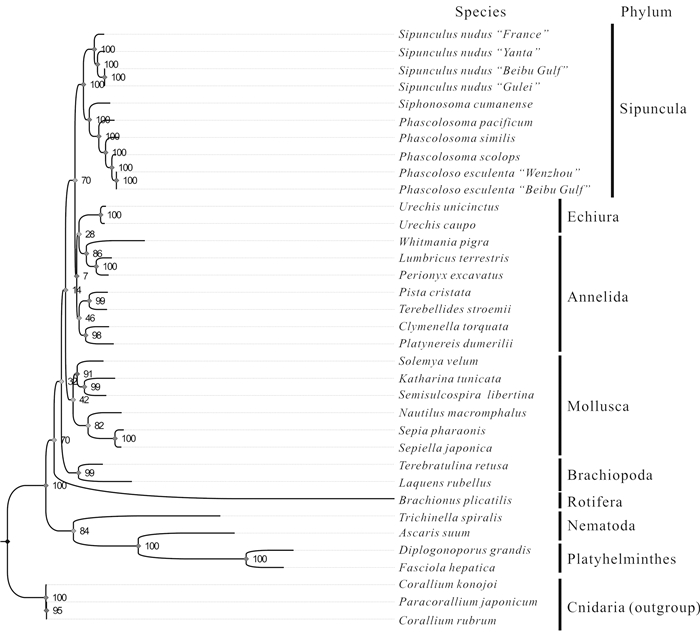
|
| 图 6 基于线粒体基因组构建的星虫动物系统进化树 Fig. 6 Phylogenetic tree based on analysis of mitochondrial genome from Sipuncula |
3 讨论 3.1 星虫动物线粒体基因组的基因序列特征
本研究结果显示,广西北部湾5种常见星虫动物线粒体基因组长度为15—17 kb,符合无脊椎动物线粒体基因组的长度特征,其长度的差异主要是由非编码的控制区长度变异导致的[21]。大多数后生动物线粒体基因组的碱基组成有偏好性,一般都是富含A、T碱基[22],本研究中的星虫动物线粒体基因组的碱基组成也呈A、T偏好,其A、T碱基含量为57.5%—65.6%,并且可口革囊星虫属的A、T碱基含量普遍高于方格星虫属,可能与可口革囊星虫属线粒体基因组中含有比例较高的富含A、T碱基的编码控制区有关,是否与可口革囊星虫属耐受低氧的进化选择有关尚需进一步的研究分析。无脊椎动物线粒体典型基因组成包括13个蛋白质编码基因,22个转运RNA基因和2个核糖体RNA基因,以及一些非编码区域,并且线粒体基因组的基因排列在长期进化过程中一般是保持稳定的,特别是蛋白质编码基因[22],本研究的结果类似。5种星虫动物的线粒体基因均编码在H链上,13个蛋白质编码基因排列高度保守,存在3个高度保守的基因排列区块,尚未发现蛋白质编码基因有易位的进化现象,仅在管体星虫属和口革囊星虫属星虫动物中发现tRNAMet基因重排进化。在环节动物和螠虫动物也有类似的现象报道[13, 23],环节动物和螠虫动物的线粒体基因组基因组成和排列在进化过程中高度保守,进化过程中可能存在“棘齿”现象,导致环节动物和螠虫动物的线粒体所有的基因均在同一条链上编码,并且环节动物线粒体基因组存在6个保守的基因区块,螠虫动物线粒体基因组存在4个保守的基因区块,它们的线粒体基因排列仅有部分tRNAs基因发生易位,可能与其相对原始的进化地位有关。星虫动物较为原始的进化地位可能导致其线粒体基因组的基因重排频率低于其他高等无脊椎动物,尚需进一步的研究证实。
3.2 星虫动物线粒体基因组的遗传变异线粒体基因组的基因由于具备遗传变异信息丰富、序列进化相对保守和母系遗传等优点成为后生动物的起源、进化、系统发育关系和群体遗传多样性研究的理想分子标记[24, 25]。一般而言,进化速率较慢的基因适合种、属、科等高级分类阶元之间系统进化分析。而同一物种内,不同种群间的遗传多样性分析则需要选择进化速率较快、遗传变异水平较高基因作为分子标记。本研究结果表明,在5种星虫动物中cox1、cox2和cob等3个基因进化速率最慢、遗传变异水平最低,适合作为星虫动物种属系统进化研究以及不同种间生物条形码构建的分子标记。nad6、nad4、nad5和nad2等4个基因的遗传变异水平比较高(大于60%),变异位点数量较多,适合作为星虫动物种群遗传多样性研究的分子标记。此外,线粒体基因组基因的进化速率一般受纯化(负)选择、突变和定向(正)选择的控制,非同义替换率和同义替换率的比值(Ka/Ks)是确定线粒体基因组蛋白质编码基因进化选择压力的一个重要指标[25]。本研究结果显示,星虫动物线粒体13个蛋白质编码基因的Ka/Ks均低于1(0.058 2—0.726 6),表明星虫动物线粒体的蛋白质编码在基因遗传进化中存在一定的纯化(负)选择。其中,cox1、cox3和cob等3个基因的Ka/Ks最低(均低于0.1),表明在星虫动物线粒体的遗传进化中,这3种蛋白质编码基因承受强烈的自然选择压力和功能束缚。在环节动物和节肢动物等无脊椎动物中也有研究报道指出[13],线粒体基因组中细胞色素氧化酶亚基基因(cox1、cox2、cox3和cob)的Ka/Ks最低,承受最强的自然选择压力和功能束缚;腺苷三磷酸酶亚基atp8基因的Ka/Ks最高,承受最小的自然选择压力和功能束缚[26],甚至在某些软体动物中线粒体的atp8基因发生进化丢失现象[27]。而在星虫动物线粒体中,nad2基因的Ka/Ks为0.726 6,表明星虫动物线粒体中nad2基因承受最弱的自然选择压力和功能束缚,其次才是atp8基因(Ka/Ks为0.577 3)。该研究结果与环节动物和节肢动物研究结果稍有不同,是否与星虫动物耗能较低的穴居生存方式有关,尚需更多的研究分析来证实。
3.3 星虫动物的系统进化星虫动物的分类地位至今还存在不少争议,因为星虫动物和软体动物具有相似的胚胎发育过程和担轮幼虫发育期,不少研究者认为星虫动物和软体动物具有较近的亲缘关系,再加上缺乏环节动物的典型特征(身体分节和具有几丁质刚毛),星虫动物通常被认为与环节动物亲缘关系较远[8]。然而,本研究中,基于线粒体全基因蛋白质编码基因构建的系统进化树显示,星虫动物与环节动物的分类地位和亲缘关系较近,与软体动物的亲缘关系较远,这与Shen等[13]的研究结果一致。有研究显示,星虫动物和环节动物可能起源于分节的共同祖先。虽然星虫动幼体和成体都不分节,但是其脑神经具有分节的特征,表明星虫动物可能由有分节的祖先进化而来,但在其长期穴居生存的进化过程中分节的特征逐渐退化,仅在神经系统中保持分节的特征[1]。此外,星虫动物的线粒体基因组基因排列与环节动物的更为相似,星虫动物的基因排列可能是从环节动物基因排列重组衍生而来,两者具有相似的保守基因排列区块,仅部分tRNAs基因发生易位;而软体动物线粒体基因组进化重组率较高,基因排列高度异化,与星虫动物和环节动物明显不同[27]。该结果亦说明星虫动物和环节动物的亲缘关系较近,而与软体动物的亲缘关系较远。当然,这一结论的证据尚不够充分,为了深入研究星虫动物的系统进化关系,尚需要挖掘更多其他星虫动物种类的线粒体基因组数据以及核基因数据来联合研究分析。
4 结论本研究分析了广西北部湾5种常见星虫动物线粒体基因组序列信息,研究结果显示,星虫动物线粒体基因组具有典型的无脊椎动物线粒体基因组的特征,其编码基因包括13个蛋白质编码基因,22个转运RNA基因和2个核糖体RNA基因。星虫动物线粒体基因组的基因均编码在H链上,存在3个高度保守的基因排列区块,特别是其13个蛋白质编码基因排列高度保守,尚未发现蛋白质编码基因有易位的进化现象,仅在管体星虫属和口革囊星虫属星虫动物中发现tRNAMet基因重排进化,与环节动物和螠虫动物线粒体基因组特征较为相似,表明星虫动物进化地位较为原始。在星虫动物中,cox1、cox2和cob等3个基因进化速率最慢、遗传变异水平最低,适合作为其种属系统进化研究以及不同种间生物条形码构建的分子标记;nad6、nad4、nad5和nad2等4个基因的遗传变异水平比较高,变异位点数量较多,适合作为其种群遗传多样性研究的分子标记。星虫动物线粒体遗传进化过程存在较强的纯化(负)选择,cox1、cox3和cob等3个基因在星虫动物线粒体遗传进化过程承受强烈的自然选择压力和功能束缚。系统进化树研究表明,星虫动物分为方格星虫纲和革囊星虫纲两个进化分支,星虫动物与环节动物的进化地位、亲缘关系较近,与软体动物的亲缘关系较远。
| [1] |
CUTLER E B. The Sipuncula:Their systematics, biology, and evolution[M]. Ithaca, New York: Cornell University Press, 2018.
|
| [2] |
李凤鲁. 中国沿海革囊星虫属(星虫动物门)的研究[J]. 青岛海洋大学学报, 1989, 19(3): 78-90. |
| [3] |
李凤鲁, 周红, 王玮. 中国沿海星虫动物门名录[J]. 青岛海洋大学学报, 1992, 22(2): 72-88. |
| [4] |
孙仁杰, 范航清, 吴斌, 等. 广西红树林生态系统的常见物种[J]. 广西科学, 2018, 25(4): 372-387. |
| [5] |
徐艳, 王慧芹, 邹杰, 等. 方格星虫生物活性成分的研究进展[J]. 广西科学学院学报, 2015, 31(4): 273-280. |
| [6] |
王帅.北部湾革囊星虫属资源初探及遗传多样性分析[D].南宁: 广西大学, 2018.
|
| [7] |
彭银辉, 黄国强, 刘旭佳, 等. 方格星虫种质资源及人工增养殖研究进展[J]. 广西科学学院学报, 2015, 31(1): 9-15. |
| [8] |
兰国宝, 杨素芳, 谢体三, 等. 星虫动物门系统发生研究进展[J]. 广西科学, 2007, 14(2): 186-192. DOI:10.3969/j.issn.1005-9164.2007.02.029 |
| [9] |
匡卫民, 于黎. 基因组时代线粒体基因组拼装策略及软件应用现状[J]. 遗传, 2019, 41(11): 979-993. |
| [10] |
CHESTERS D. The phylogeny of insects in the data-driven era[J]. Systematic Entomology, 2020, 45(3): 540-551. DOI:10.1111/syen.12414 |
| [11] |
GISSI C, IANNELLI F, PESOLE G. Evolution of the mitochondrial genome of Metazoa as exemplified by comparison of congeneric species[J]. Heredity, 2008, 101(4): 301-320. DOI:10.1038/hdy.2008.62 |
| [12] |
KAYAL E, ROURE B, PHILIPPE H, et al. Cnidarian phylogenetic relationships as revealed by mitogenomics[J]. BMC Evolutionary Biology, 2013, 13(1): 5. DOI:10.1186/1471-2148-13-5 |
| [13] |
SHEN X, MA X Y, REN J F, et al. A close phylogenetic relationship between Sipuncula and Annelida evidenced from the complete mitochondrial genome sequence of Phascolosoma esculenta[J]. BMC Genomics, 2009, 10(1): 136. DOI:10.1186/1471-2164-10-136 |
| [14] |
ZHONG S P, ZHAO Y F, ZHANG Q, et al. The complete mitochondrial genome of the cryptic species in peanut worm Sipunculus nudus (Sipuncula, Sipunculidae) from Beibu Bay[J]. Mitochondrial DNA Part B, 2018, 3(2): 484-485. DOI:10.1080/23802359.2018.1463830 |
| [15] |
周红, 李凤鲁, 王玮. 中国动物志:无脊椎动物第四十六卷星虫动物门螠虫动物门[M]. 北京: 科学出版社, 2007.
|
| [16] |
陈建琴, 张振华, 邢雨辉, 等. 光滑异装蟹(Heteropanope glabra)线粒体基因组测定及其在系统发生研究中的意义[J]. 南京师大学报:自然科学版, 2018, 41(4): 108-114. DOI:10.3969/j.issn.1001-4616.2018.04.017 |
| [17] |
BOLGER A M, LOHSE M, USADEL B. Trimmomatic:A flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [18] |
HAHN C, BACHMANN L, CHEVREUX B. Recon-structing mitochondrial genomes directly from genomic next-generation sequencing reads-A baiting and iterative mapping approach[J]. Nucleic Acids Research, 2013, 41(13): e129. DOI:10.1093/nar/gkt371 |
| [19] |
BERNT M, DONATH A, JVHLING F, et al. MITOS:Improved de novo metazoan mitochondrial genome annotation[J]. Molecular Phylogenetics and Evolution, 2013, 69(2): 313-319. |
| [20] |
GUINDON S, DUFAYARD J F, LEFORT V, et al. New algorithms and methods to estimate maximum-likelihood phylogenies:Assessing the performance of PhyML 3.0[J]. Systematic Biology, 2010, 59(3): 307-321. |
| [21] |
BOORE J L. Animal mitochondrial genomes[J]. Nucleic Acids Research, 1999, 27(8): 1767-1780. DOI:10.1093/nar/27.8.1767 |
| [22] |
ARMSTRONG K F, BALL S L. DNA barcodes for biosecurity:Invasive species identification[J]. Philosophical Transactions of the Royal Society B:Biological Sciences, 2005, 360(1462): 1813-1823. DOI:10.1098/rstb.2005.1713 |
| [23] |
MASTA S E, BOORE J L. The complete mitochondrial genome sequence of the spider Habronattus oregonensis reveals rearranged and extremely truncated tRNAs[J]. Molecular Biology and Evolution, 2004, 21(5): 893-902. |
| [24] |
申欣, 田美, 孟学平, 等. 尾索动物线粒体基因组特征比较及分子系统发育[J]. 海洋科学, 2012, 36(11): 30-37. |
| [25] |
HURST G D, JIGGINS F M. Problems with mitocho-ndrial DNA as a marker in population, phylogeographic and phylogenetic studies:The effects of inherited symbionts[J]. Proceedings of the Royal Society B:Biological Sciences, 2005, 272(1572): 1525-1534. |
| [26] |
申欣, 李晓, 沙忠利, 等. 日本鼓虾线粒体基因组:真虾下目内部的基因重排与系统发育[J]. 中国科学:生命科学, 2012, 42(7): 595-602. |
| [27] |
宋文涛, 高祥刚, 李云峰, 等. 双壳贝类线粒体基因组结构的比较[J]. 遗传, 2009, 31(11): 1127-1134. |



