传统抗生素的无节制使用,导致病原菌抗药性问题日益突出,挖掘新型结构、高活性的抗生素已成为解决病原菌抗药性问题的重要手段。随着从陆地资源挖掘新型化合物的潜力下降,人们逐渐将目光转向海洋生物资源,种群丰富的海洋微生物将是挖掘新型化合物的聚宝盆。海洋生境是一个复杂的生态系统,其高盐、无光、低温(局部高温)、低营养及高压的特殊环境孕育了海洋生物物种的多样性。物种种类优势以及复杂的海洋环境造就了微生物独特的代谢方式,赋予了人类从海洋微生物获取新型化合物的可能。截至2017年,已完成分离鉴定的海洋微生物来源的新型天然产物达1 490种,来源微生物集中在细菌、霉菌、放线菌和蓝藻[1]。二十四元大环内酯化合物Macrolactins (MLNs)主要来源于海生Bacillus sp.和Actinomadura sp.,包含超过32种Macrolactin (MLN)单体化合物[2]。自Fenical首次从深海芽孢杆菌中分离鉴定MLN A-F 6种单体以来,MLNs因具备抗病毒、抗肿瘤和抗病菌等多种生物抑制活性而备受关注。MLN A除了可抑制HIV外,还对哺乳动物疱疹病毒和肿瘤细胞表现出抑制作用;MLN F对鲨烯合成酶具有抑制作用,可作为调控胆固醇水平的潜在药物[3, 4]。在抑菌活性方面,MLN W可有效抑制革兰氏阳性菌和部分革兰氏阴性菌[5]。MLN T不仅能够对常见病原菌耐甲氧西林金黄色葡萄球菌(Methicillin-resistant Staphylococcus aureus)表现出抑制作用,而且对植物致病菌交链孢(Alternaria spp.)和稻瘟霉(Pyricularia oryzae)也表现出较明显的抑制效果[6]。可见MLNs是一类具有广谱生物抑制活性的化合物,在治疗疾病、防治农业病虫害生物等领域具有广阔的应用潜力[7]。但野生菌株代谢产生的MLNs产量较低,限制了该类化合物的进一步研究。鉴于此,本文对MLNs生物合成研究现状进行综述,包括MLNs合成基因簇、代谢菌株遗传改造、发酵工艺优化等,以期为MLNs的批量生产提供研究资料。
1 MLNs合成基因簇MLNs是一类聚酮化合物,是由生物体通过聚酮合酶(Polyketide Synthase, PKS)基因簇逐级缩合产生的天然化合物。根据结构和功能的差异,PKS酶系可分为Ⅰ型、Ⅱ型和Ⅲ型。Ⅰ型PKS是由多个模块按线性排列而成的酶催化体系,每个模块含有一个特有的催化结构域,常用来催化形成具有大环结构的硫酯化合物[8];Ⅱ型PKS是一个多功能酶复合体,由一套独立的4—6个可重复使用的酶组成,每一种酶在重复的反应步骤中可多次催化相同的反应[9];Ⅲ型PKS与前两种类型的明显区别是,可在不需要ACP的情况下直接催化泛酞辅酶A的缩合[10]。
随着生物信息学的快速发展,生物遗传密码被不断破译[11]。Borriss研究团队通过分析Bacillus amyloliquefaciens FZB42基因组,首次确认了Ⅰ型PKS基因簇参与MLNs合成[12]。整个基因簇大小约54 kb,共由11个重复模块组成,每个模块至少包括酰基转移酶、酮基合成酶和酰基载体蛋白3个元件。每个元件在MLNs碳骨架的合成过程中承担着不同的生物功能:酰基转移酶负责底物的激活和运转,酮基合成酶负责延伸聚酮链,酰基载体蛋白负责将完成延伸2个碳原子的聚酮链运转到下一模块。MLNs聚酮链的合成过程与脂肪酸碳骨架的合成过程类似,具体过程如下:延伸单元丙二酰辅酶A被酰基转移酶激活后,在酮基合成酶作用下,激活态的丙二酰辅酶A与酰基转移酶结合的底物分子发生克莱森(Claisen)反应,底物延伸2个碳原子;延伸产物由酰基载体蛋白运转到下一模块的酰基载体蛋白,完成一次二碳单元的延伸。经过11轮重复“激活、延伸、运转”3个步骤,完成二十四元环骨架的合成(图 1)。随后,脱水酶、酮还原酶、烯醇还原酶进一步对二十四元环骨架侧链基团进行修饰,修饰模块多样化决定了MLNs化合物结构的多样化[13]。
2 PKS基因簇多样化
PKS基因簇根据结构和功能可分为Ⅰ型、Ⅱ型和Ⅲ型,各类型的PKS基因簇由于序列、调控元件等因素差异也呈现多样化状态。海洋生态系统中微生物种类丰富,孕育着多样化的基因资源,董晓毅[14]根据Ⅰ型PKS保守序列设计简并引物,扩增海水、沿岸土壤、海洋沉积物等不同生境的微生物基因组,获得多样化的Ⅰ型PKS,为挖掘新型结构的MLNs化合物提供理论基础。在PKS结构分析过程中,研究者发现酮缩合酶(KS)片段可能来源于PKS-NRPS杂合基因簇,Moffitt等[15]研究表明,PKS-NRPS杂合基因簇的KS结构域在活性半胱氨酸N-末端方向的第三个氨基酸残基被谷氨酰胺替代,突变后的KS可以以氨基酸作为起始单元参与生物合成。据文献报道,PKS-NRPS杂合基因簇包括两种类型,一是整体PKS模块和整体NRPS模块相连接(介导杂合化合物的合成),二是由PKS和NRPS的单一模块交替相接形成杂合体[14]。由于PKS-NRPS杂合基因簇的特殊性,对PKS-NRPS杂合基因簇的深入研究将有利于新型MLNs化合物的挖掘[16]。
此外,陆生和海生微生物PKS基因簇也表现出差异,宋婧文[17]对陆生和海生MLNs代谢菌株的基因组进行比较分析,发现海生微生物PKS基因簇启动子区缺失25个碱基,根据MLNs代谢水平与代谢时间的线性关系,研究者推断这段缺失序列可能参与MLNs合成调控。
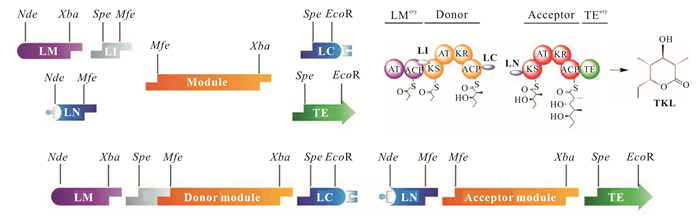
3 人工设计PKS基因簇PKS各个模块按线性排列,每个模块均具有特定的生物学功能,PKS模块重排有利于合成“人工设计”的天然产物。在酮基合成酶作用下,激活态的丙二酰辅酶A与酰基转移酶结合的底物分子发生克莱森反应,底物延伸2个碳原子,通过多次的克莱森反应完成目标化合物的碳骨架延伸,最后在硫酯酶的作用下长链碳骨架闭环合成环形结构。可见,PKS模块的排列顺序决定了化合物骨架的碳原子数目和结构,同理,改变这些模块的排列顺序也可导致新化合物的生成[18]。PKS模块这种一一的对应关系,为编辑PKS基因簇基因(具体操作包括结构域或模块的互换、突变、插入、删除)提供了可能。但是对PKS的模块编辑面临不少问题[18-20],一是对野生菌株而言导入目的DNA片段具有一定挑战性,尤其是部分不可培养或者生长极其缓慢的菌株;二是PKS基因簇具有不易操作性,具体表现为基因簇往往太长(35-200 kb)、没有保守位点,以及模块重复不利于定点编辑。针对PKS基因簇不易操作性难题,Menzella等[18]在模块和结构域两端插入限制性酶切位点,模块间或结构域间通过限制性酶切位点重新组合,高效获得重构PKS基因簇(图 2),接近一半的重构基因簇表现出正常的基因簇生物学功能,并获得结构多样的化合物。
限制性酶切位点策略很好地解决了PKS基因簇重构的难题,但在天然宿主菌种中表达PKS基因簇,产量往往较低, 不利于检测和后续的生物学研究。异源表达是快速、高效表达目的基因的重要手段,常用的异源表达宿主菌包括枯草芽孢杆菌、大肠杆菌、毕赤酵母等,这些异源宿主菌具有易于培养、遗传背景清晰、遗传操作技术成熟等优势,便于构建异源表达工程菌株。除此之外,PKS基因簇异源表达载体构建需要考虑的因素包括选择具备相当容量的载体、密码子优化等问题。目前已成功表达PKS基因簇的异源宿主菌包括酵母菌、大肠杆菌、天蓝色链霉菌、变铅青链霉菌、弗氏链霉菌、糖多孢红霉菌等[14]。
4 从遗传水平调控MLNs生物合成从遗传水平对微生物的MLNs合成途径进行调控,一方面可以挖掘新型结构的MLNs化合物,另一方面也是提高已知MLNs化合物代谢水平的主要手段。高效的遗传转化手段是进行遗传操作的重要基础,但和实验室菌株相比较,从环境中筛选获得的野生微生物由于细胞壁构造特殊且遗传背景不清晰,适用于实验室菌株的遗传操作技术对于其往往效率低下。针对野生菌株重新建立一套高效的遗传操作技术对于微生物遗传育种而言是必要的。Liu等[21]对一株产MLNs的海洋芽孢杆菌遗传转化参数进行优化,通过评价转化体系、培养基、目的DNA去甲基化、渗透压保护剂以及其他化学成分对海洋芽孢杆菌遗传转化效率的影响,首次建立了海洋芽孢杆菌的高效转化体系,转化效率可达7.1×104 cfu/μg质粒,为MLNs代谢菌株遗传改造奠定了重要基础。
Borriss研究组于2007年首次阐明了MLNs合成基因簇[12],但一直以来其分子调控机理并不清晰,合成关键基因也鲜有报道,阻碍了从遗传水平定向调控MLNs生物合成的研究进程。MLNs合成模块中,反式酰基转移酶负责延伸单元丙二酰辅酶A的运转,理论上提高丙二酰辅酶A的运转速率有利于提高MLNs合成水平。刘扬等[22]过度表达反式酰基转移酶BmmA,结果显示MLNs A的产量提高了约0.6倍,说明丙二酰辅酶A基因在MLNs生物合成过程中参与调控MLNs代谢。改变PKS基因簇主要元件的表达水平可调控MLNs代谢水平,改变修饰元件的表达水平也能影响MLNs的代谢。BmmGT1基因负责海洋芽孢杆菌B-9987的MLNs代谢产物的糖基化修饰,BmmGT1基因过度表达后,非糖基化MLNs水平迅速降低,并获得5种糖基化的MLNs衍生物[23]。
MLNs生物合成过程的调控机制和主要关键基因尚不清晰,不利于应用现代分子生物学技术构建优良工程菌株。传统微生物育种手段,如诱变[24]、适应性驯化[25, 26]、Genome shuffling[27]等,其突出优点是不需要对目标菌株遗传背景有清晰的了解。Yi等[28]运用ARTP诱变育种技术对海洋芽孢杆菌进行诱变,基于菌株的生长系数筛选获得优良突变菌,其MLNs代谢水平提高52.8%。研究者进一步对突变菌株转录组进行研究,156个差异表达基因主要涉及5个生物过程,包括组氨酸代谢、核黄素代谢、非核糖体肽结构、脂肪酸降解和脂肪酸代谢。这些基因组学研究数据为探索MLNs代谢调控机制提供了理论支撑,其中特别值得关注的生物途径是脂肪酸代谢,因为细胞膜脂肪酸在提高细胞抗生素抗性方面发挥重要作用[29, 30]。MLNs是一类广谱抗生素,当其浓度累积到一定程度后将不可避免地影响代谢菌株的生理状态,而提高细胞膜脂肪酸浓度可能有利于高浓度的MLNs积累。
5 发酵优化提高MLNs代谢水平野生菌株的MLNs代谢水平低,成为制约其进行药物开发研究的主要瓶颈。微生物培养法获得大量目标化合物需要满足两个基本条件:获得代谢性能优良的菌株和扩大培养。扩大培养的效果常受培养基组分、溶氧、pH值、温度等因素的影响,因此基于实验结果和经济因素考虑,在开展扩大培养前需要对培养条件进行优化。响应面法是发酵优化的常用技术手段,在提高MLNs产量方面也取得比较理想的结果[31-34],目前已报道的MLNs发酵优化最高产量为851 mg/L[35]。
微生物只有进入生长稳定期才开始大量合成聚酮化合物,然而进入稳定期后,细胞对初级代谢产物的需求显著下降[36],此时作为初级代谢产物的中间代谢产物丙二酰辅酶A是否与MLNs的合成存在直接联系,尚未明确。王明璐[34]尝试在培养基中添加乙酸钠、丙酸钾营养成分,通过生物转化以维持胞内丙二酰辅酶A浓度,但MLNs产量并未受到影响,而添加低浓度的豆油则能明显促进MLNs代谢。Wang等[36]结合基因组学研究阐明豆油和聚酮化合物合成间的直接联系,发现菌株在对数期细胞生长的同时还积累三酰甘油(豆油主成分),进入稳定期后胞内三酰甘油降解机制被激活,为稳定期细胞的聚酮化合物合成提供前体化合物以及还原力。
6 展望MLNs具有广谱生物抑制活性,在医药和农业具有潜在的应用前景。目前针对改善菌株MLNs代谢水平的相关问题已开展了部分工作,但受限于MLNs代谢调控机制、关键基因不清晰,菌株MLNs代谢水平低的局面至今仍然没有得到改善,一定程度上限制了MLNs的生物活性研究以及市场推广。Ⅰ型PKS基因簇由多个模块组成,这些基因共同受一个操纵子调控表达。因此,对操纵子的关键元件(如启动子)进行改造,通过改造一个位点实现对整个PKS基因簇的表达进行定向调控,这方面的研究有望进一步改善MLNs代谢水平,也能避免PKS基因簇基因元件因重复性而不利于遗传操作、可能涉及的多位点操作等难题。
| [1] |
CARROLL A R, COPP B R, DAVIS R A, et al. Marine natural products[J]. Natural Product Reports, 2020, 37(2): 175-223. |
| [2] |
LI L, SAPKOTA M, GAO M, et al. Macrolactin F inhibits RANKL-mediated osteoclastogenesis by suppressing Akt, MAPK and NFATc1 pathways and promotes osteoblastogenesis through a BMP-2/smad/Akt/Runx2 signaling pathway[J]. European Journal of Pharmacology, 2017, 815: 202-209. DOI:10.1016/j.ejphar.2017.09.015 |
| [3] |
GUSTAFSON K R, ROMAN M, FENICAL W. The macrolactins, a novel class of antiviral and cytotoxic macrolides from a deep-sea marine bacterium[J]. Journal of the American Chemical Society, 1989, 111(19): 7519-7524. DOI:10.1021/ja00201a036 |
| [4] |
JIN J, HWANG K, JOO J D, et al. Combination therapy of 7-O-succinyl macrolactin A tromethamine salt and temozolomide against experimental glioblastoma[J]. Oncotarget, 2018, 9(2): 2140-2147. DOI:10.18632/oncotarget.23295 |
| [5] |
MONDOL M A, KIM J H, LEE H S, et al. Macrolactin W, a new antibacterial macrolide from a marine Bacillus sp.[J]. Bioorganic & Medicinal Chemistry Letters, 2011, 21(12): 3832-3835. |
| [6] |
CHUNMEI X, LI T, MINJUAN X, et al. A new 24-membered lactone and a new polyene delta-lactone from the marine bacterium Bacillus marinus[J]. The Journal of Antibiotics, 2008, 61(11): 668-674. DOI:10.1038/ja.2008.94 |
| [7] |
FAN B, WANG C, SONG X, et al. Bacillus velezensis FZB42 in 2018:The gram-positive model strain for plant growth promotion and biocontrol[J]. Frontiers in Microbiology, 2018, 9: 2491. DOI:10.3389/fmicb.2018.02491 |
| [8] |
DONADIO S, STAVER M J, MCALPINE J B, et al. Modular organization of genes required for complex polyketide biosynthesis[J]. Science, 1991, 252(5006): 675-679. DOI:10.1126/science.2024119 |
| [9] |
WESTLEY J W, BLOUNT J F, EVANS R H, et al. Biosynthesis of lasalocid.Ⅱ.X-ray analysis of a naturally occurring isomer of lasalocid A[J]. The Journal of Antibiotics, 1974, 27(8): 597-604. DOI:10.7164/antibiotics.27.597 |
| [10] |
FUNA N, OHNISHI Y, FUJII I, et al. A new pathway for polyketide synthesis in microorganisms[J]. Nature, 1999, 400(6747): 897-899. DOI:10.1038/23748 |
| [11] |
WANG G L, SHI T, CHEN T, et al. Integrated whole-genome and transcriptome sequence analysis reveals the genetic characteristics of a riboflavin-overproducing Bacillus subtilis[J]. Metabolic Engineering, 2018, 48: 138-149. DOI:10.1016/j.ymben.2018.05.022 |
| [12] |
CHEN X H, KOUMOUTSI A, SCHOLZ R, et al. Co-mparative analysis of the complete genome sequence of the plant growth-promoting bacterium Bacillus amyloliquefaciens FZB42[J]. Nature Biotechnology, 2007, 25(9): 1007-1014. DOI:10.1038/nbt1325 |
| [13] |
张偲. 中国海洋微生物多样性[M]. 北京: 科学出版社, 2012: 474-475.
|
| [14] |
董晓毅.海洋微生物Ⅰ型PKS基因资源的筛选与鉴定[D].上海: 第二军医大学, 2007.
|
| [15] |
MOFFITT M C, NEILAN B A. Evolutionary affiliations within the superfamily of ketosynthases reflect complex pathway associations[J]. Journal of Molecular Evolution, 2003, 56(4): 446-457. DOI:10.1007/s00239-002-2415-0 |
| [16] |
赵彬.极地海洋放线菌天然产物合成潜力及其基因簇挖掘研究[D].上海: 华东理工大学, 2018.
|
| [17] |
宋婧文.Macrolactin生物合成基因簇序列与功能分析[D].上海: 第二军医大学, 2013.
|
| [18] |
MENZELLA H G, REID R, CARNEY J R, et al. Com-binatorial polyketide biosynthesis by de novo design and rearrangement of modular polyketide synthase genes[J]. Nature Biotechnology, 2005, 23(9): 1171-1176. DOI:10.1038/nbt1128 |
| [19] |
KAO C M, KATZ L, KHOSLA C. Engineered biosynthesis of a complete macrolactone in a heterologous host[J]. Science, 1994, 265(5171): 509-512. DOI:10.1126/science.8036492 |
| [20] |
MCDANIEL R, THAMCHAIPENET A, GUSTAFS-SON C, et al. Multiple genetic modifications of the erythromycin polyketide synthase to produce a library of novel "unnatural" natural products[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(5): 1846-1851. DOI:10.1073/pnas.96.5.1846 |
| [21] |
LIU Y, ZHENG H, ZHAN G, et al. Establishment of an efficient transformation protocol and its application in marine-derived Bacillus strain[J]. Science China Life Sciences, 2014, 57(6): 627-635. |
| [22] |
刘扬, 郑华, 姚婷婷, 等. 海洋芽孢杆菌B-9987中macrolactin生物合成基因簇分析及反式酰基转移酶高表达[J]. 中国海洋药物, 2014, 33(3): 69-75. |
| [23] |
QIN W, LIU Y, REN P, et al. Uncovering a glycosyltransferase provides insights into the glycosylation step during macrolactin and bacillaene biosynthesis[J]. Chembiochem:A European Journal of Chemical Biology, 2014, 15(18): 2747-2753. DOI:10.1002/cbic.201402384 |
| [24] |
SONG C W, CHELLADURAI R, PARK J M, et al. Engineering a newly isolated Bacillus licheniformis strain for the production of (2R, 3R)-butanediol[J]. Journal of Industrial Microbiology & Biotechnology, 2020, 47(1): 97-108. |
| [25] |
YUAN K, SONG P, LI S, et al. Combining metabolic flux analysis and adaptive evolution to enhance lipase production in Bacillus subtilis[J]. Journal of Industrial Microbiology & Biotechnology, 2019, 46(8): 1091-1101. |
| [26] |
NSHIMIYIMANA P, LIU L, DU G. Engineering of L-amino acid deaminases for the production of alpha-keto acids from L-amino acids[J]. Bioengineered, 2019, 10(1): 43-51. |
| [27] |
CHEN L, CHONG X Y, ZHANG Y Y, et al. Genome shuffling of Bacillus velezensis for enhanced surfactin production and variation analysis[J]. Current Microbiology, 2020, 77(1): 71-78. |
| [28] |
YI X X, GAN Y M, JIANG L, et al. Rapid improvement in the macrolactins production of Bacillus sp.combining atmospheric room temperature plasma with the specific growth rate index[J]. Journal of Bioscience and Bioengineering, 2020, 130(1): 48-53. |
| [29] |
EIJKELKAMP B A, BEGG S L, PEDERICK V G, et al. Arachidonic acid stress impacts pneumococcal fatty acid homeostasis[J]. Frontiers in Microbiology, 2018, 9: 813. |
| [30] |
GONCALVES F D, DE CARVALHO C C. Phenotypic modifications in staphylococcus aureus cells exposed to high concentrations of vancomycin and teicoplanin[J]. Frontiers in Microbiology, 2016, 7: 13. |
| [31] |
HE S, WANG H, WU B, et al. Response surface methodology optimization of fermentation conditions for rapid and efficient accumulation of macrolactin A by marine Bacillus amyloliquefaciens ESB-2[J]. Molecules, 2012, 18(1): 408-417. |
| [32] |
杨桥, 韩文菊, 张文俊, 等. 产Macrolactin A抗生素海洋解淀粉芽孢杆菌的鉴定及发酵条件优化[J]. 药物生物技术, 2009, 16(4): 311-315. |
| [33] |
杜瑜欣.高产24元大环内酯的海洋芽孢杆菌突变株Bacillus sp.A72发酵工艺优化[D].南宁: 广西中医药大学, 2019.
|
| [34] |
王明璐.一种新型大环内酯类抗真菌抗生素的发酵工艺及稳定性研究[D].杭州: 浙江大学, 2013.
|
| [35] |
CHEN H, WU M B, CHEN Z J, et al. Enhancing production of a 24-membered ring macrolide compound by a marine bacterium using response surface methodology[J]. Journal of Zhejiang University Science B, 2013, 14(4): 346-354. |
| [36] |
WANG W S, LI S S, LI Z L, et al. Harnessing the intracellular triacylglycerols for titer improvement of polyketides in Streptomyces[J]. Nature Biotechnology, 2020, 38(1): 76-83. |