2. 广西科学院, 国家非粮生物质能源工程技术研究中心, 非粮生物质酶解国家重点实验室, 广西生物质工程技术研究中心, 广西生物炼制重点实验室, 广西南宁 530007
2. National Engineering Research Center for Non-food Biorefinery, State Key Laboratory of Non-food Biomass and Enzyme Technology, Guangxi Biomass Engineering Technology Research Center, Guangxi Key Laboratory of Biorefinery, Guangxi Academy of Sciences, Nanning, Guangxi, 530007, China
乙偶姻(Acetoin, AC),化学品名为3-羟基-2-丁酮,又名3-羟基丁酮或甲基乙酰甲醇,具有强烈的奶油、脂肪、黄油样香气,是多种天然食品和发酵食品的重要风味物质。我国标准GB 2760—2014规定乙偶姻为允许使用的食用香料,FEMA安全号是2008[1-3]。而且,乙偶姻作为一种重要的四碳平台化合物,已经被美国能源部指定为优先开发的平台化合物之一,在食品、化工、医药、烟草、化妆品、调酒等行业具有广泛的用途[4-5]。乙偶姻具有(R)-乙偶姻和(S)-乙偶姻两种旋光异构体,单一构型的(R)-乙偶姻在合成高附加值手性药物中间体、化学中间体、液晶材料等方面具有重要应用,因此光学纯(R)-乙偶姻的价值要远远高于混合型乙偶姻[3-4]。
目前生产乙偶姻的方法主要为化学合成法[6]和微生物合成法[7-14]。化学合成乙偶姻产品中容易掺杂致癌致病化合物,限制了乙偶姻作为食品香精香料的使用。微生物合成的乙偶姻属于天然香料,其市场价格要远远高于化学合成的产品。自然界能够合成乙偶姻的微生物有很多,如粘质沙雷氏菌(Serratia marcescens)、产酸克雷伯氏菌(Klebsiella oxytaca)、肺炎克雷伯氏菌(Klebsiella pneumoniae)、阴沟肠杆菌(Enterobacter cloacae)、多粘类芽孢杆菌(Paenibacillus polymyxa)和部分芽孢杆菌属(Bacillus)等[15-19],但天然菌株往往合成乙偶姻的能力较弱、(R)-乙偶姻光学纯度低或菌株具有致病性,不利于(R)-乙偶姻的微生物发酵生产,导致光学纯(R)-乙偶姻的市场价格居高不下,极大地限制了其发展和应用。
近年来,选育和改造天然微生物合成乙偶姻取得较多的研究成果。Xu等[20]通过诱变筛选得到高产菌株B. subtilis TH-49,以葡萄糖为碳源得到乙偶姻产量46.9 g/L;Sun等[21]利用紫外及LiCl进行复合诱变后筛选得到S.marcescens H32,再以蔗糖为碳源进行补料分批发酵,乙偶姻产量能达到60.5 g/L。诱变育种随机性较高,通过代谢工程手段对细胞合成(R)-乙偶姻进行定向改造逐渐成为研究的主流。Bai等[22]和Lv等[23]分别对菌株S.marcescens进行遗传操作,通过敲除meso-2, 3-丁二醇脱氢酶基因slaC,或过量表达转录调控因子基因slaR,使其具有利用蔗糖生产(R)-乙偶姻的能力,产量分别为21.8 g/L和39.9 g/L;Wang等[24]敲除K.pneumoniae CGMCC 1.6366的2, 3-丁二醇脱氢酶编码基因budC,可阻断菌株将乙偶姻转化为2, 3-丁二醇;进一步敲除乙偶姻脱氢酶编码基因acoABCD,工程菌株利用葡萄糖在补料发酵条件下生产(R)-乙偶姻可达62.3 g/L,光学纯度为98.0%。大肠杆菌Escherichia coli属于非致病菌株,并且其本身不合成乙偶姻,因此有利于定向改造成生产光学纯(R)-乙偶姻的工程菌株。Xiao等[25]在E.coli BL21(DE3)中过量表达来自B. subtilis的2, 3-丁二醇脱氢酶基因ydjL (bdhA)和来自Lactobacillus brevis的NADH氧化酶基因noxE,以(2R, 3R)-2, 3-丁二醇为原料进行全细胞催化得到41.8 g/L(R)-乙偶姻,光学纯度为96.0%;Xu等[3]克隆S.marcescens的budR、budA、budB基因以及L. brevis的NADH氧化酶基因,将其导入E. coli DH5α中进行表达,葡萄糖补料分批发酵的(R)-乙偶姻产量为60.3 g/L,光学纯度为97.3%。
在微生物细胞中,α-乙酰乳酸合成酶(α-acetolactate Synthase,AlsS)和α-乙酰乳酸脱羧酶(α-acetolactate Decarboxylase,AlsD)是发酵丙酮酸生成(R)-乙偶姻的两个关键酶。另外,糖酵解和三羧酸循环会产生大量的还原型辅酶NADH,而由丙酮酸到(R)-乙偶姻的合成并没有消耗NADH,因此如果单一增强(R)-乙偶姻的合成会造成NADH累积,细胞将启动各种杂醇杂酸合成途径消耗过量的NADH来实现氧化型辅酶NAD+再生,必然会降低(R)-乙偶姻的合成效率和光学纯度。本研究通过对细胞(R)-乙偶姻代谢途径进行系统分析,利用辅酶工程精确调控异源NADH氧化酶进行合理表达来消耗胞内过量的NADH,使胞内NADH/NAD+氧化还原平衡,从而提高(R)-乙偶姻光学纯度。以大肠杆菌E.coli MG1655为宿主细胞,将来源于E. cloacae的α-乙酰乳酸合成酶基因budB、α-乙酰乳酸脱羧酶基因budA和来源于L. brevis的NADH氧化酶基因noxE的核苷酸序列进行密码子优化,利用人工合成的方法获得包含3个基因的基因簇,构建表达质粒pTrc99A-budB-budA-noxE并导入大肠杆菌获得工程菌株,达到提高目标产物转化率和维持胞内NADH/NAD+氧化还原平衡的双重目的,进一步优化工程菌的培养基成分和发酵条件。
1 材料与方法 1.1 材料 1.1.1 菌株及载体宿主菌株E.coli MG1655和表达载体pTrc99A由作者所在实验室保藏。
1.1.2 试剂T4 DNA连接酶和各种限制性内切酶(NEB);质粒提取试剂盒、凝胶回收试剂盒和DNA纯化试剂盒(Promega);蛋白胨和酵母粉(OXOID,分析纯);葡萄糖、氯化钠(分析纯)、乙腈(国药集团化学试剂有限公司,HPLC级别);乙酸乙酯(成都市科龙化工试剂厂,分析纯);乙偶姻(>98.0%)和2, 3-丁二醇(>97.0%)(TCA);氨苄(Amp, 北京索莱宝科技有限公司);甜菜碱(阿拉丁公司);维生素B1(生工生物工程(上海)股份有限公司)。
1.1.3 仪器恒温水浴摇床ZWY-110X50(上海智城分析仪器制造有限公司), 生物传感分析仪SBA-40D(山东省科学院生物研究所), 3 L发酵罐(上海保兴生物设备工程有限公司), 旋转蒸发器RE-5000(上海贤德实验仪器有限公司), 气相色谱仪7890A(安捷伦科技有限公司), 紫外可见光分光光度计DU800(美国贝克曼库尔特公司), MIKRO 200离心机(德国Hettich科学仪器公司)。
1.1.4 培养基种子培养基(LB培养基):酵母粉5 g/L,蛋白胨10 g/L,氯化钠10 g/L,pH值调至7.0,转化子培养时添加氨苄0.1 g/L,固体培养基另加20 g/L琼脂粉。
初始发酵培养基:葡萄糖80 g/L,酵母粉7 g/L,蛋白胨10 g/L,氯化钠0.5 g/L,氨苄0.1 g/L,甜菜碱4 mmol/L,维生素B1 0.1 g/L,pH值调至7.0。
1.2 方法 1.2.1 产(R)-乙偶姻工程菌株的构建将来源于E. cloacae的α-乙酰乳酸合成酶基因budB、α-乙酰乳酸脱羧酶基因budA和来源于L. brevis的NADH氧化酶基因noxE的核苷酸序列进行密码子优化,在每个基因前面添加含核糖体结合位点和间隔序列TAAGGAGGATATACA连接成基因簇budB-budA-noxE,并由苏州金唯智生物科技有限公司进行人工合成,利用酶切位点SacⅠ和BamHⅠ将基因簇budB-budA-noxE插入质粒pTrc99A的启动子后面,获得多顺反子重组质粒pTrc99A-budB-budA-noxE,再将重组质粒pTrc99A-budB-budA-noxE转化宿主菌E.coli MG1655;以含质粒pTrc99A的E.coli MG1655作为空白对照。分别挑取两种转化子提取质粒进行质粒酶切验证、PCR验证和测序验证。
1.2.2 产(R)-乙偶姻工程菌株的发酵将验证正确的转化子接到含Amp+的LB液体培养基中,37℃培养12 h,然后分别按照10%的接种量转接于含有50 mL初始发酵培养基的三角瓶(250 mL)中,实验组与对照组各3瓶,37℃,250 r/min,培养36 h。取发酵液,经离心机12 000 r/min离心2 min后,取上清液,经过稀释一定倍数后测残糖浓度以及(R)-乙偶姻含量。其中,(R)-乙偶姻得率为产物(R)-乙偶姻质量与消耗葡萄糖质量之比,生产强度为产物(R)-乙偶姻浓度与发酵周期之比。
1.2.3 产(R)-乙偶姻工程菌株发酵条件的优化利用摇瓶发酵分别对葡萄糖浓度、酵母粉浓度、蛋白胨浓度、最佳接种量以及最适初始pH值进行单因素实验,优化工程菌株发酵培养基和培养条件。
1.2.4 产(R)-乙偶姻工程菌株发酵罐补料发酵优化初始发酵培养基后,将菌种接种于3 L发酵罐中,装液量1.5 L,控制通气量1.0 vvm,搅拌转速500 r/min,温度37℃。当残糖浓度为30-40 g/L时补加葡萄糖至150-170 g/L,待发酵至残糖浓度降到5-8 g/L时停止发酵。
1.2.5 残糖浓度的检测取发酵液12 000 r/min离心处理2 min,取上清液稀释100倍后,用生物传感分析仪SBA-40D检测残糖浓度。
1.2.6 乙偶姻以及2, 3-丁二醇浓度的检测使用气相色谱仪测定乙偶姻和2, 3-丁二醇的浓度。色谱柱为Phenomenex ZB-WAXplus毛细管色谱柱(30 m×0.32 mm×0.25 μm),进样口温度和检测器温度均为250℃,使用内标法并以乙腈作内标来进行检测。
1.2.7 (R)-乙偶姻光学纯度的测定采用气相色谱仪测定(R)-乙偶姻的光学纯。色谱柱为Agilent Cyclosil-B毛细管手性柱(30 m×0.32 mm×0.25 μm);FID氢火焰检测器,载气为氮气,流速1.6 mL/min。起始柱温度100℃,保留1 min,然后以10℃/min的速度升温至120℃,以6℃/min的速度升温至130℃,以20℃/min的速度升温至230℃,进样口温度和检测器温度均为240℃,以乙酸乙酯为萃取剂。
2 结果与分析 2.1 产(R)-乙偶姻工程菌株GXASR的构建与发酵将合成的基因簇budB-budA-noxE亚克隆至表达载体pTrc99A,构建重组质粒pTrc99A-budB-budA-noxE,将重组质粒转化E. coli MG1655,转化子提取质粒进行质粒酶切、PCR条带电泳及DNA测序结果均验证正确,证明工程菌株构建成功,命名为GXASR。同时本研究在各个基因前引入RBS序列来增强与核糖体间的亲和力,提高蛋白翻译效率[26],以此来提高α-乙酰乳酸脱羧酶、α-乙酰乳酸合成酶以及氧化还原酶的表达,调控代谢流,以增加(R)-乙偶姻产量。
将对照菌株与工程菌株GXASR进行摇瓶培养,36 h后发酵结束,对发酵液进行检测,对照菌株发酵液中没有检测到(R)-乙偶姻产生,工程菌株GXASR能够有效地合成(R)-乙偶姻(表 1)。
| 菌种 Strains |
残还原糖浓度 Concentration of residual sugar (g/L) |
(R)-乙偶姻浓度 Concentration of (R)-Acetoin (g/L) |
2, 3-丁二醇浓度 Concentration of 2, 3-BD (g/L) |
得率 Yield (g/g) |
生产强度 Productivity(g/(L·h)) |
| MG1655/pTrc99A | 74.10±0.77 | ND | ND | - | - |
| GXASR | 0.00 | 26.5±0.63 | 2.49±0.55 | 0.33 | 0.73 |
|
注:ND表示未检测到 Note:ND means not detected | |||||
2.2 产(R)-乙偶姻工程菌株GXASR发酵条件优化 2.2.1 葡萄糖浓度对GXASR工程菌株合成(R)-乙偶姻的影响
工程菌株GXASR在葡萄糖浓度80-120 g/L范围内,随着碳源浓度的升高,(R)-乙偶姻产量也随着提高。但是,随着碳源浓度的升高,培养基变得更加黏稠使得传质效率变低,且较高的碳源浓度,使得菌体所受的渗透压变得更大,不利于菌体生长以及产物的合成。当葡萄糖浓度>100 g/L时,发酵周期明显延长,并且残糖浓度较高,乙偶姻产量提高幅度较小而副产物2, 3-丁二醇浓度提高幅度较大(表 2),从发酵周期、葡萄糖利用程度、乙偶姻产量、副产物生成量以及生产强度综合考虑,将发酵培养基碳源浓度定为100 g/L。
| 葡萄糖浓度 Concentration of glucose (g/L) | 发酵周期 Fermentation time (h) |
残还原糖浓度 Concentration of residual sugar (g/L) |
(R)-乙偶姻浓度 Concentration of (R)-Acetoin (g/L) |
2, 3-丁二醇浓度 Concentration of 2, 3-BD (g/L) |
得率 Yield (g/g) |
生产强度 Productivity (g/(L·h)) |
| 80 | 36 | 0.35±0.03 | 26.10±0.86 | 2.17±0.34 | 0.33 | 0.73 |
| 90 | 38 | 0.43±0.08 | 28.44±0.77 | 2.31±0.25 | 0.32 | 0.75 |
| 100 | 40 | 6.60±0.73 | 30.49±0.82 | 3.54±0.46 | 0.33 | 0.76 |
| 110 | 48 | 16.47±0.84 | 31.16±0.61 | 9.10±0.38 | 0.33 | 0.65 |
| 120 | 54 | 29.40±0.61 | 33.24±0.64 | 12.24±0.77 | 0.37 | 0.62 |
2.2.2 蛋白胨浓度对工程菌株GXASR合成(R)-乙偶姻的影响
工程菌株GXASR在蛋白胨浓度为15 g/L时,(R)-乙偶姻浓度、得率、生产强度都达到了最大;当蛋白胨浓度超过15 g/L时,(R)-乙偶姻合成效率反而变低,得率以及生产效率也跟着下降(表 3),这可能是由于蛋白胨浓度不断变高,使得氮源浓度过高导致营养富余,菌体将多余的营养物质用于合成自身所需的其他物质,从而致使(R)-乙偶姻合成效率低下,故将发酵培养基蛋白胨浓度定为15 g/L。
| 蛋白胨浓度 Concentration of peptone (g/L) | 发酵周期 Fermentation time (h) |
残还原糖浓度 Concentration of residual sugar (g/L) |
(R)-乙偶姻浓度 Concentration of (R)-Acetoin (g/L) |
2, 3-丁二醇浓度 Concentration of 2, 3-BD (g/L) |
得率 Yield (g/g) |
生产强度 Productivity (g/(L·h)) |
| 5.0 | 36 | 7.73±0.25 | 28.01±0.26 | 0.98±0.13 | 0.30 | 0.78 |
| 7.5 | 36 | 6.73±0.43 | 29.73±0.17 | 1.21±0.06 | 0.32 | 0.83 |
| 10.0 | 36 | 1.73±0.36 | 32.41±0.82 | 1.48±0.22 | 0.33 | 0.90 |
| 12.5 | 36 | 3.67±0.54 | 33.31±0.61 | 1.62±0.04 | 0.35 | 0.93 |
| 15.0 | 36 | 6.23±0.61 | 34.05±0.05 | 1.56±0.14 | 0.36 | 0.95 |
| 17.5 | 36 | 3.63±0.71 | 33.48±0.88 | 1.62±0.25 | 0.35 | 0.93 |
| 20.0 | 36 | 8.97±0.83 | 31.27±0.34 | 1.39±0.30 | 0.34 | 0.87 |
2.2.3 酵母粉浓度对工程菌株GXASR合成(R)-乙偶姻的影响
酵母粉浓度为15 g/L时,(R)-乙偶姻浓度、得率和生产强度最高;随着酵母粉浓度继续增大,3项指标都略有下降(表 4),这是由于菌株将富余的营养物质以及能量用于副产物的合成,从而降低(R)-乙偶姻合成效率,因此将发酵培养基酵母粉浓度定为15 g/L。
| 酵母粉浓度 Concentration of yeast extract (g/L) | 发酵周期 Fermentation time (h) |
残还原糖浓度 Concentration of residual sugar (g/L) |
(R)-乙偶姻浓度 Concentration of (R)-Acetoin (g/L) |
2, 3-丁二醇浓度 Concentration of 2, 3-BD (g/L) |
得率 Yield (g/g) |
生产强度 Productivity (g/(L·h)) |
| 5.0 | 36 | 7.45±0.32 | 30.74±0.58 | 1.91±0.03 | 0.31 | 0.85 |
| 7.5 | 36 | 1.70±0.53 | 32.68±0.83 | 2.71±0.21 | 0.33 | 0.90 |
| 10.0 | 36 | 2.83±0.66 | 33.00±0.80 | 2.43±0.33 | 0.34 | 0.92 |
| 12.5 | 36 | 2.55±0.82 | 34.36±0.24 | 2.86±0.54 | 0.37 | 0.95 |
| 15.0 | 36 | 0.20±0.10 | 35.51±0.33 | 2.85±0.18 | 0.38 | 0.99 |
| 17.5 | 36 | 3.77±0.75 | 35.13±0.22 | 2.52±0.39 | 0.37 | 0.98 |
| 20.0 | 36 | 0.27±0.16 | 35.01±0.67 | 3.11±0.26 | 0.35 | 0.97 |
2.2.4 发酵初始pH值以及接种量对工程菌株GXASR合成(R)-乙偶姻的影响
pH值为6.0-7.5时,工程菌株耗糖效率均比较好,残糖浓度小于10 g/L,当pH值为6.5时,菌株的(R)-乙偶姻产量最高(图 1)。首先菌株在pH值为7.0的种子培养基中培养后,转接到pH值为6.5的发酵培养基中,由于pH值相近,菌种很容易适应pH值的变化;其次菌种在发酵过程中会产生有机酸,培养基会呈现弱酸性的趋势,选择pH值为6.5的发酵条件,在发酵过程中不需要补加过多的碱以维持培养基呈中性,减少了碱液对发酵过程的影响。接种量为10%时菌株耗糖效率最快、(R)-乙偶姻产量最高。接种量过低,菌种增殖速度慢,(R)-乙偶姻产量不理想;而接种量过高,菌种增殖速度快,细胞生长额外消耗了较多的碳源,导致用于合成(R)-乙偶姻的葡萄糖减少,故(R)-乙偶姻不升反降(图 2)。因此,确定菌种最适发酵培养基pH值为6.5,最适接种量为10%。在优化的条件下摇瓶发酵(R)-乙偶姻产量为36.82 g/L,光学纯为99.1%。
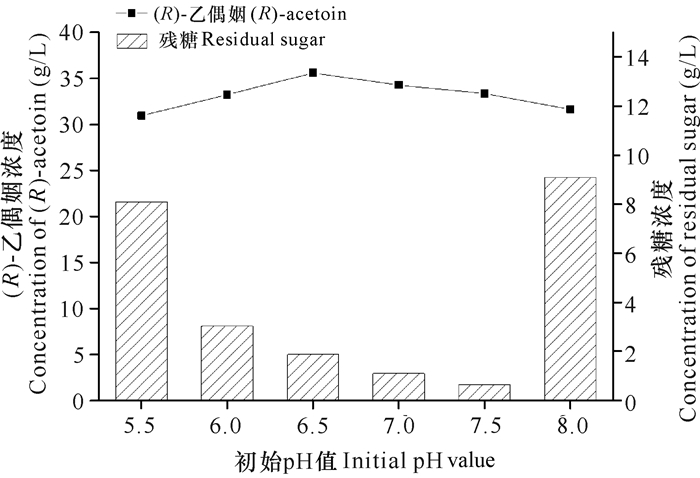
|
| 图 1 初始pH值对菌株GXASR合成(R)-乙偶姻的影响 Fig. 1 Effect of initial pH value on (R)-acetoin produced by GXASR |
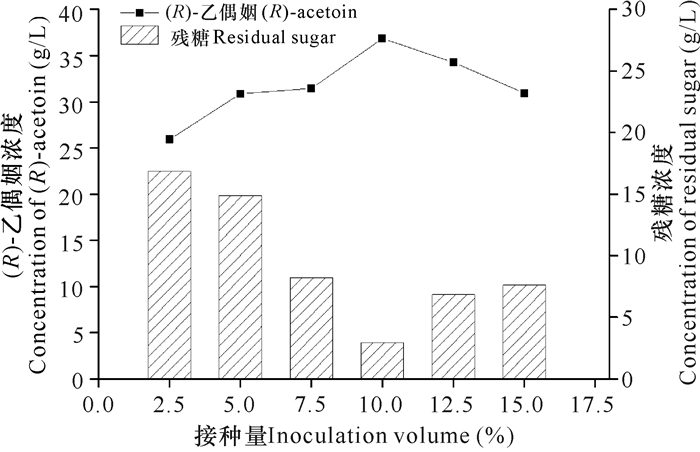
|
| 图 2 接种量对菌株GXASR合成(R)-乙偶姻的影响 Fig. 2 Effect of inoculate volume on (R)-acetoin produced by GXASR |
2.3 工程菌株GXASR发酵罐补料发酵
经过发酵条件的优化,确定以100 g/L葡萄糖为碳源、15 g/L蛋白胨和15 g/L酵母粉为氮源的发酵培养基,pH值为6.5以及接种量10%进行3 L发酵罐补料发酵实验。在发酵过程中的不同时间点进行取样,检测发酵液菌体浓度、(R)-乙偶姻浓度、2, 3-丁二醇浓度以及残糖浓度。当残糖浓度降至30-40 g/L时一次补加葡萄糖至160 g/L。发酵初期菌体OD600增长迅速,耗糖较快,发酵7.5 h时残糖浓度降低到40.2 g/L,菌体OD600达17.4,此时菌体浓度高、代谢活力强,通过补加葡萄糖,细胞可以高效合成(R)-乙偶姻,发酵液中(R)-乙偶姻浓度快速升高。发酵36 h时,残糖浓度降至8.5 g/L,发酵液中(R)-乙偶姻浓度不再增加,发酵结束,(R)-乙偶姻产量达到67.65 g/L,生产强度为1.88 g/(L·h),得率为0.40 g/g(图 3)。
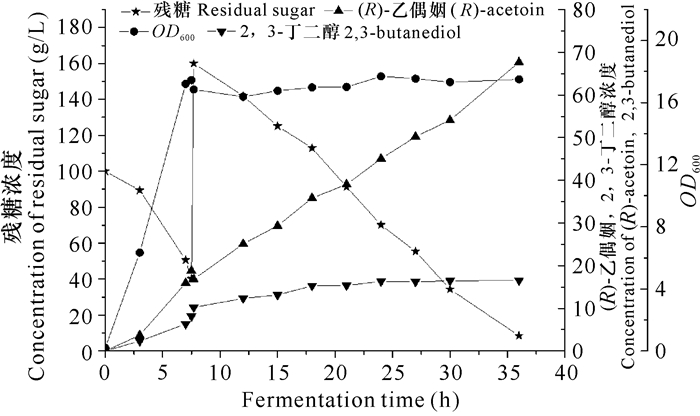
|
| 图 3 工程菌株GXASR补料发酵 Fig. 3 The fed-batch fermentation of engineering strain GXASR |
3 讨论
化学法及天然菌种产生的乙偶姻都是(R)-乙偶姻和(S)-乙偶姻的混合物,两者的物理化学性质相近,手性拆分十分困难,存在高成本、高能耗、污染严重等问题,极大地限制了光学纯(R)-乙偶姻开发和应用。因此,开展微生物代谢网络改造及高效生产光学纯(R)-乙偶姻的研究具有重要的意义,也是近年来研究热点之一。Kochius等[27]利用酶催化氧化meso-2, 3-丁二醇的方法合成(R)-乙偶姻,浓度为48 mmol/L,反应需要的辅酶NAD+由电化学方法再生;2016年,Guo等[28]在E.coli BL21(DE3)中表达meso-2, 3丁二醇脱氢酶、NADH氧化酶和血红蛋白的基因,将获得的E.coli/pET-mbdh-nox-vgb作为全细胞生物催化剂,可以高效催化meso-2, 3丁二醇还原为(R)-乙偶姻,(R)-乙偶姻的产量为86.7 g/L,光学纯度为97.9%。但全细胞催化法需要采用meso-2, 3-丁二醇、(R, R)-2, 3-丁二醇等作为底物,光学纯的底物成本高昂,无法应用于工业生产(R)-乙偶姻。有相当多的报道[3, 22-25]通过合成生物学方法和代谢工程方法在微生物细胞中高效表达(R)-乙偶姻合成的关键酶基因或敲除(R)-乙偶姻降解的相关基因,对菌株进行代谢网络改造,采用葡萄糖、蔗糖等碳源合成(R)-乙偶姻,其产量和光学纯度都有较大的提高。但是工程菌株中代谢网络的碳架物质流量往往会发生重大改变,导致(R)-乙偶姻合成与细胞内氧化还原平衡关系的失调,进而限制(R)-乙偶姻产量和光学纯度的进一步提高,(R)-乙偶姻光学纯度通常在98%以下。大肠杆菌遗传背景清晰、转化效率高、遗传操作技术成熟,使其在代谢工程等研究领域备受青睐,是用于构建微生物细胞工厂合成生物基化学品首选宿主之一。本研究以大肠杆菌E.coli MG1655为宿主细胞,将来源于E. cloacae的α-乙酰乳酸合成酶基因budB、α-乙酰乳酸脱羧酶基因budA和来源于L. brevis的NADH氧化酶基因noxE组成基因簇,构建大肠杆菌生产(R)-乙偶姻工程菌株,实现大肠杆菌高效合成(R)-乙偶姻和胞内NADH/NAD+氧化还原平衡,通过优化发酵过程,经摇瓶发酵的(R)-乙偶姻产量为36.82 g/L,光学纯度高达99.1%,发酵罐补料发酵的(R)-乙偶姻产量达到67.65 g/L,生产强度为1.88 g/(L·h),得率为0.40 g/g,发酵周期短,生产强度高,是具有潜力的(R)-乙偶姻生产方法。
同时在研究中发现,以葡萄糖、酵母粉和蛋白胨等作为原料时成本仍较高,在大规模发酵生产时不利于提高效益;发酵过程中随着(R)-乙偶姻的积累,会出现2, 3-丁二醇、乙醇、乳酸、乙酸等副产物的产出,副产物浓度升高时会降低目的产物的转化率并导致生物毒性,对菌体的生长速率、菌体浓度以及外源基因表达都会产生明显的抑制作用。本研究下一步将探讨利用廉价原料替代高价碳源氮源,并通过代谢工程、基因组学等手段对细胞内(R)-乙偶姻的合成和分解代谢网络进行系统解析与优化,为低成本、高效率生产高光学纯(R)-乙偶姻提供操作性良好的技术支持。
4 结论本研究成功构建了产光学纯(R)-乙偶姻的大肠杆菌工程菌株GXASR,并通过摇瓶发酵对发酵培养基和培养条件进行优化,使得工程菌株GXASR能高效地合成(R)-乙偶姻。GXASR工程菌株在优化后的培养基以及发酵条件中,经摇瓶发酵的(R)-乙偶姻产量为36.82 g/L,通过发酵罐补料发酵36 h,(R)-乙偶姻产量能够达到67.65 g/L,光学纯为99.1%,生产强度为1.88 g/(L·h),得率为0.40 g/g,显著提高了(R)-乙偶姻合成效率。本研究为人工途径合成(R)-乙偶姻提供了现实指导意义。
| [1] |
XIAO Z J, LU J R. Strategies for enhancing fermentative production of acetoin:A review[J]. Biotechnology Advance, 2014, 32(2): 492-503. DOI:10.1016/j.biotechadv.2014.01.002 |
| [2] |
XIAO Z J, LU J R. Generation of acetoin and its derivatives in foods[J]. Journal of Agricultural and Food Chemistry, 2014, 62(28): 6487-6497. DOI:10.1021/jf5013902 |
| [3] |
XU Q M, XIE L X, LI Y Y, et al. Metabolic engineering of Escherichia coli for efficient production of (3R)-acetoin[J]. Journal of Chemical Technology & Biotechnology, 2015, 90(1): 93-100. |
| [4] |
GAO C, ZHANG L, XIE Y, et al. Production of (3S)-acetoin from diacetyl by using stereoselective NADPH-dependent carbonyl reductase and glucose dehydrogenase[J]. Bioresource Technology, 2013, 137: 111-115. DOI:10.1016/j.biortech.2013.02.115 |
| [5] |
WERPY T, PETERSEN G.Top value added chemicals from biomass: Volume I-Results of screening for potential candidates from sugars and synthesis gas[R].Washington DC: US Department of Energy, 2004. https://www.osti.gov/biblio/15008859
|
| [6] |
甄德帅. 香料乙偶姻(3-羟基-2-丁酮)化学工艺合成现状[J]. 黔南民族师范学院学报, 2015, 35(4): 121-124. |
| [7] |
李欣, 张显, 杨套伟, 等. 代谢工程改造枯草芽孢杆菌发酵生产乙偶姻[J]. 基因组学与应用生物学, 2017(12): 5159-5166. |
| [8] |
BAE S J, KIM S, HAHN J S. Efficient production of acetoin in Saccharomyces cerevisiae by disruption of 2, 3-butanediol dehydrogenase and expression of NADH oxidase[J]. Scientific Reports, 2016, 6: 27667. DOI:10.1038/srep27667 |
| [9] |
WANG S, LUO Q, LIU J, et al. Mutation and fermentation optimization of Bacillus amyloliquefaciens for acetoin production[J]. Chinese Journal of Biotechnology, 2018, 34(5): 803-811. |
| [10] |
DAI J, WANG Z, XIU Z L. High production of optically pure (3R)-acetoin by a newly isolated marine strain of Bacillus subtilis CGMCC 13141[J]. Bioprocess and Biosystems Engineering, 2019, 42(3): 475-483. DOI:10.1007/s00449-018-2051-8 |
| [11] |
JIA X, KELLY R M, HAN Y. Simultaneous biosynthesis of (R)-acetoin and ethylene glycol from, D-xylose through, in vitro, metabolic engineering[J]. Metabolic Engineering Communications, 2018, 7: e00074. DOI:10.1016/j.mec.2018.e00074 |
| [12] |
ZHANG G, WANG Q, SU Y, et al. Genome sequence of Staphylococcus aureus PX03, an acetoin-producing strain with a small-sized genome[J]. Genome Announcements, 2017, 5(37): e00753-17. |
| [13] |
CHO S, KIM K D, AHN J H, et al. Selective production of 2, 3-butanediol and acetoin by a newly isolated bacterium Klebsiella oxytoca M1[J]. Applied Biochemistry and Biotechnology, 2013, 170(8): 1922-1933. DOI:10.1007/s12010-013-0291-2 |
| [14] |
TIAN Y, XU H, LIU J, et al. Construction of acetoin high-producing Bacillus subtilis strain[J]. Biotechnology & Biotechnological Equipment, 2016, 30(4): 700-705. |
| [15] |
SHI L T, GAO S S, YU Y, et al. Microbial production of 2, 3-butanediol by a newly-isolated strain of Serratia marcescens[J]. Biotechnology Letters, 2014, 36: 969-973. DOI:10.1007/s10529-013-1433-x |
| [16] |
JANTAMA K, POLYIAM P, KHUNNONKWAO P, et al. Efficient reduction of the formation of by-products and improvement of production yield of 2, 3-butanediol by a combined deletion of alcohol dehydrogenase, acetate kinase-phosphotransacetylase, and lactate dehydrogenase genes in metabolically engineered Klebsiella oxytoca in mineral salts medium[J]. Metabolic Engineering, 2015, 30: 16-26. DOI:10.1016/j.ymben.2015.04.004 |
| [17] |
RATHNASINGH C, PARK J M, KIM D K, et al. Metabolic engineering of Klebsiella pneumoniae and in silico investigation for enhanced 2, 3-butanediol production[J]. Biotechnology Letters, 2016, 38(6): 975-982. DOI:10.1007/s10529-016-2062-y |
| [18] |
LI L X, LI K, WANG Y, et al. Metabolic engineering of Enterobacter cloacae for high-yield production of enantiopure (2R, 3R)-butanediol from lignocellulose-derived sugars[J]. Metabolic Engineering, 2015, 28: 19-27. DOI:10.1016/j.ymben.2014.11.010 |
| [19] |
YU B, SUN J B, BOMMAREDDY R R, et al. Novel (2R, 3R)-2, 3-butanediol dehydrogenase from potential industrial strain Paenibacillus polymyxa ATCC 12321[J]. Applied and Environmental Microbiology, 2011, 77(12): 4230-4233. DOI:10.1128/AEM.02998-10 |
| [20] |
XU H, JIA S, LIU J. Development of a mutant strain of Bacillus subtilis showing enhanced production of acetoin[J]. African Journal of Biotechnology, 2013, 10(5): 779-799. |
| [21] |
SUN J, ZHANG L, RAO B, et a1. Enhanced acetoin production by Serratia marcescens H32 using statistical optimization and a two-stage agitation speed control strategy[J]. Biotechnology and Bioprocess Engineering, 2012, 17(3): 598-605. DOI:10.1007/s12257-011-0587-4 |
| [22] |
BAI F, DAI L, FAN J, et al. Engineered Serratia mar-cescens for efficient (3R)-acetoin and (2R, 3R)-2, 3-butanediol production[J]. Journal of Industrial Microbiology & Biotechnology, 2015, 42(5): 779-786. |
| [23] |
LV X, DAI L, BAI F, et al. Metabolic engineering of Serratia marcescens MG1 for enhanced production of (3R)-acetoin[J]. Bioresources Bioprocessing, 2016, 3(1): 52. DOI:10.1186/s40643-016-0128-2 |
| [24] |
WANG D, ZHOU J, CHEN C, et al. R-acetoin accumulation and dissimilation in Klebsiella pneumoniae[J]. Journal of Industrial Microbiology & Biotechnology, 2015, 42(8): 1105-1115. |
| [25] |
XIAO Z J, LV C, GAO C, et al. A novel whole-cell biocatalyst with NAD+ regeneration for production of chiral chemicals[J]. PLoS One, 2010, 5(1): e8860. DOI:10.1371/journal.pone.0008860 |
| [26] |
OLINS P O, RANGWALA S H. A novel sequence element derived from bacteriophage T7 mRNA acts as an enhancer of translation of the lacZ gene in Escherichia coli[J]. The Journal of Biological Chemistry, 1989, 264(29): 16973-16976. |
| [27] |
KOCHIUS S, PAETZOLD M, SCHOLZ A, et al. Enantioselective enzymatic synthesis of the α-hydroxy ketone (R)-acetoin from meso-2, 3-butanediol[J]. Journal of Molecular Catalysis B:Enzymatic, 2014, 103: 61-66. DOI:10.1016/j.molcatb.2013.08.016 |
| [28] |
GUO Z, ZHAO X, HE Y, et al. Efficient (3R)-acetoin production from meso-2, 3-butanediol using a new whole-cell biocatalyst with co-expression of meso-2, 3-butanediol dehydrogenase, NADH oxidase and Vitreoscilla hemoglobin[J]. Journal of Microbiology and Biotechnology, 2017, 27(1): 92-100. DOI:10.4014/jmb.1608.08063 |



