2. 广西大学计算机与电子信息学院, 广西南宁 530004;
3. 广西高校并行与分布式计算技术重点实验室, 广西南宁 530004
2. School of Computer, Electronics and Information, Guangxi University, Nanning, Guangxi, 530004, China;
3. Guangxi Colleges and Universities Key Laboratory of Parallel and Distributed Computing Technology, Nanning, Guangxi, 530004, China
蛋白质在各类生物的生命活动中扮演着至关重要的作用,是生命构成的基本单位。研究表明,各类蛋白质所具有的特定生物功能与其结构、属性等有着紧密的联系。特别是,同源蛋白质虽然可能来源于不同的生物或不相同的分子,但是他们具有相同或相似的结构,具有相同或相似的功能。因此,通过获取与学习蛋白质的结构信息、属性信息,探测和识别蛋白质的同源性,对于预测未知蛋白质的功能有着重要的科研价值和广泛的应用需求。随着蛋白质氨基酸序列检测技术的发展,越来越多的蛋白质氨基酸序列正逐步被人们所认识。蛋白质氨基酸序列的获取变得越来越快捷、准确。与之形成鲜明对比的是,蛋白质的结构特征、属性信息(蛋白质二级结构、跨膜结构、疏水性、溶剂可及性)等的获取与识别方法仍然没有较大的突破。目前,90%已知的蛋白质结构信息、属性信息是通过X射线、核磁共振、生物化学实验等方法获得的[1-2]。这些获取方法不仅对实验条件要求高,需要大量的人力物力,而且所需时间比较长[2-4]。获取一个蛋白质分子的结构信息、属性信息平均需要20多天。因此,构建一种能够从蛋白质氨基酸序列中自动快速地学习蛋白质的结构和属性特征,并对蛋白质的同源性进行准确探测和识别的人工智能方法,将对蛋白质的结构探测与分析、蛋白质功能的识别与分析,以及相关的生物医学研究的发展,起到十分重要的促进作用。
在最近的一些研究中,研究人员尝试以一种或少数几种特定的蛋白质属性信息作为研究对象,探测识别蛋白质的结构,但是由于忽略了许多蛋白质的属性信息,识别准确度和召回率等测试结果并不理想[4-5]。有的研究人员将蛋白质同源性预测,转化为基于氨基酸链匹配的检索任务,通过搜索具有排序相似性的氨基酸链,探测同源蛋白质。最近几年,研究人员开始尝试使用机器学习的方法,采用特征获取与统计学习的策略,期望能提升蛋白质探测与识别的效能[6-7]。常用方法有隐马尔科夫模型(Hidden Markov model,HMM)、神经网络(Artificial neural network,ANN)、支持向量机(Support vector machine,SVM),等等。
目前,神经网络等机器学习技术已经广泛用于各种领域的数据特征学习、信息分类等研究,表现优异[8-9]。根据蛋白质的结构相似、功能相近的原则,基于相似性比对的方法是目前最为流行的一种同源蛋白质探测识别技术。一些研究人员使用基于模板匹配技术,构建机器学习模型,以期实现同源蛋白质预测[4, 9-10]。其中,1-TASSER是目前最为有名的基于模板匹配技术的同源蛋白质预测模型[4]。该模型使用CAS和CABS算法提取蛋白质结构特征信息,然后结合HMM和SVM方法,进行基于模板的相似性匹配[9]。Webb教授的科研团队[11]使用神经网络提取蛋白质特征,然后使用MODELLER算法进行蛋白质模板匹配分类,其研究成果证明,使用神经网络结构可以获取一定量符合同源蛋白质探测需求的数据特征,但是将数据特征获取与同源性探测分开进行,导致重要的、最合适的特征数据丢失;而且浅层的神经网络无法获取到蛋白质的高级不变性特征。
为解决这些方法在探测同源蛋白质方面的局限,研究人员针对蛋白质的某些属性,采取有针对性的特征获取方法,期望获取蛋白质特定属性的高级不变性特征,进而提升蛋白质的探测识别准确度。Wu等[12]和Zahiri等[13]针对蛋白质的残基结构,进行有针对性的特征提取,提升蛋白质的探测识别效能。He等[14]通过构建蛋白质的PSSM矩阵,获取同源蛋白质的氨基酸结合特征,提高蛋白质识别效率。Blaszczyk等[15]通过统计分析蛋白质氨基酸链的物理属性特征,构造残基的等价矩阵,进行同源蛋白质的相似性匹配。另外,部分研究人员尝试使用浅层神经网络获取蛋白质的高级结构和属性特征,例如疏水性、蛋白质二级结构、进化相关性等信息,再使用分类算法进行蛋白质分类识别[3, 9]。最近,一些研究中采用了深度学习技术来提取蛋白质高级特征。例如,Eickholt等[16]和Di等[17]使用递归神经网络(Recurrent neural network, RNN)学习蛋白质的高级特征,然后使用Softmax分类器对获取的特征进行分类。实验结果表明深度网络架构可以提取到更高级、更有效的数据特征。
目前基于深度学习的方法普遍采用浅层分类器对所学特征进行分类,分类准确率不高。而且均是将蛋白质特征提取过程和分类识别过程分开进行学习训练,导致深度网络所学到的蛋白质特征,不是最适合当前识别任务需求的特征信息,不能依据分类任务的特点,有针对性地学习最合适、最有效的特征。为了解决这些问题,本文构建了一种新的混合深度学习模型(Hybrid deep learning model for protein homology prediction, HDLM-PHP),该模型主要由多组深度卷积神经网络和多层RBM结构组成,按照自底向上全连接的方式构建,旨在将蛋白质特征的获取和同源蛋白质探测任务目标融为一个整体,获取对于同源蛋白质探测最有效、最合适的蛋白质特征信息,从本质上提升同源蛋白质的探测与识别。模型通过对待检测蛋白质和靶蛋白质的相似性识别,将同源蛋白质探测问题转化为蛋白质相似性识别问题,令抽象的探测问题可以通过条件概率预测解决。模型的每一组深度卷积网络有针对性地学习蛋白质的特征信息,在模型高层汇聚成全局高级不变性的蛋白质特征,使得所提取的蛋白质特征内容丰富、不变性强。模型还采用统一的“管道式”训练算法,以识别任务为导向,联合训练特征提取过程和识别分类过程,保证了模型能获取到对于当前识别任务最合适、最有效的蛋白质全局高级不变性特征。模型顶层的多层RBM结构则最终保证同源蛋白质分类识别的效力。
1 材料与方法 1.1 模型结构基于混合深度学习技术设计面向蛋白质结构检测的模型(HDLM-HPH)。该模型整合了多种深度学习架构的优点,将蛋白质特征的获取和同源蛋白质探测任务目标融为一个整体,获取对于同源蛋白质探测最有效、最合适的蛋白质特征信息,从本质上提升同源蛋白质的探测与识别。模型采用多组深度卷积神经网络构造深度学习架构,学习蛋白质特征信息,然后通过全连接方式将蛋白质高级不变性特征与多层分类模块相连接。最后,以同源蛋白质探测作为任务目标,采用统一的模型训练方式,将特征获取模块和分类识别模块融为一个整体。模型构建的目的是能学习到适合同源蛋白质探测的蛋白质特征,从本质上提高同源蛋白质探测和识别的效能。混合深度学习模型的架构如图 1所示。

|
| 图 1 混合深度学习模型示意图 Fig. 1 Schematic diagram of hybrid deep learning model |
HDLM-HPH具备3大特点:
(1) HDLM-HPH通过自底向上的深度层次结构,直接从待测蛋白质和对比蛋白质中学习高级相似性特征,可以最大程度地保证特征学习的一致性,保证所学到的特征是最能反映两个蛋白质相似度的特征。传统深度学习方法的解决方案是,分别使用深度学习算法学习待测蛋白质特征和对比蛋白质特征,然后再进行特征比对。这种解决方案并不能保证两个蛋白质的特征学习处于同一步调,不能保证所学特征与任务导向的一致性,从而没有办法保证所学特征适合同源蛋白质探测任务需求。
(2) HDLM-HPH采用多组深度卷积神经网络结构,组成复合型的数据特征学习网络,每一组深度卷积神经网络采用滑动窗口策略学习对应的蛋白质链段特征,从而保证模型能应对各种长度、各种结构的蛋白质探测与识别情况。传统深度学习方法的解决方案只采用一套深度学习架构,解决各种数据特征学习问题,无法根据数据的不同属性特性,进行有针对性的特征学习,无法获得全局高级不变性特征。而多组深度卷积神经网络,能逐层学习到蛋白质链高级特征,将通过模型高层的全连接结构重新组合成完整的蛋白质高级特征,从数据的不同属性特征出发,尽可能地学习蛋白质的全局高级不变性特征。
(3) HDLM-HPH采用统一的“管道式”模型训练方法,以同源蛋白质检测任务为导向,将特征提取和任务目标统一进行训练,保证模型能提取到最有效、最合适的蛋白质特征信息,从本质上提升同源蛋白质探测与识别的效能。
1.2 模型的具体实现 1.2.1 模型的内部架构借鉴机器翻译最新研究成果,HDLM-HPH构造了一个基于语句特征的判别式模型,通过最大化翻译语句的条件概率,实现机器自动翻译。同源蛋白质探测任务可以视为根据待检测蛋白质的特征预测与靶蛋白质同源的概率。因此,模型可以通过深度混合网络架构来学习待检测蛋白质和靶蛋白质的相关性高级特征(High-level relational feature),并根据此特征使用多层RBM结构实现待检测蛋白质和靶蛋白质的相似度估计,实现蛋白质同源性检测。相应的公式如下:
| $ {S^*} = \mathop {\arg \max }\limits_{S,\theta } \log p(S|Q,\theta ), $ | (1) |
式中,S表示靶蛋白质,Q表示混合深度学习网络获得的待检测蛋白质和靶蛋白质的相关性高级特征,S*是检测到的最有可能的同源蛋白质,θ是模型参数。Bai等[18-19]的研究表明,公式(1)中的log似然函数可以转换为基于能量函数的算法进行推理和学习,如下面公式所示:
| $ p(S)=\frac{\sum_{Q} e^{-E(S, Q)}}{\sum_{S, Q} e^{-E(S, Q)}}, $ | (2) |
| $ p(S | Q)=\frac{e^{-E(S, Q)}}{\sum_{S, Q} e^{-E(S, Q)}}, $ | (3) |
式中,E(S, Q)是混合深度网络表示的系统能量函数,其值越大,表示待检测蛋白质和靶蛋白质同源相似度越大。E(S, Q)的计算公式如下:
| $ E(S, Q)=-\sum_{i, j}{{{w}_{s}}}\psi \left( {{s}_{i}}, {{q}_{j}} \right)-\sum_{i}{{{b}_{i}}}{{s}_{i}}-\sum_{j}{{{c}_{j}}}{{q}_{j}}, $ | (4) |
其中,ws表示待检测蛋白质和靶蛋白质相关性权值参数,φ(si, qi)表示待检测蛋白质和靶蛋白质的相关性高级特征,bi和cj是模型的偏移量参数。
HDLM-HPH由多组深度卷积神经网络(CNNs)、多层RBM结构以及softmax分类器组成。模块之间采用全连接方式结合(图 2)。模型底层由并行的5组深度卷积神经网络构成,每一组深度卷积神经网络由4套“卷积层—池化层”模块构成。每一组网络会有针对性地学习蛋白质的各种属性特征,例如蛋白质二级结构特征、跨膜结构特征、疏水性特征、信息序列特征、残基特征等。因为蛋白质有着很强的局部相关性,所以深度卷积神经网络,采用N-windows滑动窗口的方式处理蛋白质链。深度卷积神经网络架构可以保证随着层次的不断升高,可以逐步学习到待检测蛋白质和靶蛋白质的高级不变性特征。

|
| 图 2 深度网络架构示意图 Fig. 2 Diagram of deep network architecture |
这些高级不变性特征将会传入多层RBM结构进行特征的融合,多层RBM结构首先通过全连接方式,将5组深度卷积神经网络提取到的蛋白质特征融为一个整体,然后通过逐层收拢学习方式,获取全局不变性特征,并与最高层的softmax分类器共同实现待检测蛋白质和靶蛋白质的同源性探测和识别。
由于混合深度学习模型含有多种深度学习模块,而且所含深度卷积神经网络组数多、较为复杂,为了防止过拟合的情况,首先, 需要对每一组深度卷积神经网络进行有针对性的预训练,保证每一组网络都能针对特定的蛋白质属性进行学习;其次,采用CD-K算法[19]对多层RBM结构和softmax分类器进行预训练;最后,采用反向传播训练算法(Back propagation,BP)对整个混合后深度学习模型进行训练,以保证模型能根据识别任务的固有特点进行特征提取,从而提高模型的探测识别效能。
1.2.2 深度卷积神经网络混合深度学习模型包含5组深度卷积神经网络,每一组有针对性地学习待检测蛋白质和靶蛋白质的某些属性特征。模型采用并行的训练方式,采用统一的“管道式”连接方式,以识别任务为导向,训练深度卷积神经网络。避免特征提取过程与分类识别过程分开训练造成的负面影响。
每一组深度卷积神经网络由4组“卷积层—池化层”模块构成。每一个卷积层都能使用经过训练的卷积核,从前面的池化层中学习更高级、不变性更强的特征信息,如下面公式所示:
| $ y_{j}^{C}=\sum_{i} k_{i j} \times x_{i}+b_{j}, $ | (5) |
其中,yj表示第j个卷积映射层,kij是对应的卷积核,他从前面的池化层xi学习到本卷积映射层的数据特征,bj是第j个卷积映射层的运算偏移参数。每一个卷积层会使用多个卷积核,计算得到多个卷积映射层,从而能提取到广泛有效的各种数据特征。
每一个卷积层后会紧跟一个池化层,池化层将采用平均运算子,对前一个卷积映射层学习到的数据特征进行聚合收拢,把重要的特征保留加强,噪声和无用特征滤除。计算推理公式为
| $ y_{j}^{p}=S\left(\beta \sum x_{i}^{n \times n}+\alpha\right), $ | (6) |
其中,xin×n是前一个卷积映射层n×n的图像块,β是可训练参数,表示池化的训练期望权重,α是模型偏移参数,S是激发函数。
1.2.3 多层RBM分类结构混合深度学习模型的高层是由多层RBM结构和softmax分类器构成,这两种模块通过全连接方式逐层将多组深度卷积神经网络提取的特征数据进行融合与再精炼,最后根据精炼的特征向量进行联合概率推理,估计待检测蛋白质和靶蛋白质的同源相似度。联合概率计算正定于多层RBM结构和softmax分类器组成的系统能量函数,即:
| $ p({y_c}|v) = \frac{{{e^{{d_c}}}{\prod _j}(1 + {e^{{c_j} + {U_{jc}} + {\sum _k}{w_{jk}}{v_k}}})}}{{{\sum _i}{e^{{d_i}}}{\prod _j}({\rm{ }}1 + {e^{{c_j} + {U_{jc}} + {\sum _k}{w_{jk}}{v_k}}})}}。$ | (7) |
本文将在国际标准数据集SCOPe中进行实验,实验方法采用目前较为流行的启发式实验评价体系[20-22],通过与当前表现优异的蛋白质同源检测方法进行比较,说明本文提出的混合深度学习模型的性能。这些方法是CoMOGPHOG-Score[20], TM-Score[21], 和SP-Score[22]。
SCOPe数据集是Structural classification of protein (SCOP)数据集的最新扩展版,是当前蛋白质研究最常用的一个标准数据集[23]。他以类比分类学为理论基础,将收集的蛋白质数据按照逻辑范畴和蛋白质相关研究的需求,自顶向下分为6层领域,分别是:物种、蛋白质、族、超族、折叠、类。“族”领域是根据蛋白质的结构相似性,将蛋白质序列进行聚类所得。因此,本文的实验主要是在“族”领域中开展。
1.3.2 实验评价体系为了更系统地评估本文提出的模型,实验将统计多项指标数据,包括马修斯相关系数(Mathews correlation coefficient,MCC)、受试者工作特征曲线(Receiver operating characteristics,ROC)、敏感性(Sensitivity)、精确度(Precision)和特异性(Specificity)。这些实验统计指标数据均是当前蛋白质相关研究的标准测试指标数据。对比模型的代码均来自作者公开的研究成果,实验结果均为各个对比模型的最佳实验输出。
马修斯相关系数(MCC)是一种十分著名的相似度和分类度量方法,广泛应用于蛋白质研究等生物信息学领域。他系统地考虑正阳性、假阳性、真阴性、假阴性之间的相关性,是一种鲁棒性很强的评价方法,可以有效处理测试数据集大小不均、实验数据和实验结果不平衡等问题。MCC通过统计实验结果与真实数据之间的二元分类相关系数(Correlation coefficient),来评估模型在同源蛋白质分类方面的效能。二元分类相关系数是一个处于-1到1区间内的实数,当取值越接近1,则表明模型的预测能力越好;若取值等于0,则表明模型的性能与随机猜测的效果差不多;若取值越接近-1,则表明模型的结果与实际结果完全相反。马修斯相关系数评测计算方法如公式(8)所示。
| $ \begin{array}{c}{\mathrm{MCC}=} \\ {\frac{T P \times T N-F P \times F N}{\sqrt{(T P+F P) \times(T P+F N) \times(T N+F P) \times(T N+F N)}}}。\end{array} $ | (8) |
精确度-召回率曲线是相似度和分类研究中最为常用的一种评价指标。他可以联立分析同源蛋白质检测的精确度和召回率,而且直观描述出精确度在召回率变化时模型的鲁棒性与泛化性能。
受试者工作特征曲线(ROC)又称为感受性曲线(Sensitivity curve)是生物信息学研究领域最常用的一种评价标准。他以真阳性率(True positive rate)为纵坐标,以假阳性率(False positive rate)为横坐标,在二维平面上绘制模型对于测试样本集中各个测试样本的真阳性和假阳性的判断概率,并用光滑的曲线将这些点连接起来。图左上角(0, 1)点表示理想情况的最佳分类效果,图右下角(1, 0)点表示最糟糕分类情况。从点(0, 0)至点(1, 1)的对角线,表示随机分类的效果。因此,ROC曲线处于对角线上方,并且曲线转角约接近(0, 1)点,表明模型的分类效能越好。
2 结果与分析 2.1 马修斯相关系数分析图 3描述了HDLM-HPH与3个对比模型(CoMOGPHOG-Score, TM-Score, 和SP-Score)在马修斯相关系数评测中的表现。通过观察曲线的走势,可以发现HDLM-HPH在进行了150次训练后,取得了最优的实验结果,马修斯相关系数最终达到0.923 5;其他3个对比模型最好的实验结果是马修斯相关系数0.864 0,最低的马修斯相关系数是0.688 0。可见,HDLM-HPH取得了0.059 5至0.235 5的提升。分析上述模型的算法特点发现,3个对比模型均只是使用了有限的蛋白质氨基酸链特征信息。例如CoMOGPHOG-Score方法采用“方向梯度金字塔直方图”提取蛋白质的空间特征信息;SP-Score方法主要使用氨基酸残基各种原子之间的距离信息。而与对比模型不同,混合深度学习模型能根据探测识别任务的特点,有针对性地学习到最合适、最有效的待检测蛋白质和靶蛋白质的相关性特征,这些高级特征对于提高同源蛋白质的检测起到十分有益的促进作用。
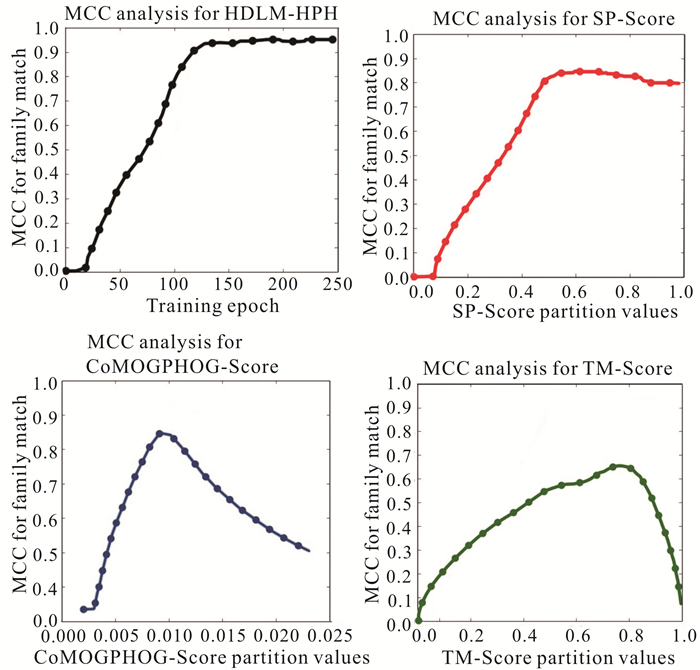
|
| 图 3 马修斯相关系数实验结果图 Fig. 3 Experimental result chart of Mathews correlation coefficient |
2.2 精确度与召回率分析
图 4展示了本文提出的HDLM-HPH和3个对比模型的精确度-召回率曲线。4条曲线直观地展示出4个模型在精确度和召回率统计分析指标上的表现,HDLM-HPH在精确度和召回率上均取得了最优的表现,精确度-召回率曲线一直优于其他3个对比模型。而且在精确度指标随召回率变化方面,HDLM-HPH的精确度指标变化也是最缓慢的。更加值得注意的是,混合深度学习模型在召回率达到100%时,仍能保持38.3%以上的精确度;其他3个对比模型在召回率达到70%~80%时,精确度就已经降到1%~5%。分析4个模型的探测识别算法,HDLM-HPH的优势体现在两个方面:1)HDLM-HPH采用多组深度卷积神经网络学习蛋白质的各种属性特征,并且使用多层RBM结构对这些属性特征进行聚合和精炼,保证能使用待检测蛋白质和靶蛋白质丰富的高级相关性特征,促进同源蛋白质的检测和识别。2)HDLM-HPH采用统一的“管道式”训练方式,将特征提取过程和检测识别过程作为一个整体进行学习训练,保证深度学习架构能根据检测识别任务的特点,学习最有效、最合适的蛋白质特征,保证模型能达到最优检测和识别性能。
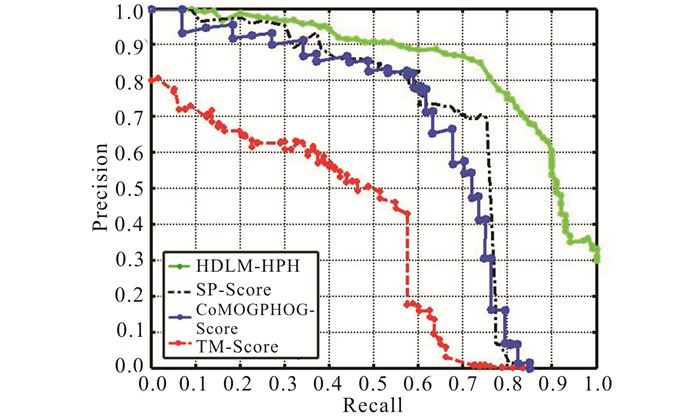
|
| 图 4 精确度-召回率实验结果图 Fig. 4 Experimental result chart of precision-recall |
2.3 受试者工作特征曲线分析
图 5描述了HDLM-HPH和3个对比模型的ROC曲线。图 5直观地展示出HDLM-HPH的ROC曲线处于其他3个模型ROC曲线之上,并且曲线的转角也更加靠近(0, 1)点,可见HDLM-HPH优于其他3个模型。通过仔细分析图 5中的数据,在相同的假阳性点上,HDLM-HPH比其他3个模型,均能获得更高的真阳性值。而且HDLM-HPH获得0.91的ROC值,比其他3个方法中表现最好的CoMOGPHOG-Score(ROC值=0.85)高出6%。而且相比于其他3个模型,HDLM-HPH以最低的假阳性值0.035,获得超过0.8的真阳性值,以8%的优势超越其他3个模型。分析4个模型的探测识别过程,HDLM-HPH能在上述评价指标中取得最好表现,主要是因为所设计的混合深度学习模型将特征提取过程和检测识别过程作为一个整体,采用了统一的“管道式”训练方法,进行从头至尾的反馈传播训练,保证深度学习架构能根据检测识别任务的特点,学习最合适的蛋白质相关性高级特征,从本质上提升了深度学习模型的检测识别性能。
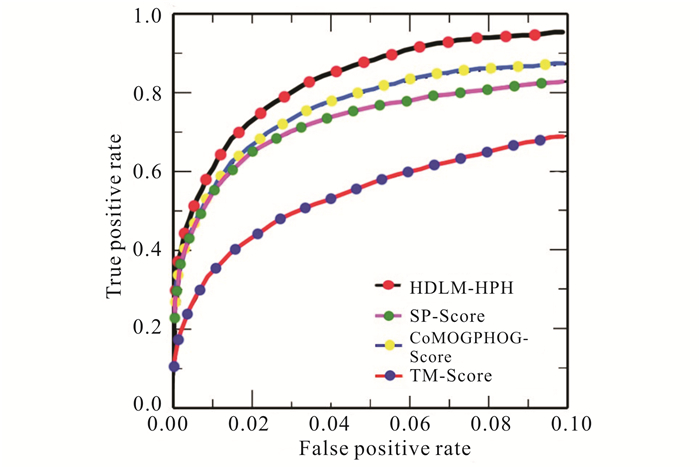
|
| 图 5 受试者工作特征曲线分析图 Fig. 5 Analysis chart of receiver operating characteristic curve |
3 结论
探测和识别待测蛋白质的同源性、所属族类,是现代计算生物学研究的一个关键难点,是未来进一步将人工智能技术应用生物医学和生物信息学的基础。本文提出一种新的混合深度学习模型(HDLM-HPH),旨在解决同源蛋白质探测识别过程中的关键问题:如何学习最有效、最适合同源蛋白质预测的特征信息;如何更好地运用蛋白质特征信息,提升同源蛋白质的探测与识别。HDLM-HPH继承了深度卷积网络架构(ConvNet)和多层RBM结构的优点,采用多组并行的深度卷积神经网络,学习蛋白质的各种属性信息,以期获得丰富的待检测蛋白质和靶蛋白质的高级相关性特征。模型通过全连接方式使用多层RBM结构融合和精炼这些相关性特征为全局相关性特征,通过统一的深度网络连接方式,将蛋白质特征获取与同源蛋白质预测统一为一个整体,以探测和识别任务为导向,学习到对于同源蛋白质预测最有效、最全面的蛋白质特征信息。在标准数据集上进行的对比实验结果说明本文提出的模型在马修斯相关系数、受试者工作特征曲线、敏感性、精确度等评价指标方面均优于当前公开发表的研究成果。HDLM-HPH可以扩展到蛋白质研究的其他领域。
| [1] |
MÁRQUEZ-CHAMORRO A E, ASENCIO-CORTÉS G, SANTIESTEBAN-TOCA C E, et al. Soft computing methods for the prediction of protein tertiary structures:A survey[J]. Applied Soft Computing, 2015, 35: 398-410. DOI:10.1016/j.asoc.2015.06.024 |
| [2] |
KC D B. Recent advances in sequence-based protein structure prediction[J]. Briefings in Bioinformatics, 2016, 18(6): 1021-1032. |
| [3] |
UPADHYAY V P, PANWAR S, MERUGU R.Protein sequence structure prediction using artificial intelligent techniques[C]//Proceedings of the International Conference on Advances in Information Communication Technology & Computing.Bikaner, India: ACM, 2016.
|
| [4] |
ROY A, KUCUKURAL A, ZHANG Y. I-TASSER:A unified platform for automated protein structure and function prediction[J]. Nature Protocols, 2010, 5(4): 725-738. DOI:10.1038/nprot.2010.5 |
| [5] |
YANG J, ZHANG W, HE B, et al. Template-based protein structure prediction in CASP11 and retrospect of I-TASSER in the last decade[J]. Proteins, 2016, 84. |
| [6] |
BAI L, YANG L.A unified deep learning model for protein structure prediction[C]//20173rd IEEE International Conference on Cybernetics (CYBCONF).Exeter, UK: IEEE, 2017: 1-6.
|
| [7] |
YANG L, LIN B, PAN J, et al.Indirect method-potential theory in the harmonic transformation model[C]//20173rd IEEE International Conference on Cybernetics (CYBCONF).Exeter, UK: IEEE, 2017: 1-6.
|
| [8] |
BAI L, CHEN Q. Visual phrase recognition by modeling 3D spatial context of multiple objects[J]. Neurocomputing, 2017, 253(C): 183-192. |
| [9] |
REMMERT M, BIEGERT A, HAUSER A, et al. HHblits:Lightning-fast iterative protein sequence searching by HMM-HMM alignment[J]. Nature Methods, 2012, 9(2): 173-175. DOI:10.1038/nmeth.1818 |
| [10] |
LIN Z, LANCHANTIN J, QI Y.MUST-CNN: A multilayer shift-and-stitch deep convolutional architecture for sequence-based protein structure prediction[C]//AAAI'16 Proceedings of the Thirtieth AAAI Conference on Artificial Intelligence.Phoenix, Arizona: AAAI Press, 2016: 27-34.
|
| [11] |
ESWAR N, ERAMIAN D, WEBB B, et al. Protein structure modeling with MODELLER[M]. Switzerland AG: Springer Nature, 2008: 145-159.
|
| [12] |
WU S, SZILAGYI A, ZHANG Y. Improving protein structure prediction using multiple sequence-based contact predictions[J]. Structure, 2011, 19(8): 1182-1191. DOI:10.1016/j.str.2011.05.004 |
| [13] |
ZAHIRI J, YAGHOUBI O, MOHAMMAD-NOORI M, et al. PPIevo:Protein-protein interaction prediction from PSSM based evolutionary information[J]. Genomics, 2013, 102(4): 237-242. DOI:10.1016/j.ygeno.2013.05.006 |
| [14] |
HE Y, RACKOVSKY S, YIN Y, et al. Alternative approach to protein structure prediction based on sequential similarity of physical properties[J]. PNAS, 2015, 112(16): 5029-5032. DOI:10.1073/pnas.1504806112 |
| [15] |
BLASZCZYK M, JAMROZ M, KMIECIK S, et al. CABS-fold:Server for the de novo and consensus-based prediction of protein structure[J]. Nucleic Acids Research, 2013, 41(Web Server issu): W406-W411. |
| [16] |
EICKHOLT J, CHENG J. Predicting protein residue-residue contacts using deep networks and boosting[J]. Bioinformatics, 2012, 28(23): 3066-3072. DOI:10.1093/bioinformatics/bts598 |
| [17] |
DI L P, NAGATA K, BALDI P. Deep architectures for protein contact map prediction[J]. Bioinformatics, 2012, 28(19): 2449-2457. DOI:10.1093/bioinformatics/bts475 |
| [18] |
BAI L, LI K.Predicting image caption by a unified hierarchical model[C]//2015 IEEE International Conference on Multimedia and Expo (ICME).Turin, Italy: IEEE, 2015: 1-6.
|
| [19] |
BAI L, LI K, PEI J, et al. Main objects interaction activity recognition in real images[J]. Neural Computing and Applications, 2016, 27(2): 335-348. |
| [20] |
KARIM R, AZIZ M M A, SHATABDA S, et al.A novel and effective scoring scheme for structure classification and pairwise similarity measurement[J]. arXiv preprint arXiv: 1610.01052, 2016.
|
| [21] |
YANG Y, ZHAN J, ZHAO H, et al. A new size-independent score for pairwise protein structure alignment and its application to structure classification and nucleic-acid binding prediction[J]. Proteins:Structure Function and Bioinformatics, 2012, 80(8): 2080-2088. |
| [22] |
ZHANG L, BAILEY J, KONAGURTHU A S, et al. A fast indexing approach for protein structure comparison[J]. BMC Bioinformatics, 2010, 11(1): S46. DOI:10.1186/1471-2105-11-46 |
| [23] |
FOX N K, BRENNER S E, CHANDONIA J M. SCOPe:Structural classification of proteins-extended, integrating SCOP and ASTRAL data and classification of new structures[J]. Nucleic Acids Research, 2013, 42(D1): D304-D309. |



