包括动物、植物及低等单细胞真核生物在内的大多数真核生物为多倍体(Polyploid),即一个细胞内包含多条染色体。近年来,关于真核生物多倍性的产生机制及基因组生物学方面的研究已取得了相当大的进展[1-4],但关于多倍性是否对真核生物的成功进化有积极效应仍未知[5]。
与真核生物相比,长时间以来,人们一直认为原核生物(细菌和古细菌)为典型的单倍体(Monoploid),意味着在绝大多数生长周期内其单个细胞只含有单条染色体。该概念甚至还出现在目前有的教科书中[6]。但是,最近十几年来,越来越多的研究发现多倍性实际上也相当广泛地存在于细菌和古细菌中[7-16]。这些物种主要从属于变形菌门(Proteobacteria)、蓝藻细菌门(Cyanobacteria)、异常球菌-栖热菌门(Deinococcus-Thermus)以及广古菌门(Euryarchaeota)。在这些物种中,有的基因组的拷贝数是2~9条(寡倍性Oligoploid),有的是10条或以上(多倍性),有的甚至达到了上百、上千条, 如巨型的内共生细菌Epulopiscium sp.[16]。因此,与先前的观点恰好相反,目前对原核生物的倍性研究结果表明寡倍体或多倍体的细菌比单倍体细菌更加常见[17-19],实际上只有少数细菌如枯草芽孢杆菌(Bacillus subtilis)、新月柄杆菌(Caulobacter crescentus)以及处于缓慢生长时期的大肠杆菌(Escherichia coli)才是真正的单倍体[20]。多倍体细菌广泛分布于原核生物系统发育树上,但是在同一个属的不同种之间,倍性水平却可能有很大差异,因此,细菌多倍性的产生应该是在细菌进化过程中环境选择压力的结果,即多倍性拥有单倍性无法比拟的进化优势[17]。本文重点总结目前国内外学者实验研究所获得的关于多倍体细菌和古细菌的生长及生存的6点优势,为多倍体原核生物的研究提供参考依据。
1 多倍体原核生物具有高的同源重组率耐辐射奇球菌(Deinococcus radiodurans)是最早被鉴定为寡倍体的细菌,其单个细胞含有5~8条染色体[21]。研究发现该菌对X射线及干燥的环境的抵抗性比单倍体细菌高出极多[22-23]。与其相似,多倍体古细菌如嗜盐杆菌(Halobacterium salinarum)也被发现对能引起DNA双链断裂的环境(如X射线)具有极强抵抗力[24]。在对以上两个微生物所进行的实验中,染色体都被X射线辐射断裂成了无数个片段,但是超乎寻常的是,这些断裂的染色体片段能迅速地重新组装成完整的染色体。在进一步的实验中,通过对H.salinarum施加更高强度的X射线,从而筛选到了抗辐射能力更强的突变株。在该突变株中,两个单链DNA结合蛋白基因(ssB)被发现重度上调表达。由此证明,该突变株能在高强度的辐射环境中生存下来,是因为在其细胞内发生了超高效的同源重组[25]。与此一致,在多倍体的沃氏嗜盐富饶菌(Haloferax volcanii)中过量表达rpaC基因(单链DNA结合蛋白基因同源物),其基因组DNA抗毁坏的能力大幅度增强[26];另外,在D.radiodurans中,ssB基因的表达水平直接与菌体抗辐射水平呈正相关[27]。以上的研究结果均证明,寡倍性或多倍性能为基因组提供高效率的同源重组功能,从而赋予原核生物更强的抗逆性能力(即基因组DNA的抗毁坏能力)。
2 多倍体原核生物拥有低的自发突变率某个基因位点在单个细胞的多条染色体上存在,称为基因冗余性(Gene redundancy)。这意味着,在特殊情况下,该位点可拥有至少2个等位基因,从而形成杂合细胞(Heterozygous cells)。实验证明,杂合细胞确实存在于多倍体原核生物中,如H.volcanii的突变体可在缺乏色氨酸和亮氨酸的培养基上生长,说明该突变体细胞中实际上还存在能表达这2种氨基酸合成途径中的关键酶的野生型等位基因[28]。杂合细胞也被发现存在于几种多倍体蓝藻细菌中[29-31]。属于栖热菌属(Thermus)的噬热栖热细菌(Thermus thermophilus)也被鉴定为寡倍体,Ohtani等[11]及笔者的研究(数据已提交发表)表明,在有适当筛选条件的存在下,染色体上的2个等位基因可稳定地存在于杂合细胞中。这些研究结果说明多倍性为原核生物提供基因冗余性。
基因冗余性能使原核生物自发突变率大幅度降低。例如,与单倍体古细菌相比,H.volcanii的pyrE基因的自发突变率能低数个数量级[32]。这么低的自发突变率正是基因冗余性造成的,即突变的等位基因与野生型的等位基因同时存在,从而野生型等位基因信息能被利用于修复突变的等位基因。可以预料,自然生长的原核细菌或者古细菌,经常会遇到一些不利的生长环境,由基因组多倍性产生的杂合突变型细胞将拥有比纯合的野生型细胞更强的适应能力。
3 多倍性赋予原核生物在极端环境下生存的能力辐射、干燥、高温、高渗透压等环境因素对原核生物的生存是极大威胁。而有些原核生物经过了长期的进化,最终在这些极端环境中得以生存下来,因此被称为极端微生物。例如,耐辐射奇球菌(D.radiodurans)能在极度干燥与强电离辐射的环境中生存[21-23];噬热栖热菌(T.thermophilus)能在高达85℃的温度下生存[11]。越来越多的噬盐古菌(Haloarchaea)被报道分离自从地质时代就产生的沉积盐中,说明这些古菌已在这种高渗环境下生存了上万年[33-37];Wierzchos等[38]报道噬盐古菌不仅能在沉积盐中生存,还能在极度缺水的沙漠岩石上生存;Fendrihan等[39]将噬盐球菌(Halococcus dombrowskii)置于模拟的火星紫外射线下辐射,其生存的概率比在被盐晶体包被的液体中的还大。另外,有研究发现相当一部分Halococcus sp.细胞能在太空中生存14 d[40]。有趣的是,以上列举的这些极端原核生物均为多倍体[17],可以说明正是由于多倍性,这些微生物才能在极端环境下生存,最可能的原因是多倍体具有高效的双链DNA断裂修复系统,从而使被环境破坏的基因组DNA得以迅速修复(见上文述)。
4 多倍体原核生物不需要严格地控制染色体分离及细胞分裂理论上,大部分的细菌和古细菌的细胞周期如下:核DNA复制一次,同时细胞质量增加一倍并在细胞正中间产生隔膜,随后复制的姐妹基因组DNA转移到细胞末端,细胞在细胞隔膜处分裂,复制的2个姐妹DNA分别分离到2个子细胞中(图 1)。在单倍体原核生物中,细胞周期必须精确严格控制,以确保每个子细胞都能得到母细胞基因组DNA,因为缺失母细胞基因组DNA的细胞即无核细胞没有生长优势,最终会死亡。与此相反,理论上,在多倍体原核生物中,这个过程可以不必要严格控制,因为子细胞得不到母细胞基因组DNA的概率是极低的(图 1)。事实上,产生不同大小以及不同DNA含量的子细胞对多倍体原核生物似乎更加有利,因为可形成群体多样性,以利于适应不同的生存环境。目前,已经有多项研究证明,在多倍体细菌或者古细菌中,染色体能随机分离到子细胞中。例如,詹氏甲烷球菌(Methanocaldococcus jannaschii)不需要严格控制DNA子细胞分离和细胞分裂过程[41]。在其指数生长时期,细胞群体的平均染色体拷贝数在3~15内变化,而在稳定生长时期,平均每个细胞只含有2~4条染色体。与DNA含量的上下浮动相对应的是,在指数生长时期,细胞分裂通常是不均等的,即细胞隔膜不是在预分裂的母细胞的正中间产生的。不需要严格进行DNA分离及细胞分裂的现象也在一些细菌中发现,如多倍体的蓝藻菌Synechocystis sp.PCC6803以及Anabaena sp.PCC 7120[42-43]。值得提出的是,多倍性只是允许原核生物不需要严格的细胞分裂及染色体分离机制,并不是强制其遵循这一机制。如嗜盐杆菌H.salinarum及另外一种蓝藻菌即细长聚球藻(Synechococcus elongatus)能进行对称的、精确的DNA子细胞分离[8, 44]。
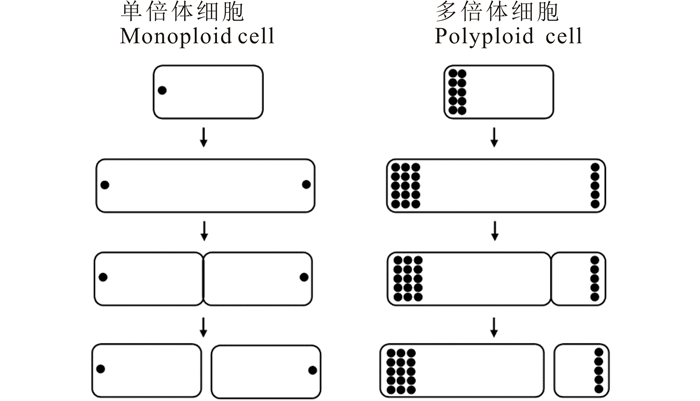
|
单倍体细胞进行严谨的染色体分离与细胞分裂,多倍体细胞进行随机的染色体分离与细胞分裂;黑色圆点代表染色体 Stringent and random chromosome segregations and cell divisions are carried out in the monoploid and polyploid cells, respectively; Black-filled circles represent chromosomes 图 1 单倍体原核生物与多倍体原核生物的单个细胞周期示意图 Fig.1 Schematic drawings of the cell cycles in monoploid and polyploid prokaryotes |
在H.salinarum中,处于对数生长时期的细胞染色体拷贝数明显多于处于稳定生长时期的细胞染色体拷贝数[8]。相似的情况也发生在另一种古细菌即地中海富盐菌(Haloferax mediterranei)中[45],以及一些多倍体蓝藻菌中,如对数生长时期的Synechocystis sp.PCC6803含有高达218条染色体,而稳定生长时期的细胞只含有58条染色体[46]。另外,笔者的研究也显示T.thermophilus的染色体拷贝数也是随着生长环境变化而变化的(数据未发表)。以上研究说明多倍体细菌或古细菌的拷贝数并非固定不变,而是随着细胞群体的生长状态或者生长条件相应被调节。由此可见,在多倍体原核生物中,在无外界调节因素的情况下,某个复制子上基因的总体表达水平取决于其拷贝数水平,也就是说,多倍体细菌或古细菌具备通过调节复制单位的拷贝数来整体性地控制基因表达水平的生理基础,这一优势是单倍性无法给予的。当然,多倍体原核生物是否采用该机制来控制基因表达及其具体机理,还需要大量后续实验数据来验证。
6 多倍体原核生物的基因组DNA可能为细胞生长提供能源物质以上几点论述均从遗传学角度论证多倍性的优势,近期,一个非遗传学方面的优势也被提出,即在多倍体原核生物中,其本身的基因组DNA可能为细胞生长提供碳源、氮源以及磷源。目前,该观点已在H.volcanii中得到证实。Zerulla等[47]首先发现H.volcanii可在只添加外源基因组DNA的培养基上生长,说明其能利用基因组DNA作为能源物质。进一步,该研究组发现H.volcanii还能在未添加任何磷酸盐的培养基上生长,说明细胞本身能为其生长提供磷源。在此培养基上生长的细胞中染色体的拷贝数从30降到了2,与此同时,细胞内核糖体的数目却保持不变,证明提供细胞生长的磷酸盐来源于DNA而非RNA。这些染色体DNA被降解释放出磷酸盐为其他生物分子如ATP、NADP+、磷脂、磷蛋白等提供磷酸基团,从而维持细胞持续生长。其他多倍体细菌或古细菌是否也能像H.volcanii一样能将基因组DNA作为磷酸盐储存聚合物,还待后续的研究揭示。
7 展望近十年来,聚倍性或多倍性被陆续发现于多种原核生物中。目前研究学者们已分析的多倍体细菌或古细菌多数是在实验室环境中被培养过数十年的,或许会引起相关质疑,即多倍性不是原核生物自然的属性。但是多倍性也被发现于原核生物的新鲜分离物中,甚至是无法实现实验室培养的原核生物中[37],因此,多倍体原核生物在自然界中应该是广泛存在的。本文阐述了多倍性赋予原核生物生存和生长的优势,所论述6项中的5项已至少在一个物种中得到实验验证(表 1)。其中,最突出的优势是高效的DNA修复功能以及极低的自发突变率,也正是由于这些特性的存在,多倍体细菌或古细菌能在极端环境下长期生存。关于多倍体细菌或古细菌是否能通过调节拷贝数来整体性地控制基因表达水平,目前还处于理论假设阶段,但不是没有可能性,在单倍体细胞中,基因的表达调控完全是靠转录因子结合到相关的操纵序列上进行调节的,属于单个随机事件;但在多倍体细胞中,总体基因的表达水平是需要遵循复制子的拷贝数水平的,因此,该调节方式更具有统一性[17]。
| 表 1 多倍体细菌及古细菌的生存及生长优势 Table 1 The survival and growth advantages of polyploid bacteria and archaea |
总结起来,关于多倍体原核生物的研究虽然已取得了一些进展,但内在的生理调节机制仍未知。比如,不同生长的生长状态、不同的生长条件下,原核生物基因组拷贝数不同的影响因素和调控机制;同一原核生物细胞中,不同的复制单位具有不同的拷贝数变化的成因;对极端环境的生存与生长优势是否为多倍体原核生物共有的现象;等等。这些问题都应该是多倍体原核生物的下一步研究方向。
| [1] |
CHOLEVA L, JANKO K. Rise and persistence of animal polyploidy:Evolutionary constraints and potential[J]. Cytogenet Genome Res, 2013, 140(2/3/4): 151-170. |
| [2] |
WEISS-SCHNEEWEISS H, EMADZADE K, JANG T S, et al. Evolutionary consequences, constraints and potential of polyploidy in plants[J]. Cytogenet Genome Res, 2013, 140(2/3/4): 137-150. |
| [3] |
HARVEY A C, FJELLDAL P G, SOLBERG M F, et al. Ploidy elicits a whole-genome dosage effect:Growth of triploid Atlantic salmon is linked to the genetic origin of the second maternal chromosome set[J]. BMC Genetics, 2017, 18: 34. |
| [4] |
PERUZZI S, PUVANENDRAN V, RIESEN G, et al. Growth and development of skeletal anomalies in diploid and triploid Atlantic salmon (Salmo salar) fed phosphorus-rich diets with fish meal and hydrolyzed fish protein[J]. PLoS One, 2018, 13(3): e0194340. DOI:10.1371/journal.pone.0194340 |
| [5] |
MADLUNG A. Polyploidy and its effect on evolutionary success:Old questions revisited with new tools[J]. Heredity (Edinb), 2013, 110(2): 99-104. DOI:10.1038/hdy.2012.79 |
| [6] |
MADIGAN M T, MARTINKO J. Brock biology of micro-organisms[M]. San Francisco, USA: Benjamin-Cummings Publishing Company, 2012.
|
| [7] |
KOMAKI K, ISHIKAWA H. Genomic copy number of intracellular bacterial symbionts of aphids varies in response to developmental stage and morph of their host[J]. Insect Biochemistry and Molecular Biology, 2000, 30(3): 253-258. DOI:10.1016/S0965-1748(99)00125-3 |
| [8] |
BREUERT S, ALLERS T, SPOHN G, et al. Regulated polyploidy in halophilic archaea[J]. PLoS ONE, 2006, 1: e92. DOI:10.1371/journal.pone.0000092 |
| [9] |
MICHELSEN O, HANSEN F G, ALBRECHTSEN B, et al. The MG1363 and IL1403 laboratory strains of Lactococcus lactis and several dairy strains are diploid[J]. J Bacteriol, 2010, 192(4): 1058-1065. DOI:10.1128/JB.00900-09 |
| [10] |
TOBIASON D M, SEIFERT H S. Genomic content of Neisseria specie[J]. J Bacteriol, 2010, 192(8): 2160-2168. DOI:10.1128/JB.01593-09 |
| [11] |
OHTANI N, TOMITA M, ITAYA M. An extreme thermophile, Thermus thermophilus, is a polyploid bacterium[J]. J Bacteriol, 2010, 192(20): 5499-5505. DOI:10.1128/JB.00662-10 |
| [12] |
GRIESE M, LANGE C, SOPPA J. Ploidy in cyanobacteria[J]. FEMS Microbiol Lett, 2011, 323(2): 124-131. DOI:10.1111/fml.2011.323.issue-2 |
| [13] |
HILDENBRAND C, STOCK T, LANGE C, et al. Genome copy numbers and gene conversion in methanogenic archaea[J]. J Bacteriol, 2011, 193(3): 734-743. DOI:10.1128/JB.01016-10 |
| [14] |
PECORARO V, ZERULLA K, LANGE C, et al. Quantification of ploidy in proteobacteria revealed the existence of monoploid, (mero-) oligoploid and polyploid species[J]. PLoS ONE, 2011, 6(1): e16392. DOI:10.1371/journal.pone.0016392 |
| [15] |
SPAANS S K, OOST J V D, KENGEN S W. The chromosome copy number of the hyperthermophilic archaeon Thermococcus kodakarensis KOD1[J]. Extremophiles, 2015, 19(4): 741-750. DOI:10.1007/s00792-015-0750-5 |
| [16] |
MIYAKE S, NGUGI D K, STINGL U. Phylogenetic diversity, distribution, and cophylogeny of giant bacteria (Epulopiscium) with their surgeonfish hosts in the red sea[J]. Front Microbiol, 2016, 7: 285. |
| [17] |
ZERULLA K, SOPPA J. Polyploidy in haloarchaea:Advantages for growth and survival[J]. Front Microbiol, 2014, 5: 274. |
| [18] |
STAN-LOTTER H, FENDRIHAN S. Halophilic archaea:Life with desiccation, radiation and oligotrophy over geological times[J]. Life (Basel), 2015, 5(3): 1487-1496. |
| [19] |
MAURER S, LUDT K, SOPPA J. Characterization of copy number control of two Haloferax volcanii replication origins using deletion mutants and haloarchaeal artificial chromosomes[J]. J Bacteriol, 2017, 200(1): e00517-17. DOI:10.1128/JB.00517-17 |
| [20] |
BREMER H, DENNIS P P. Modulation of chemical composition and other parameters of the cell at different exponential growth rates[J]. Eco Sal Plus, 2008, 3(1). |
| [21] |
HANSEN M T. Multiplicity of genome equivalents in the radiation-resistant bacterium Micrococcus radiodurans[J]. J Bacteriol, 1978, 134(1): 71-75. |
| [22] |
ZAHRADKA K, SLADE D, BAILONE A, et al. Reassembly of shattered chromosomes in Deinococcus radiodurans[J]. Nature, 2006, 443(7111): 569-573. |
| [23] |
SLADE D, LINDNER A B, PAUL G, et al. Recombination and replication in DNA repair of heavily irradiated Deinococcus radiodurans[J]. Cell, 2009, 136(6): 1044-1055. DOI:10.1016/j.cell.2009.01.018 |
| [24] |
KOTTEMANN M, KISH A, ILOANUSI C, et al. Physiological responses of the halophilic archaeon Halobacterium sp.strain NRC1 to desiccation and gamma irradiation[J]. Extremophiles, 2005, 9(3): 219-227. DOI:10.1007/s00792-005-0437-4 |
| [25] |
EVANS J J, GYGLI P E, MCCASKILL J, et al. Divergent roles of RPA homologs of the model archaeon Halobacterium salinarum in survival of DNA damage[J]. Genes (Basel), 2018, 9(4): 223. DOI:10.3390/genes9040223 |
| [26] |
SKOWYRA A, MACNEILL S A. Identification of essential and non-essential single-stranded DNA-binding proteins in a model archaeal organism[J]. Nucleic Acids Research, 2012, 40(3): 1077-1090. DOI:10.1093/nar/gkr838 |
| [27] |
AGAPOV A A, KULBACHINSKIY A V. Mechanisms of stress resistance and gene regulation in the radioresistant bacterium Deinococcus radiodurans[J]. Biochemistry (Moscow), 2015, 80(10): 1201-1216. DOI:10.1134/S0006297915100016 |
| [28] |
LANGE C, ZERULLA K, BREUERT S, et al. Gene conversion results in the equalization of genome copies in the polyploid haloarchaeon Haloferax volcanii[J]. Molecular Microbiology, 2011, 80(3): 666-677. DOI:10.1111/mmi.2011.80.issue-3 |
| [29] |
SPENCE E, BAILEY S, NENNINGER A, et al. A homolog of Albino3/OxaⅠ is essential for thylakoid biogenesis in the cyanobacterium Synechocystis sp.PCC6803[J]. J Biol Chem, 2004, 279(53): 55792-55800. DOI:10.1074/jbc.M411041200 |
| [30] |
TAKAHAMA K, MATSUOKA M, NAGAHAMA K, et al. High-frequency gene replacement in cyanobacteria using a heterologous rps 12 gene[J]. Plant Cell Physiol, 2004, 45(3): 333-339. |
| [31] |
WANG Y, GAO Y, LI C, et al. Three substrains of the cyanobacterium Anabaena sp.strain PCC 7120 display divergence in genomic sequences and hetC function[J]. J Bacteriol, 2018, 200(13): e00076-18. |
| [32] |
MACKWAN R R, CARVER G T, DRAKE J W, et al. An unusual pattern of spontaneous mutations recovered in the halophilic archaeon Haloferax volcanii[J]. Genetics, 2007, 176(1): 697-702. |
| [33] |
FENDRIHAN S, LEGAT A, PFAFFENHUEMER M, et al. Extremely halophilic archaea and the issue of long-term microbial survival[J]. Rev Environ Sci Biotechnol, 2006, 5: 203-218. DOI:10.1007/s11157-006-0007-y |
| [34] |
PARK J S, VREELAND R H, CHO B C, et al. Haloarchaeal diversity in 23, 121 and 419 MYA salts[J]. Geobiology, 2009, 7(5): 515-523. DOI:10.1111/j.1472-4669.2009.00218.x |
| [35] |
WINTERS Y D, LOWENSTEIN T K, TIMOFEEFF M N. Starvation-survival in haloarchaea[J]. Life (Basel), 2015, 5(4): 1587-1609. |
| [36] |
GIBTAN A, PARK K, WOO M, et al. Diversity of extremely halophilic archaeal and bacterial communities from commercial salts[J]. Front Microbiol, 2017, 8: 799. DOI:10.3389/fmicb.2017.00799 |
| [37] |
JAKKOLA S T, ZERULLA K, GUO Q, et al. Halophilic archaea cultivated from surface sterilized middle-late eocene rock salt are polyploid[J]. PLoS One, 2014, 9(10): e110533. DOI:10.1371/journal.pone.0110533 |
| [38] |
WIERZCHOS J, DE LOS RÍOS A, ASCASO C. Microorganisms in desert rocks:The edge of life on earth[J]. Int Microbiol, 2012, 15(4): 173-183. |
| [39] |
FENDRIHAN S, BÉRCES A, LAMMER H, et al. Inverstigating the effects of simulated martian ultravialett radiation on Halococcus dombrowskii and other extremely halophilic archaeabacteria[J]. Astrobiology, 2009, 9(1): 104-112. DOI:10.1089/ast.2007.0234 |
| [40] |
SCHMIDT S K, GENDRON E M S, VINCENT K, et al. Life at extreme elevations on Atacama volcanoes:The closest thing to Mars on Earth?[J]. Antonie van Leeuwenhoek, 2018, 111(8): 1389-1401. DOI:10.1007/s10482-018-1066-0 |
| [41] |
BISSON-FILHO A W, ZHENG J, GARMER E. Archaeal imaging:Leading the hunt for new discoveries[J]. Mol Biol Cel, 2018, 29(13): 1675-1681. |
| [42] |
SCHNEIDER D, FUHRMANN E, SCHOLZ I, et al. Fluorescence staining of live cyanobacterial cells suggest non-stringent chromosome segregation and absence of a connection between cytoplasmic and thylakoid membranes[J]. BMC Cell Biology, 2007, 8: 39. DOI:10.1186/1471-2121-8-39 |
| [43] |
HU B, YANG G, ZHAO W. MreB is important for cell shape but not for chromosome segregation of the filamentous cyanobacterium Anabaena sp.PCC7120[J]. Mol Microbiol, 2007, 63(6): 1640-1652. DOI:10.1111/j.1365-2958.2007.05618.x |
| [44] |
JAIN I H, VIJAYAN V, O'SHEA E K. Spatial ordering of chromosomes enhances the fidelity of chromosome partitioning in cyanobacteria[J]. Proc Natl Acad Sci USA, 2012, 109(34): 13638-13643. DOI:10.1073/pnas.1211144109 |
| [45] |
YANG H B, WU Z F, LIU J F, et al. Activation of a dormant replication origin is essential for Haloferax mediterranei lacking the primary origins[J]. Nat Commun, 2015, 6: 8321. DOI:10.1038/ncomms9321 |
| [46] |
ZERULLA K, LUDT K, SOPPA J. The ploidy level of Synechocystis sp.PCC6803 is highly variable and is influenced by growth phase and by chemical and physical external parameters[J]. Microbiology, 2016, 162(5): 730-739. DOI:10.1099/mic.0.000264 |
| [47] |
ZERULLA K, CHIMILESKI S, NÄTHER D, et al. DNA as a phosphate storage polymer and the alternative advantages of polyploidy for growth or survival[J]. PLoS ONE, 2014, 9(4): e94819. DOI:10.1371/journal.pone.0094819 |
 2018, Vol. 25
2018, Vol. 25 


