【研究意义】放线菌可以产生许多有用的次生代谢物质,广泛应用的抗生素约70%是各种放线菌所产生,一些种类的放线菌还能产生各种酶制剂(蛋白酶、淀粉酶和纤维素酶等)、维生素(B12)和有机酸等[1]。因此,放线菌与人类关系密切,在医药工业上有重要意义,研究放线菌及其代谢网络以发现更多的次级代谢产物仍是当前的研究热点之一。链霉菌中某一特定次生代谢途径相关的基因通常以基因簇(Cluster)的形式存在线状染色体上,链霉菌基因组富含次生代谢物合成基因簇,典型的次生代谢途径基因簇长度约为15~120 kb[2],通常还包含调控和抗性基因,仅极少数抗性基因甚至合成基因不在同一基因簇中。大部分的生物合成基因簇表达不太强,产生的次生代谢产物不足以用现代的分析方法检测到。基于此,通常需要通过代谢工程手段以提高次生代谢物的产量[3]。放线菌属的生物活性物质基因簇的表达严格受复杂的调节网络调控,一种链霉菌中往往存在多条代谢途径均能利用的共同前体[4]。因此,可以敲除或失活那些竞争共同前体的代谢途径基因簇,或降低其表达水平,使共同前体代谢物尽可能多地“流经”目标产物代谢途径,提高目标产物的相对产量。这种敲除非目的代谢途径或解除限速代谢途径调控抑制作用,以提高目标代谢物产量的方法是利用基因组信息改造菌株的最直接方法。同时,通过对次级代谢生物合成调控基因的应用和调控机制的研究,能够明确其调节机制,进而提高次生代谢物的产量,有利于加快药物的发现及生产,为改良抗生素生产菌和创造新的药物提供重要的理论依据[5]。landomycin E(laE)是一种具有很强的抗肿瘤活性的抗生素分子,是一种具有重要应用和基础研究价值的天然产物。链霉菌属放线菌Streptomyces globisporus能产laE,但产量低,需进一步对影响其产量的代谢网络进行研究以提高次生代谢物的产量。【前人研究进展】 2001年7月,链霉菌属的代表种天蓝色链霉菌(Streptomyces coelicolor)的全基因组测序在英国剑桥Sanger中心完成,这是放线菌中第一个完成全基因组测序的种。该属菌的基因组测序分析表明,放线菌比预期含有更为丰富的能合成有生物活性的次级代谢物的资源。虽然有些放线菌基因组已经测序,但探索完成这些细菌的生物合成代谢途径不是一件简单的事情。到目前为止,国内外对laE的相关研究报道非常少,国内几乎没有查到相关文献。国外研究者Lilya Horbal通过构建含Tn5转座子的突变系统并将其整合至链霉菌S.globisporus 1912中,所获得的突变株的laE产量发生明显改变,有的降低,有的提高至3倍左右,但其产量依然很低。放线菌S.globisporus的部分参与生物合成laE的调节基因簇已经被测序,且部分基因的功能已经被确定[6-9],但其产laE的生物合成途径和代谢网络的调节机理还需进一步深入研究,如通过研究不同突变基因对laE产量的影响,识别影响laE产量的调节因子的功能。Tn7转座基因整合对宿主菌的功能基因产生插入失活作用,对目标菌代谢不产生有害突变,并且可产生很高的突变率,其重组几率平均可达到79%。突变后经过简单的测序就可知道整合突变位点的位置。放线菌的次级代谢产物的合成途径和调节网络相对比较复杂而庞大,很难通过理性手段对代谢途径的某一环节进行修饰,而通过随机突变的手段通常能克服这一困难。【本研究切入点】本研究在转座子Tn5的基础上进行优化创新,利用美国Johns Hopkins University新发展的Tn7转座子遗传重组法构建转座子整合突变系统。【拟解决的关键问题】本研究利用转座子Tn7为基础构建带有强启动子的转座子系统,并整合至S.globisporus染色体产生突变库,从中筛选比原始菌株产量高的突变株,并分析基因失活对次级代谢物产量的影响。
1 材料与方法 1.1 材料 1.1.1 菌种和质粒E.coli DH5α用于常规克隆,E.coil ET2567/pUB307用于转移性供体菌,质粒pAcDZ1、pUC57TetRA均为本实验室保存,转座子质粒pGRG25为周礼芹博士惠赠,菌株Streptomyces globisporus(DSM 40047)购自德国菌种DSMZ保藏中心。
1.1.2 酶和主要试剂DNA聚合酶、T4 DNA连接酶、限制性内切酶EcoRⅠ、XhoⅠ等均购自TaKaRa公司,阿泊拉霉素(Apramycin)、卡那霉素、氨苄、红霉素和X-gal等购自上海生物工程有限公司,标样landomycin E购自Sigma公司,凝胶回收试剂盒、纯化试剂盒、质粒提取试剂盒为天根生化科技(北京)有限公司产品。
1.1.3 培养基LB培养基(g/L):酵母粉5,胰蛋白胨10,NaCl 10,pH值7.0,添加2%琼脂为固体培养基。
SG培养基(g/L):葡萄糖4.0,酵母粉4.0,麦芽浸膏10.0,CaCO3 2.0,Agar 12.0 g,pH值7.2。液体培养基除去CaCO3。
燕麦培养基(g/L):燕麦粉20,琼脂18,七水合硫酸亚铁0.001,四水合氯化亚锰0.001,七水合硫酸锌0.001,pH值7.3±0.2。
1.2 方法 1.2.1 DNA操作链霉菌基因组和大肠杆质粒DNA的提取、克隆等参考文献[10],DNA限制性酶切及连接等分子生物学试剂的使用参考供应商的使用说明。
1.2.2 带有链霉菌表达框架的Tn7转座子的构建以XbaⅠ酶切质粒pUC57TetRA得到含tcp830启动子的DNA片段,克隆至经同样酶切的质粒pGRG25,得到质粒pGRG-P;以双酶KpnⅠ-EcoRⅠ从质粒pUC57TetRA切下含红霉素抗性启动子基因ermEp1的DNA片段,克隆至经同样双酶切的质粒pGRG-P,得到质粒pGREry;用PstⅠ和KpnⅠ双酶切质粒pKC1218,分离回收大小为1.3 Kb的DNA片段,克隆至经同样双酶切的质粒pGREry,得到质粒pGRTEA,用于链霉菌的转座突变。
1.2.3 细胞转化和培养条件接种受体菌S.globisporus至5 mL含SG液体培养基的指型瓶中,30℃振荡培养约20 h。接种供体菌E.coil ET2567/pUB307至5 mL LB+Km液体培养基中,37℃振荡培养约16 h。用同一个1.5 mL离心管收集适量供体菌和受体菌,12 000 r/min离心30 s,弃上清,用移液器轻轻吹打将菌泥悬浮混匀后移入SG平板中央,28℃正置培养过夜。液体SG培养基用于转座突变后菌株培养发酵产LaE,原始菌株和突变株用100 mL SG液体培养基于30℃振荡培养48 h。
1.2.4 突变文库的构建和产LaE菌株的预筛选利用转座载体pGRTEA在链霉菌体内转座子突变(in vivo transposon mutagenesis)产生突变库。由于产laE的菌株在燕麦平板上显橘黄色或棕色,因此,可通过平板预筛选获得产生LaE的突变株,初步构建突变库。由于laE的最大吸收峰在450 nm,突变株和野生菌株用SG液体培养基培养48 h后,培养液用乙酸乙酯进行抽提,用分光光度计(BECKMAN DU800)于450 nm测定其吸收值OD450,计算laE含量。
1.2.5 突变株产LaE的HPLC分析为了定性定量分析链霉菌S.globisporus的laE产量,将上述乙酸乙酯抽提液重新溶解于甲醇,获得样品用戴安Utimat3000HPLC进行分析,分析柱是Hewlett Packard ZORBAX SB C-18(粒度5 μm,4.6×150 mm),柱温维持在30℃,进样量10 μL。A液为0.5%的乙酸溶液,B液为乙腈,以非线性梯度洗脱(浓度为20%~95%乙腈),流速为0.6 mL/min。示差检测器(Shodex),检测波长为254 nm。通过峰值计算laE含量,以标样laE作对照。
1.2.6 转座子插入位点分析根据转座子序列设计向上游和向下游的引物(TEF:5′-ACGACTACGCACTAGCCAACA-3′和TER:5′-TCGGCATGGACGAGCTGTAC-3′),以突变菌株的总DNA为模板进行PCR反应,对阳性PCR产物进行序列分析,并将测序结果与野生型菌株对照,从而确定了突变链霉菌中转座子的插入位点。
2 结果与分析 2.1 含两个外向启动子的Tn7基转座子载体pGRTEA的构建一般情况下,转座子插入会导致目标基因突变失活。因此,本研究构建的转座子含有两个反方向的启动子,ermEp1是组成型的强启动子,tcp830是诱导型的启动子,将这两个不同类型的启动子插入Tn7衍生而来的含有转座酶基因te的载体pGRG25,可使相邻的基因高效表达,获得转座载体pGRTEA(图 1),该载体含有阿泊拉抗性基因和原核复制子,将其转化链霉菌S.globisporus后,在含阿泊拉抗性平板上能生长,表明该tcp830启动子表达框架可以在链霉菌中表达(图未显示),证明转座子已经成功插入链霉菌基因组。此外,这个载体含有温度敏感型的复制子(ori pSG25),当培养温度提高至37℃以上质粒将丢失。
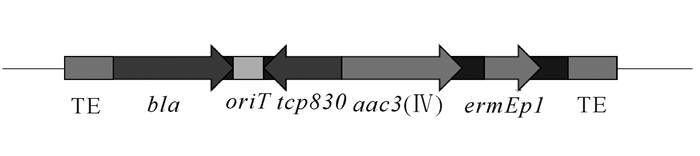
|
TE:转座酶识别序列;oriT:大肠杆菌复制子;acc 3(Ⅳ):阿伯拉霉素抗性基因;tcp830:四环素基因启动子;ermEp1红霉素基因启动子 TE:recognition sequence for transosase; oriT:origin of replication sequence in E.coli cells; acc 3 (Ⅳ):apramycin gene; tcp830: tetracycline resistence gene promoter; ermEp1: erythromycin gene promoter 图 1 经修饰的含两个启动子的Tn7转座子基载体构建框架 Fig.1 Schematic representation of the modified Tn7 transposon with two promoters |
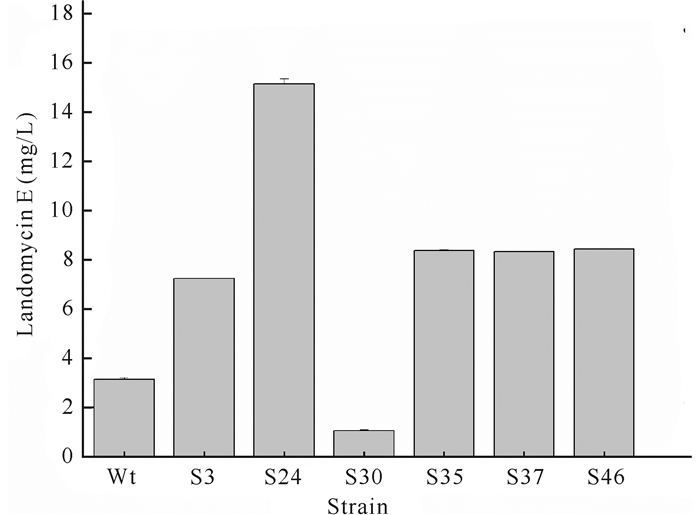
在燕麦平板上挑取显橘黄色或棕色的单菌落即为产laE的S.globisporus突变株(图 2)。先后筛选了将近1 000株的转座子突变株,获得了5株laE产量有所提高的突变株,如图 3所示,突变株S24的laE产量最高,为15.13 mg/L,是野生菌株wt的5倍;其他4株S3、S35、S37和S46的产量相近,均为8.3 mg/L左右;S30的产量最低,为1.1 mg/L,比野生菌株低。

|
(a)野生型;(b)产landomycin E的突变菌株S24 (a)Wild-type; (b)Mutant strain S24 that produce landomycin E 图 2 S.globisporus在燕麦培养基上的表型 Fig.2 Phenotyoe of S.globisporus on oatmeal agar |
由HPLC分析结果可知,抽提液的主要成分出峰时间是16.767 min,与标样landomycin E出峰时间16.787 min基本一致,因此,可确定乙酸乙酯抽提液的主要成分为landomycin E (图 4)。
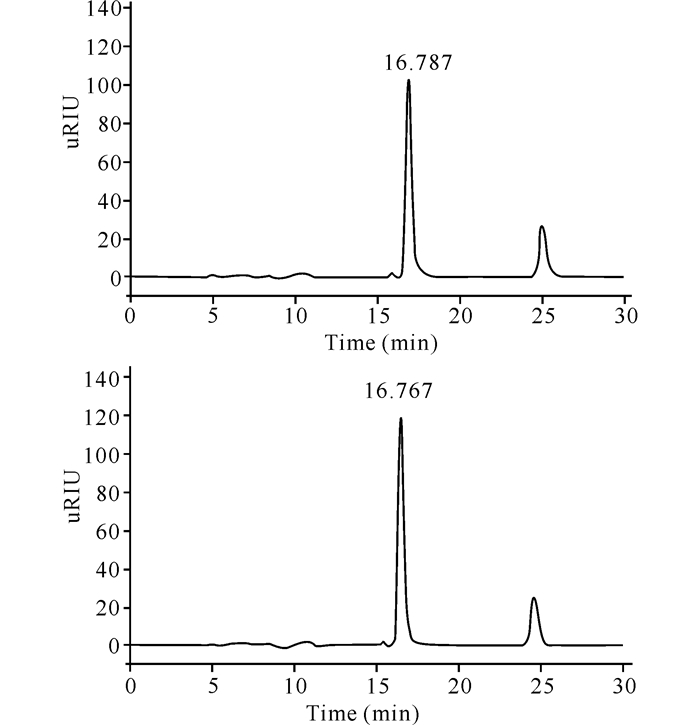
|
图 4 链霉菌S.globisporus突变株产landomycin E的HPLC分析 Fig.4 HPLC analysis of landomycin E from S.globisporus mutant strain |
将链霉菌S.globisporus突变株S24和S30的转座子双臂衔接插入区域的DNA进行克隆和DNA测序分析,以鉴定影响landomycin E合成的代谢流的关键限速步骤。突变整合插入位点的序列分析表明,突变株S24插入位点的基因序列与来自S.griseus的ZP_08237327的同源性最高,突变株S30转座子整合入S.globisporus染色体上的编码TetR家族蛋白和铜分子伴侣蛋白之间的基因。
3 讨论长久以来,次生代谢物产量的提高一般通过物理方法(如紫外线)和化学方法产生随机突变。对于传统的诱变育种,虽然能缓慢提高发酵水平,但获得的突变株品质较差,在传代和保存过程中很容易发生性状丢失,且不易了解其代谢的机制。利用基于全基因组的系统代谢网络调控策略对关键代谢节点进行合理的遗传改造,通过多途径优化的方法阻断各种竞争支路来强化laE的生物合成代谢途径,从而有效克服细胞代谢网络的刚性,实现胞内碳架物质流量分布的重大改变,有望快速获得遗传性质稳定,发酵水平提高的细胞工厂,这些研究思路已经被Lilya Horbal初步验证[3, 5],在其他细胞系统也得到了很好的验证[11-12]。
DNA转座(Transposition)是指一段DNA顺序可以从原位上单独复制或断裂下来,环化后插入另一位点,并对其后的基因起调控作用。因此,转座子在基因组分析、基因插入敲除突变的应用、基因融合表达方面是一个强有力的工具[13-16]。本研究所构建的经过修饰的微型转座子载体pGRTEA在链霉菌体内转座子突变(in vivo transposon mutagenesis)产生突变库,经过筛选获得了6株laE产量变化较大的突变株。其中突变株S24的laE产量最高(图 3),为原始菌株产量的5倍,突变整合插入位点的序列分析表明,插入位点基因与来自S.griseus的ZP_08237327的同源性最高,该基因编码GntR家族调控因子,GntR家族调控因子影响链霉菌属的表型和抗生素的产生[17-18],转座子的插入改变了该基因的功能,从而影响laE的产量。突变株S30的laE产量最低(图 3),插入位点的序列分析表明,转座子整合入S.globisporus染色体上编码TetR家族蛋白和铜分子伴侣蛋白之间的基因,转座子上的ermEp1启动子插入TetR蛋白抑制因子基因之前,这也许加强了该抑制基因的转录,从而影响了laE的产量[19]。
|
(a)野生型;(b)产landomycin E的突变菌株S24 (a)Wild-type; (b)Mutant strain S24 that produce landomycin E 图 3 链霉菌S.globisporus野生菌株和突变株的landomycin E产量 Fig.3 Level of landomycin E production of S.globisporus wild-type and mutant strains |
利用转座子随机突变技术研究放线菌次生代谢物的产量,可以帮助我们更加深入地认识链霉菌的代谢网络和次生代谢产物的合成机制,从而提高次生代谢物的产量,为改良抗生素生产菌提供理论依据。如何更加高效地获得产量更高的突变株,并确定转座子的插入位点,还需要通过进一步的研究加以解决。
4 结论本研究所构建的经过修饰的基于Tn7转座子的微型转座载体不仅带有抗性标记且带有外向型的强启动子,可整合插入链霉菌染色体产生突变,影响次生代谢产物laE的产量。该载体含温度敏感型元素,可通过提高培养温度而使质粒丢失消除抗性。通过所构建的转座子突变载体产生的突变库,筛选获得5株laE产量提高的突变株,其中突变株S24的laE产量最高,为原始菌株的5倍。同时也分析识别了链霉菌S.globisporus中一些影响次生代谢物laE合成的新的调节因子。另外,本研究也证明转座子基载体可应用于非模式菌链霉菌的转座整合。随着研究的深入,转座子技术或将成为提高放线菌天然产物产量的实用策略。
| [1] |
孙肇暘, 杨秀萍. 放线菌素研究进展[J]. 首都师范大学学报:自然科学版, 2011, 32(1): 54-58. SUN Z Y, YANG X P. The research progress of actinomycins[J]. Journal of Capital Normal University:Natural Science Edition, 2011, 32(1): 54-58. |
| [2] |
黄敏红, 刘田甜, 陆群, 等. 链霉菌抗生素生物合成的基因调控[J]. 中国抗生素杂志, 2009, 34(7): 392-398. HUANG M H, LIU T T, LU Q, et al. Genetic regulation of antibiotic biosynthesis in Streptomyces[J]. Chinese Journal of Antibiotics, 2009, 34(7): 392-398. |
| [3] |
HORBAL L, FEDORENKO V, BECHTHOLD A, et al. A transposon-based strategy to identify the regulatory gene network responsible for landomycin E biosynthesis[J]. FEMS Microbiol Lett, 2013, 342(2): 138-146. DOI:10.1111/1574-6968.12117 |
| [4] |
KUTAS P, FECKOVA L, REHAKOVA A, et al. Strict control of auricin production in Streptomyces aureofaciens CCM 3239 involves a feedback mechanism[J]. Appl Microbiol Biotechnol, 2013, 97(6): 2413-2421. DOI:10.1007/s00253-012-4505-2 |
| [5] |
ZOTCHEV S B, SEKUROVA O N, KATZ L. Genome-based bioprospecting of microbes for new therapeutics[J]. Curr Opin Biotechnol, 2012, 23(6): 941-947. DOI:10.1016/j.copbio.2012.04.002 |
| [6] |
REBETS Y, OSTASH B, LUZHETSKYY A, et al. Production of landomycins in Streptomyces globisporus 1912 and S.cyanogenus S136 is regulated by genes encoding putative transcriptional activators[J]. FEMS Microbiol Lett, 2003, 222(1): 149-153. DOI:10.1016/S0378-1097(03)00258-1 |
| [7] |
REBETS Y, OSTASH B, LUZHETSKYY A, et al. DNA-binding activity of LndI protein and temporal expression of the gene that upregulates landomycin E production in Streptomyces globisporus 1912[J]. Microbiology, 2005, 151: 281-290. DOI:10.1099/mic.0.27244-0 |
| [8] |
DUTKO L, REBETS Y, OSTASH B, et al. A putative proteinase gene is involved in regulation of landomycin E biosynthesis in Streptomyces globisporus 1912[J]. Fems Microbiol Lett, 2006, 255: 280-285. DOI:10.1111/fml.2006.255.issue-2 |
| [9] |
OSTASH B, REBETS Y, MYRONOVSKYY M. Identification and characterization of the Streptomyces globisporus 1912 regulatory gene lndYR that affects sporulation and antibiotic production[J]. Microbiology, 2011, 157: 1240-1249. DOI:10.1099/mic.0.045088-0 |
| [10] |
萨姆布鲁克J, 拉塞尔D W. 分子克隆实验指南[M]. 黄培堂, 译. 第3版. 北京: 科学出版社, 2002. SAM BROOK J, RUSSELL D W. Molecular cloning-A laboratory manual[M]. HUANG P T(trans). 3rd edition. Beijing: Science Press, 2002. |
| [11] |
WANG Q, CHEN T, ZHAO X, et al. Metabolic engineering of thermophilic Bacillus licheniformis for chiral pure D-2, 3-butanediol production[J]. Biotechnol Bioeng, 2012, 109: 1610-1621. DOI:10.1002/bit.v109.7 |
| [12] |
QI G, KANG Y, LI L, et al. Deletion of meso-2, 3-butanediol dehydrogenase gene budC for enhanced D-2, 3-butanediol production in Bacillus licheniformis[J]. Biotechnol Biofuels, 2014, 7: 16. DOI:10.1186/1754-6834-7-16 |
| [13] |
BOHDAN B, STEPHEN W, MAKSYM M, et al. In vivo random mutagenesis of streptomycetes using mariner-based transposon Himar1[J]. Appl Microbiol Biotechnol, 2013, 97: 351-359. DOI:10.1007/s00253-012-4550-x |
| [14] |
DAMASCENO J D, BEVERLEY S M, TOSI L R. A transposon toolkit for gene transfer and mutagenesis in protozoan parasites[J]. Genetica, 2010, 38: 301-311. |
| [15] |
PETZKE L, LUZHETSKYY A. In vivo Tn5-based transposon mutagenesis of Streptomycetes[J]. Appl Microbiol Biotechnol, 2010, 86: 1593. DOI:10.1007/s00253-010-2511-9 |
| [16] |
WEADEN J, DYSON P. Transposon mutagenesis with IS6100 in the avermectin-producer Streptomyces avermitilis[J]. Microbiology, 1998, 144: 1963-1970. DOI:10.1099/00221287-144-7-1963 |
| [17] |
OSTASH B, REBETS Y, MYRONOVSKYY M. Identification and characterization of the Streptomyces globisporus 1912 regulatory gene lndYR that affects sporulation and antibiotic production[J]. Microbiology, 2011, 157(4): 1240-1249. DOI:10.1099/mic.0.045088-0 |
| [18] |
RIGALI S, TITGEMEYER F, BARENDS S, et al. Feast or famine:The global regulator DasR links nutrient stress to antibiotic production by Streptomyces[J]. EMBO Rep, 2008, 9: 670-675. DOI:10.1038/embor.2008.83 |
| [19] |
RAMOS J L, MARTINEZ-BUENO M, MOLINA-HENARES A J, et al. The TetR family of transcriptional repressors[J]. Microbiol Mol Biol Rev, 2005, 69: 326-356. DOI:10.1128/MMBR.69.2.326-356.2005 |
 2018, Vol. 25
2018, Vol. 25 

