2. 亚热带农业生物资源保护与利用国家重点实验室,广西南宁 530005
2. State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources, Nanning, Guangxi, 530005, China
【研究意义】 α-L-鼠李糖苷酶是一种水解酶,可以作用于α-1,2、α-1,3、α-1,4、α-1,6以及α-1连接的糖苷键[1],不同来源的鼠李糖苷酶其结构不同,催化特性也有所不同[2]。α-L-鼠李糖苷酶可分为GH13、GH28、GH78、GH106家族[3]。α-L-鼠李糖苷酶在很多工业中具有潜在的应用价值,主要表现在水解果汁饮品中的柚皮苷或橙皮苷,去除其苦味,改善果汁饮品的口味[4];其次在医药领域,水解芦丁生成异槲皮苷,而异槲皮苷相较于芦丁具有更好的医药应用前景[5]。【前人研究进展】目前关于动物组织和植物来源的α-L-鼠李糖苷酶的报道相对较少,主要是通过细菌和真菌(如曲霉属、青霉属等)发酵来获取α-L-鼠李糖苷酶。国外关于真菌α-L-鼠李糖苷酶基因的克隆研究主要集中在来源于曲霉属的α-L-鼠李糖苷酶。2008年,韩冰等[5]从Absidia sp.G3g中筛选出可将人参皂苷Re水解成Rg1[6]的人参皂苷-α-鼠李糖苷酶以及将芦丁水解成异槲皮苷的芦丁-α-鼠李糖苷酶[7];2016年,Li等[8]、Sarita等[9]分别从Aspergillus niger JMU-TS528和Penicillium griseoroseum中发现一株能够耐葡萄糖和乙醇的鼠李糖苷酶。2012年,Daniela等[10]从Aspergillus terreus中重组构建了一株能够高效转化芦丁生成异槲皮素的重组菌株。2015年Sebastian等[11]从Aspergillus terreus菌株中构建了一个重组菌株,它能够与β-葡萄糖苷酶共同作用于甜菊糖类物质。2016年Federica等[12]从Novospingobium sp.PP1Y中得到一个对α-1,2、α-1,6鼠李糖糖苷键均有较好水解效果的鼠李糖苷酶。【本研究切入点】目前大多数鼠李糖苷酶是从真菌曲霉中分离得到的,从细菌中寻找鼠李糖苷酶,为研究鼠李糖苷酶提供了新的方向。【拟解决的关键问题】本研究从实验室保藏的克雷伯氏菌中克隆得到一个α-L-鼠李糖苷酶基因,并构建重组质粒,对其进行诱导表达,进一步研究其酶学性质及其底物特异性,发掘其潜在的理论及应用价值。
1 材料与方法 1.1 材料菌株:克雷伯氏菌Klebsiella sp.GXK-1、大肠杆菌E.coli XL-blue由本实验室保藏。
试剂:限制性内切酶NcoⅠ、SacⅠ等,PrimeSTARTMHS DNA聚合酶、dNTP、T4DNA连接酶、λ/HindⅢ DNA Marker等均购买自TaKaRa公司;质粒DNA小量提取试剂盒、PCR纯化试剂盒、胶回收试剂盒均购自BioFlux公司;Ni-NTA填料购自Qiagen公司;对硝基苯基-α-L-吡喃鼠李糖苷(pNPR)等对硝基苯基底物均购自Sigma公司;各种糖类及其他试剂均为国产分析纯;柚皮苷、新橙皮苷购自阿拉丁生化科技股份有限公司;橙皮苷、芦丁购自生工生物工程(上海)股份有限公司;槲皮苷、杨梅苷、柴胡皂苷C购买自源叶生物。
仪器:PCR仪(Biometra),离心机(Eppendorf Centrifuge 5415D),高效液相色谱仪(Agilent G1314F 1260VWD、Agilent G1314F 1260RID),恒温培养箱(Binder),恒温摇床(ZHWY-211B),超声波细胞破碎机(新芝JY92-2D)。
1.2 方法 1.2.1 α-L-鼠李糖苷酶基因保守序列的克隆在GenBank数据库中查找与克雷伯氏菌(Klebsiella sp.GXK-1)同属的鼠李糖苷酶基因序列,利用SMART软件对这些同属的基因所编码的蛋白质进行结构组件分析,然后根据这些蛋白质结构组件的不同进行分类,用生物软件Vector NTI Advance 11.5对这些基因进行比对分析,查找其同源区并最终设计出二对简并引物。
第一类简并引物:
K1-1:5′-ACCTTCGGCGAR ACGCTGAGCG-AAG-3′
K1-2:5′-TTAAAGTCCGTACTGGCGAATA-AACC-3′
第二类简并引物:
K2-1:5′-ATGTTAAGTATY AATTACCAA-ACG-3′
K2-2:5′-TTACAATAGTACTTTTATRTC-ACC-3′
PCR反应程序:95℃ 3 min;95℃ 30 s,56.5℃ 30 s,72℃ 2 min,进行30个循环;72℃ 10 min。PCR扩增出的DNA片段连接至pMD19-T,转化到E.coli XL1-Blue,进行测序。根据测序结果,在NCBI上BLAST进行比对分析。
1.2.2 α-L-鼠李糖苷酶基因rha1的克隆根据保守区的比对分析结果,以pSE380为表达载体,设计含有6×His标签的引物,以Klebsiella sp.GXK-1的总DNA为模板,PCR扩增rhamnosidase完整基因rha1。rha1-1:5′-ACGCCATG-GAA CATCATCATCATCATCATTCACAAGCA-ATTCAATACAATTCC-3′(引入酶切位点NcoⅠ和组氨酸标签); rha1-2:5′-GGCGAGCTCTTA-AAGTCCGTACTGGCGAATAAAC-3′(引入酶切位点SacⅠ)。PCR反应程序:98℃ 3 min;98℃ 15 s,56.5℃ 10 s,72℃ 2 min,进行30个循环;72℃ 10 min。将PCR扩增所得的目的片段及提取的pSE380质粒分别经NcoⅠ和SacⅠ酶切处理后,进行连接,转化到E.coli XL1-Blue,进行测序。将成功构建的重组质粒命名为pSE-rha1。
1.2.3 蛋白质RHA1的生物信息学分析(1) RHA1的结构域预测及信号肽分析
在网站(http://smart.embl-heidelberg.de/)对α-L-鼠李糖苷酶基因rha1编码的氨基酸序列进行SMART Main page分析。在网站(http://www.cbs.dtu.dk/services/SignalP/)对RHA1进行SignalP4.1信号肽分析。
(2) RHA1的疏水性预测
使用ExPASy的ProtScale工具对重组酶RHA1进行疏水性分析。分析结果对每个氨基酸位点均有亲疏水性的评分。以0为分界线,亲水性的氨基酸评分为负值,疏水性氨基酸评分为正值。
(3) RHA1的二级结构预测
将RHA1的蛋白质序列提交到PSIPRED网站进行蛋白质二级结构预测。
(4) RHA1的三级结构的预测
利用SWISS-MODEL网站对RHA1进行建模分析,以4xhc.1.A(PDB-ID)为模板,它与RHA1序列相似性达到76.82%,达到同源建模的要求。
1.2.4 重组酶RHA1的诱导表达及纯化将菌种37℃培养使菌液OD600至0.4~0.6,再加入终浓度为0.5 mmol/L的IPTG,于20℃、180 r/min诱导20 h,离心收集菌体。超声波破胞后镍亲和层析纯化重组酶RHA1。并对纯化后的重组酶进行变性聚丙烯酰胺凝胶电泳(SDS-PAGE)。
1.2.5 重组酶RHA1酶学性质研究鼠李糖苷酶酶活力单位(U)的定义:在最适反应条件下,每分钟内催化1 μmol底物(pNPR)转化为产物(pNP)所需要的酶量。
酶活力测定方法:酶标准反应体系是由缓冲液、底物、酶液组成200 μL体系(除特别说明外)。取170 μL柠檬酸-磷酸氢二钠缓冲液和20 μL 10 mmol/L的pNPR先在最适温度下预热2 min,加入10 μL适当稀释倍数的酶液,准确反应20 min后,加入50 μL 2 mol/L Na2CO3终止酶反应,混匀,以不加酶的反应管为空白对照。取200 μL反应液至酶标板中,在405 nm波长下读取吸光值。
(1) 重组酶最适pH值测定
在37℃条件下,测定一系列不同pH值(4.0,4.5,5.0,5.5,6.0,6.5,7.0,7.5,8.0)的0.1 mol/L柠檬酸-0.2 mol/L磷酸氢二钠缓冲液对重组酶酶活力的影响,并以最高酶活力为100%,计算在各个pH值下重组酶的相对酶活力,相对活力最高对应的pH值即为该酶的最适反应pH值。
(2) 重组酶最适温度测定
在酶的最适pH值条件下,测定重组酶在一系列不同温度(25℃,30℃,35℃,40℃,45℃,50℃,55℃,60℃,65℃)下的酶活力,并以最高活力为100%,计算重组酶在各个温度下的相对酶活力,相对酶活力最高对应的温度即为该酶的最适反应温度。
(3) 酶pH稳定性的测定
将重组酶保存在一系列不同pH值(3.0,3.5,4.0,4.5,5.0,5.5,6.0,6.5,7.0,7.5,8.0的磷酸氢二钠-柠檬酸、8.0,8.5,9.0的硼酸-硼砂、9.0,9.5,10.0,10.5的甘氨酸-氢氧化钠)的缓冲液中,于4℃保存12 h。然后在最适反应条件下测定其残存酶活力,以保存在pH值7.0的磷酸氢二钠-磷酸二氢钠缓冲液中的纯酶的酶活力为100%,来计算各个pH值下的重组酶的相对酶活力。
(4) 酶热稳定性的测定
将重组酶保存在一系列不同温度(25~65℃,每个间隔为5℃)下1 h,立即取出放于冰上,然后在最适反应条件下测定其残存酶活力,以保存在冰箱4℃的纯酶的酶活力为100%,来计算各个温度下的重组酶的相对酶活力。
(5) 重组酶的动力学常数测定
在最适反应条件下,测定酶在以一系列不同终浓度(0.1 mmol/L,0.2 mmol/L,0.4 mmol/L,0.5 mmol/L,0.6 mmol/L,0.8 mmol/L,1.0 mmol/L,2.0 mmol/L)的pNPR为底物时的比活力,使用软件GraphPad Prism 5通过非线性回归分析拟合出重组酶的Km、Vmax值。
(6) 鼠李糖和葡萄糖对酶的影响
按照酶活力测定方法,测定重组酶分别在添加0~20 mmol/L不同浓度的鼠李糖以及0~200 mmol/L不同浓度葡萄糖时的酶活力,以不添加鼠李糖和葡萄糖的反应管为对照,其酶活力为100%,计算一系列不同浓度鼠李糖和葡萄糖下重组酶的相对活力。
(7) 金属离子对酶活力的影响
在最适反应条件下,在反应体系中添加终浓度为5 mmol/L的各种金属离子溶液,测定重组酶的残存酶活力。以不添加金属离子溶液的反应管的酶活力为100%,计算各种金属离子下重组酶的相对酶活力。实验测定的金属离子包括LiCl、KCl、RbCl、NaCl、CsCl、AgNO3、BaCl2、CaCl2、MnCl2、MgCl2、NiCl2、CoCl2、HgCl2、ZnSO4、CuSO4、FeSO4、Pb(NO3)2、FeCl3、CrCl3、AlCl3。
(8) 化学试剂对酶活力的影响
在最适反应条件下,在反应体系中分别添加终浓度为0.05% SDS(M/V)、1% TritonX-100(V/V)、4 mol/L脲、0.01 mol/L Imidazole、1%巯基乙醇(V/V)、5% Tween-80(V/V),测定重组酶的残存酶活力。以不添加化学试剂的反应管的酶活力为100%,计算各种化学试剂下重组酶的相对酶活力。
(9) 有机试剂对重组酶酶活力的影响
在最适反应条件下,在反应体系中分别添加5%、10%、15%、20%、25%、30%(V/V)的一元醇、二元醇及有机溶剂(一元醇类:甲醇、乙醇、正丙醇、异丙醇、正丁醇;二元醇类:乙二醇、1, 3-丙二醇;有机溶剂:DMSO、DMF、吡啶、四氢呋喃、乙腈),测定重组酶的残存酶活力。以不添加有机试剂的反应管的酶活力为100 %,计算各个有机试剂下重组酶的相对酶活力。
(10)酶底物特异性的测定
人工底物:取合适的底物浓度,以及适当稀释的酶液,在最适反应条件下进行酶活力的测定。以pNP或oNP的生成量来计算酶活力,以pNPR作为底物时的酶活力为100%。测定的底物包括对硝基苯基-α-L-吡喃鼠李糖苷(pNPR)、对硝基苯基-β-D-吡喃葡萄糖苷(pNPG)、对硝基苯基-β-D-吡喃木糖苷(pNPX)、4-硝基苯基-β-D-吡喃半乳糖苷(pNPGal)、4-硝基苯基-2-乙酰氨基-2-脱氧-β-D-吡喃葡糖苷(pNPNAG)、4-甲基伞形酮-β-D-葡糖苷酸(MU-Glc)、邻硝基苯- β-D-半乳糖苷(oNPG)、4-硝基苯基-β-D-吡喃半乳糖苷(pNPGal)、4-硝基苯基-β-D-阿拉伯糖苷(pNPA)、4-硝基苯基-β-D-纤维二糖苷(pNPC)、4-硝基苯基-α-D-葡萄糖苷(α- pNPC)。
二糖及糖苷类:以终浓度为1%(W/V)的麦芽糖、异麦芽糖、海藻糖、乳糖、纤维二糖、蔗糖、苦杏仁苷、水杨苷分别作为底物,于纯酶在最适酶反应条件下测定酶活力。反应20 min后,煮沸终止5 min,利用葡萄糖氧化试剂盒测定反应管中葡萄糖的生成量,具体操作步骤按照试剂盒的说明书进行,依次计算酶活力。
多糖类物质:以终浓度为1%(W/V)的羧甲基纤维素钠、淀粉、山毛榉木聚糖分别作为底物,于纯酶在最适酶反应条件下测定酶活力。反应6 h后,加入400 μL DNS终止酶反应,再将反应管煮沸显色5 min。取200 μL反应液至酶标板中,在540 nm波长下读取吸光值。根据还原糖的生成量来计算酶活力。
天然类底物:以终浓度为0.1%(W/V)的橙皮苷、芦丁、槲皮苷分别作为底物,与纯酶在最适酶反应条件反应12 h后,煮沸终止5 min。用50%乙腈稀释10倍,12 000 r/min离心30 min,取上清利用HPLC分析酶与这3种底物的反应产物。HPLC检测条件:仪器设备为安捷伦1260系列液相色谱仪,色谱柱为C18柱,进样量10 μL,柱温35℃,流速1 mL/min,检测波长280 nm。流动相:water(A)、methanol(B)、acetonitrile(C)。梯度洗脱:A:B:C=62:12:26(0~7 min)、A:B:C=15:35:50(7~9 min)、A:B:C=15:35:50(9~15 min)、A:B:C=62:12:26(15~17 min)、A:B:C=62:12:26(17~20 min)[8]以终浓度为0.1%(M/V)的杨梅苷作为底物,于纯酶在最适酶反应条件下反应12 h后,煮沸终止5 min。用50%乙腈稀释3倍,12 000 r/min离心30 min,取上清用HPLC分析反应产物。HPLC检测条件:仪器设备为安捷伦1260系列液相色谱仪,色谱柱为C18柱,进样量10 μL,柱温30℃,流速1 mL/min,检测波长358 nm。流动相:acetonitrile(A)、0.1%磷酸水溶液(B)。梯度洗脱:A:B=20:80(0 min)、A:B=30:70(0~10 min)、A:B=50:50(10~15 min)、A:B=80:20(15~20 min)、A:B=80:20(20~25 min)、A:B=20:80(20~30 min)[13]。
以终浓度为0.1%(W/V)的柚皮苷、新橙皮苷分别作为底物,于纯酶在最适酶反应条件下反应12 h后,煮沸终止5 min。用50%乙腈稀释10倍,12 000 r/min离心30 min,取上清用HPLC分析反应产物。HPLC检测条件:仪器设备为安捷伦1260系列液相色谱仪,色谱柱为C18柱,进样量10 μL,柱温30℃,流速1 mL/min,检测波长285 nm。流动相:0.1%磷酸水溶液(A)、acetonitrile(B)。梯度洗脱:A:B=77:23(0~5 min)、A:B=45:55(5~10 min)、A:B=45:55(10~25 min)、A:B=77:23(25~30 min)。
以终浓度为0.1%(W/V)的柴胡皂苷C作为底物,于纯酶在最适酶反应条件下反应12 h后,煮沸终止5 min。用50%乙腈稀释3倍,12 000 r/min离心30 min,取上清用HPLC分析反应产物。HPLC检测条件:仪器设备为安捷伦1260系列液相色谱仪,色谱柱为C18柱,进样量10 μL,柱温25℃,流速1 mL/min,检测波长210 nm。流动相:acetonitrile(A)、0.1%磷酸水溶液(B)。梯度洗脱:A:B=30:70(0 min)、A:B=60:40(0~22 min)、A:B=60:40(22~27 min)、A:B=30:70(0~5 min)。
2 结果与分析 2.1 基因保守区的扩增及rha1的克隆以提取的克雷伯氏菌的总DNA为模板,利用引物K2-1和K2-2能够扩增出基因的保守区,大小约为1.3 Kb。将测序结果在NCBI上进行BLAST分析,相似性高达99%的结果有40多个,对这些结果进行分析,发现测序结果及这些序列的开始和结尾是一样的、高度保守的。rha1的基因大小为1 569 bp,G+C百分含量为61.12%,可编码由522个氨基酸组成的蛋白质。该蛋白质的预计分子量为59 707.84 Da,等电点pI为5.85。
2.2 蛋白质RHA1的生物信息学分析 2.2.1 RHA1的结构域预测及信号肽分析对RHA1进行蛋白质结构组件SMART分析,发现RHA1只含有一个Bac_rhamnosid功能域(图 1a)。结合SignalP4.1分析,结果表明蛋白质RHA1是不含信号肽序列的(图 1b)。
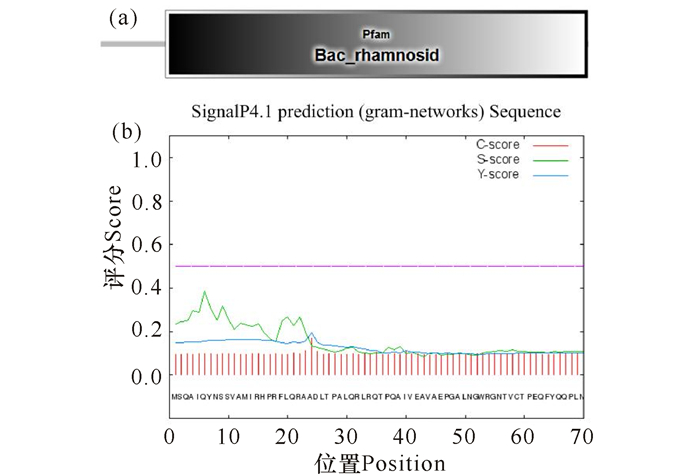
|
图 1 RHA1的结构组件及信号肽分析 Fig.1 Structural components and signal peptide analysis of RHA1 |
使用ExPASy的ProtScale工具对重组酶RHA1进行疏水性分析,根据分析结果显示,RHA1蛋白质具有较为明显的亲水性(图 2)。
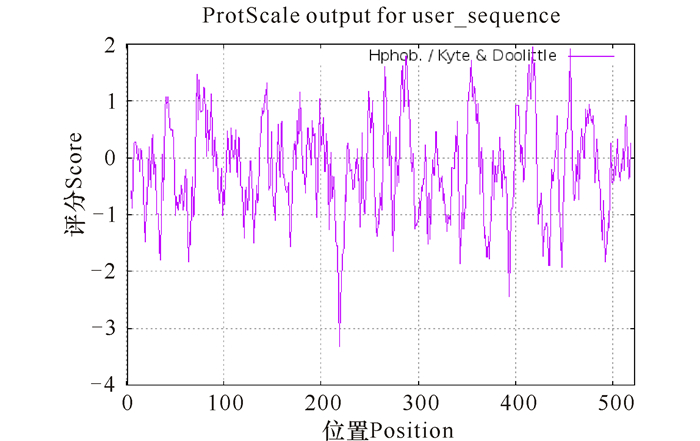
|
图 2 RHA1的疏水性分析 Fig.2 Hydrophobic analysis of RHA1 |
将RHA1的蛋白质序列提交到PSIPRED网站进行蛋白质二级结构预测,结果如图 3所示。结果中显示了RHA1二级结构中可能出现α螺旋区、β折叠区以及无规卷曲的分布,其中α螺旋区占36.78%,β折叠区占15.33%,无规卷曲占47.89%。
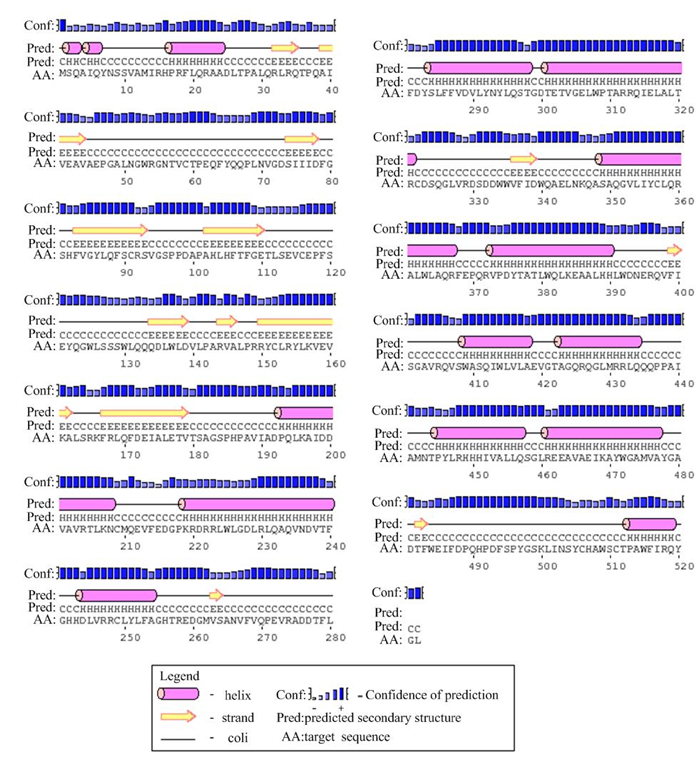
|
图 3 RHA1的二级结构分析 Fig.3 Secondary structure analysis of RHA1 |
利用SWISS-MODEL网站对RHA1进行建模分析,结果是以4xhc.1.A(PDB-ID)为模板,它与RHA1序列相似性达到76.82%,达到同源建模的要求。根据建模分析的结果(图 4)预测,RHA1具有α螺旋、β折叠以及无规卷曲等二级结构。活性中心涉及的氨基酸残基可能为221位的Asp和485位的Glu[14]。

|
图 4 RHA1三级结构的预测 Fig.4 The prediction tertiary structure of RHA1 |
重组质粒转化到E.coli XL1-Blue进行诱导表达,镍亲和层析纯化目的蛋白RHA1。纯化产物进行SDS-PAGE。在约60 kDa位置处出现特征条带,与理论分子量一致。且纯化条带明显,可以进行下一步的鉴定(图 5)。
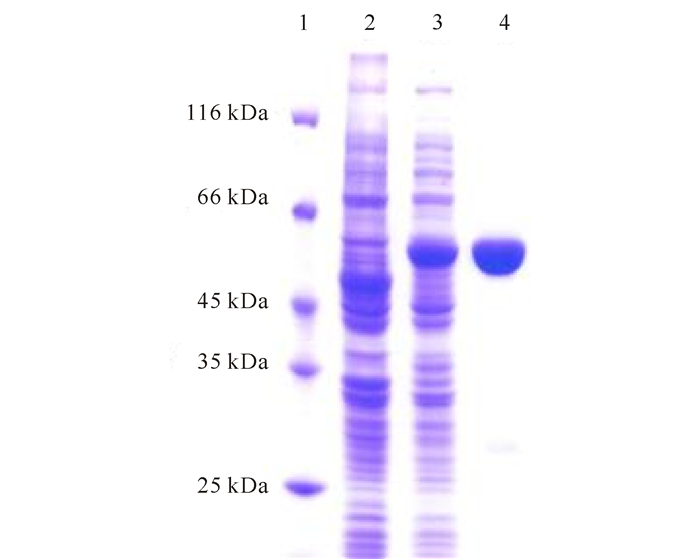
|
1:Standard protein marker; 2:E.coli/pSE380诱导菌体的裂解物; 3:E.coli/pSE-rha1诱导菌体的裂解物; 4:纯化的RHA1 1:Standard protein marker; 2:Inclued cell lysate of E.coli/pSE380;3:Inclued cell lysate of E.coli/pSE-rha1;4:Purified protein RHA1 图 5 纯化产物RHA1的SDS-PAGE分析 Fig.5 SDS-PAGE analysis of purified product RHA1 |
RHA1的最适pH值为5.0,在pH值为4.5~6.0时能保持80 %以上的酶活力(图 6a)。RHA1的最适温度为45℃,在40~50℃时能保持80%以上的酶活力(图 6b)。
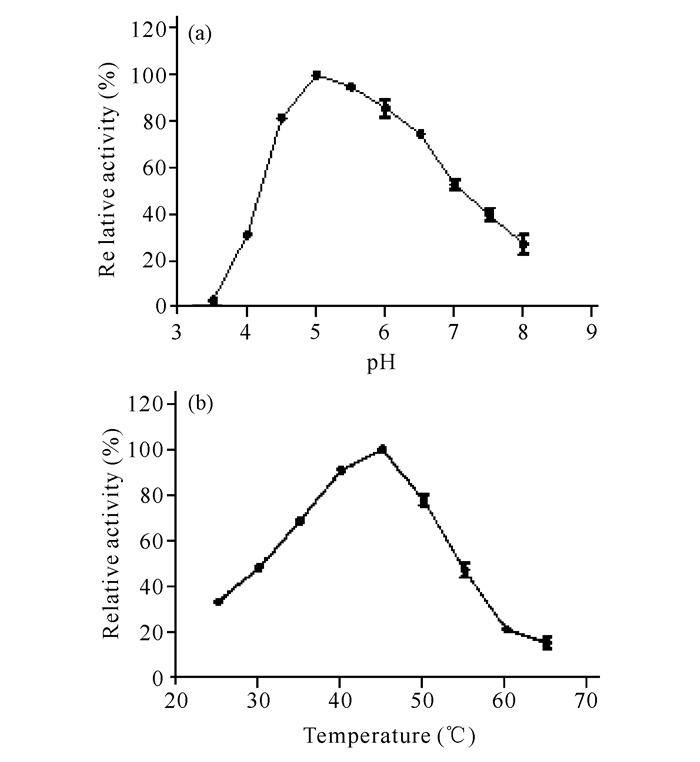
|
图 6 pH值和温度对RHA1酶活的影响 Fig.6 The effects of pH and temperature on the RHA1 enzyme activity |
pH稳定性测定:在pH值6~10的缓冲液内4℃放置12 h后,酶活力仍保持在80%以上(图 7a),说明在此范围内该酶具有较好的pH稳定性,在高pH值缓冲液环境中仍能保持较高的活力。
温度稳定性测定:在温度为25~40℃时,酶活力较为稳定,保温1 h后仍保持80%以上的活力(图 7b),但在温度高于40℃时酶活力迅速下降。
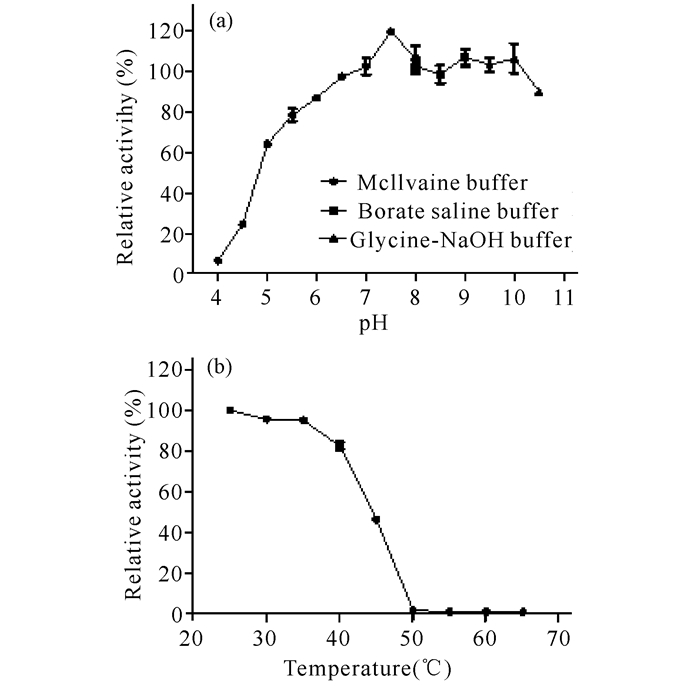
|
图 7 pH稳定性及温度稳定性的测定 Fig.7 The pH stability and temperature stability of RHA1 |
如图 8所示,以一系列不同终浓度的pNPR为底物,利用软件GraphPad Prism 5通过非线性回归拟合出RHA1的Km值为(0.223±0.030) mmol/L,Vmax值为(1.272±0.121) μmol/(mg·min)。
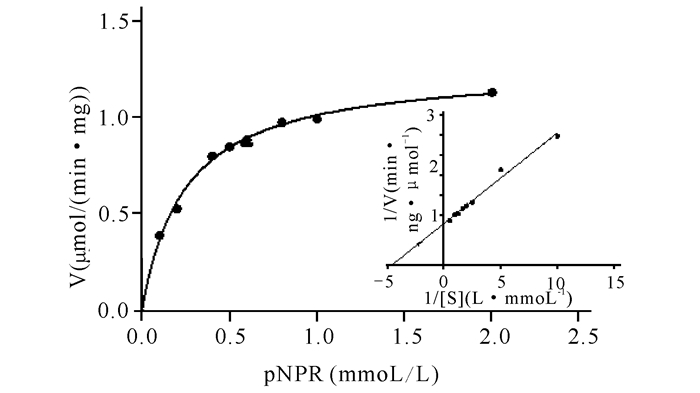
|
图 8 RHA1的Km和Vmax值 Fig.8 The determination of Km and Vmax |
RHA1对鼠李糖较为敏感(图 9a),在鼠李糖浓度为20 mmol/L时RHA1的活力仅为不添加鼠李糖时的40%左右。RHA1对葡萄糖不是很敏感(图 9b),在葡萄糖浓度为100 mmol/L时RHA1的活力为不添加葡萄糖时的40%左右。

|
图 9 鼠李糖和葡萄糖对RHA1酶活力的影响 Fig.9 The effects of rhamnose and glucose on the RHA1 enzyme activity |
从表 1可以看出:Ag+和Hg2+完全抑制重组酶活性,Pb2+、Fe3+对重组酶有较强的激活作用,Rb+、K+、Fe2+、Li+对重组酶略有激活作用,其他所测的金属离子对重组酶有不同程度的抑制作用。
| 表 1 金属离子对RHA1酶活的影响 Table 1 The effects of metal ions on the RHA1 enzyme activity |
从图 10可以看出,0.05% SDS和4 mol/L脲能够使酶失活,0.01 mol/L Imidazole对酶活力影响不大,1% TritonX-100、5% Tween-80和1%巯基乙醇对酶活略有抑制作用。
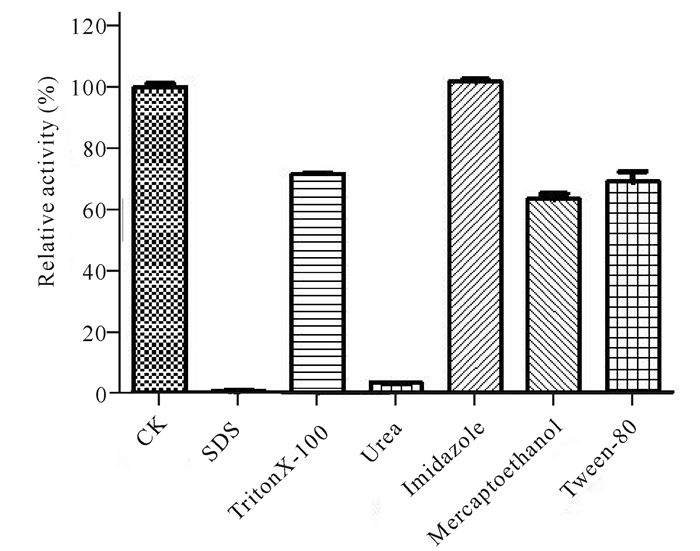
|
图 10 化学试剂对RHA1酶活的影响 Fig.10 The effects of chemical reagents on the RHA1 enzyme activity |
由图 11可知,乙二醇和1, 3-丙二醇在5%浓度时对酶活力有一定的促进作用,在浓度为30%(V/V)时,对RHA1酶活的抑制没有那么强烈,仍保持有一半的酶活力,而其他几种有机试剂对RHA1酶活的抑制较为明显,且抑制程度与浓度相关,浓度越高,对RHA1的抑制作用越强。
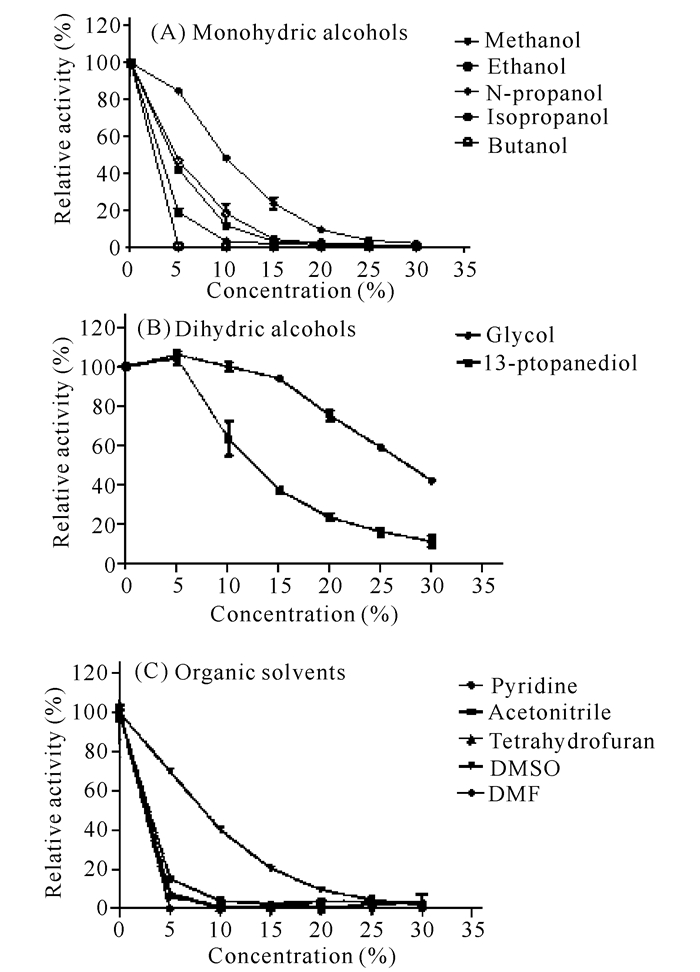
|
图 11 有机试剂对RHA1酶活的影响 Fig.11 The effects of organic reagents on the RHA1 enzyme activity |
随着有机溶剂的浓度增大,其对RHA1的抑制作用越强。其中RHA1对DMSO表现出较高的耐受性,在10%的DMSO下仍能保持40%以上的活力。其余几种有机溶剂对RHA1有强烈的抑制作用。
2.3.8 RHA1的底物特异性RHA1仅能水解pNPR,对其他人工底物没有水解能力;对蔗糖有较弱的水解能力,对其他二糖及糖苷类没有水解能力;对于天然类底物RHA1仅能够水解α-1,6糖苷键的橙皮苷及芦丁(表 2)。
| 表 2 天然底物特异性 Table 2 The natural substrates specificity |
鼠李糖苷酶一般以L-鼠李糖苷酶为主,L-鼠李糖苷酶可分为α-L-鼠李糖苷酶(EC 3.2.1.40)和β-L-鼠李糖苷酶(EC 3.2.1.43),这两种酶催化水解非还原性末端L-鼠李糖残基。本文研究的RHA1是属于GH78家族的α-L-鼠李糖苷酶。重组酶RHA1的最适反应pH值和最适反应温度为5.0和45℃。大多数鼠李糖苷酶的最适反应pH值为4~7,少数pH值大于7;最适酶反应温度差异较大,多数在40~60℃,少数在70℃或以上[2, 15]。Mai等[15]从Aspergillus oryzae中克隆得到的α-L-鼠李糖苷酶最适酶反应温度为70℃,来源于Aspergillus terreus的α-L-鼠李糖苷酶在温度为70℃时仍具有较好的耐受性[10]。
本研究中,RHA1对橙皮苷和芦丁均具有一定的水解效果。橙皮苷在水中的溶解度极低,鼠李糖苷酶水解橙皮苷生成橙皮素葡萄糖苷和鼠李糖,而橙皮素葡萄糖苷是生产甜味剂的前体物质。芦丁在自然界中的含量非常丰富,而异槲皮素作为芦丁水解掉一个鼠李糖基后的产物,在自然界中的含量是十分低的[7]。但异槲皮素相较于芦丁具有更好的药理活性,更容易被人体吸收。因此,水解橙皮苷和芦丁具有一定的实际应用价值。
4 结论本研究通过设计简并引物PCR扩增获得α-L-鼠李糖苷酶基因rha1,其基因大小为1 569 bp,编码522个氨基酸。以pSE380为表达载体,并在大肠杆菌E.coli XL-blue表达,使用镍亲和层析纯化重组蛋白,研究目的蛋白RHA1的酶学性质。以pNPR为底物,测得重组酶RHA1的最适反应pH值和最适反应温度为5.0和45℃。Km值为(0.223±0.030)mmol/L,Vmax值为(1.272±0.121) μmol/(min·mg);重组酶RHA1对高pH值缓冲液有较好的耐受性;对于天然类底物RHA1能够水解α-1-6糖苷键的橙皮苷及芦丁,水解芦丁生成的异槲皮苷具有较好的医药功能。
| [1] |
YADAV V, YADAV P K, YADAV S, et al. α-L-Rhamnosidase:A review[J]. Process Biochemistry, 2010, 45(8): 1226-1235. DOI:10.1016/j.procbio.2010.05.025 |
| [2] |
王艳君, 刘同军, 曹涛, 等. α-L鼠李糖苷酶的研究进展[J]. 中国酿造, 2010, 233(10): 11-15. WANG Y G, LIU T J, CAO T, et al. Research progress of α-L-rhamnosidase[J]. China Brewing, 2010, 233(10): 11-15. DOI:10.3969/j.issn.0254-5071.2010.10.004 |
| [3] |
张霞, 李利君, 倪辉, 等. 微生物来源α-L-鼠李糖苷酶的分子和结构生物学研究进展[J]. 生命科学研究, 2015, 19(1): 68-74. ZHANG X, LI L J, NI H, et al. Progress on molecular biology and structural biology of α-L-rhamnosidase from microbial source[J]. Life Science Research, 2015, 19(1): 68-74. |
| [4] |
LI L J, WU Z Y, YU Y, et al. Development and characterization of an α-L-rhamnosidase mutant with improved thermostability and a higher efficiency for debittering orange juice[J]. Food Chemistry, 2018, 245: 1070-1078. DOI:10.1016/j.foodchem.2017.11.064 |
| [5] |
韩冰, 付绍平, 金凤燮, 等. 两种菌产两种不同天然苷类α-鼠李糖苷酶的研究[J]. 大连工业大学学报, 2008, 27(2): 105-109. HAN B, FU S P, JIN F X, et al. Characterization of glycoside-α-rhamnosidase from Absidia sp.[J]. Journal of Dalian polytechnic University, 2008, 27(2): 105-109. |
| [6] |
金赞敏, 鱼红闪, 金凤燮. 人参皂甙-α-鼠李糖苷酶分离提纯及其酶性质[J]. 大连轻工业学院学报, 2003, 22(2): 103-106. JIN Z M, YU H S, JIN F X. Purification and characterization of ginsenoside-α-rhamnosidase[J]. Journal of Dalian Institute of Light Industry, 2003, 22(2): 103-106. |
| [7] |
王侃, 鱼红闪, 金凤燮. 芦丁-α-鼠李糖苷酶分离提纯及其酶性质[J]. 大连轻工业学院学报, 2004, 23(1): 30-33. WANG K, YU H S, JIN F X. Purification and characterization of rutin-α-rhamnosidase[J]. Journal of Dalian Institute of Light Industry, 2004, 23(1): 30-33. |
| [8] |
LI L J, YU Y, ZHANG X, et al. Expression and bio- chemical characterization of recombinant α-L-rhamnosidase r-Rha1 from Aspergillus niger JMU-TS528[J]. International Journal of Biological Macromolecules, 2016, 85: 391-399. DOI:10.1016/j.ijbiomac.2015.12.093 |
| [9] |
YADAV S, YADAVA S, YADAV K D S. α-L-rhamnosidase selective for rutin to isoquercitrin transformation from Penicillium griseoroseum MTCC-9224[J]. Bioorganic Chemistry, 2017, 70: 222-228. DOI:10.1016/j.bioorg.2017.01.002 |
| [10] |
GERSTORFEROVÁ D, FLIEDROVÁ B, HALADA P, et al. Recombinant α-L-rhamnosidase from Aspergillus terreus in selective trimming of rutin[J]. Process Biochemistry, 2012, 47(5): 828-835. DOI:10.1016/j.procbio.2012.02.014 |
| [11] |
SPOHNERS C, ZAHN D, SCHAUM V, et al. Recombinant α-L-rhamnosidase from Aspergillus terreus in selective trimming of α-L-rhamnose from steviol glycosides[J]. Journal of Molecular Catalysis B:Enzymatic, 2015, 122: 248-254. DOI:10.1016/j.molcatb.2015.09.009 |
| [12] |
FEDERICA D L, FRANCESCA M, VINCENZO T, et al. RHA-P:Isolation, expression and characterization of a bacterial α-L-rhamnosidase from Novosphingobium sp.PP1Y[J]. Journal of Molecular Catalysis B:Enzymatic, 2016, 134: 136-147. DOI:10.1016/j.molcatb.2016.10.002 |
| [13] |
齐洁, 万竹青, 李墨影, 等. HPLC法测定杨梅叶中杨梅苷的含量[J]. 中国野生植物资源, 2012, 31(1): 35-37. QI J, WAN Z Q, LI M Y, et al. Determination of myricitrin in leaves of Myrica rubra Sieb.et Zuce.by HPLC[J]. Chinese Wild Plant Resources, 2012, 31(1): 35-37. |
| [14] |
O'NEILL E C, STEVENSON C E, PATERSON M J, et al. Crystal structure of a novel two domain GH78 family α-rhamnosidase from Klebsiella oxytoca with rhamnose bound[J]. Proteins, 2015, 83(9): 1742-1749. DOI:10.1002/prot.24807 |
| [15] |
ISHIKAWA M, SHIONO Y, KOSEKI T. Biochemical characterization of Aspergillus oryzae recombinant α-L-rhamnosidase expressed in Pichia pastoris[J]. Jouranl of Bioscience and Bioengineering, 2017, 124(6): 630-634. DOI:10.1016/j.jbiosc.2017.07.007 |
 2018, Vol. 25
2018, Vol. 25 


