【研究意义】木聚糖是植物细胞壁半纤维素的重要组成部分,它是自然界中含量仅次于纤维素的第二大丰富的多聚糖,几乎占地球可再生碳源的三分之一[1]。β-1,4-内切木聚糖酶 (Endo-β-1,4-xylanase,EC 3.2.1.8) 主要从糖链内部作用于β-1,4-糖苷键,能够专一降解木聚糖为低聚木糖和木寡糖,是木聚糖降解过程中最重要的酶之一[1]。自然界能够产生木聚糖酶的微生物有很多,包括丝状真菌、细菌、放线菌等。不同微生物来源的木聚糖酶,其理化性质和分子量不尽相同,分子量从7.7 kDa到150 kDa不等,绝大多数为20~40 kDa,最适pH值在2~11都有分布[2-4]。木聚糖酶在造纸、食品、饲料、纺织和能源等工业领域都有着广泛的应用前景[5],特别是在造纸行业。木聚糖酶可以解聚凝结于纤维表面的半纤维素,提高漂白剂在纸浆中的扩散速度,提高漂白效率,减少氯的使用量[6],因此可用于纸浆的预漂白、二次纤维的脱墨等。近几年,国家倡导“绿色工艺”,要求工业上减少能源消耗,降低污水排放;而造纸行业由于大量使用氯漂白、酸碱清洗的缘故,污染比较严重,环保及节能减排压力很大,技术更新需求迫切。在造纸上添加木聚糖酶可减少氯的排放和酸碱的使用,有利于绿色工艺,然而并不是所有的木聚糖酶能够满足造纸行业碱性高温的苛刻环境,只有极端耐热耐碱的木聚糖酶才能满足这样的条件[6-10]。【前人研究进展】2016年Lu等[11]将来源于Bacillus pumilus HBP8的耐碱木聚糖酶188位点的天冬酰胺突变为丙氨酸后,酶的热稳定性提高,通过基因优化并在毕赤酵母中实现高效表达;在纸张漂白过程中补加15 U/g的木聚糖酶可提高纸张2%的亮度,13%的强度,6.5%的破裂系数。因此,在纸张漂白过程中添加耐热耐碱的木聚糖酶有助于纸张的漂白及性质的改善。【本研究切入点】来源于嗜碱芽孢杆菌S7(Bacillus halodurans S7) 的木聚糖酶最适温度为75℃,最适pH值为9,并且该酶在很广的pH值范围内都有酶活力[12],因此该酶能够满足造纸行业碱性高温的苛刻环境。【拟解决的关键问题】根据该酶的基因序列 (GenBank:AY687345) 进行密码子优化后合成新的基因 (GenBank:KX712250),并将其克隆到pHBM905BDM载体上,然后通过电转法转入毕赤酵母GS115;最后通过交联木聚糖底物平板水解圈法对重组菌进行初筛以及摇瓶诱导后比较酶活力大小进行复筛,筛选得到一株产酶量较高的高产菌株。
1 材料和方法 1.1 材料 1.1.1 菌株及质粒大肠杆菌 (Escherichia coli) DH5α克隆菌株,用于重组质粒的构建;毕赤酵母 (Pichia pastoris) GS115菌株,用于目的蛋白的发酵表达;pHBM905BDM质粒 (来源于本实验室[8])。
1.1.2 试剂NotⅠ、CopⅠ、SalⅠ等限制性内切酶,T4 DNA polymerase、Solution Ⅰ连接酶,Ex Taq酶均购自TaKaRa公司;Endo H购自BioLabs公司;DNA分子量标准λ/EcoT14与DL 2000、dNTP购自TaKaRa公司;预染蛋白Marker购自Thermo公司;木聚糖、YNB、考马斯亮蓝以及溴化乙锭 (EB) 购自Sigma公司。引物合成及测序由上海生工生物工程技术服务有限公司完成,密码子优化及基因合成由武汉金开瑞生物工程有限公司完成。
1.1.3 培养基LB培养基,用于大肠杆菌的培养;YPD、MD、BMGY和BMMY培养基,用于毕赤酵母的培养、筛选和诱导表达,具体配方见Invitrogen公司的毕赤酵母操作手册。
1.2 方法 1.2.1 密码子优化许多数据证明,优化外源基因密码子对提高外源蛋白表达量有显著的作用,可以使表达量提高几倍甚至数十倍[13-15]。因此在保证氨基酸序列不变的前体下,尽量使用毕赤酵母偏爱密码子对外源基因序列进行改造。对毕赤酵母密码子的偏好性有不同的算法[16-17],很多研究也给出酵母稀有密码子及偏爱密码子表,可以根据这些算法及偏爱密码子统计表对目的基因密码子序列进行改造[13-15]。此外,还可以通过一些密码子优化网站 (http://helixweb.nih.gov/dnaworks) 对密码子序列进行改造。
1.2.2 pHBM905BDM-xyn质粒的构建用限制酶CopⅠ和NotⅠ双酶切处理pHBM905BDM质粒,琼脂糖凝胶回收大片段作为载体。以武汉金开瑞生物工程有限公司合成的基因构建的pMD18T-xyn质粒为模板,用木聚糖酶基因 (大小为1 116 bp) 的前引物xynF (5′-GTCAAACGTTGCTGCTGCTCAA-3′) 和后引物xynR (5′-GGCCATTAATCGATGATTCTCCAAT-3′) 进行PCR扩增,扩增条件:95℃预变性3 min;95℃变性30 s,56℃退火30 s,72℃延伸70 s,25个循环;72℃再延伸5 min。PCR扩增后胶回收PCR片段,用T4 DNA聚合酶与dTTP于12℃条件下处理20 min,得到与限制酶CopⅠ和NotⅠ酶切位点有相同粘性末端的目的片段,再用SolutionⅠ连接酶将质粒与目的片段按1:3的摩尔比在16℃条件下连接2 h,将连接产物转化E.coliDH5α克隆感受态细胞,涂布于含氨苄青霉素的LB固体平板,37℃过夜培养。pHBM905BDM-xyn质粒的构建流程具体如图 1所示。

|
图 1 重组质粒pHBM905BDM-xyn的构建流程 Fig.1 Theflow chart of constructrecombinant vector pHBM905BDM-xyn |
待平板上长出单菌落后,挑选部分转化子分别作为模板,以引物xynF和xynR进行菌落PCR验证,PCR扩增条件:95℃预变性5 min;95℃变性30 s,56℃退火30 s,72℃延伸70 s,30个循环;72℃再延伸5 min。将能扩增得到目的条带的转化子摇瓶培养后抽提质粒并测序验证,得到的重组质粒命名为pHBM905BDM-xyn。
1.2.4 目的基因的转化用限制酶SalⅠ将pHBM905BDM-xyn质粒进行线性化并回收大片段。取80 μL毕赤酵母GS115感受态细胞与5~20 ng线性化DNA (溶于10 μL TE缓冲液) 混合后,转入预冷的0.2 cm电转杯中,冰上放置5 min,根据参数电击,最后加入1 mL 1 mol/L PBST 30℃静置培养2 h后涂布MD平板。28℃培养2~3 d直至长出单菌落,挑选单菌落以引物xynF和xynR进行菌落PCR验证,PCR扩增条件:95℃预变性10 min;95℃变性30 s,56℃退火30 s,72℃延伸70 s,30个循环;72℃再延伸5 min。
1.2.5 木聚糖酶基因的表达与高产菌株的初筛挑MD平板上筛选出的重组子接种在含有桦木木聚糖的BMMY底物平板上,每12 h加400 μL甲醇诱导木聚糖酶基因表达。产木聚糖酶的重组菌落会水解底物平板上的交联木聚糖从而产生水解圈,根据产生水解圈的速度和大小可以初步筛选出酶活力较高的重组菌落。
1.2.6 高产菌株的复筛将初筛得到的重组菌落做好标记,分别接种到50 mL BMGY种子培养基中,28℃摇瓶培养至OD600=20~30时,将菌体量调一致后分别离心收集菌体并转接至25 mL BMMY诱导培养基中,同时添加400 μL甲醇,于28℃摇瓶诱导发酵。每隔24 h在超净工作台中取出400 μL发酵液并添加400 μL甲醇,诱导10 d。将发酵液于12 000 r/min条件下离心1 min,所得上清液即为粗酶液。用3,5-二硝基水杨酸 (DNS) 法[8, 13]测定木聚糖酶酶活力,具体步骤如下:取100 μL用glycine-NaOH缓冲液 (pH值为9.0) 稀释适当倍数的粗酶液和100 μL溶解在glycine-NaOH缓冲液 (pH值为9.0) 中的1%(W/V) 桦木木聚糖溶液于70℃反应10 min,然后100℃灭活5 min,冷却后加入250 μL DNS溶液并在100℃反应5 min,冷却至室温后加入500 μL H2O混匀后在540 nm处测其吸光度。酶活力单位 (U) 定义:在该测试条件下每分钟生成1 μmol的木糖所需要的酶量作为一个酶活力单位 (U)。比较不同重组子的酶活力大小,进一步筛选出酶活力较高的重组菌。
2 结果与分析 2.1 密码子优化为提高木聚糖酶xyn10A在毕赤酵母P.pastoris GS115中的表达量,依据毕赤酵母的密码子偏好性对该木聚糖酶基因 (GenBank:AY687345) 的密码子进行优化 (新登录号:KX712250)。原始基因序列中含有的稀有密码子会降低蛋白翻译的效率,甚至终止翻译过程。因此,对xyn10A基因序列中的228个稀有密码子进行优化,提高毕赤酵母的最适密码子使用频率。通过DNAMEN软件分析优化后的xyn10A序列与原始基因序列比对一致性为76.70%(图 2)。
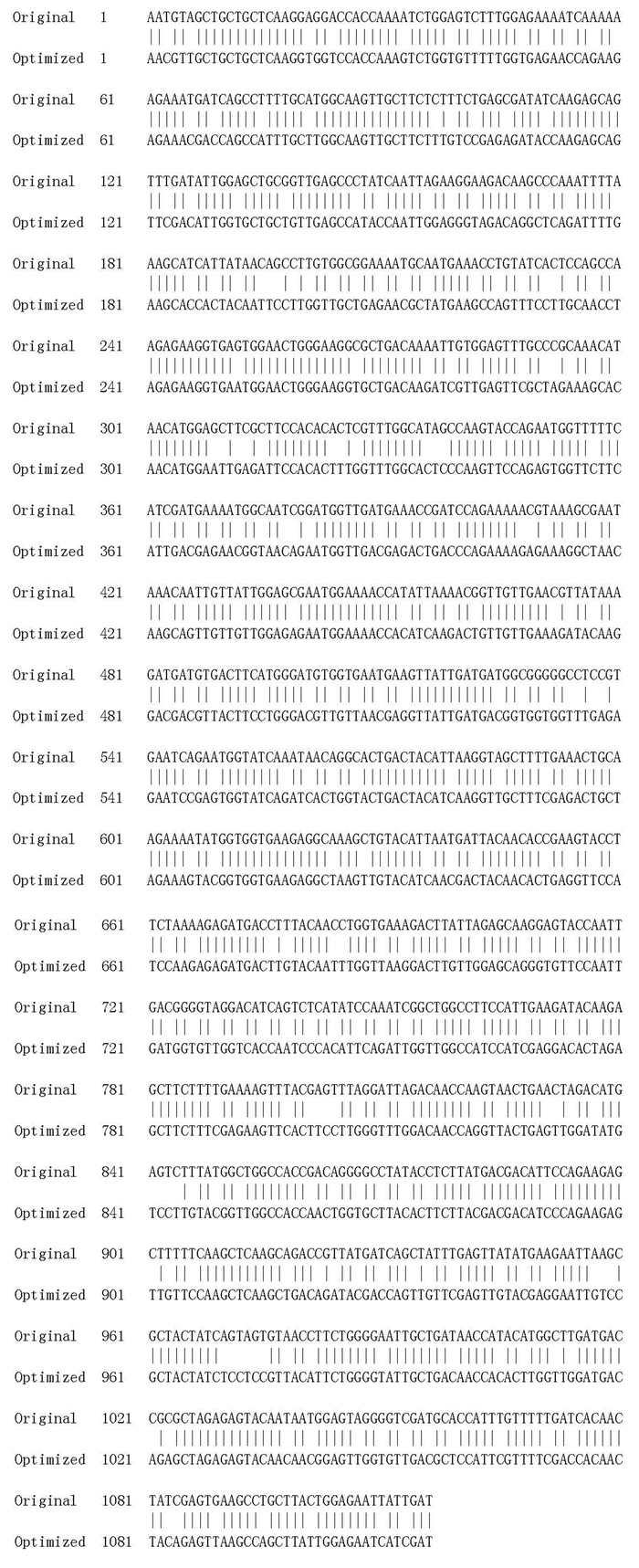
|
图 2 xyn10A序列与原始基因序列比对 Fig.2 xyn10A alignment with original gene sequence |
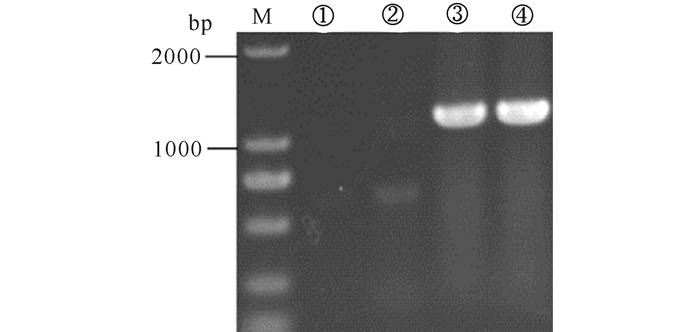
分别以pHBM905BDM质粒和构建好的pHBM905BDM-xyn质粒为模板,使用引物对木聚糖酶基因 (大小为1 116 bp) 进行PCR扩增 (图 3),从图中可以看出以pHBM905BDM-xyn质粒为模板的PCR在1 100 bp左右处出现一条亮带,证明重组质粒pHBM905BDM-xyn构建成功。
|
M:DNA Marker DL 2000;①、②:以pHBM905BDM质粒为模板;③、④:以pHBM905BDM-xyn质粒为模板 M: DNA Marker DL 2000; ①, ②: pHBM905BDM plasmid as template; ③, ④: pHBM905BDM-xyn plasmid as template 图 3 pHBM905BDM-xyn重组质粒的PCR验证 Fig.3 The PCR verification of pHBM905BDM-xynrecombinant plasmid |
将测序验证后的pHBM905BDM-xyn重组质粒用限制酶SalⅠ线性化后电转化毕赤酵母GS115感受态细胞,涂在MD平板上筛选重组子,挑取转化子进行菌落PCR验证后,挑取MD平板上的单菌落转接到含桦木木聚糖底物的平板BMMY上并加甲醇诱导,根据产生水解圈的快慢和大小初步筛选酶活力较高的重组菌,从图 4中可以看出③~⑩都产生水解圈,可以进行下一步实验。

|
①GS115;②GS115/ pHBM905BDM;③~⑩重组酵母 ①GS115; ②GS115/ pHBM905BDM; ③~⑩The recombinant strains 图 4 重组子的初步筛选 Fig.4 Preliminary screening of the recombinants |
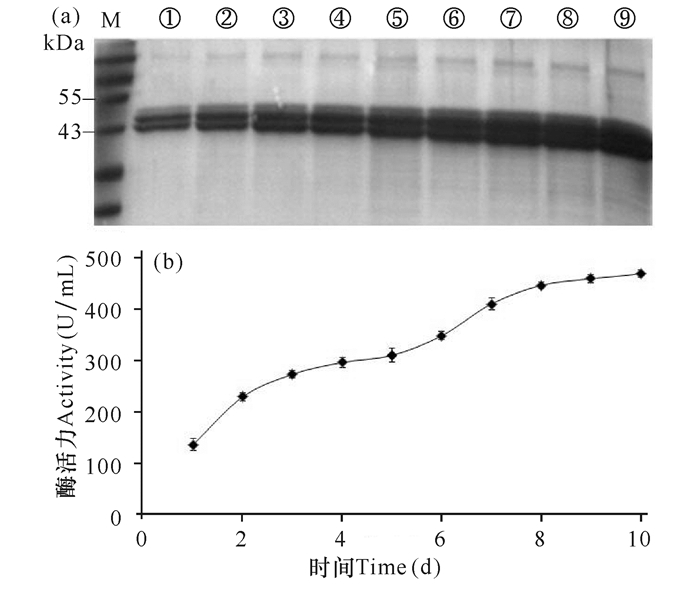
通过初筛,挑选6株产生水解圈较快、较大的重组菌。将这6株菌进行摇瓶诱导复筛得一株酶活力相对较高的菌株。甲醇诱导10 d,每隔24 h取其发酵上清液测活并用SDS-PAGE检测表达量,结果如图 5。由图 5b可知,随着诱导时间的延长,木聚糖酶的表达量和酶活力在逐步提高,诱导8 d以后酶的表达量和酶活力基本趋于稳定,在第10天最高活力达到495 U/mL。
|
(a) SDS-PAGE检测木聚糖酶的表达,其中M为Marker, ①~⑨泳道分别为2号菌加甲醇诱导10 d,每24 h的表达量;(b) 重组木聚糖酶的产酶曲线 (a) SDS-PAGE analysis of high-yield strain 2, M: Marker; Lane ①~⑨, the samples at 24-interval for 10 d; (b) Enzyme production of recombinant xylanase 图 5 2号菌不同诱导时间木聚糖酶的表达量和产酶曲线 Fig.5 Expression of xylanase and its enzyme production curve at different induction time |
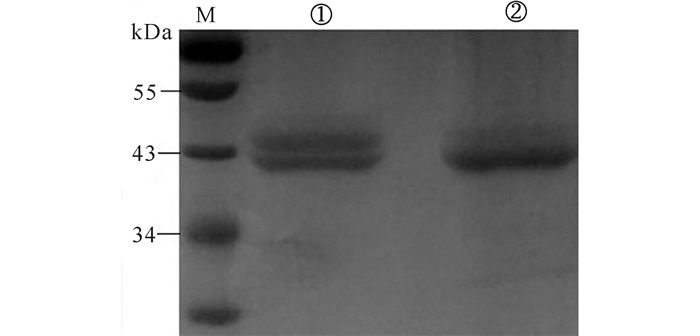
该木聚糖基因长度为1 116 bp,预测蛋白质分子量约为43 kDa,由图 5a可以看出表达的蛋白大小基本正确。然而图中有两条带,推测较大分子量的蛋白质可能是木聚糖酶在酵母中表达糖基化修饰造成的。因此,用去糖基化酶Endo H处理,验证是否发生糖基化修饰[18],结果如图 6所示。SDS-PAGE表明,经去糖基化酶Endo H处理后发酵液中的木聚糖酶条带由两条变为一条,且分子量变小,证明该酶在毕赤酵母中表达时的确发生糖基化修饰。
|
M:预染蛋白Marker;①未用Endo H糖基化酶处理的粗酶液;②用Endo H糖基化酶处理后的酶液 M:marker; ①The crude enzyme solution was not treated with Endo H; ②Xylanase treated with Endo H 图 6 木聚糖酶糖基化验证 Fig.6 Glycosylation analysis of xylanase |
毕赤酵母表达系统由于遗传背景清晰、安全性好,具有较强的醇氧化酶基因 (Alcohol Oxidase,AOX1) 启动子,并能严格调控外源蛋白的表达,获得外源蛋白的表达量高,因此得到广泛应用,并已成功表达数百种外源蛋白[19-21]。由于巴斯德毕赤酵母存在特殊的密码子使用偏好,在所有61个密码子中有25个是毕赤酵母所偏爱的[22-24],如果外源基因密码子偏爱性不同于宿主毕赤酵母,将导致外源蛋白表达量低或表达出没有生物学功能的外源蛋白[25]。研究表明,密码子优化是提高外源蛋白在毕赤酵母中表达量的有效策略之一[11, 26]。本研究成功地将一种来源于嗜碱芽孢杆菌S7的内切木聚糖酶基因 (xyn10A) 经密码子优化后克隆到pHBM905BDM载体上并转化毕赤酵母GS115菌株,实现该酶的分泌表达,并通过底物平板水解圈法和摇瓶发酵比较酶活力大小筛选出产酶量较高的菌株。
基因剂量效应是影响蛋白表达量的重要因素。重组质粒在毕赤酵母染色体上整合一般为单拷贝,但是在甲醇诱导的PAOX1强启动子下,即使单拷贝也能获得外源蛋白高水平表达。一般情况下外源基因整合的拷贝数越高,蛋白的表达量就越高。为进一步提高该酶的表达量,可以用“生物砖”法构建多拷贝质粒[27],进一步筛选出产酶量更高的菌株。
虽然该酶在毕赤酵母表达系统中成功表达,但该酶的活性不高,摇瓶发酵只有495 U/mL,而现在很多木聚糖酶的活力可以很高,如Lu等[11]在毕赤酵母中表达的木聚糖酶的活力可达到6 043 U/mL。由于该酶耐热耐碱,可以进一步采用体外定向进化技术改造该酶基因,例如根据该酶已经解析过的结构对其进行一些理性改造,合理构建突变体库筛选出热稳定性更好,酶活力更高的重组木聚糖酶,进一步满足工业化大规模生产的要求。
4 结论本研究将来源于嗜碱芽孢杆菌S7的耐热耐碱内切木聚糖酶基因 (xyn10A) 经密码子优化合成后克隆到pHBM905BDM载体上,成功转化毕赤酵母GS115菌株后,通过交联木聚糖底物平板水解圈法筛选得到一株产酶量较高的菌株,可用于后续造纸工业纸浆的漂白预处理。
| [1] |
PRADE R A. Xylanases:From biology to biotechnology[J]. Biotechnology and Genetic Engineering Reviews, 1996, 13(1): 101-132. DOI:10.1080/02648725.1996.10647925 |
| [2] |
YAMAURA I, MATSUMOTO T, FUNATSU M, et al. Purification and some properties of endo-1, 3-β-D-xylanase from Pseudomonas sp.PT-5[J]. Agric Biol Chem, 1990, 54(4): 921-926. |
| [3] |
COLLINS T, GERDAY C, FELLER G. Xylanases, xylanase families and extremophilic xylanases[J]. FEMS Microbiol Rev, 2005, 29(1): 3-23. DOI:10.1016/j.femsre.2004.06.005 |
| [4] |
邵婷婷, 王春明, 李蘅香, 等. Bacillus sublitis JH-1木聚糖酶的纯化及酶学性质[J]. 生物加工过程, 2016, 14(5): 51-55. SHAO T T, WANG C M, LI H X, et al. Purification and characterization of xylanase from Bacillus sublitis JH-1[J]. Chinese Journal of Bioprecess Engineering, 2016, 14(5): 51-55. |
| [5] |
阮同琦, 赵祥颖, 刘建军. 木聚糖酶及其应用研究进展[J]. 山东食品发酵, 2008(1): 42-45. RUAN T Q, ZHAO X Y, LIU J J. Research progress of xylanase and its application[J]. Shandong Food Ferment, 2008(1): 42-45. |
| [6] |
ZHAO J, LI X Z, QU Y B. Application of enzymes in producing bleached pulp from wheat straw[J]. Bioresource Technology, 2006, 97(13): 1470-1476. DOI:10.1016/j.biortech.2005.07.012 |
| [7] |
COSSON T, VENDRELL A M P, TERESA B G, et al. Enzymatic assays for xylanase and β-glucanase feed enzymes[J]. Animal Feed Science and Technology, 1999, 77(3/4): 345-353. |
| [8] |
ZHANG G M, HUANG J, HUANG G R, et al. Molecular cloning and heterologous expression of a new xylanase gene from Plectosphaerella cucumerina[J]. Appl Microbiol Biotechnol, 2007, 74(2): 339-346. DOI:10.1007/s00253-006-0648-3 |
| [9] |
聂国兴, 王俊丽, 明红. 木聚糖酶的应用现状与研发热点[J]. 工业微生物, 2008, 38(1): 53-59. NIE G X, WANG J L, MING H. Application status of xylanase and its hottopics of researches and developments[J]. Industrial Microbiology, 2008, 38(1): 53-59. |
| [10] |
徐晓丽. 麦草浆预处理用木聚糖酶的制备与应用[D]. 南京: 南京农业大学, 2005. XU X L.Pretreatment of wheat straw pulp with xylanase preparation and application[D].Nanjing:Nanjing Agricultural University, 2005. |
| [11] |
LU Y H, FANG C, WANG Q H, et al. High-level expression of improved thermo-stable alkaline xylanase variant in Pichia pastoris through codon optimization, multiple gene insertion and high-density fermentation[J]. Scientific Reports, 2016, 6: 37869. DOI:10.1038/srep37869 |
| [12] |
MAMO G, HATTI-KAUL R, MATTIASSON B. A thermostable alkaline active endo-β-1-4-xylanase from Bacillus halodurans S7:Purification and characterization[J]. Enzyme Microb Technol, 2006, 39(7): 1492-1498. DOI:10.1016/j.enzmictec.2006.03.040 |
| [13] |
YANG J K, LIU L Y. Codon optimization through a two-step gene synthesis leads to a high-level expression of Aspergillus niger lip2 gene in Pichia pastoris[J]. Journal of Molecular Catalysis B:Enzymatic, 2010, 63(3/4): 164-169. |
| [14] |
WU A B, CHEN H D, TANG Z Z, et al. Synthesis of Drosophila melanogaster acetylcholinesterase gene using yeast preferred codons and its expression in Pichia pastoris[J]. Chemico-Biological Interactions, 2008, 175(1/2/3): 403-405. |
| [15] |
陈惠, 赵海霞, 王红宁, 等. 植酸酶基因中稀有密码子的改造提高其在毕赤酵母中的表达量[J]. 中国生物化学与分子生物学报, 2005, 21(2): 171-175. CHEN H, ZHAO H X, WANG H N, et al. Increasing expression level of phytase gene (phyA) in Pichia pastoris by changing rare codons[J]. Chinese Journal of Biochemistry and Molecular Biology, 2005, 21(2): 171-175. |
| [16] |
LLOYD A T, SHARP P M. Synonymous codon usage in Kluyveromyces lactis[J]. Yeast, 1993, 9(11): 1219-1228. DOI:10.1002/(ISSN)1097-0061 |
| [17] |
SHARP P M, COWE E. Synonymous codon usage in Saccharomyces cerevisiae[J]. Yeast, 1991, 7(7): 657-678. DOI:10.1002/(ISSN)1097-0061 |
| [18] |
MILLER G L. Use of dinitrosalicylic acid reagent for determination of reducing sugar[J]. Analytical Chemistry, 1959, 31(3): 426-428. DOI:10.1021/ac60147a030 |
| [19] |
CREGG J M, VEDVICK T S, RASCHKE W C. Recent advances in the expression of foreign genes in Pichia pastoris[J]. Nature Biotechnology, 1993, 11(8): 905-910. DOI:10.1038/nbt0893-905 |
| [20] |
PENG Y C, ACHESON N H. Production of active polyomavirus large T antigen in yeast Pichia pastoris[J]. Virus Research, 1997, 49(1): 41-47. DOI:10.1016/S0168-1702(97)01455-X |
| [21] |
CREGG J M, CEREGHINO J L, SHI J Y, et al. Reco-mbinant protein expression in Pichia pastoris[J]. Moleculer Biotechnology, 2000, 16(1): 23-25. DOI:10.1385/MB:16:1 |
| [22] |
赵翔, 霍克克, 李育阳. 毕赤酵母的密码子用法分析[J]. 生物工程学报, 2000, 16(3): 308-311. ZHAO X, HUO K K, LI Y Y. Synonymous codon usage in Pichia pastoris[J]. Chinese Journal of Biotechnology, 2000, 16(3): 308-311. |
| [23] |
GRANTHAM R, GAUTIER C, GOUY M, et al. Codon catalog usage and the genome hypothesis[J]. Nucleic Acids Research, 1980, 8(1): r49-r62. |
| [24] |
SHARP P M, TUOHY T M F, MOSURSKI K R. Codon usage in yeast:Cluster analysis clearly differentiates highly and lowly expressed genes[J]. Nucleic Acids Research, 1986, 14(13): 5125-5143. DOI:10.1093/nar/14.13.5125 |
| [25] |
SCORER C A, BUCKHOLZ R G, CLARE J J, et al. The intracellular production and secretion of HIV-1 envelope protein in the methylotrophic yeast Pichia pastoris[J]. Gene, 1993, 136(1/2): 111-119. |
| [26] |
CHANG S W, SHIEH C J, LEE G C, et al. Multiple mutagenesis of the Candida rugosa LIP1 gene and optimum production of recombinant LIP1 expressed in Pichia pastoris[J]. Applied and Microbiology Biotechnology, 2005, 67(2): 215-224. DOI:10.1007/s00253-004-1815-z |
| [27] |
CREGG J M, TSCHOPP J F, STILLMAN C, et al. High-level expression and efficient assembly of hepatitis B surface antigen in the methylotrophic yeast, Pichia pastoris[J]. Nature Biotechnology, 1987, 5(5): 479-485. DOI:10.1038/nbt0587-479 |
 2017, Vol. 24
2017, Vol. 24 


