酮酶是广泛存在于各类生物体中,负责碳碳键形成的酶,是脂肪酸、聚酮以及甲羟戊酸途径的基础代谢酶[1]。它一般通过克莱森缩合反应添加碳二单元,如此反复缩合,完成一个复杂分子碳骨架的生物合成[2]。酮酶通常以两种形式存在:一种是作为大型复合酶的一个结合域,如Ⅰ型脂肪酸合成酶和Ⅰ型聚酮合酶;另一种是作为单独的酶起催化作用,如Ⅱ型脂肪酸合成酶,Ⅱ型和Ⅲ型聚酮合酶。另外,它的生物合成产物除了长链脂肪酸外,还有大量具有生物活性的次级代谢产物,如具有抗菌作用的红霉素,抗癌作用的多柔比星以及具有抗氧化作用的白藜芦醇。因此,无论是在初级代谢还是在次级代谢中,该类酶都具有重要的生理作用。
根据如何生成亲电的阴离子,可以将该类酶分为脱羧的和非脱羧的。脱羧的克莱森反应是通过丙二酰辅酶A上双酮基团中的β碳脱羧完成阴离子的生成;而非脱羧的克莱森反应是通过乙酰辅酶A上α碳的质子消除,完成阴离子的生成 (图 1)。最为常见的两种脂肪酸合成酶和3种聚酮合酶都是属于脱羧的缩合反应,目前,这类酶的研究已经相当广泛和深入[3-6]。这类酶在反应过程中,一般是由较活泼的丙二酰辅酶A触发反应,通过脱羧产生活性的阴离子,对羰基进行进攻,完成碳碳键的缩合。然而,非脱羧的酮酶研究仅限于Ⅱ型硫解酶[7],这类酶对底物的激活是通过活性中心的第二个半胱氨酸来实现,这种方式也是较为独特的。

|
图 1 脱羧和非脱羧克莱森缩合反应的阴离子生产机制 Fig.1 Anion generation of decarboxylative and non-decarboxylative Claisen condensation |
近年来,随着合成生物学研究的深入,催化非脱羧克莱森反应的多个新型酮酶相继被发现,它们都在天然产物的合成中起到关键的作用,其中包括CsyB[8],MxnB/CorB[9-10],Ppys[11]和StlD。它们之间催化的反应即很类似,又有不同点 (图 2)。CsyB是第一个已知的同时催化脱羧和非脱羧缩合反应的Ⅲ型聚酮合酶;MxnB/CorB是负责合成α-吡喃酮类抗生素myxopyronin A/corallopyronin A的关键酶,在合成的最后一步,非脱羧的缩合左右两条链;Ppys是一类广泛存在的,负责合成新型群体效应分子photopyrone的酮酶;StlD是同时催化克莱森反应和麦克尔加成反应的新型酮酶。这些新型酮酶无一例外的都是完成非脱羧的克莱森缩合反应,接受的底物也与之前的脱羧反应不同,拥有新颖的催化机制,并能合成出全新的分子骨架。近年来,研究者对这些酶进行了详细的研究,包括酶学特性、结构基础、催化机制以及酶学应用。本文就这些最新的进展进行总结和归纳。

|
图 2 新型非脱羧酮酶的酶反应 Fig.2 Enzymatic reaction of novel non-decarboxylative ketosynthases |
CsyB是一种来源于真菌米曲霉的Ⅲ型聚酮合酶,最早是在2005年通过米曲霉菌株全基因组测序发现的[12]。当时,在全基因组中发现了4个疑似Ⅲ型聚酮合酶的CsyA,CsyB,CsyC和CsyD基因,但未发现相关的代谢产物。因此,其功能并未确定。2011年,CsyB的功能研究是通过在米曲霉中同源表达进行的,其产物是带有酰基侧链的吡喃酮,这个结构非常新颖,尤其是酰基的引入。由于是体内表达,它所接受的底物并不十分明确,但可以肯定的是它至少接受一个不同于之前的丙二酰辅酶A的底物[13]。2014年,它的催化功能得到了进一步明确,它可以催化两分子双酮分子的分子内聚合反应 (图 2A),接受非丙二酰辅酶A的新型底物,这些特性都是之前Ⅲ型聚酮合酶所不具备的。同时,它是第一个可以同时催化脱羧缩合和非脱羧缩合的Ⅲ型聚酮合酶[14]。
1.2 MxnB/CorBmyxopyronin A和corallopyronin A是吡喃酮类的抗生素,由于它们是细菌RNA polymerase (RNA聚合酶) 的非竞争性抑制剂,并且与之前的RNA酶抑制剂是作用于不同部位而广受关注[15]。另外,吡喃酮结构在抗生素中的重要性也得到了证实。喂养试验证明,这两个抗生素的生物合成由Ⅰ型聚酮合酶完成。MxnB和CorB是抗生素myxopyronin A和corallopyronin A生物合成中负责左右两部分缩合的关键酶[16-17],它们具有一定的同源性,功能上也十分接近,只是在接受的底物的左链部分有一些不同,因此,在本文中,这两个酶放在一起进行说明。它们的生物学功能通过体外试验完成,底物合成通过有机合成的方法进行,底物是两个完全不同的双酮单元[9-10](图 2B)。
1.3 PpysPpys是负责催化合成群体效应信号分子photopyrone的酮酶。这类酶的发现过程是伴随着对该信号分子的生物合成途径的研究的深入。该酶首先是通过基因敲除试验证实的,当基因ppys被敲除后,photopyrone分子消失[18];接着,利用异源表达技术,将ppys,bkdABC和ngrA导入大肠杆菌中表达,重构了photopyrone的生物合成途径[19]。这个酶的功能被彻底证实。它的底物是一个脂肪链的单酮和一个脂肪链的双酮 (图 2C),脂肪链的侧链经常被修饰,可以产生如pseudopyronine这样的分子,这类酶也广泛存在于细菌中[11]。
1.4 StlDStlD是一类全新的,属于DarB亚家族的酮酶,它的功能已经完成鉴定,主要是通过晶体结构、突变体活性试验和非天然底物的体外试验进行。StlD的发现是基于前期对一种广泛存在的DarB家族酮酶的研究[20],DarB酶和DarA酶联合作用,一个负责缩合两分子底物,另一个负责脱氢脱羧,可以产生之前只有植物中才能产生的1, 3-环己二酮和苯二酚类化合物。它们可以接受一个双酮分子和一个不饱和的单酮分子作为底物 (图 2D),完成一个同时具有克莱森缩合反应和麦克尔加成的酶催化反应。这类反应是非常少见的催化类型。
2 新型酮酶的结构基础近两年,新型酮酶的结构基础陆续得到解析,其中主要包括CsyB、MxnB/CorB和StlD (详见表 1)。
| 表 1 新型非脱羧酮酶的晶体结构 Table 1 Crystal structure of novel non-decarboxylative ketosynthases |
2014年,CsyB的晶体结构得到了解析[21]。野生型CsyB和辅酶A的复合体用分子替换的方法解析度可以达到1.7 Å。二聚体CsyB的整体结构维持了保守的αβαβα的折叠,以C155、H310和N343 3个氨基酸形成催化中心,该中心深埋在每个单体的内部。这些都是Ⅲ型聚酮合酶的典型特点。因此,该酶仍保留了Ⅲ型聚酮合酶的催化性能。CsyB的晶体结构主要分为3个部分:长链脂肪酸的活性口袋、中心催化区和一个特有的活性口袋。值得注意的是,在CsyB的晶体结构中发现一个其他Ⅲ型聚酮合酶所不具有的特异性的活性口袋。初步推测该口袋与其新的催化性能有关,通过突变体活性试验、非天然底物活性试验和突变体结晶试验,确定了该活性口袋具有接受脂肪链酰基的作用,同时,改变该活性口袋的关键氨基酸的大小可以改变CsyB接受底物的特异性。在晶体结构中,还发现了一个氢键网络,同位素喂养试验结果表明,该氢键网络参与到酶催化的过程中[8]。
2.2 MxnB/CorB2015年,MxnB/CorB的晶体结构同时得到了解析,这两个酶由不同的两个研究小组完成,并都得到了较高的分辨率。MxnB的结构是以大肠杆菌FabH为模板,通过分子替换的方法得到的。CorB也是通过分子替换的方法得到的。它们的整体结构维持了保守的αβαβα的折叠结构,可以清楚地看到左右两个活性口袋,且口袋的体积较大,催化三联体氨基酸是C121、H264和N294。从氨基酸序列看,它们保留了脱羧催化的关键氨基酸,但通过对催化三联体氨基酸精确位置的分析,发现它们之间的距离已经发生了微妙的变化,这些变化很可能使得它们的脱羧催化活力丧失。突变体活性试验显示这些氨基酸在催化过程中都在发挥作用[9-10]。
2.3 StlD2016年,StlD的晶体结构得到了解析,这是一个新的结构亚类,是通过硒代甲硫氨酸的方法获得的晶体结构。它的整体结构也维持了保守的αβαβα的折叠结构。关键的催化氨基酸为C126、H302和N337,序列依然保守。但突变体活性试验结果显示,突变体H302A仍然具有与野生型相似的活力,这是在之前的酮酶中没有见到的,这说明催化三联体在非脱羧的反应中已经失去了催化脱羧的作用。而突变体H302A的晶体结构也证实了这一点。
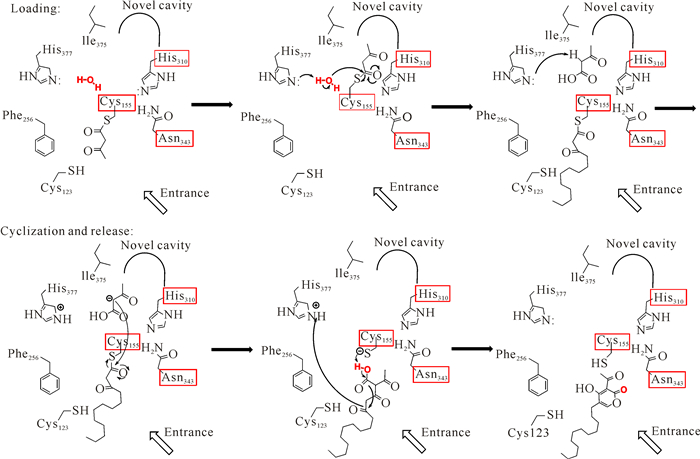
3 新型酮酶的催化机制 3.1 CysBCsyB的催化机制研究较为完整,通过野生型结构、突变体酶学分析、非天然底物活性试验、突变体结构和同位素活性试验,推测出了较为合理的催化机制。具体如下:首先,第一个二酮底物进入活性口袋,结合到活性中心155位的半胱氨酸;接着,转移到底物特异性的活性口袋,同时,将原有的活性口袋让出,然后,通过由一分子水分子、155位的半胱氨酸和377位的组氨酸形成的氢键网络,将第一个底物从155位水解下来;这时,第二个二酮底物进入活性口袋,第一个二酮底物被377位的组氨酸激活,完成对第一个底物的攻击,形成一个碳碳键,接着,中间产物自身环化,完成反应,新产物从活性口袋释放出来,反应结束 (图 3)[8]。
3.2 MxnB/CorB从催化反应中不难看出,MxnB及CorB的反应类型和CsyB相同,不同点是它们接受的底物的侧链不同,因此,这两类酶很可能拥有相同的催化机制。然而,通过氨基酸序列分析,在CsyB中起到关键催化作用的377位的组氨酸,在MxnB及CorB的氨基酸序列中并未发现,通过这两个酶的晶体结构的进一步分析,在其活性中心也并未发现起类似作用的氨基酸残基。因此,它们的催化机制不同于CsyB。初步推测,它们的底物的激活是依靠其他机制,即借助292位的天冬酰胺完成的。另外,通过底物优先试验,MxnB的催化顺序也已被确定,它是先接受左链,然后再缩合右链完成缩合反应的。同时,在反应的过程中有两个ACP蛋白的参与,帮助底物结合在蛋白上,添加ACP蛋白可以明显的提高反应速率[9-10]。
3.3 Ppys在Ppys的催化机制中,它所接受的底物与CsyB和MxnB/CorB相比,在侧链上的结构更加简单,而且它的底物是一个单酮基团和一个双酮基团;不是两个双酮基团。在底物的激活方式上,推测是与其105位的谷氨酸有关[11]。激活的位点推测是单酮基团的碳2位置,就这一步上来说,激活方式有点类似OleA的激活方式[22]。OleA的激活就是由活性中心的酸性氨基酸完成的,激活的底物位点也是单酮基团的碳2位置。
3.4 StlD目前,StlD的具体催化机制已经通过晶体结构、突变体活性试验和非天然底物活性试验进行研究。由于StlD的活性口袋较小且对突变较为敏感,虽然完整的催化机制未得到全面的解析,但一些关键特性已经得到了证实。不同于激活碳2位置的CsyB,StlD激活的是更加不活泼的碳4位置,基于晶体结构和活性口袋氨基酸分析,推测154位的谷氨酸负责激活,突变体活性试验也证实了这个结果。另外,值得注意的是,催化中心的302位的组氨酸突变体仍然保持活力,这在之前的所有酮酶中是没有发现的,说明该氨基酸彻底失去了催化活力,无论是脱羧的,还是不脱羧的反应[23]。
4 结束语CsyB所展现出的对不同类型双酮的缩合能力和其较大的活性口袋,尤其是在侧链的部分上,这些特质都使其在酶法应用上具有更多的想象空间。它是否可以一步法合成不同类型的吡喃酮类抗生素,也是值得深入研究的。就目前的结果看,它是可以通过化学合成前体底物,再经CsyB一步法合成的。
之前的芪类化合物都是来源于植物,StlD的发现给我们利用细菌来源的酶进行酶法合成提供了可能。单独的StlD只能完成芪类化合物前体的合成,但要直接得到芪类化合物,要通过StlD和StlC的联合作用才可以。通过体外实验,对非天然底物,StlD和StlC表现出良好的底物耐受性。目前,已经通过酶法合成出多种芪类化合物的结构类似物,相关的活性正在检测中[23]。
这两年,新型非脱羧的克莱森缩合反应的酮酶不断被报道。对这些全新的酮酶的研究,丰富了酮酶的亚类种类的知识,了解了其更多的催化机制,揭示了许多普遍存在于细菌体内的重要催化反应,给酶法合成提供了可能的思路,为我们全面了解催化非脱羧的克莱森缩合反应的酮酶奠定了理论基础。
| [1] |
HEATH R J, ROCK C O. The Claisen condensation in biology[J]. Natural Product Report, 2002, 19(5): 581-596. DOI:10.1039/b110221b |
| [2] |
HERTWECK C. The biosynthetic logic of polyketide diversity[J]. Angewandte Chemie International Edition, 2009, 48(26): 4688-4716. DOI:10.1002/anie.v48:26 |
| [3] |
MORITA H, SHIMOKAWA Y, TANIO M, et al. A structure-based mechanism for benzalacetone synthase from Rheum palmatum[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(2): 669-673. DOI:10.1073/pnas.0909982107 |
| [4] |
MORITA H, WANIBUCHI K, NII H, et al. Structural basis for the one-pot formation of the diarylheptanoid scaffold by curcuminoid synthase from Oryza sativa[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(46): 19778-19783. DOI:10.1073/pnas.1011499107 |
| [5] |
MORITA H, YAMASHITA M, SHI S P, et al. Synthesis of unnatural alkaloid scaffolds by exploiting plant polyketide synthase[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(33): 13504-13509. DOI:10.1073/pnas.1107782108 |
| [6] |
WHICHER J R, DUTTA S, HANSEN D A, et al. Structural rearrangements of a polyketide synthase module during its catalytic cycle[J]. Nature, 2014, 510(7506): 560-564. DOI:10.1038/nature13409 |
| [7] |
MODIS Y, WIERENG R K. Crystallographic analysis of the reaction pathway of Zoogloea ramigera biosynthetic thiolase[J]. Journal of Molecular Biology, 2000, 297(5): 1171-1182. DOI:10.1006/jmbi.2000.3638 |
| [8] |
MORI T, YANG D F, MATSUI T, et al. Structural basis for the formation of acylalkylpyrones from two β-ketoacyl units by the fungal type Ⅲ polyketide synthase CsyB[J]. Journal of Biological Chemistry, 2015, 290(8): 5214-5225. DOI:10.1074/jbc.M114.626416 |
| [9] |
SUCIPTO H, SAHNER J H, PRUSOV E, et al. In vitro reconstitution of α-pyrone ring formation in myxopyronin biosynthesis[J]. Chemical Science, 2015, 6(8): 5076-5085. DOI:10.1039/C5SC01013F |
| [10] |
ZOCHER G, VILSTRUP J, HEINE D, et al. Structural basis of head to head polyketide fusion by CorB[J]. Chemical Science, 2015, 6(11): 6525-6536. DOI:10.1039/C5SC02488A |
| [11] |
KRESOVIC D, SCHEMPP F, CHEIKH-ALI Z, et al. A novel and widespread class of ketosynthase is responsible for the head-to-head condensation of two acyl moieties in bacterial pyrone biosynthesis[J]. Beilstein Journal of Organic Chemistry, 2015, 11: 1142-1147. |
| [12] |
SESHIME Y, JUVVADI P R, FUJII I, et al. Discovery of a novel superfamily of type Ⅲ polyketide synthases in Aspergillus oryzae[J]. Biochemical and Biophysical Research Communications, 2005, 331(1): 253-260. DOI:10.1016/j.bbrc.2005.03.160 |
| [13] |
SESHIME Y, JUVVADI P R, KITAMOTO K, et al. Identification of csypyrone B1 as the novel product of Aspergillus oryzaetype Ⅲ polyketide synthase CsyB[J]. Bioorganic & Medicinal Chemistry, 2010, 18(12): 4542-4546. |
| [14] |
HASHIMOTO M, KOEN T, TAKAHASHI H, et al. Aspergillus oryzae CsyB catalyzes the condensation of two β-ketoacyl-CoAs to form 3-acetyl-4-hydroxy-6-alkyl-α-pyrone[J]. Journal of Biological Chemistry, 2014, 289(29): 19976-19984. DOI:10.1074/jbc.M114.569095 |
| [15] |
MUKHOPADHYAY J, DAS K, ISMAIL S, et al. The RNA polymerase "Switch Region" is a target for inhibitors[J]. Cell, 2008, 135(2): 295-307. DOI:10.1016/j.cell.2008.09.033 |
| [16] |
EROL Ö, SCHÄBERLE T F, SCHMITZ A, et al. Biosynthesis of the myxobacterial antibiotic corallopyronin A[J]. Chembiochem, 2010, 11(9): 1253-1265. DOI:10.1002/cbic.v11:9 |
| [17] |
SUCIPTO H, WENZEL S C, MULLER R. Exploring chemical diversity of α-pyrone antibiotics:Molecular basis of myxopyronin biosynthesis[J]. Chembiochem, 2013, 14(13): 1581-1589. DOI:10.1002/cbic.v14.13 |
| [18] |
BRACHMANN A O, BRAMEYER S, KRESOVIC D, et al. Pyrones as bacterial signaling molecules[J]. Nature Chemical Biology, 2013, 9(9): 573-578. DOI:10.1038/nchembio.1295 |
| [19] |
BRACHMANN A O, REIMER D, LORENZEN W, et al. Reciprocal cross talk between fatty acid and antibiotic biosynthesis in a nematode symbiont[J]. Angewandte Chemie International Edition, 2012, 51(48): 12086-12089. DOI:10.1002/anie.201205384 |
| [20] |
FUCHS S W, BOZHÜYÜK K A, KRESOVIC D, et al. Formation of 1, 3-cyclohexanediones and resorcinols catalyzed by a widely occuring ketosynthase[J]. Angewandte Chemie International Edition, 2013, 52(15): 4108-4112. DOI:10.1002/anie.201210116 |
| [21] |
YANG D F, MORI T, MATSUI T, et al. Expression, purification and crystallization of a fungal type Ⅲ polyketide synthase that produces the csypyrones[J]. Acta Crystallographica Section F:Structural Biology Communications, 2014, 70(6): 730-733. DOI:10.1107/S2053230X14008516 |
| [22] |
GOBLIRSCH B R, FRIAS J A, WACKETT L P, et al. Crystal structures of Xanthomonas campestris OleA reveal features that promote head-to-head condensation of two long-chain fatty acids[J]. Biochemistry, 2012, 51(20): 4138-4146. DOI:10.1021/bi300386m |
| [23] |
MORI T, AWAKAWA T, SHIMOMURA K, et al. St-ructural insight into the enzymatic formation of bacterial stilbene[J]. Cell Chemical Biology, 2016, 23(12): 1468-1479. DOI:10.1016/j.chembiol.2016.10.010 |
 2017, Vol. 24
2017, Vol. 24