全球每年需要消耗约3 000万t的聚酰胺产品,且消耗量呈现逐年增长的趋势。利用传统的化学法生产聚酰胺产品,将会面临石油资源枯竭,油价上涨,环境污染等众多问题,而利用生物法生产高分子聚合物单体的方法可实现资源的可持续利用,且具有绿色环保、污染少等优点,备受人们的关注。目前,使用最广泛的聚酰胺产品是聚酰胺PA6和聚酰胺PA66,约占全球聚酰胺产品总量的90%[1]。其中,聚酰胺PA66是由己二酸和己二胺聚合而成,己二酸可以通过微生物法合成,己二胺依赖于传统的石油化工合成[2]。人们发现与己二胺互为同系物的1, 5-戊二胺与癸二酸聚合成的聚酰胺PA510,其性能与聚酰胺PA6和聚酰胺PA66相似,可替代其在工业中的应用;1, 5-戊二胺与己二酸聚合成聚酰胺PA56,经处理后得到的PA56纤维,与PA6纤维相比,具有相同的染色性和耐磨性,且回潮率较高[3]。由1, 5-戊二胺单体聚合成的聚氨酯、螯合剂以及固化剂等高分子聚合物在工业中也都有广泛的用途。因此,利用生物法合成1, 5-戊二胺,对于合成聚酰胺、聚氨酯等高分子聚合物具有十分重要的意义。
1, 5-戊二胺,又名尸胺,是一种具有生物活性的含氮碱,可以作为一种正常生理物质存在于生物体中,同时,也可以作为一种肉毒胺存在于腐败物中。通常情况下,细胞内的1, 5-戊二胺是由赖氨酸在赖氨酸脱羧酶的催化脱羧作用后产生。1, 5-戊二胺在农业、医药等领域中也具有广泛应用。在农业上,1, 5-戊二胺参与多种生理过程,如植物细胞分裂与伸长、胚胎产生、开花坐果、果实发育以及胁迫反应等[4-5];在医药方面,1, 5-戊二胺是合成喹嗪碱的前体物质,喹嗪碱可用于治疗心律不齐、催产、缓解低血糖等功效[6],同时,1, 5-戊二胺也可作为一种治疗痢疾的特效药物。此外,1, 5-戊二胺有许多重要的生理学功能,如它是微生物细胞内调控铁载体同化铁离子能力的“铁亲和系统”的组成成分[7],也是解脂厌氧弧菌 (Anaerovibrio lipolytica)、嗜碱性韦荣球菌 (Veillonella alcalescens)、反刍兽新月单胞菌 (Selenomonas ruminantium)、小韦荣球菌 (Veillonella parvula) 等革兰氏阴性菌细胞壁的组成成分[8],对维持细胞的正常生长具有非常重要的作用,同时,它在关闭孔道蛋白以及保护细胞免受活性氧伤害等方面也具有重要作用[9-10]。本文将重点阐述1, 5-戊二胺的最新研究进展。
1 生物法合成1, 5-戊二胺的关键酶 1.1 赖氨酸脱羧酶L-赖氨酸脱羧酶 (L-lysine decarboxylase,简称LDC,EC 4.1.1.18),是生物体内催化L-赖氨酸脱羧生成1, 5-戊二胺的高度专一性酶,是生物合成1, 5-戊二胺的限速酶,其酶促反应需要磷酸吡哆醛 (Pyridoxal phosphate) 作为辅因子[11]。L-赖氨酸脱羧酶分布广泛,存在于大多数微生物中,如大肠杆菌 (Escherichia coli)、蜂房哈夫尼菌 (Hafnia alvei)、尸杆菌 (Bacterium cadaveris)、耐碱芽胞杆菌 (Bacillus halodurans)、伯克霍尔德氏菌 (Burkholderia vietnamensia)、霍乱弧菌 (Vibrio cholerae) 等[9, 12]。L-赖氨酸脱羧酶也存在于高等植物中,如家山黧豆、黄瓜、拟南芥等[13-14]。目前,在NCBI数据库中,赖氨酸脱羧酶基因序列有很多,其中绝大多数都是通过细菌全基因组测序得到,缺乏与其相关的酶学性质报道。有研究报道的细菌来源的L-赖氨酸脱羧酶主要是大肠杆菌 (Escherichia coli)、蜂房哈夫尼菌 (Hafnia alvei)、反刍兽新月单胞菌 (Selenomonas ruminantium)、肺炎克雷伯氏菌 (Klebsiella pneumoniae) 以及伯克霍尔德氏菌 (Burkholderia vietnamensia)。
大肠杆菌中存在两种赖氨酸脱羧酶,诱导型酶CadA和组成型酶LdcC,磷酸吡哆醛是辅因子[15]。CadA是由cadBA操纵子中cadA基因编码,Pcad启动子和cadB基因也属于cadBA操纵子,CadC和LysA蛋白是cadBA操纵子的转录调控因子[16]。研究报道,有关诱导型酶CadA的调控机制被称为酸诱导调控机制,这种机制主要发生在有L-赖氨酸存在的低氧酸性环境条件下,CadA可以消耗细胞内质子将L-赖氨酸转化为1, 5-戊二胺和CO2,CadB蛋白将1, 5-戊二胺分泌到胞外并摄入L-赖氨酸,CO2直接释放到胞外,从而提高细胞外环境中的pH值,保证细胞存活[17]。在体外实验中发现,LdcC在较宽泛的pH值范围内有活性,当pH值为7.6时活性最高,CadA的最适pH值为5.5;LdcC与CadA的最适温度均为52℃,但是LdcC的热稳定性没有CadA稳定;在以L-赖氨酸作为底物的条件下,虽然LdcC与CadA具有相似的Km值,但是LdcC的最大反应速率高于CadA[15]。
肺炎克雷伯菌中也存在两种赖氨酸脱羧酶,一种由cadA基因 (2 148 bp) 编码的诱导型酶CadA,另一种由ldcC基因 (2 190 bp) 编码的组成型酶LdcC,磷酸吡哆醛是辅因子。CadA与LdcC在氨基酸水平上的相似性为82%,CadA与LdcC最适温度为37℃,最适pH值为7.0,以L-赖氨酸为底物的条件下,CadA的Km值为7.7 mmol/L,Kcat值为0.98/s,同时CadA的酶活力高于LdcC[18]。此外,CadA与LdcC对L-鸟氨酸有微弱的脱羧作用[18]。蜂房哈夫尼菌ldc基因的ORF长2 217 bp,编码739个氨基酸,分子量为83 kDa。该基因编码的赖氨酸脱羧酶与大肠杆菌中的LdcC在氨基酸水平上的相似性为85%,一致性为69%。体外实验表明,LdcC的最适温度为37℃,最适pH值为6.5,以L-赖氨酸为底物,Km值为4.93 mmol/L[19]。Jeong等[20]分别在大肠杆菌中克隆表达来源于Selenomonas ruminantium(srLDC), Vibrio vulnificus(vvLDC) 以及Geobacillus thermodenitrificans(gtLDC) 的3种赖氨酸脱羧酶,通过研究3种酶的最适pH值,发现srLDC的最适pH值为6.0,vvLDC为9.0,gtLDC为7.0;vvLDC不仅对L-赖氨酸有催化活性,同时对L-鸟氨酸也具有催化活性,在pH值分别为8, 9, 10时,尸胺的转化率分别为17.8%,38.9%,16%。
目前,已经克隆到的赖氨酸脱羧酶序列有很多,但是缺乏与它们相关的酶学性质报道,加大对赖氨酸脱羧酶酶学性质的研究,不仅有利于丰富赖氨酸脱羧酶基因资源库,而且为构建高产1, 5-戊二胺菌株奠定基础。
1.2 戊二胺-赖氨酸转运蛋白戊二胺-赖氨酸转运蛋白CadB是一种转运1, 5-戊二胺和赖氨酸的跨膜蛋白。Soksawatmaekhin等[21]发现在中性pH值条件下,CadB可以从细胞外摄入1, 5-戊二胺,用于细胞自身生长需求;在酸性pH值条件下,CadB从细胞外摄入L-赖氨酸,分泌1, 5-戊二胺,该过程与PotE功能类似,都参与维持细胞的生长,增加细胞外环境pH值的过程;CadB是由cadBA操纵子上的cadB基因编码的一条含有444个氨基酸残基的多肽链,体外研究发现CadB对戊二胺的摄入和分泌Km值分别为20.8 μmol/L和303 μmol/L。PotE是腐胺-鸟氨酸转运蛋白,在酸性条件下,PotE蛋白在细胞内分别起到分泌腐胺,摄入鸟氨酸的作用,该过程需要消耗H质子,可以增加细胞外环境pH值,增强细胞对酸性环境的耐受性[21]。CadB与PotE在氨基酸水平上一致性超过30.7%,其空间结构也具有一定的相似性。因此,Soksawatmaekhin等[22]根据PotE的结构特点,利用定点突变的技术研究CadB中参与戊二胺摄入与戊二胺-赖氨酸转运相关的关键氨基酸残基位点,结果如下:Tyr73、Tyr89、Tyr90、Glu204、Tyr235、Asp303和Tyr423残基强烈影响戊二胺-赖氨酸转运功能;Tyr55、Glu76、Tyr246、Tyr310、Cys370和Glu377残基对戊二胺-赖氨酸转运功能有一定的影响;Trp43、Tyr57、Tyr107、Tyr366和Tyr368残基强烈影响CadB对戊二胺的摄入;Trp41、Tyr174、Asp185和Glu408残基对戊二胺的摄入功能影响较弱;Arg299残基主要影响CadB对戊二胺的分泌,推测Arg299可能涉及到赖氨酸α-羧基的识别;在中性pH值条件下,CadB在摄入1, 5-戊二胺的同时可能会转运出H+,Cys370不仅能识别1, 5-戊二胺的氨基基团,还能识别H+,因此,Cys370对于1, 5-戊二胺的摄入具有重要作用。研究戊二胺-赖氨酸转运蛋白CadB,了解其转运机制,有利于对CadB进行功能改造,提高1, 5-戊二胺转运效率,为构建高产1, 5-戊二胺菌株奠定基础。
2 大肠杆菌中1, 5-戊二胺的代谢途径及改造 2.1 大肠杆菌中1, 5-戊二胺的代谢途径在大肠杆菌中,1, 5-戊二胺的代谢途径包括1, 5-戊二胺的合成、降解、胞外摄入以及胞内分泌[23]。大肠杆菌中1, 5-戊二胺的代谢途径如图 1所示[24-25]。大肠杆菌中戊二胺的合成途径通过L-赖氨酸的合成途径,由柠檬酸循环 (TCA) 中的草酰乙酸开始,在天冬氨酸转氨酶 (AspC) 的作用下形成L-天冬氨酸,在3种天冬氨酸激酶ThrA、MetL和LysC的作用下使L-天冬氨酸磷酸化,再经过9步反应将L-天冬氨酸转化为L-赖氨酸。天冬氨酸激酶LysC是生物体内合成L-赖氨酸的特异性酶,而ThrA和MetL是一种双功能酶,可将天冬氨酸半醛转化为L-高丝氨酸,从而使碳循环进入合成L-苏氨酸、L-甲硫氨酸以及L-异亮氨酸分支途径[12]。二氨基庚二酸盐是细胞壁肽聚糖的组成成分,也是生物合成L-赖氨酸途径中的一个分支途径。lysC、asd、dapB、dapD以及lysA基因在转录水平上可调控L-赖氨酸的合成,LysC和DapA对L-赖氨酸的合成具有反馈调节作用。大肠杆菌自身合成的赖氨酸脱羧酶 (CadA和LdcC),可以生物催化L-赖氨酸脱羧合成1, 5-戊二胺,由戊二胺-赖氨酸转运蛋白 (CadB) 对其进行转运。大肠杆菌中存在1, 5-戊二胺的分支代谢途径,如speE编码的氨丙基转移酶,speG编码的乙酰转移酶,ygjG编码的氨基转移酶都可以将1, 5-戊二胺转化为其它副产物[12]。
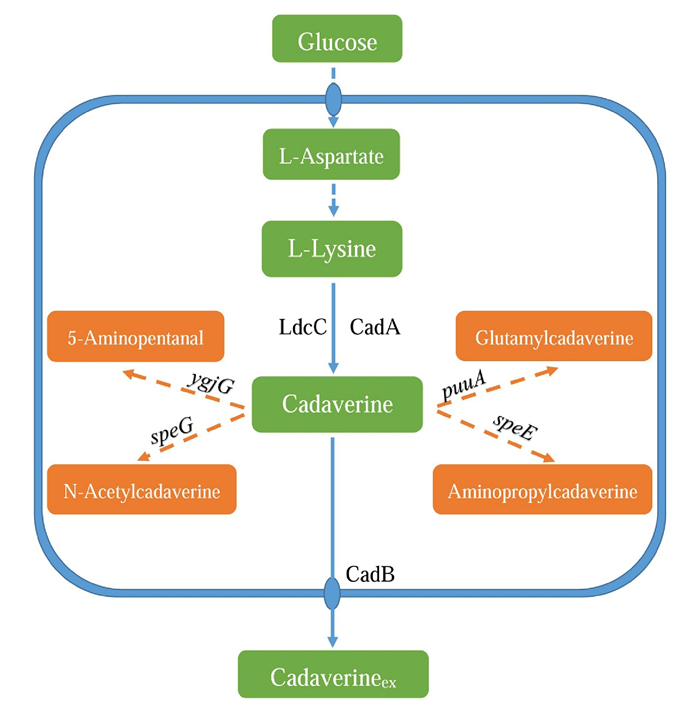
|
橘黄色框表示副产物,橘黄色箭头表示1, 5-戊二胺代谢分支途径,蓝色虚线表示经过多步反应 Orangeboxes represent by-products, orange arrows represent the branched metabolic pathway of 1, 5-diaminopentane, bluedashed lines representmulti-step reactions 图 1 大肠杆菌中1, 5-戊二胺的代谢途径 Fig.1 Metabolic pathway of 1, 5-diaminopentane in Escherichia coli |
大肠杆菌是一种遗传背景清楚、细胞构造简单的模式菌株,且该菌株对1, 5-戊二胺有一定的耐受性,是研究生物合成1, 5-戊二胺的理想宿主之一。研究者们通过对1, 5-戊二胺代谢途径进行代谢工程改造来提高1, 5-戊二胺的合成量。Qian等[24]对大肠杆菌K12 W3110菌株进行代谢工程改造,通过敲除宿主菌株染色体上的speE、speG、puuA以及ygjG基因,利用强启动子tac过量表达赖氨酸脱羧酶基因cadA,用trc启动子替换掉dapA、dapB、lysA基因自身的启动子并整合谷氨酸棒状杆菌中的ddh基因到宿主菌的染色体上,得到的重组菌株的1, 5-戊二胺产量为9.61 g/L。Wang等[19]通过克隆来源于蜂房哈夫尼菌中的赖氨酸脱羧酶基因 (ldc),成功构建的E.coli JM109/pTrc99a-ldc重组菌株,其1, 5-戊二胺的生成量为60.5 g/L,L-赖氨酸的转化率为88.5%;通过易错PCR和DNA洗牌技术对ldc基因进行体外定向进化,筛选到一株1, 5-戊二胺生成量为63.9 g/L,转化率为93.4%的突变株。Li等[15]通过构建含有密码子优化过的产酸克雷伯氏杆菌的赖氨酸脱羧酶基因 (ldc) 和tac启动子的重组表达质粒,构建大肠杆菌突变体LN24菌株,通过补料培养的方式,1, 5-戊二胺的生成率为0.133%/min/g。Li等[26]通过易错PCR技术对ldc基因进行体外定向进化,筛选到LN3014菌株,与LN24菌株相比,其1, 5-戊二胺的生成量提高了50%,L-赖氨酸盐酸盐的转化效率为80%,1, 5-戊二胺的生成率为0.156%/min/g,无副产物生成。研究表明,通过体外定向进化技术提高赖氨酸脱羧酶的活性,可以提高1, 5-戊二胺的生成量;研究其它来源的赖氨酸脱羧酶基因有利于对1, 5-戊二胺合成途径的改造。
全细胞生物转化是一种高效转化L-赖氨酸为1, 5-戊二胺的方法,此方法普遍存在渗透率问题。细菌的细胞壁和细胞膜会成为原材料和代谢产物进出细胞的障碍物,使得代谢产物在细胞内大量积累,大肠杆菌中的CadB蛋白可以分泌1, 5-戊二胺的同时摄入L-赖氨酸,且这种转运方式完全依赖于浓度梯度,不需要消耗能量[24]。Ma等[27]构建的pETDuet-pelB-CadB-CadA质粒含有两个T7启动子控制基因,使得CadA和pelB-CadB融合蛋白表达效果最佳。根据pETDuet-pelB-CadB-CadA建立了一个生物转化L-赖氨酸为1, 5-戊二胺的体系,通过补加L-赖氨酸的方式可以消除底物抑制,经16 h的生物转化,1, 5-戊二胺的生成量为221 g/L,摩尔转化率为92%。Kim等[18]构建克隆有克雷伯氏肺炎杆菌赖氨酸脱羧酶基因 (ldc) 的大肠杆菌重组菌株,通过全细胞生物转化法,L-赖氨酸的摩尔转化率为90%。Oh等[28]采用直接稀释高浓度工业生产的L-赖氨酸溶液作为底物,成功构建出6株克隆有不同赖氨酸脱羧酶基因的大肠杆菌重组菌株,利用生物转化法从6株重组菌中筛选到一株1, 5-戊二胺高产菌株,通过底物pH值、底物浓度等条件优化,1, 5-戊二胺的生成量达到133.7 g/L,摩尔转化率为99.9%。
3 谷氨酸棒状杆菌中1, 5-戊二胺的代谢途径及改造 3.1 谷氨酸棒状杆菌中1, 5-戊二胺的代谢途径谷氨酸棒状杆菌自身不合成1, 5-戊二胺,但该菌株是合成1, 5-戊二胺前体物质L-赖氨酸的高产菌株,有研究表明,谷氨酸棒状杆菌每年生产L-赖氨酸超过100万t,并对1, 5-戊二胺有一定的耐受性,可作为合成1, 5-戊二胺的宿主菌之一[29]。因此,研究者们通过对谷氨酸棒状杆菌中L-赖氨酸代谢途径进行改造,使谷氨酸棒状杆菌具有合成1, 5-戊二胺的能力。谷氨酸棒状杆菌中L-赖氨酸的代谢途径与大肠杆菌极为相似,同样是由柠檬酸循环 (TCA) 中的草酰乙酸开始,在天冬氨酸转氨酶 (AspC) 的作用下形成L-天冬氨酸,再经过9步反应将L-天冬氨酸转化为L-赖氨酸[12]。不同之处在于谷氨酸棒状杆菌中只存在一种天冬氨酸激酶LysC使L-天冬氨酸磷酸化;天冬氨酸半醛是在高丝氨酸脱氢酶的作用下转化为L-高丝氨酸,从而使碳流进入分支途径;四氢二吡啶二羧酸转化为二氨基庚二酸有两种途径,一种是直接经过二氨基庚二酸脱氢酶 (Ddh) 催化完成,另外一种是经过4种酶 (DapD、DapC、DapE、DapF) 催化4种反应完成[12]。在谷氨酸棒状杆菌中表达外源赖氨酸脱羧酶,可以将L-赖氨酸转化为1, 5-戊二胺,将外源赖氨酸脱羧酶与CadB进行共表达,发现谷氨酸棒状杆菌具有分泌1, 5-戊二胺的作用[30]。谷氨酸棒状杆菌中1, 5-戊二胺的代谢途径如图 2所示[24-25]。谷氨酸棒状杆菌中的LysE蛋白能够分泌L-赖氨酸到胞外,不利于细胞内1, 5-戊二胺的积累,此外,Ncgl1469基因编码的乙酰转移酶催化1, 5-戊二胺转化为N-乙酰戊二胺,谷氨酸棒状杆菌自身表达的Cg2893蛋白具有微弱的分泌作用。
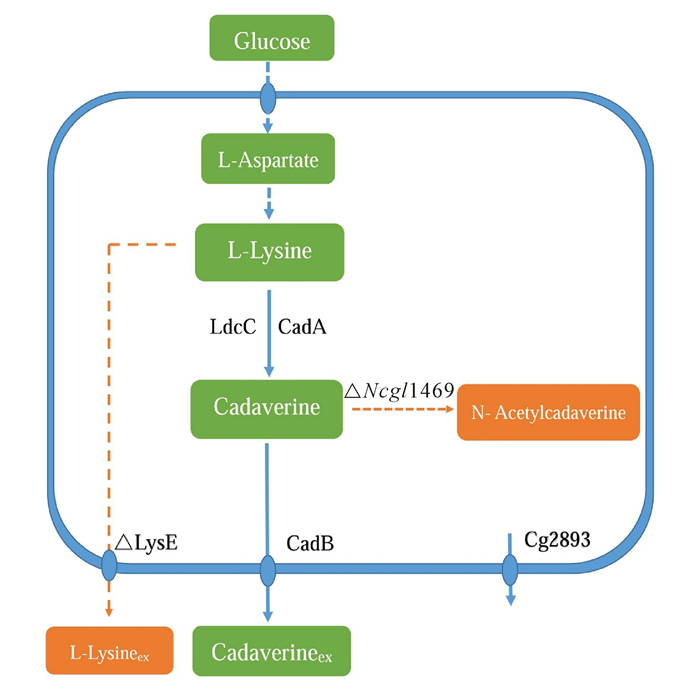
|
橘黄色框表示副产物,橘黄色箭头表示1, 5-戊二胺代谢分支途径和L-赖氨酸的胞外分泌途径,蓝色虚线表示经过多步反应 Orange boxes represent by-products, orange arrows represent the branched metabolic pathway of 1, 5-diaminopentaneand the exocytosis pathway of L-lysine, blue dashed lines represent multi-step reactions 图 2 谷氨酸棒状杆菌中1, 5-戊二胺的代谢途径 Fig.2 Metabolic pathway of 1, 5-diaminopentane in Corynebacterium glutamicum |
研究者们通过对L-赖氨酸在谷氨酸棒状杆菌中的代谢途径进行代谢工程改造,期望获得1, 5-戊二胺高产菌株。Mimitsuka等[29]通过克隆来源于大肠杆菌中的赖氨酸脱羧酶基因 (cadA) 替换谷氨酸棒状杆菌中高丝氨酸脱氢酶基因 (hom),成功构建出第一株能利用葡萄糖发酵合成1, 5-戊二胺的重组菌株TM45,经发酵培养,上清液中1, 5-戊二胺的含量为2.6 g/L,葡萄糖转化率为9.1%。Tateno等[31]克隆来源于链球菌中的α-淀粉酶基因 (amyA) 以及来源于大肠杆菌中的赖氨酸脱羧酶基因 (cadA),实现两个基因在谷氨酸棒状杆菌中共表达,成功构建出的工程菌株利用可溶性淀粉发酵合成1, 5-戊二胺,在不添加磷酸吡哆醛作为辅酶的条件下,1, 5-戊二胺产量为23.4 mmol/L。Buschke等[32]将来源于大肠杆菌中的木糖异构酶基因xylA、木酮糖激酶基因xylB与赖氨酸脱羧酶基因cadA在谷氨酸棒状杆菌中共表达,构建出的工程菌株以木糖为碳源,经发酵培养后,发酵液中1, 5-戊二胺的产量为13.9 mmol/L。为提高戊二胺产量,Buschke等[33]敲除木糖代谢途径中的lysE和act基因,构建出C.glutamicumDAP-Xyl1 icdGTG PeftufbpPsodtktΔactΔlysE突变株,其1, 5-戊二胺产量高达103 g/L。上述3个研究均通过对谷氨酸棒状杆菌中代谢途径的改造,实现谷氨酸棒状杆菌利用除葡萄糖以外的糖类物质生物合成1, 5-戊二胺。牛涛等[34]通过穿梭质粒pXMJl9将蜂房哈夫尼菌中赖氨酸脱羧酶基因 (ldc) 克隆至谷氨酸棒状杆菌中,构建出C.glutamicumTK260512/pXMJl9-ldc菌株,通过摇瓶发酵培养,发酵上清液中1, 5-戊二胺的浓度为0.96 g/L。
谷氨酸棒状杆菌中1, 5-戊二胺的分泌量很低,需要对其代谢途径中不同关键节点进行改造或大量修饰。Kind等[35]通过敲除pepck基因,过量表达aspC、dapA、ddh和lysA基因,利用强启动子tuf表达ldcC基因,得到的DAP-3c菌株经发酵培养后,1, 5-戊二胺的生成量为0.11 g/g葡萄糖,加入辅因子磷酸吡哆醛,1, 5-戊二胺的生成量提高了55%。此外,在发酵过程中检测到25%的副产物N-乙酰戊二胺,通过敲除NCgl1469基因,1, 5-戊二胺产量增加11%。Kind等[25]选取L-赖氨酸高产菌株LYS-12进行代谢途径改造,通过将密码子优化的ldcC基因插入到bioD基因位点,用tuf启动子替代自身启动子,缺失NCgl1469基因,缺失lysE基因,强启动子sod替代cg2893自身启动子,最终得到的DAP-16菌株发酵50 h后,1, 5-戊二胺的累积量为88 g/L,其副产物N-乙酰戊二胺的浓度低于1 g/L。谷氨酸棒状杆菌中与1, 5-戊二胺分泌相关的机制目前仍不清楚,将编码透性酶基因 (cg2893) 缺失,1, 5-戊二胺的分泌量降低,有关该基因与1, 5-戊二胺的分泌调节机制有待深入研究。1, 5-戊二胺分泌量过低会引起细胞内出现1, 5-戊二胺滞留现象,抑制赖氨酸脱羧酶活性,降低1, 5-戊二胺的合成量。因此,提高谷氨酸棒状杆菌中1, 5-戊二胺的分泌量是增加1, 5-戊二胺产量的关键因素之一。Li等[30]通过克隆蜂房哈夫尼菌中的赖氨酸脱羧酶基因 (ldc),实现ldc与cadB基因在谷氨酸棒状杆菌中共表达,发现1, 5-戊二胺的分泌率提高了22%,1, 5-戊二胺的生成量提高了30%。
除了上述对谷氨酸棒状杆菌代谢途径改造外,还可以通过添加某些物质或开发新的表达载体,增强1, 5-戊二胺的合成量。Matsushima等[36]构建了谷氨酸棒状杆菌ATCC 13287/pHT1-cadBA重组菌株以及ATCC 13287/pHT1-ldcC重组菌株,其中ATCC 13287/pHT1-ldcC菌株1, 5-戊二胺合成能力较强,培养16 h后,1, 5-戊二胺浓度为7 mmol/L,若将培养基中硫酸铵的初始浓度由76 mmol/L增加到152 mmol/L,在培养16 h时添加适量的吐温40,培养48 h后,1, 5-戊二胺的生成量为60 mmol/L。该研究表明,增加硫酸铵的初始浓度以及添加适量的吐温40有利于1, 5-戊二胺的合成。Oh等[37]在谷氨酸棒状杆菌中分别构建以PL10、PL26、PI16、PI64、PH30和PH36作为启动子表达ldcC基因的6个重组菌株,结果显示ldcC基因的表达强度不一样,其中含有强启动子PH36构建的重组菌在摇床培养时检测不到1, 5-戊二胺生成量,而含有强启动子PH30的重组菌株1, 5-戊二胺生成量最高,通过补料培养的方式培养64 h后,1, 5-戊二胺累积浓度达到40.91 g/L。由此可见,开发新的表达载体有利于提高谷氨酸棒状杆菌中1, 5-戊二胺的合成量。
4 甲醇芽孢杆菌中1, 5-戊二胺的代谢途径及改造 4.1 甲醇芽孢杆菌中1, 5-戊二胺的代谢途径甲醇芽孢杆菌是一种适温型的甲基营养菌,能够利用甲醇作为碳源,在37~60℃范围内可以正常生长,最适生长温度为50℃。一些甲醇芽孢杆菌在自然条件下可以合成L-赖氨酸,经人工改造获得的高产L-赖氨酸的甲醇芽孢杆菌,通过高密度细胞补料培养的方式L-赖氨酸的产量为65 g/L[38]。在理论水平上,甲醇芽孢杆菌利用甲醇合成L-赖氨酸为0.82 g/g,与谷氨酸棒状杆菌理论上产生L-赖氨酸相似[39]。甲醇芽孢杆菌虽然自身不合1, 5-戊二胺,但是该菌株对1, 5-戊二胺有一定的耐受性,可作为合成1, 5-戊二胺的宿主菌之一[39]。因此,通过表达外源赖氨酸脱羧酶,使甲醇芽孢杆菌具有合成1, 5-戊二胺的能力。甲醇芽孢杆菌中L-赖氨酸的合成也是从柠檬酸循环 (TCA) 中的草酰乙酸开始,合成L-天冬氨酸,在天冬氨酸激酶 (DapG、LysC、YclM)、天冬氨酸半醛脱氢酶Asd、二氢甲基吡啶酸合成酶DapA等酶的作用下形成L-Δ1-四氢吡啶二酸,在二氨基庚二酸脱羧酶LysA作用下,生成L-赖氨酸,表达外源赖氨酸脱羧酶可以将L-赖氨酸转化为1, 5-戊二胺,图 3为甲醇芽孢杆菌中1, 5-戊二胺的代谢途径[39-40]。目前,研究利用甲醇芽孢杆菌合成1, 5-戊二胺的报道较少,有关其代谢途径中1, 5-戊二胺的合成、降解及分泌有待进一步研究。
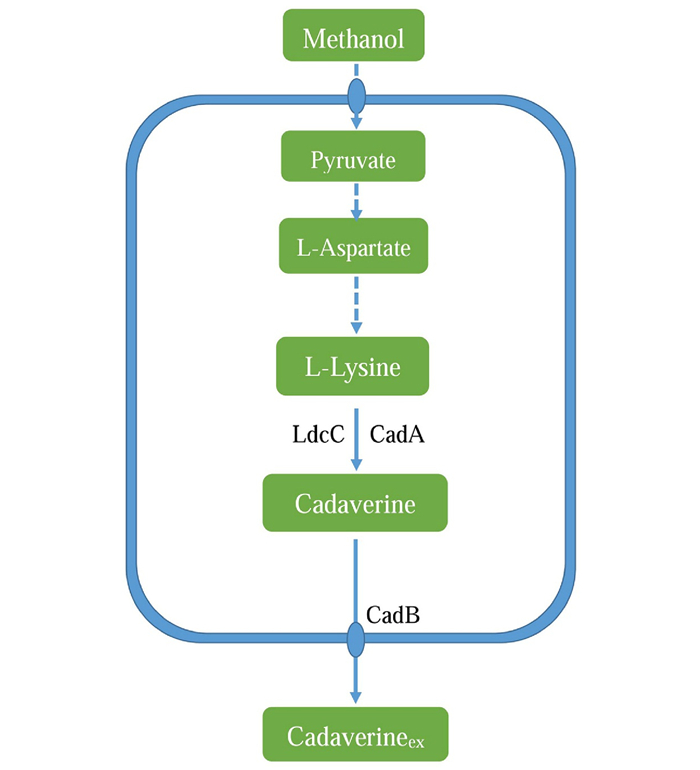
|
蓝色虚线表示经过多步反应 Bluedashed lines represent multi-step reactions 图 3 甲醇芽孢杆菌中1, 5-戊二胺的代谢途径 Fig.3 Metabolic pathway of 1, 5-diaminopentane in Bacillus methanolicus |
甲醇芽孢杆菌可利用非粮物质甲醇作为唯一碳源合成1, 5-戊二胺,这使得其受到一些研究学者的关注,他们期望通过对L-赖氨酸在甲醇芽孢杆菌中的代谢途径进行代谢工程改造,获得1, 5-戊二胺高产菌株。Nærdal等[39]成功构建出含有pTH1mp-cadA重组质粒的甲醇芽孢杆菌菌株,其1, 5-戊二胺的生成量要高于含有pTH1mp-ldcC重组质粒的甲醇芽孢杆菌菌株,对含有pTH1mp-cadA重组质粒的甲醇芽孢杆菌菌株采用高密度细胞补料培养的方式,可获得11.3 g/L的1, 5-戊二胺。目前,适用于研究甲醇芽孢杆菌的表达载体主要有两种,分别为pNW33N和pHP13,pTH1mp质粒来源于pHP13。Irla等[41]认为开发新的基因遗传工具有利于增强甲醇芽孢杆菌在工业生产中的应用范围,他们成功构建出含有pBV2mp-cadA重组质粒的甲醇芽孢杆菌MGA3菌株,该菌株通过高密度细胞补料培养的方式,1, 5-戊二胺的生成量为17.5 g/L。与Nærdal等[39]构建含有pTH1mp-cadA重组质粒的甲醇芽孢杆菌MGA3菌株相比,含有pBV2mp-cadA重组质粒的甲醇芽孢杆菌MGA3菌株获得的1, 5-戊二胺产量提高了55%。
5 展望本文主要综述了已报道的赖氨酸脱羧酶的相关酶学特性,以及利用代谢工程对大肠杆菌、谷氨酸棒状杆菌和甲醇芽孢杆菌中1, 5-戊二胺代谢途径进行改造的研究工作。目前,在大肠杆菌和谷氨酸棒状杆菌中采用强化1, 5-戊二胺合成途径,抑制或者消除分支代谢途径等方法,在提高1, 5-戊二胺产量和L-赖氨酸转化率方面取得了一定的进展。但是,改造后的菌株生产1, 5-戊二胺的水平仍达不到工业化大规模生产的要求,还需进行大量的科学研究。赖氨酸脱羧酶酶活力不够高、酶稳定性不强是合成1, 5-戊二胺产量低的主要原因。针对这一现象可通过研究新的赖氨酸脱羧酶及相关酶学性质,丰富赖氨酸脱羧酶基因资源库,从而选取酶活力更高更稳定的赖氨酸脱羧酶基因,利用定向进化技术,如易错PCR技术、DNA洗牌技术、杂合酶等体外定向进化的方法对赖氨酸脱羧酶基因进行改造。
目前,已有报道的赖氨酸脱羧酶主要来源于纯培养技术培养的微生物菌株,而在自然界中超过99%以上的微生物在现有条件下尚未获得纯培养,这些未培养微生物中所蕴含的基因资源十分丰富,具有潜在的应用价值。宏基因组学技术是探寻微生物多样性、发现新功能基因的一种有效手段,而利用宏基因组技术研究赖氨酸脱羧酶的报道较少。本实验室利用宏基因组文库技术,构建了广西亚热带山区土壤微生物宏基因组文库,利用功能筛选和随机测序相结合的筛选策略,获得多个赖氨酸脱羧酶基因 (ldc1E,ldc1K等),发现这些基因长度约为1 kb或2.2 kb,并完成部分基因序列的生物信息学分析及相关酶学特性研究,其中Ldc1E具有较高的比活力,对底物L-赖氨酸的亲和能力以及催化效率方面优于已报道的蜂房哈夫尼菌中的赖氨酸脱羧酶。
此外,高浓度的1, 5-戊二胺对细胞具有毒害作用是造成戊二胺产量低的另一重要原因。针对这一原因可通过增加1, 5-戊二胺的胞外分泌量,减少胞内累积对宿主产生的毒害作用;提高大肠杆菌、谷氨酸棒状杆菌、甲醇芽孢杆菌等宿主菌株对1, 5-戊二胺的耐受性;强化1, 5-戊二胺代谢途径以及开发新的宿主菌株等策略,有利于实现高效合成1, 5-戊二胺工程菌株的构建。目前,研究者对大肠杆菌以及谷氨酸棒状杆菌中1, 5-戊二胺代谢途径的改造研究较为深入,但是有关利用甲醇芽孢杆菌生产1, 5-戊二胺的研究报道较少。相比于大肠杆菌以及谷氨酸棒状杆菌均以糖类物质作为碳源,甲醇芽孢杆菌可利用非粮物质甲醇作为唯一碳源具有一定的优势。由于适用于甲醇芽孢杆菌中对外源基因进行高效表达的工具盒子较少,可选择性较低,因此,开发更多能应用在甲醇芽孢杆菌中的工具盒子将有利于1, 5-戊二胺合成菌株的构建。此外,深入研究甲醇芽孢杆菌中1, 5-戊二胺代谢途径以及对该途径的进一步改造将更有利于构建高效合成1, 5-戊二胺的工程菌株。
| [1] |
钱伯章. 尼龙新产品的开发应用[J]. 国外塑料, 2011, 29(2): 36-43. QIAN B Z. New developments and new applications of Nylon products[J]. World Plastic, 2011, 29(2): 36-43. |
| [2] |
SANDERS J, SCOTT E, WEUSTHUIS R, et al. Bio-refinery as the bio-inspired process to bulk chemicals[J]. Macromolecular Bioscience, 2007, 7(2): 105-117. DOI:10.1002/(ISSN)1616-5195 |
| [3] |
黄正强, 崔喆, 张鹤鸣, 等. 生物基聚酰胺研究进展[J]. 生物工程学报, 2016, 32(6): 761-774. HUANG Z Q, CUI Z, ZHANG H M, et al. Progress in bio-based polyamides[J]. Chinese Journal of Biotechnology, 2016, 32(6): 761-774. |
| [4] |
王晓云, 邹琦. 多胺与植物衰老关系研究进展[J]. 植物学通报, 2002, 19(1): 11-20. WANG X Y, ZOU Q. Advances in studies on relationship between polyamines and plant senescence[J]. Chinese Bulletin of Botany, 2002, 19(1): 11-20. |
| [5] |
陈学好, 于杰, 李伶利. 高等植物开花结实的多胺研究进展[J]. 植物学通报, 2003, 20(1): 36-42. CHEN X H, YU J, LI L L. Advances in study on polyamines during flowering and fruit setting and development in higher plants[J]. Chinese Bulletin of Botany, 2003, 20(1): 36-42. |
| [6] |
BUNSUPA S, KATAYAMA K, IKEURA E, et al. Lysine decarboxylase catalyzes the first step of quinolizidine alkaloid biosynthesis and coevolved with alkaloid production in lequminosae[J]. The Plant Cell, 2012, 24(3): 1202-1216. DOI:10.1105/tpc.112.095885 |
| [7] |
FLORES F J, RINCÓN J, MARTÍN J F. Characterization of the iron-regulated desA promoter of Streptomyces pilosus as a system for controlled gene expression in actinomycetes[J]. Microbial Cell Factories, 2003, 2: 5. DOI:10.1186/1475-2859-2-5 |
| [8] |
TAKATSUKA Y, KAMIO Y. Molecular dissection of the Selenomonas ruminantium cell envelope and lysine decarboxylase involved in the biosynthesis of a polyamine covalently linked to the cell wall peptidoglycan layer[J]. Bioscience, Biotechnology, and Biochemistry, 2004, 68(1): 1-19. DOI:10.1271/bbb.68.1 |
| [9] |
SCHNEIDER J, WENDISCH V F. Biotechnological production of polyamines by bacteria:Recent achievements and future perspectives[J]. Applied Microbiology and Biotechnology, 2011, 91(1): 17-30. DOI:10.1007/s00253-011-3252-0 |
| [10] |
姚响文, 周密, 曹颖瑛, 等. 多胺在微生物中的研究进展[J]. 中国真菌学杂志, 2014, 9(2): 114-118. YAO X W, ZHOU M, CAO Y Y, et al. Recent progress of polyamines in microorganisms[J]. Chinese Journal of Mycology, 2014, 9(2): 114-118. |
| [11] |
蒋丽丽, 吴晓燕, 刘毅, 等. 赖氨酸脱羧酶发酵工艺及酶学性质[J]. 精细化工, 2006, 23(11): 1060-1063, 1067. JIANG L L, WU X Y, LIU Y, et al. Fermentation and properties of lysine decarboxylase[J]. Fine Chemicals, 2006, 23(11): 1060-1063, 1067. |
| [12] |
KIND S, WITTMANN C. Bio-based production of the platform chemical 1, 5-diaminopentane[J]. Applied Microbiology and Biotechnology, 2011, 91(5): 1287-1296. DOI:10.1007/s00253-011-3457-2 |
| [13] |
逯明辉, 李晓明, 陈劲枫, 等. 黄瓜发芽期耐冷性与赖氨酸脱羧酶基因表达[J]. 中国农业科学, 2005, 38(12): 2492-2495. LU M H, LI X M, CHEN J F, et al. Study on chilling tolerance of cucumber during germination and expression of lysine decarboxylase gene[J]. Scientia Agricultura Sinica, 2005, 38(12): 2492-2495. |
| [14] |
张凯, 蔡恒, 汪晨, 等. 生物法制备1, 5-戊二胺的研究进展[J]. 现代化工, 2012, 32(10): 21-24. ZHANG K, CAI H, WANG C, et al. Research advances in bio-based production of the platform chemical 1, 5-diaminopentane[J]. Modern Chemical Industry, 2012, 32(10): 21-24. |
| [15] |
LI N Q, CHOU H, YU L J, et al. Cadaverine production by heterologous expression of Klebsiella oxytoca lysine decarboxylase[J]. Biotechnology and Bioprocess Engineering, 2014, 19(6): 965-972. DOI:10.1007/s12257-014-0352-6 |
| [16] |
HANEBURGER I, FRITZ G, JURKSCHAT N, et al. Deactivation of the E.coli pH stress sensor CadC by cadaverine[[J]. Journal of Molecular Biology, 2012, 424(1/2): 15-27. |
| [17] |
RAUSCHMEIER M, SCHVPPEL V, TETSCH L, et al. New insights into the interplay between the lysine transporter LysP and the pH sensor CadC in Escherichia Coli[J]. Journal of Molecular Biology, 2014, 426(1): 215-229. DOI:10.1016/j.jmb.2013.09.017 |
| [18] |
KIM J H, KIM H J, KIM Y H, et al. Functional study of lysine decarboxylases from Klebsiella pneumoniae in Escherichia coli and application of whole cell bioconversion for cadaverine production[J]. Journal of Microbiology and Biotechnology, 2016, 26(9): 1586-1592. DOI:10.4014/jmb.1602.02030 |
| [19] |
WANG C, ZHANG K, CHEN Z J, et al. Directed evolution and mutagenesis of lysine decarboxylase fromHafnia alvei AS1.1009 to improve its activity toward efficient cadaverine production[J]. Biotechnology and Bioprocess Engineering, 2015, 20(3): 439-446. DOI:10.1007/s12257-014-0690-4 |
| [20] |
JEONG S, YEON Y J, CHOI E G, et al. Alkaliphilic lysine decarboxylases for effective synthesis of cadaverine from L-lysine[J]. Korean Journal of Chemical Engineering, 2016, 33(5): 1530-1533. DOI:10.1007/s11814-016-0079-5 |
| [21] |
SOKSAWATMAEKHIN W, KURAISHI A, SAKAT-A K, et al. Excretion and uptake of cadaverine by CadB and its physiological functions inEscherichia coli[J]. Molecular Microbiology, 2004, 51(5): 1401-1412. DOI:10.1046/j.1365-2958.2003.03913.x |
| [22] |
SOKSAWATMAEKHIN W, UEMURA T, FUKIWA-KE N, et al. Identification of the cadaverine recognition site on the cadaverine-lysine antiporter CadB[J]. Journal of Biological Chemistry, 2006, 281(39): 29213-29220. DOI:10.1074/jbc.M600754200 |
| [23] |
李东霞, 黎明, 王洪鑫, 等. 生物法合成戊二胺研究进展[J]. 生物工程学报, 2014, 30(2): 161-174. LI D X, LI M, WANG H X, et al. Progress in biosythesis of diaminopentane[J]. Chinese Journal of Biotechnology, 2014, 30(2): 161-174. |
| [24] |
QIAN Z G, XIA X X, LEE S Y. Metabolic engineering of Escherichia coli for the production of cadaverine:A five carbon diamine[J]. Biotechnology and Bioengineering, 2011, 108(1): 93-103. DOI:10.1002/bit.22918 |
| [25] |
KIND S, NEUBAUER S, BECKER J, et al. From zero to hero-production of bio-based nylon from renewable resources using engineered Corynebacterium glutamicum[J]. Metabolic Engineering, 2014, 25: 113-123. DOI:10.1016/j.ymben.2014.05.007 |
| [26] |
LI N Q, CHOU H, XU Y. Improved cadaverine produ-ction from mutant Klebsiella oxytoca lysine decarboxylase[J]. Engineering in Life Sciences, 2016, 16(3): 299-305. DOI:10.1002/elsc.v16.3 |
| [27] |
MA W C, CAO W J, ZHANG H, et al. Enhanced cadaverine production from L-lysine using recombinant Escherichia coli co-overexpressing CadA and CadB[J]. Biotechnology Letters, 2015, 37(4): 799-806. DOI:10.1007/s10529-014-1753-5 |
| [28] |
OH Y H, KANG K H, KWON M J, et al. Development of engineered Escherichia coli whole-cell biocatalysts for high-level conversion of L-lysine into cadaverine[J]. Journal of Industrial Microbiology & Biotechnology, 2015, 42(11): 1481-1491. |
| [29] |
MIMITSUKA T, SAWAI H, HATSU M, et al. Metabolic engineering ofCorynebacterium glutamicum for cadaverine fermentation[J]. Bioscience, Biotechnology, and Biochemistry, 2007, 71(9): 2130-2135. DOI:10.1271/bbb.60699 |
| [30] |
LI M, LI D X, HUANG Y Y, et al. Improving the secretion of cadaverine in Corynebacterium glutamicum by cadaverine-lysine antiporter[J]. Journal of Industrial Microbiology & Biotechnology, 2014, 41(4): 701-709. |
| [31] |
TATENO T, OKADA Y, TSUCHIDATE T, et al. Direct production of cadaverine from soluble starch using Corynebacterium glutamicum coexpressing α-amylase and lysine decarboxylase[J]. Applied Microbiology and Biotechnology, 2009, 82(1): 115-121. DOI:10.1007/s00253-008-1751-4 |
| [32] |
BUSCHKE N, SCHRÖDER H, WITTMANN C. Metabolic engineering of Corynebacterium glutamicum for production of 1, 5-diaminopentane from hemicellulose[J]. Biotechnology Journal, 2011, 6(3): 306-317. DOI:10.1002/biot.v6.3 |
| [33] |
BUSCHKE N, BECKER J, SCHÄFER R, et al. Syste-ms metabolic engineering of xylose-utilizing Corynebacterium glutamicum for production of 1, 5-diaminopentane[J]. Biotechnology Journal, 2013, 8(5): 557-570. DOI:10.1002/biot.201200367 |
| [34] |
牛涛, 黎明, 张俊环, 等. 一步法生产1, 5-戊二胺谷氨酸棒杆菌基因工程菌的构建[J]. 中国生物工程杂志, 2010, 30(8): 93-99. NIU T, LI M, ZHANG J H, et al. Construction of recombinant Corynebacterium glutamicum producing 1, 5-pentanediamine by one step method[J]. China Biotechnology, 2010, 30(8): 93-99. |
| [35] |
KIND S, JEONG W K, SCHRÖDER H, et al. Systems-wide metabolic pathway engineering in Corynebacterium glutamicum for bio-based production of diaminopentane[J]. Metabolic Engineering, 2010, 12(4): 341-351. DOI:10.1016/j.ymben.2010.03.005 |
| [36] |
MATSUSHIMA Y, HIRASAWA T, SHIMIZU H. Enhancement of 1, 5-diaminopentane production in a recombinant strain of Corynebacterium glutamicum by Tween 40 addition[J]. The Journal of General and Applied Microbiology, 2016, 62(1): 42-45. DOI:10.2323/jgam.62.42 |
| [37] |
OH Y H, CHOI J W, KIM E Y, et al. Construction of synthetic promoter-based expression cassettes for the production of cadaverine in recombinant Corynebacterium glutamicum[J]. Applied Biochemistry and Biotechnology, 2015, 176(7): 2065-2075. DOI:10.1007/s12010-015-1701-4 |
| [38] |
BRAUTASET T, JAKOBSEN Ø M, DEGNES K F, et al. Bacillus methanolicus pyruvate carboxylase and homoserine dehydrogenase Ⅰ and Ⅱ and their roles for L-lysine production from methanol at 50℃[J]. Applied Microbiology and Biotechnology, 2010, 87(3): 951-964. DOI:10.1007/s00253-010-2559-6 |
| [39] |
NÆRDAL I, PFEIFENSCHNEIDER J, BRAUTASET T, et al. Methanol-based cadaverine production by genetically engineered Bacillus methanolicus strains[J]. Microbial Biotechnology, 2015, 8(2): 342-350. DOI:10.1111/mbt2.2015.8.issue-2 |
| [40] |
MVLLER J E N, LITSANOV B, BORTFELD-MILLER M, et al. Proteomic analysis of the thermophilic methylotroph Bacillus methanolicus MGA3[J]. Proteomics, 2014, 14(6): 725-737. DOI:10.1002/pmic.v14.6 |
| [41] |
IRLA M, HEGGESET T M B, NÆRDAL I, et al. Genome-based genetic tool development for Bacillus methanolicus:Theta-and rolling circle-replicating plasmids for inducible gene expression and application to methanol-based cadaverine production[J]. Frontiers in Microbiology, 2016, 7: 1481. |
 2017, Vol. 24
2017, Vol. 24 


