2. 钦州市产品质量监督检验所,广西钦州 535008;
3. 广西大学生命科学与技术学院,广西南宁 530004
2. Qinzhou Products Quality Supervision and Inspection Institute, Qinzhou, Guangxi, 535008, China;
3. College of Life Science and Technology, Guangxi University, Nanning, Guangxi, 530004, China
乙偶姻 (acetoin),化学名为3-羟基-2-丁酮,自然存在于玉米、葡萄、可可、苹果、香蕉、干酪、肉类等食品中,具有强烈的奶油、脂肪、白脱样香气,高度稀释后有令人愉快的奶香气。国家标准GB 2760—1986规定乙偶姻为允许使用的食用香料,美国食品香料和萃取物制造者协会 (FEMA) 安全号为2008,国内外常用于奶油、干酪、咖啡、果实的香味增强剂,还用于配置奶香型、肉香型、草莓香型的香精,是一种应用广泛的食用香料[1-3]。同时,乙偶姻作为一种重要的四碳平台化合物,被美国能源部列为30种优先开发利用的平台化合物之一,在食品、医药、烟草、化妆品、化工等行业具有广泛的用途[1-2, 4-5]。近年来,微生物发酵生产天然乙偶姻取得了丰硕的研究成果,并已实现商业化生产。
乙偶姻分子内含有手性碳原子,因此存在 (R)-乙偶姻、(S)-乙偶姻两种手性异构体。如图 1所示,细菌合成手性乙偶姻的主要途径如下:微生物以糖质原料经糖酵解途径生成丙酮酸;α-乙酰乳酸合成酶 (α-Acetolactate synthase) 催化两分子丙酮酸结合,然后经过脱羧反应得到α-乙酰乳酸;α-乙酰乳酸在α-乙酰乳酸脱羧酶 (α-Acetolactate decarboxylase) 的作用下脱羧产生 (R)-乙偶姻。此外,α-乙酰乳酸在有氧条件下,能够自发氧化脱羧产生丁二酮,然后在 (S)-乙偶姻生成型的丁二酮还原酶 (Diacetyl reductase) 的作用下还原产生 (S)-乙偶姻 (图 1)[6-8]。因此,在有氧条件下天然微生物发酵产生的乙偶姻通常是 (R)-乙偶姻和 (S)-乙偶姻两种手性异构体的混合物[3-4],不同异构体所占的比例由微生物的种类和发酵条件决定。由于α-乙酰乳酸自发氧化脱羧产生丁二酮的效率很低,乙偶姻生产菌株的主要产物是 (R)-乙偶姻,(S)-乙偶姻所占的比例通常很低。
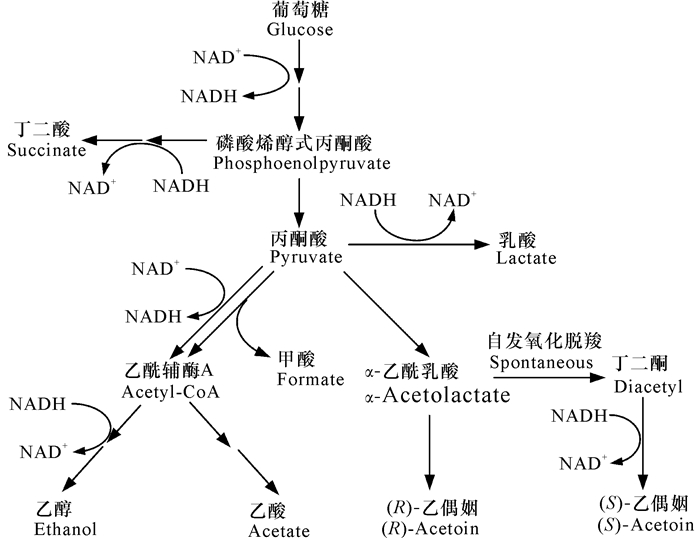
|
图 1 细菌合成乙偶姻手性异构体的典型代谢途径[6-8] Fig.1 Typical metabolic pathways of acetoin stereoisomers in bacteria[6-8] |
乙偶姻手性异构体不仅具有乙偶姻的基本功能,而且由于其具有独特的立体结构,在不对称合成方面优势突出,在合成高附加值手性药物中间体、化学中间体、液晶材料等方面具有特殊的应用[3-4]。化学合成或者天然微生物发酵得到的乙偶姻通常需要经过工艺复杂的手性拆分才能获得一定旋光度的手性异构体,由于 (R)-乙偶姻和 (S)-乙偶姻的物理化学性质相近,手性拆分十分困难,成本高昂,极大地限制了手性乙偶姻的应用。因此,开展生物法合成手性乙偶姻的研究具有重要的意义,也是近年来研究热点之一。本文重点关注了近年来生物法合成手性乙偶姻的研究成果,并讨论了目前所存在的问题和难点,以及今后应该重点关注的研究方向,为后续研究提供参考。
1 酶法催化合成手性乙偶姻在还原型辅酶NADH/NADPH存在的条件下,不同立体选择性的丁二酮还原酶可将丁二酮还原为 (R)-乙偶姻[9-10]或 (S)-乙偶姻[11],每催化丁二酮产生1 mol乙偶姻,需要消耗1 mol NADH/NADPH。由于丁二酮是非手性化合物,可通过微生物发酵或者化学合成获得,常被用作合成手性乙偶姻和2, 3-丁二醇的前体[4]。Takeda等[12]分离纯化获得Mycobacterium sp.B-009的一个醇脱氢酶,该酶具有乙偶姻/丁二酮还原酶的活性。利用偶联反应的方法,该酶可催化反应体系中的正丁醇和氧化型辅酶NAD+产生正丁醛和还原型辅酶NADH,而NADH可作为该酶催化丁二酮还原为 (S)-乙偶姻的还原动力,从而实现辅酶NADH和NAD+的循环。Gao等[4]报道Gluconobacter oxydans DSM 2003的一个NADPH依赖型羰基还原酶也具有丁二酮还原酶的活性。由于该酶催化丁二酮产生 (S)-乙偶姻需要消耗NADPH,他们在反应体系中加入葡萄糖脱氢酶及其底物葡萄糖,葡萄糖脱氢酶可催化葡萄糖和NADP+产生NADPH。在最优条件下反应75 min,14.3 g/L底物丁二酮可转化为12.2 g/L的 (S)-乙偶姻,产物光学纯度为96.9%。Kochius等[13]报道了利用酶催化氧化meso-2, 3-丁二醇的方法合成 (R)-乙偶姻,产物浓度为48 mmol/L,反应所需的辅酶NAD+通过电化学的方法进行再生。Loschonsky等[14]发现Azoarcus sp.strain 22 Lin的二磷酸硫胺依赖型1, 2-环己二酮水解酶 (Thiamine diphosphate dependent cyclohexane-1, 2-dione hydrolase) 可催化丙酮酸、乙醛合成 (S)-乙偶姻。以丙酮酸或乙醛为单独底物,或者两种底物同时添加,产物 (S)-乙偶姻的光学纯度最高分别为93%、95%和92%。该酶的催化效率较低,而且产物光学纯度不够高。
2 全细胞生物催化合成手性乙偶姻随着基因工程技术的发展,全细胞生物催化 (Whole-cell biocatalysis) 成为了合成手性乙偶姻的有效途径。全细胞催化法的基本原理是利用完整的生物有机体作为催化剂,其本质是利用细胞内的酶进行催化[15]。相比纯酶催化制备 (S)-乙偶姻,胞内酶能够更好地保持生理活性,稳定性更好,无需添加昂贵的辅酶,而且省去繁琐的酶分离纯化过程,生产成本更低。
Klebsiella pneumoniae IAM 1063的meso-2, 3-丁二醇脱氢酶 (meso-2,3-Butanediol dehydrogenase) 具有 (R)-乙偶姻还原酶的活性,Ui等[16]将其编码基因导入Escherichia coli JM109进行表达,当培养基中加入外消旋乙偶姻,工程菌株可对 (R)-乙偶姻和 (S)-乙偶姻进行手性拆分,即将其中的 (R)-乙偶姻转化为meso-2, 3-丁二醇,最终可得到0.70 g/L单一构型的 (S)-乙偶姻。由于该meso-2, 3-丁二醇脱氢酶还具有丁二酮还原酶的活性,当培养基加入3 g/L底物丁二酮,上述工程菌株可将丁二酮转化为 (S)-乙偶姻,转化率约为70%[17]。有些Saccharomyces cerevisiae也具有还原丁二酮产生乙偶姻的能力[18],刘培海等[19]发现S.cerevisiae XG-4-2所催化的丁二酮还原反应具有较高的立体选择性,以其休止细胞为全细胞生物催化剂,通过加入辅助底物乙醇来再生NADH,丁二酮转化率可达82%,(S)-乙偶姻纯度为91.2%。
2, 3-丁二醇的光学异构体也可作为前体来合成 (R)-乙偶姻和 (S)-乙偶姻。Yamada-Onodera等[20]将Hansenula polymorpha的甘油脱氢酶编码基因导入E.coli,利用工程菌株的休止细胞作为全细胞生物催化剂,24 h可转化110 mmol/L的 (2R, 3R)-2, 3-丁二醇或meso-2, 3-丁二醇为光学纯 (R)-乙偶姻或 (S)-乙偶姻,转化率为99%。为降低原料成本,Liu等[21]首先利用干重28 g/L的K.pneumoniae CICC 10011休止细胞催化葡萄糖底物制备meso-2, 3-丁二醇和 (2S, 3S)-2, 3-丁二醇,然后利用减压蒸馏的方法去除反应体系中的乙偶姻,适当稀释后加入干重15.4 g/L的Bacillus subtilis 168休止细胞作为催化剂,可将meso-2, 3-丁二醇转化为 (S)-乙偶姻,反应24 h后 (S)-乙偶姻产量为56.7 g/L,光学纯度为96.2%。Gao等[22]在E.coli Rosetta (DE3) 中克隆表达了Paenibacillus polymyxaZJ-9的丁二酮还原酶,获得能够有效转化丁二酮制备 (S)-乙偶姻的工程菌株,反应所需的还原型辅酶NADH通过宿主内源酶系催化辅助底物葡萄糖产生。由于丁二酮对微生物有一定的毒性,通过流加补料的方式控制反应体系中丁二酮浓度,在最优反应条件下,批式补料实验的 (S)-乙偶姻产量为39.4 g/L。Xiao等[23]在E.coli BL21(DE3) 中过量表达Lactobacillus brevis的NADH氧化酶基因nox,构建了高效的原位辅酶再生系统。辅助表达B.subtilis 168的 (2R, 3R)-2, 3-丁二醇脱氢酶基因ydjL,获得了能够有效催化meso-2, 3-丁二醇或 (2R, 3R)-2, 3-丁二醇为 (S)-乙偶姻或 (R)-乙偶姻的基因工程菌E.coli BL21(DE3) (pETDuet-ydjL-nox)。在最优反应条件下,(S)-乙偶姻和 (R)-乙偶姻的产量分别是36.7 g/L和41.8 g/L (光学纯度均为96%)。而在没有表达NADH氧化酶的情况下,工程菌株E.coli BL21(DE3) (pETDuet-ydjL) 的 (S)-乙偶姻产量仅为17.8 g/L。2016年,Guo等[24]在E.coli BL21(DE3) 中表达了meso-2, 3-丁二醇脱氢酶、NADH氧化酶和血红蛋白的基因,所获得的E.coli/pET-mbdh-nox-vgb作为全细胞生物催化剂,可以高效催化meso-2, 3-丁二醇还原为 (R)-乙偶姻。在最优条件下,(R)-乙偶姻的产量为86.7 g/L,光学纯度为97.9%。
3 基因工程微生物发酵制备手性乙偶姻2, 3-丁二醇高产菌株的 (R)-乙偶姻、2, 3-丁二醇代谢途径具有较高的碳架物质流量,通过敲除2, 3-丁二醇脱氢酶的方式可快速获得发酵葡萄糖制备手性乙偶姻的工程菌株。Serratia marcescens MG1发酵的主要代谢产物是meso-2, 3-丁二醇,同时会产生少量的 (2R, 3R)-2, 3-丁二醇和 (2S, 3S)-2, 3-丁二醇,乙偶姻的产量很低。敲除该菌株的meso-2, 3-丁二醇脱氢酶基因slaC,可完全阻断乙偶姻还原为 (2S, 3S)-2, 3-丁二醇,并且meso-2, 3-丁二醇的浓度也由30.8 g/L降低至1.5 g/L,从而获得有效积累 (R)-乙偶姻的工程菌株,(R)-乙偶姻产量为21.8 g/L[25]。Wang等[26]通过敲除K.pneumoniae CGMCC 1.6366的2, 3-丁二醇脱氢酶基因budC来阻断 (R)-乙偶姻转化为2, 3-丁二醇,获得有效积累 (R)-乙偶姻的工程菌株。然而,当发酵液中的葡萄糖耗尽时,(R)-乙偶姻会被作为碳源利用而逐渐降解,相关的分解酶属于乙偶姻脱氢酶系 (Acetoin dehydrogenase enzyme system)。鉴于此,他们进一步敲除编码乙偶姻脱氢酶系的基因簇acoABCD,通过发酵参数的优化,补料发酵条件下 (R)-乙偶姻的产量最高可达62.3 g/L (光学纯度98.0%),底物葡萄糖转化率为28.7%。
然而,2, 3-丁二醇高产菌株大多属于K.pneu-moniae、S.marcescens、Enterobacter aerogenes、Klebsiella oxytoca,这些菌株被世界卫生组织列为致病微生物 (Risk group 2,pathogenic microorganisms),大规模工业化生产的安全性原则在很大程度上限制了其应用[6-7, 27-28]。合成生物学的飞速发展大大促进了生物制造的进步[29],研究者尝试在安全模式微生物E.coli中构建人工合成途径来制备手性乙偶姻。Ui等[30]在宿主E.coli JM109中过量表达来源于K.pneumoniae IAM 1063的α-乙酰乳酸合成酶、α-乙酰乳酸脱羧酶,从而有效地将糖代谢的中心产物丙酮酸导入 (R)-乙偶姻合成途径,工程菌株利用40 g/L葡萄糖发酵时,(R)-乙偶姻的产量为17.5 g/L。Xu等[3]克隆了S.marcescens H30的 (R)-乙偶姻合成基因簇budRAB,其中budR编码转录调控因子,budA编码α-乙酰乳酸脱羧酶,而budB编码α-乙酰乳酸合成酶。将budRAB基因簇导入宿主E.coli DH5α,工程菌株能够产生9.8 g/L的 (R)-乙偶姻。鉴于 (R)-乙偶姻的合成并不能消耗糖酵解产生的大量还原型辅酶NADH (图 1),为进一步提高 (R)-乙偶姻的产量,需要使用辅酶工程策略优化胞内NADH/NAD+的比例。通过辅助表达来源于L.brevis的NADH氧化酶,实现了氧化型辅酶NAD+的再生,(R)-乙偶姻的摇瓶发酵产量从9.8 g/L提高到了23.4 g/L,补料分批发酵产量为60.3 g/L,转化率为86.3%。
相比 (R)-乙偶姻,(S)-乙偶姻很难通过发酵法来制备,原因是 (R)-乙偶姻可直接通过α-乙酰乳酸脱羧酶催化的反应高效合成,而 (S)-乙偶姻前体丁二酮在胞内的合成涉及到非酶介导的自发氧化脱羧反应,该反应的效率非常低。在培养基中添加三价铁离子,可以提高α-乙酰乳酸自发氧化脱羧产生丁二酮的效率[31-32]。Liu等[31]利用基因敲除方法阻断了丁二酮的各种竞争途径,工程菌株Lactococcus lactis CS4701m的丁二酮产量可以达到8.2 g/L。在该菌株中过量表达Enterobacter cloacae的丁二酮脱氢酶,可将丁二酮转化为 (S)-乙偶姻,产量为5.8 g/L[32]。
4 总结与展望综上,手性乙偶姻具有广泛的用途,特别是在不对称合成中具有独特的优势。酶催化法可以制备高纯度的手性乙偶姻,但反应体系中需要加入昂贵的辅酶NAD (H)/NADP (H),而且酶的分离纯化成本较高;全细胞生物催化制备 (S)-乙偶姻具有较大的优势,发酵法由于 (S)-乙偶姻的前体丁二酮的合成涉及到低效的非酶介导氧化脱羧反应,难以获得高浓度 (S)-乙偶姻。由于 (R)-乙偶姻在胞内可通过α-乙酰乳酸脱羧酶介导的高效专一的酶促反应直接产生,利用发酵法可制备较高浓度的 (R)-乙偶姻,例如K.pneumoniae工程菌株发酵 (R)-乙偶姻产量可以达到62.3 g/L,光学纯度98.0%[26]。发酵法制备 (R)-乙偶姻可以利用廉价的淀粉质或纤维素作为原料,具有产量高、成本低等优势,未来有望实现产业化生产。尽管近几年取得一系列的研究成果,但仍存在许多问题和难点,今后研究中应该重点关注以下两个方面。
4.1 菌种选育对2, 3-丁二醇高产菌株进行改造无疑是实现 (R)-乙偶姻高产的捷径,但通常这些高产菌株都属于致病微生物[6-7]。从工业生产安全性原则出发,使用非致病微生物将会成为今后研究的主流方向。部分食品安全的芽胞杆菌能够在高温条件下生长,如Bacillus licheniformis BL1可以在50℃条件下发酵制备光学纯 (2R, 3R)-2, 3-丁二醇[33],将这些嗜热微生物改造为 (R)-乙偶姻生产菌株,不但可以大大降低染菌的机会,同时有利于使用淀粉和纤维素为原料进行同步糖化发酵以降低生产成本。P.polymyxa也是理想的候选菌株之一,其具有高效合成高光学纯 (2R, 3R)-2, 3-丁二醇的能力[6-7, 34-35]。此外,模式微生物E.coli具有诸多优点,是用于构建微生物细胞工厂合成 (R)-乙偶姻的首选底盘细胞之一。
乙偶姻的发酵过程中,通常会产生乳酸、甲酸、乙酸、乙醇、丁二酸等副产物 (图 1),抑制了菌体的生长和发酵,降低了原料的转化率。利用基于全基因组的系统代谢工程和辅酶调控策略,通过多途径优化的方法对代谢网络进行合理的改造,可以有效提高细胞工厂的发酵性能。此外,质粒表达的方式需要使用抗生素、诱导剂[36],而且表达强度往往难以达到最优状态,产生代谢负荷 (Metabolic burden)[37],因此,需要将目标基因整合在大肠杆菌的染色体中,同时结合使用启动子工程 (Promoter engineering)、RBS工程 (Ribosome binding site engineering) 来精确调控目标基因的表达强度[38-40]。
在工业生产手性乙偶姻的过程中,菌体会面临多种胁迫作用,导致菌体生长缓慢,发酵性能下降;此外,对菌种进行大量的遗传改造来定向改变代谢流的方向并使通量最大化,也往往会影响菌体的正常生长和发酵[37]。对胁迫抗性相关的关键基因进行表达或调控,结合使用代谢进化 (Metabolic evolution)[41-42]和反向代谢工程 (Inverse metabolic engineering)[43],从细胞水平上提高菌体的抗逆性能,可以构建出更能适应工业生产条件的生产菌株。此外,高强度的发酵速率还可以结合过程工艺的优化来实现[44],例如使用原位分离技术可以解除高浓度产物乙偶姻对菌株的抑制。
4.2 非粮生物质原料的综合利用微生物发酵生产化学品的培养基成分对生产成本影响很大,高价原料如上述的meso-2, 3-丁二醇、(2R, 3R)-2, 3-丁二醇的使用限制了手性乙偶姻的商业化生产。此外,酵母粉由于富含氨基酸、维生素、微量元素等营养成分,常被用作氮源来发酵生产手性乙偶姻,但丰富培养基的使用通常会大大增加生产成本,例如某些乳酸发酵中酵母粉成本可占总发酵成本的30%[45]。作为重要的四碳平台化学品,开发利用廉价又有效的非粮生物质原料是推进产业化生产手性乙偶姻的必经之路。木薯、甘蔗渣、秸秆、玉米芯是较常见的非粮生物质原料,其中木薯作为发酵原料具有诸多优点:木薯产量高、耐贫瘠、种植面积大,适宜在干旱的山坡地种植,不与粮食作物争地,在我国通常被大量用于生产低附加值的淀粉和乙醇[46-47];按干重计算,其块根淀粉含量可达70%以上,有“淀粉之王”的美称,晒干打粉后可以直接进行高浓度同步糖化发酵 (Simultaneous saccharification and fermentation),无需经过工艺复杂的预处理。此外,发酵工艺优化时还需要开发利用廉价氮源 (如棉籽粉、无机氮源) 来代替酵母粉等高价氮源。通过上述思路的整合应用,有望实现以廉价生物质为原料高效生产高纯度手性乙偶姻。
| [1] |
XIAO Z J, LU J R. Strategies for enhancing fermentative production of acetoin:A review[J]. Biotechnol Adv, 2014, 32(2): 492-503. DOI:10.1016/j.biotechadv.2014.01.002 |
| [2] |
XIAO Z J, LU J R. Generation of acetoin and its derivatives in foods[J]. J Agric Food Chem, 2014, 62(28): 6487-6497. DOI:10.1021/jf5013902 |
| [3] |
XU Q M, XIE L X, LI Y Y, et al. Metabolic engineering of Escherichia coli for efficient production of (3R)-acetoin[J]. J Chem Technol Biotechnol, 2015, 90(1): 93-100. DOI:10.1002/jctb.2015.90.issue-1 |
| [4] |
GAO C, ZHANG L J, XIE Y J, et al. Production of (3S)-acetoin from diacetyl by using stereoselective NADPH-dependent carbonyl reductase and glucose dehydrogenase[J]. Bioresour Technol, 2013, 137: 111-115. DOI:10.1016/j.biortech.2013.02.115 |
| [5] |
WERPY T A, HOLLADAY J E, WHITE J F.Top value added chemicals from biomass:Vol.Ⅰ.results of screening for potential candidates from sugars and synthesis gas[R].Richland, WA:Pacific Northwest National Laboratory, 2004.
|
| [6] |
JI X J, HUANG H, OUYANG P K. Microbial 2, 3-butanediol production:A state-of-the-art review[J]. Biotechnol Adv, 2011, 29(3): 351-364. DOI:10.1016/j.biotechadv.2011.01.007 |
| [7] |
CELI  SKA E, GRAJEK W. Biotechnological production of 2, 3-butanediol-current state and prospects[J]. Biotechnol Adv, 2009, 27(6): 715-725. DOI:10.1016/j.biotechadv.2009.05.002 SKA E, GRAJEK W. Biotechnological production of 2, 3-butanediol-current state and prospects[J]. Biotechnol Adv, 2009, 27(6): 715-725. DOI:10.1016/j.biotechadv.2009.05.002 |
| [8] |
王青艳, 谢能中, 黎贞崇, 等. 微生物法合成 (R, R)-2, 3-丁二醇的研究进展与展望[J]. 基因组学与应用生物学, 2014, 33(6): 1367-1373. WANG Q Y, XIE N Z, LI Z C, et al. Progress and prospect on microbial production of (R, R)-2, 3-butanediol[J]. Genomics and Applied Biology, 2014, 33(6): 1367-1373. |
| [9] |
HEIDLAS J, TRESSL R. Purification and characterization of a (R)-2, 3-butanediol dehydrogenase from Saccharomyces cerevisiae[J]. Arch Microbiol, 1990, 154(3): 267-273. DOI:10.1007/BF00248966 |
| [10] |
YU M L, HUANG M J, SONG Q Q, et al. Characterization of a (2R, 3R)-2, 3-butanediol dehydrogenase from Rhodococcus erythropolis WZ010[J]. Molecules, 2015, 20(4): 7156-7173. DOI:10.3390/molecules20047156 |
| [11] |
WANG Z, SONG Q Q, YU M L, et al. Characterization of a stereospecific acetoin (diacetyl) reductase from Rhodococcus erythropolis WZ010 and its application for the synthesis of (2S, 3S)-2, 3-butanediol[J]. Appl Microbiol Biotechnol, 2014, 98(2): 641-650. DOI:10.1007/s00253-013-4870-5 |
| [12] |
TAKEDA M, ANAMIZU S, MOTOMATSU S, et al. Identification and characterization of a mycobacterial NAD+-dependent alcohol dehydrogenase with superior reduction of diacetyl to (S)-acetoin[J]. Biosci Biotechnol Biochem, 2014, 78(11): 1879-1886. DOI:10.1080/09168451.2014.943649 |
| [13] |
KOCHIUS S, PAETZOLD M, SCHOLZ A, et al. Enantioselective enzymatic synthesis of the α-hydroxy ketone (R)-acetoin from meso-2, 3-butanediol[J]. J Mol Catal B-Enzym, 2014, 103: 61-66. DOI:10.1016/j.molcatb.2013.08.016 |
| [14] |
LOSCHONSKY S, WALTZER S, BRECHT V, et al. Elucidation of the enantioselective cyclohexane-1, 2-dione hydrolase catalyzed formation of (S)-acetoin[J]. Chemcatchem, 2014, 6(4): 969-972. DOI:10.1002/cctc.v6.4 |
| [15] |
WACHTMEISTER J, ROTHER D. Recent advances in whole cell biocatalysis techniques bridging from investigative to industrial scale[J]. Curr Opin Biotechnol, 2016, 42: 169-177. DOI:10.1016/j.copbio.2016.05.005 |
| [16] |
UI S, ODAGIRI M, MIMURA A, et al. Preparation of a chiral acetoinic compound using transgenic Escherichia coli expressing the 2, 3-butanediol dehydrogenase gene[J]. J Ferment Bioeng, 1996, 81(5): 386-389. DOI:10.1016/0922-338X(96)85137-3 |
| [17] |
UI S, MIMURA A, OHKUMA M, et al. Formation of a chiral acetoinic compound from diacetyl by Escherichia coli expressing meso-2, 3-butanediol dehydrogenase[J]. Lett Appl Microbiol, 1999, 28(6): 457-460. DOI:10.1046/j.1365-2672.1999.00560.x |
| [18] |
蒙健宗, 李晓明, 王青艳, 等. 高密度培养基因工程大肠杆菌生产α-乙酰乳酸脱羧酶[J]. 广西科学, 2001, 8(4): 284-286. MENG J Z, LI X M, WANG Q Y, et al. Produce of α-acetolactate decarboxylase in engineering Escherichia coli by high density culture[J]. Guangxi Sciences, 2001, 8(4): 284-286. |
| [19] |
刘培海, 谢能中, 卢志晓, 等. 面包酵母还原丁二酮合成 (S)-乙偶姻的研究[J]. 食品研究与开发, 2013, 34(23): 5-8. LIU P H, XIE N Z, LU Z X, et al. Asymmetric synthesis of (S)-acetoin by reduction of 2, 3-diacetyl with resting cells[J]. Food Research and Development, 2013, 34(23): 5-8. |
| [20] |
YAMADA-ONODERA K, YAMAMOTO H, KAWAHARA N, et al. Expression of the gene of glycerol dehydrogenase from Hansenula polymorpha D1-1 in Escherichia coli for the production of chiral compounds[J]. Acta Biotechnol, 2002, 22(3/4): 355-362. |
| [21] |
LIU Z, QIN J Y, GAO C, et al. Production of (2S, 3S)-2, 3-butanediol and (3S)-acetoin from glucose using resting cells of Klebsiella pneumonia and Bacillus subtilis[J]. Bioresour Technol, 2011, 102(22): 10741-10744. DOI:10.1016/j.biortech.2011.08.110 |
| [22] |
GAO J, XU Y Y, LI F W, et al. Production of S-acetoin from diacetyl by Escherichia coli transformant cells that express the diacetyl reductase gene of Paenibacillus polymyxa ZJ-9[J]. Lett Appl Microbiol, 2013, 57(4): 274-281. |
| [23] |
XIAO Z J, LV C J, GAO C, et al. A novel whole-cell biocatalyst with NAD+ regeneration for production of chiral chemicals[J]. PLoS One, 2010, 5(1): e8860. DOI:10.1371/journal.pone.0008860 |
| [24] |
GUO Z W, ZHAO X H, HE Y Z, et al. Efficient (3R)-acetoin production from meso-2, 3-butanediol using a new whole-cell biocatalyst with co-expression of meso-2, 3-butanediol dehydrogenase, NADH oxidase and Vitreoscilla hemoglobin[J]. J Microbiol Biotechnol, 2017, 27(1): 92-100. DOI:10.4014/jmb.1608.08063 |
| [25] |
BAI F M, DAI L, FAN J Y, et al. Engineered Serratia marcescens for efficient (3R)-acetoin and (2R, 3R)-2, 3-butanediol production[J]. J Ind Microbiol Biotechnol, 2015, 42(5): 779-786. DOI:10.1007/s10295-015-1598-5 |
| [26] |
WANG D X, ZHOU J D, CHEN C, et al. R-acetoin accumulation and dissimilation in Klebsiella pneumoniae[J]. J Ind Microbiol Biotechnol, 2015, 42(8): 1105-1115. DOI:10.1007/s10295-015-1638-1 |
| [27] |
JI X J, LIU L G, SHEN M Q, et al. Constructing a synthetic metabolic pathway in Escherichia coli to produce the enantiomerically pure (R, R)-2, 3-butanediol[J]. Biotechnol Bioeng, 2015, 112(5): 1056-1059. DOI:10.1002/bit.v112.5 |
| [28] |
QIN J Y, XIAO Z J, MA C Q, et al. Production of 2, 3-butanediol by Klebsiella pneumoniae using glucose and ammonium phosphate[J]. Chin J Chem Eng, 2006, 14(1): 132-136. DOI:10.1016/S1004-9541(06)60050-5 |
| [29] |
欧阳平凯, 冯娇, 许晟, 等. 生物制造研究进展[J]. 广西科学, 2016, 23(2): 97-101. OUYANG P K, FENG J, XU S, et al. Recent advances in biological manufacturing[J]. Guangxi Sciences, 2016, 23(2): 97-101. |
| [30] |
UI S, MIMURA A, OKUMA M, et al. The production of D-acetoin by a transgenic Escherichia coli[J]. Lett Appl Microbiol, 1998, 26(4): 275-278. DOI:10.1046/j.1472-765X.1998.00313.x |
| [31] |
LIU J M, CHAN S H J, BROCK-NANNESTAD T, et al. Combining metabolic engineering and biocompatible chemistry for high-yield production of homo-diacetyl and homo-(S, S)-2, 3-butanediol[J]. Metab Eng, 2016, 36: 57-67. DOI:10.1016/j.ymben.2016.02.008 |
| [32] |
LIU J M, SOLEM C, JENSEN P R. Integrating bioco-mpatible chemistry and manipulating cofactor partitioning in metabolically engineered Lactococcus lactis for fermentative production of (3S)-acetoin[J]. Biotechnol Bioeng, 2016, 113(12): 2744-2748. DOI:10.1002/bit.v113.12 |
| [33] |
WANG Q Z, CHEN T, ZHAO X M, et al. Metabolic engineering of thermophilic Bacillus licheniformis for chiral pure D-2, 3-butanediol production[J]. Biotechnol Bioeng, 2012, 109(7): 1610-1621. DOI:10.1002/bit.v109.7 |
| [34] |
李亿, 李检秀, 刘海余, 等. 多粘类芽孢杆菌同步糖化发酵玉米粉生产 (R, R)-2, 3-丁二醇[J]. 广西科学, 2016, 23(1): 41-46. LI Y, LI J X, LIU H Y, et al. Simultaneous sccharification and (R, R)-2, 3-butanediol fermentation from corn flour by Paenibacillus polymyxa[J]. Guangxi Sciences, 2016, 23(1): 41-46. |
| [35] |
XIE N Z, LI J X, SONG L F, et al. Genome sequence of type strain Paenibacillus polymyxa DSM 365, a highly efficient producer of optically active (R, R)-2, 3-butanediol[J]. J Biotechnol, 2015, 195: 72-73. DOI:10.1016/j.jbiotec.2014.07.441 |
| [36] |
XIE N Z, LIANG H, HUANG R B, et al. Biotechnological production of muconic acid:Current status and future prospects[J]. Biotechnol Adv, 2014, 32(3): 615-622. DOI:10.1016/j.biotechadv.2014.04.001 |
| [37] |
JIANG M, ZHANG H R. Engineering the shikimate pathway for biosynthesis of molecules with pharmaceutical activities in E.coli[J]. Curr Opin Biotechnol, 2016, 42: 1-6. |
| [38] |
JONES J A, TOPARLAK Ö D, KOFFAS M A G. Metabolic pathway balancing and its role in the production of biofuels and chemicals[J]. Curr Opin Biotechnol, 2015, 33: 52-59. DOI:10.1016/j.copbio.2014.11.013 |
| [39] |
MARTÍNEZ J A, BOLÍVAR F, ESCALANTE A. Shikimic acid production in Escherichia coli:From classical metabolic engineering strategies to omics applied to improve its production[J]. Front Bioeng Biotechnol, 2015, 3: 145. |
| [40] |
LIU P P, ZHU X N, TAN Z G, et al.Construction of Escherichia coli cell factories for production of organic acids and alcohols[M]//YE Q, BAO J, ZHONG J J (eds.).Bioreactor engineering research and industrial applicationsⅠ.Berlin Heidelberg:Springer, 2016:107-140.
|
| [41] |
ZHU X N, TAN Z G, XU H T, et al. Metabolic evolution of two reducing equivalent-conserving pathways for high-yield succinate production in Escherichia coli[J]. Metab Eng, 2014, 24: 87-96. DOI:10.1016/j.ymben.2014.05.003 |
| [42] |
ZHANG X L, JANTAMA K, MOORE J C, et al. Metabolic evolution of energy-conserving pathways for succinate production in Escherichia coli[J]. P Natl Acad Sci USA, 2009, 106(48): 20180-20185. DOI:10.1073/pnas.0905396106 |
| [43] |
BAILEY J E, SBURLATI A, HATZIMANIKATIS V, et al. Inverse metabolic engineering:A strategy for directed genetic engineering of useful phenotypes[J]. Biotechnol Bioeng, 2002, 79(5): 568-579. DOI:10.1002/(ISSN)1097-0290 |
| [44] |
谢能中, 王青艳, 朱绮霞, 等. 微生物催化合成丙烯酸的研究进展[J]. 广西科学院学报, 2013, 29(3): 149-153. XIE N Z, WANG Q Y, ZHU Q X, et al. Advances in the research of acrylic acid production by microbial catalysis[J]. Journal of Guangxi Academy of Sciences, 2013, 29(3): 149-153. |
| [45] |
KWON S, LEE P C, LEE E G, et al. Production of lactic acid by Lactobacillus rhamnosus with vitamin-supplemented soybean hydrolysate[J]. Enzyme Microb Technol, 2000, 26(2/3/4): 209-215. |
| [46] |
WANG A L, XU Y Q, MA C Q, et al. Efficient 2, 3-butanediol production from cassava powder by a crop-biomass-utilizer, Enterobacter cloacae subsp.dissolvens SDM[J]. PLoS One, 2012, 7(7): e40442. DOI:10.1371/journal.pone.0040442 |
| [47] |
ZHANG Y, ZHANG T, CHI Z, et al. Conversion of cassava starch to trehalose by Saccharomycopsis fibuligera A11 and purification of trehalose[J]. Carbohyd Polym, 2010, 80(1): 13-18. DOI:10.1016/j.carbpol.2009.10.059 |
 2017, Vol. 24
2017, Vol. 24 


