随着我国工业化、城镇化的加速发展和经济全球化的不断深入,高污染、高能耗的化学催化所带来的环境问题日益严峻和突出。加大能源结构调整力度,利用低污染生物催化剂代替化学催化剂以满足工业生产需求已经刻不容缓,受到全社会的广泛关注。酶作为天然生物催化剂,相比于传统化学催化剂具有高效无污染、专一性较强等优点,在工业生产、食品医药等领域具有巨大的应用潜力和良好的发展前景[1]。大量研究发现,经过长期的自然进化,酶已适应所处的细胞微环境,但对于一些苛刻的工业生产条件仍缺乏相对耐受性,如高浓度有机溶剂、极端pH值、高温及化学变性剂等[2-4]。因此,对酶分子选择性、稳定性等理化性质进行改造,提高催化效率,不仅是科学研究的重点,也是工业生产的迫切需求。迄今为止,利用定向进化策略提高酶活性、稳定性、有机溶剂耐受力,以及改变底物范围、创造新功能酶等方面已经获得一定成效[5-6]。但是,由于突变体文库构建数量庞大,并且需要多轮定向进化,定向进化方法难以完成对序列空间的全面搜索。除此之外,突变技术中固有的密码子简并性和碱基突变倾向性使突变文库多样性受到极大限制。随着计算生物学、分子生物学、结构生物学及各种辅助检测技术的发展,分子改造也经历了从定向进化到计算机辅助改造的演变过程,计算机辅助蛋白结构预测以及新功能酶设计策略得到前所未有的重视和发展,成为生物学最为热门的研究领域之一[7-8]。
1 De novo三维结构预测蛋白质的生物学功能很大程度上由其三维结构决定,结构预测是了解酶功能的一种重要途径。早在2005年《Sicence》杂志就将蛋白质折叠问题称为125个还没有解决的问题之一[9]。目前,实验中获得蛋白质三维结构的方法主要是利用X射线衍射方法或核磁共振方法。但是,这种方法成本高昂。更重要的是,近年来,随着蛋白质工程技术的不断发展,发现新蛋白质序列的速度远超过结晶蛋白质的速度,目前仅有不到6万的蛋白质结构被录入蛋白质数据库,而检测到的蛋白质序列已超过700万。此外,20世纪90年代发现一部分蛋白质由于缺乏与其他生物分子的相互作用而无法折叠为确定结构,被称为无序蛋白,更增加了解析蛋白质三维功能的复杂性[10]。因此,面对基因组测序计划中已掌握的大量蛋白质一级序列,了解和主宰蛋白质错综复杂的折叠规律逐渐成为当今生物学的核心问题[11]。
1.1 折叠驱动力最近,Koga等[12]在《Nature》杂志上报道关于设计二级结构α-螺旋和β-折叠的一般规律。通过对蛋白质数据库结构的系统分析,疏水相互作用被认为是蛋白质折叠的主要驱动力。暴露于溶剂表面的疏水氨基酸无法与溶剂形成氢键,不利于蛋白质稳定。为降低蛋白内部疏水核心空隙占有率,同时增大范德华相互作用,疏水氨基酸的侧链极易在非原子重叠的情况下在蛋白内部发生分子间聚集,形成疏水簇 (hydrophobic cluster),以此稳定蛋白质的三维结构[13-14]。相比之下,极性氨基酸更易位于蛋白质表面与水分子相互作用。氢键键能一般为1~7 kcal/mol,比一般的共价键、离子键和金属键键能小,但强于静电相互作用。因此,这些极性基团在折叠后需以氢键形式稳定折叠结构,否则过大的脱离溶剂能将不利于折叠进行[15]。Vogt等[16]对16个家族中具有不同热稳定性的蛋白质进行极性原子表面积、蛋白氢键和盐桥分析,发现80%的家族中,蛋白质的热稳定性与氢键数目呈正比。除疏水相互作用和氢键之外,盐桥相互作用、骨架角度倾向性、二硫键以及芳香环π-π相互作用和阳离子-π相互作用等因素也对蛋白质折叠具有较大贡献。
1.2 预测方法随着计算机技术的发展,越来越多的蛋白质结构预测程序为研究者提供了蛋白质结构与功能认知的参考和依据,尤其是在1994年发起的Critical Assessment of Techniques for Structure Prediction (CASP) 竞赛的推动下发展了很多计算机预测蛋白质结构的方法。20世纪70年代,Anfinsen等[17]在实验基础上提出,蛋白在体外的一定条件下解聚失活后可自动折叠恢复空间结构与活性,其天然构象对应自由能最低时的结构。基于Anfinsen提出的热力学假说,蛋白质结构预测方法通常分为以下3类:针对高相似序列的同源模建方法[18],针对低相似序列的折叠识别方法[19],以及不依赖模板而利用物理学原理直接进行计算的从头预测方法[20]。自CASP7开始,又将前两种方法统称为模板依赖模型方法 (Template-Based Modeling, TBM)。最近的CASP竞赛结果表明,多种预测方法联用结果相比于单一预测方法更为准确[21]。
近些年,一些结构预测软件精度逐步上升。高精度模型可以直接适用于许多领域,例如由美国华盛顿大学的Baker实验组开发的Rosetta,由密歇根大学张阳实验室开发的I-TASSER (Iterative Threading Assemble Refinement)[22],由加利福尼亚大学Andrej Sali编写并维护的Modeller[23],以及开源编码蛋白质序列搜索程序HHsearch[24]等。除这些结构预测软件,Shaw课题组通过Anton计算机对12个小型蛋白进行动力学模拟,观测到400次以上的可逆折叠、去折叠过程,其结构与实验结构的RSMD值在4.5 Å之内[25]。
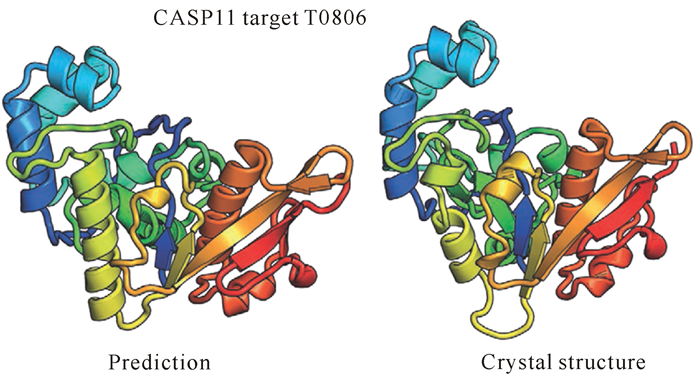
Baker实验室提供的Rosetta[26]主要使用从头预测和比对模建 (comparative modeling) 联用的方法,具有很高的准确度,在CASP大赛中一直名列前茅,从2002年的CASP5开始便成为竞赛中最好的自动预测服务器之一 (图 1)。与固定蛋白骨架的结构预测不同,Rosetta预测方法从超过10 000蛋白构象片段开始计算,采用的主要策略称为“片段插入法”。对于每个需要预测的序列,首先从已知蛋白质结构数据库 (BLAST、PSI-BLAST、3D-Jury) 中搜索与目标蛋白相关的蛋白质片段并随机组合,形成一个粗粒化蛋白质构象空间片段库。对于每个插入片段,选择一个包含3个或9个氨基酸的连续窗口,这些氨基酸的扭转角度被取代为已获得的已知结构片段的扭转角度。对于所有未知结构蛋白序列的3个或9个氨基酸窗口通过与已知结构蛋白组成的无重复数据库匹配进行打分。对于具有结构同源体的结构域,则需要遵循比较模建方法。
|
图 1 Rosetta在CASP 11大赛中T0806 de novo结构预测结果 (左) 与晶体结构 (PDB ID:5CJA)(右) 对比 Fig.1 Rosetta de novo structure prediction (left) for CASP11 target T0806, the crystal structure (PDB ID:5CJA) is shown for comparison (right) |
窗口匹配后,按照精确程度,可分为骨架结构预测和全原子结构预测。为缩小搜索空间,骨架结构预测方法将氨基酸侧链设置为质心原子,通过迭代方程来确定蛋白骨架的几何构象,随后利用联合序列优化计算来预测序列最低势能,并预测结构设计是否匹配序列设计的最低势能态。每次序列设计中都引入固定主链近似,一轮设计完成后,需对整个蛋白做结构优化,然后再进行下一轮的固定主链序列设计,直到结果收敛。在全原子结构预测中,则对所有目标蛋白侧链构象进行采样,组成旋转异构体库,其中包含侧链的扭转角度等信息。随后采用离散组合优化方法确定侧链采样中能量低、结构紧凑的旋转异构体,通过Monte Carlo模拟退火方法将侧链的构象组装到目标蛋白骨架上,并对骨架结构进行微调,达到骨架结构和侧链结构相适应的目的。2005年,Baker团队[27]基于Monte Carlo算法,辅助片段组装技术和能量最小化构象技术,得到预测精度为1.6 Å的蛋白结构。
近年来,Rosetta蛋白结构预测平台不断发展,其主要进展除修饰能量函数外,还体现在3个方面[21]:(1) 基础模拟方法:用Pfam、PSI-BALST、PsiPred、TMHMM等二级结构方法增加跨膜区域片段,减少片段选择误差,而片段取代后的重叠区域用局部运动或骨架转角来解决;(2) 从结构库中去除不理想的蛋白构象 (如能量较高的堆积结构等),提高近天然构象几率;(3) 对给定目标序列的多种独立构象聚类,通过识别最小能量值多样化蛋白质家族,一定程度上补偿能量函数带来的不精确性。
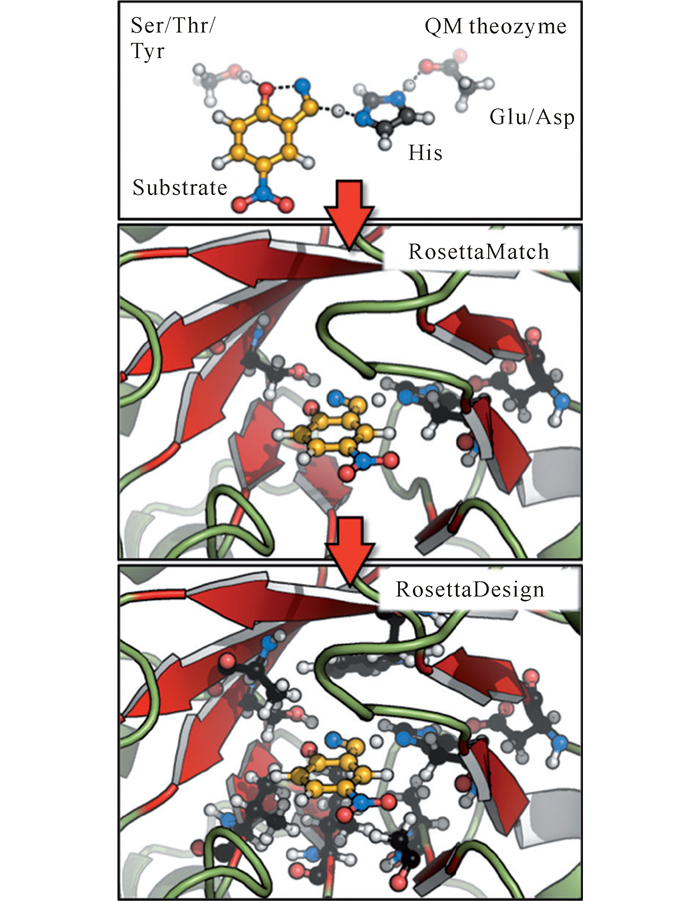
2 酶的计算设计近年来,生物酶学飞速发展,生物学家逐步将酶进化理论和折叠理论结合起来,利用计算机辅助设计特定改进酶的催化功能或其他优良特性 (高稳定性、可溶性、耐酸碱性、高表达量等),以适应某些工业生产要求,降低工业成本[28]。2008年,Baker团队首次利用计算机辅助成功设计出Retro-Aldol酶[29]和Kemp消除反应酶[30],为酶的计算设计奠定良好的基础。针对酶的计算设计改造,Baker以及Houk研究组发展了一种“Inside-out”设计策略[8]。这种策略主要基于量子化学计算方法来确定活性中心theozyme (完成催化反应所需必要氨基酸) 过渡态的稳定构象,设计突变QM区域附近的氨基酸,从而促进新酶theozyme在TS状态下的催化反应进行,同时实现折叠稳定性 (图 2)。
|
图 2 “Inside-out”策略的关键步骤 (以Kemp消除反应为例)[8] Fig.2 Key steps in the computational inside-out design protocol (shown here for the Kemp elimination)[8] |
首先,“Inside-out”策略在催化机理基础上,通过量子化学方法计算理想的活性中心过渡态构象。通过RosettaMatch[31]搜索蛋白质数据库中既可以保持过渡态紧密结合又不破坏过渡态催化作用的功能集团。对于一个既定反应,该策略将生成大量蛋白基序,每个基序由多种功能基团构成。随后,通过对每个基序的能量进行评估,生成一系列蛋白基序构象。另一个可以替代RosettaMatch的软件是Houk研究组开发的SABER[32]。SABER通过搜索蛋白质结构库中合适的功能催化位置,确定催化口袋中可以容纳新型底物的氨基酸突变位置。当theozyme确定性能优良的蛋白骨架后,通过RosettaDesign模块[33]固定催化作用中的关键氨基酸,对其他氨基酸进行最优结构计算。氨基酸侧链的旋转异构体则通过Monte Carlo模拟退火方法进行多轮采样,包括侧链、骨架、配体构象以及受体刚性结构的最小化等。采样过程保持对蛋白几何结构的限制,为减弱限制力可能造成的能量升高,对最后一轮最小化放开所有限制,调节pKa值,获得最优构象后组装至蛋白骨架。
最后,根据过渡态的能量和位置取向,遵循Rosetta能量,配体结合打分、氢键、活性位点几何构象、堆积打分等方式,对优化后的结果进行评估排序,筛选出最优设计后进行实验鉴定活性,并结合定向进化等方法提高活性。经过排序设计后的序列通常与野生型模板蛋白相比有超过10%的区别。然而,这些经过循环设计和结构优化后的新功能蛋白的构象非常容易陷入能量势阱,在相邻位置可能会存在其他极小值,从而无法获得能量最小值构象。此时一般需要引入动力学模拟或蒙特卡洛模拟增大构象采样,调整不合理结构,获得更低能量的构象 (图 3)。

|
图 3 能量函数引导的蛋白质折叠示意图[34] Fig.3 Folding occurs via alternative microscopic trajectories on potential energy surface[34] |
与传统的改造方法相比,Rosetta这种新型改造和设计方法是从设计底物反应到设计结构模型再到最终序列的逆过程,具有灵活性和创造性。目前基于Rosetta“Inside-out”策略已成功设计出可以催化Kemp消除反应的酶[30],Retro-Aldol酶[29],双烯合成酶[35],以及水解有机磷的含Zn金属酶[36]。但是,虽然Rosetta软件有非常高效的氨基酸序列搜索和优化能力,其能量函数仍然相对简单。针对能量函数的缺陷,中国科技大学的刘海燕研究员利用对于非冗余PDB数据库的搜索统计,采用自适应参数搜索策略,构建了改进的统计能量函数,在充分考虑残基局部结构特征影响的同时,兼顾空间相对位置影响,尽可能减小统计误差[37]。此外,一些研究中利用分子动力学模拟方法辅助Rosetta计算以提供更加精确的活性位点信息,并且支持引入自由能计算弥补Rosetta能量函数的缺陷,例如结合自由能、热力学积分以及自由能微扰方法等[38]。2010年,德国普朗克生物物理化学研究院的Groot团队对核糖核酸酶的109个突变体进行自由能计算,其结果与实验值的绝对误差在0.8 kal/mol之内[39]。另外,已有研究基于MD经验价键 (EVB) 方法预测Kemp消除反应[40]和二氢叶酸还原酶DHFR的催化活性[41]等,其结果与实验值比较一致。另一个比较成功的例子是北京大学来鲁华课题组基于“蛋白质关键残基嫁接”策略发展的URANTEIN搜索算法,该算法可以从PDB数据库中计算筛选出适合与铀结合的蛋白质结构并进行计算设计改造,在选出的10种蛋白质中,4种蛋白质改造后与铀酰离子结合能力达到纳摩尔,随后何川教授课题组针对其中一种蛋白质进行优化改造,得到高亲和性、选择性的铀结合蛋白,实现海水中低浓度铀的高效选择性吸附,为解决海水提铀领域中长久以来的难题开辟一条新途径[42]。我们课题组在与荷兰格罗宁根大学以及Enzypep公司合作中,采取一种“高低结合”的计算策略,利用Rosetta设计结合动态分子动力学模拟进行虚拟筛选成功设计一个经过高度改造的多肽酰胺酶突变体PAM12A (包含12个突变点)[43]。通过实验验证,该突变体显示出极高的热稳定性 (熔解温度达到76℃),并且可以在乙腈、丙酮等多种无水溶剂环境中保持数天的稳定活性。此外,PAM12A可以催化包括甲酯化、羟胺化、甲胺化、胺化在内的多种不同的多肽修饰反应,且不受多肽本身序列和C端原功能基团的限制。目前国际上蛋白酶改造和设计的前沿团队包括美国华盛顿大学的Baker团队,加州理工的Mayo团队等。
3 总结与展望设计具有特定结构或功能的蛋白质一方面可以揭示蛋白质结构与功能关系的规律,另一方面可以创造具有潜在应用价值的蛋白质。自1997年Mayo课题组成功设计锌指蛋白[44]以来,蛋白质的三维结构预测以及计算设计技术方法领域取得长足的发展,2003年Kuhlman等[45]的工作将骨架结构重新构建拓展蛋白设计目标,获得天然不存在的蛋白结构,成为全新折叠领域的里程碑。随着算法的改进和计算机科学的快速发展,蛋白设计技术方法也在经历着不断发展和完善的过程。然而,尽管目前已有一些计算设计酶的成功案例,但依然存在成功率较低和酶活性低等缺点,酶的计算设计受限于对酶分子催化机理的理解和空间构象的整体把握,一旦出现偏差,就很难获得理想的改造酶。并且,在引入自由能计算后,计算量庞大,速度较慢,无法对所有突变组合进行逐一计算,仍然需要人工排查结构不合理的改造酶。相信随着蛋白质数据库逐渐丰富,势能函数的优化以及分子动力学模拟的发展,计算的准确率以及相关性也会随之提高,并促进理性设计方法和非理性方法的融合,加快新型高效生物催化剂的发现及功能改造进程。
| [1] |
WALSH C. Enabling the chemistry of life[J]. Nature, 2001, 409(6817): 226-231. DOI:10.1038/35051697 |
| [2] |
SCHÄFER T, BORCHERT T W, NIELSEN V S, et al.Industrial enzymes[M]//ULBER R, SELL D.White biotechnology.Berlin Heidelberg:Springer, 2007:59-131.
|
| [3] |
VICK J E, SCHMIDT-DANNERT C. Expanding the enzyme toolbox for biocatalysis[J]. Angewandte Chemie International Edition, 2011, 50(33): 7476-7478. DOI:10.1002/anie.201100070 |
| [4] |
KIM J, GRATE J W, WANG P. Nanostructures for enzyme stabilization[J]. Chemical Engineering Science, 2006, 61(3): 1017-1026. DOI:10.1016/j.ces.2005.05.067 |
| [5] |
BRAKMANN S. Discovery of superior enzymes by dir-ected molecular evolution[J]. Chembiochem:A European Journal of Chemical Biology, 2001, 2(12): 865-871. DOI:10.1002/(ISSN)1439-7633 |
| [6] |
田口精一. 酵素進化工学による多様なバイオプラスチックの創製[J]. Nippon Nōgeikagaku Kaishi, 2004, 78(8): 748-750. TAGUCHI S. Biosynthesis of bioplastics with desired properties by enzyme directed evolution[J]. Japan Society for Bioscience, Biotechnology, and Agrochemistry, 2004, 78(8): 748-750. |
| [7] |
YANG W, LAI L H. Computational design of proteins with novel structure and functions[J]. Chinese Physics B, 2016, 25(1): 018702. DOI:10.1088/1674-1056/25/1/018702 |
| [8] |
KISS G, ÇELEBI-ÖLÇÜM N, MORETTI R, et al. Computational enzyme design[J]. Angewandte Chemie International Edition, 2013, 52(22): 5700-5725. DOI:10.1002/anie.201204077 |
| [9] |
The Science Editorial. So much more to know[J]. Science, 2005, 309(5731): 78-102. |
| [10] |
SUÁREZ M, JARAMILLO A. Challenges in the computational design of proteins[J]. Journal of the Royal Society Interface, 2009, 6(S4): S477-S491. |
| [11] |
ZANGHELLINI A. De novo computational enzyme design[J]. Current opinion in biotechnology, 2014, 29: 132-138. DOI:10.1016/j.copbio.2014.03.002 |
| [12] |
KOGA N, TATSUMI-KOGA R, LIU G H, et al. Principles for designing ideal protein structures[J]. Nature, 2012, 491(7423): 222-227. DOI:10.1038/nature11600 |
| [13] |
TRIKULENKO A V. Role of hydrophobic interactions in protein chain folding during biosynthesis[J]. Biochemistry, 1998, 63(5): 564-567. |
| [14] |
PACE C N, FU H L, FRYAR K L, et al. Contribution of hydrophobic interactions to protein stability[J]. Journal of Molecular Biology, 2011, 408(3): 514-528. DOI:10.1016/j.jmb.2011.02.053 |
| [15] |
FLEMING P J, ROSE G D. Do all backbone polar groups in proteins form hydrogen bonds?[J]. Protein Science, 2005, 14(7): 1911-1917. DOI:10.1110/(ISSN)1469-896X |
| [16] |
VOGT G, WOELL S, ARGOS P. Protein thermal stability, hydrogen bonds, and ion pairs[J]. Journal of Molecular Biology, 1997, 269(4): 631-643. DOI:10.1006/jmbi.1997.1042 |
| [17] |
ANFINSEN C B. Principles that govern the folding of protein chains[J]. Science, 1973, 181(4096): 223-230. DOI:10.1126/science.181.4096.223 |
| [18] |
VENSELAAR H, JOOSTEN R P, VROLING B, et al. Homology modelling and spectroscopy, a never-ending love story[J]. European Biophysics Journal, 2010, 39(4): 551-563. DOI:10.1007/s00249-009-0531-0 |
| [19] |
JONES D T, TAYLOR W R, THORNTON J M. A new approach to protein fold recognition[J]. Nature, 1992, 358(6381): 86-89. DOI:10.1038/358086a0 |
| [20] |
LIWO A, LEE J, RIPOLL D R, et al. Protein structure prediction by global optimization of a potential energy function[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(10): 5482-5485. DOI:10.1073/pnas.96.10.5482 |
| [21] |
PARK H, DIMAIO F, BAKER D. CASP11 refinement experiments with ROSETTA[J]. Proteins:Structure, Function, and Bioinformatics, 2016, 84(S1): 314-322. |
| [22] |
ZHANG Y. I-TASSER:Fully automated protein structure prediction in CASP8[J]. Proteins:Structure, Function, and Bioinformatics, 2009, 77(S9): 100-113. DOI:10.1002/prot.v77.9s |
| [23] |
WEBB B, SALI A.Protein structure modeling with MODELLER[M]//KIHARA D.Protein structure prediction.New York:Springer, 2014:1-15.
|
| [24] |
SÖDING J, BIEGERT A, LUPAS A N. The HHpred interactive server for protein homology detection and structure prediction[J]. Nucleic Acids Research, 2005, 33: W244-W248. DOI:10.1093/nar/gki408 |
| [25] |
SHAW D E, DENEROFF M M, DROR R O, et al. Anton, a special-purpose machine for molecular dynamics simulation[J]. Communications of the ACM-Web Science, 2008, 51(7): 91-97. DOI:10.1145/1364782 |
| [26] |
LEAVER-FAY A, TYKA M, LEWIS S M, et al. ROSETTA3:An object-oriented software suite for the simulation and design of macromolecules[J]. Methods in Enzymology, 2011, 487: 545-574. DOI:10.1016/B978-0-12-381270-4.00019-6 |
| [27] |
MISURA K M S, BAKER D. Progress and challenges in high-resolution refinement of protein structure models[J]. Proteins:Structure, Function, and Bioinformatics, 2005, 59(1): 15-29. DOI:10.1002/prot.20376 |
| [28] |
HUANG P S, BOYKEN S E, BAKER D. The coming of age of de novo protein design[J]. Nature, 2016, 537(7620): 320-327. DOI:10.1038/nature19946 |
| [29] |
JIANG L, ALTHOFF E A, CLEMENTE F R, et al. De novo computational design of retro-aldol enzymes[J]. Science, 2008, 319(5868): 1387-1391. DOI:10.1126/science.1152692 |
| [30] |
RÖTHLISBERGER D, KHERSONSKY O, WOLLA-COTT A M, et al. Kemp elimination catalysts by computational enzyme design[J]. Nature, 2008, 453(7192): 190-195. DOI:10.1038/nature06879 |
| [31] |
ZANGHELLINI A, JIANG L, WOLLACOTT A M, et al. New algorithms and an in silico benchmark for computational enzyme design[J]. Protein Science, 2006, 15(12): 2785-2794. DOI:10.1110/(ISSN)1469-896X |
| [32] |
NOSRATI G R, HOUK K N. SABER:A computational method for identifying active sites for new reactions[J]. Protein Science, 2012, 21(5): 697-706. DOI:10.1002/pro.2055 |
| [33] |
RICHTER F, LEAVER-FAY A, KHARE S D, et al. De novo enzyme design using Rosetta3[J]. PLoS One, 2011, 6(5): e19230. DOI:10.1371/journal.pone.0019230 |
| [34] |
DILL K A, MACCALLUM J L. The protein-folding problem, 50 years on[J]. Science, 2012, 338(6110): 1042-1046. DOI:10.1126/science.1219021 |
| [35] |
SIEGEL J B, ZANGHELLINI A, LOVICK H M, et al. Computational design of an enzyme catalyst for a stereoselective bimolecular diels-alder reaction[J]. Science, 2010, 329(5989): 309-313. DOI:10.1126/science.1190239 |
| [36] |
KHARE S D, KIPNIS Y, JR GREISEN P, et al. Computational redesign of a mononuclear zinc metalloenzyme for organophosphate hydrolysis[J]. Nature Chemical Biology, 2012, 8(3): 294-300. DOI:10.1038/nchembio.777 |
| [37] |
XIONG P, WANG M, ZHOU X Q, et al. Protein design with a comprehensive statistical energy function and boosted by experimental selection for foldability[J]. Nature Communications, 2014, 5: 5330. DOI:10.1038/ncomms6330 |
| [38] |
GUMBART J C, ROUX B, CHIPOT C. Standard binding free energies from computer simulations:What is the best strategy?[J]. Journal of Chemical Theory and Computation, 2013, 9(1): 794-802. DOI:10.1021/ct3008099 |
| [39] |
SEELIGER D, DE GROOT B L. Protein thermostability calculations using alchemical free energy simulations[J]. Biophysical Journal, 2010, 98(10): 2309-2316. DOI:10.1016/j.bpj.2010.01.051 |
| [40] |
FRUSHICHEVA M P, CAO J, WARSHEL A. Challenges and advances in validating enzyme design proposals:The case of kemp eliminase catalysis[J]. Biochemistry, 2011, 50(18): 3849-3858. DOI:10.1021/bi200063a |
| [41] |
KUMARASIRI M, BAKER G A, SOUDACKOV A V, et al. Computational approach for ranking mutant enzymes according to catalytic reaction rates[J]. The Journal of Physical Chemistry B, 2009, 113(11): 3579-3583. DOI:10.1021/jp810363k |
| [42] |
ZHOU L, BOSSCHER M, ZHANG C S, et al. A protein engineered to bind uranyl selectively and with femtomolar affinity[J]. Nature Chemistry, 2014, 6(3): 236-241. DOI:10.1038/nchem.1856 |
| [43] |
WU B, WIJMA H J, SONG L, et al. Versatile peptide C-terminal functionalization via a computationally engineered peptide amidase[J]. ACS Catalysis, 2016, 6(8): 5405-5414. DOI:10.1021/acscatal.6b01062 |
| [44] |
DAHIYAT B I, MAYO S L. De novo protein design:Fully automated sequence selection[J]. Science, 1997, 278(5335): 82-87. DOI:10.1126/science.278.5335.82 |
| [45] |
KUHLMAN B, DANTAS G, IRETON G C, et al. Design of a novel globular protein fold with atomic-level accuracy[J]. Science, 2003, 302(5649): 1364-1368. DOI:10.1126/science.1089427 |
 2017, Vol. 24
2017, Vol. 24 


